2. 南京师范大学生命科学学院 江苏省微生物与功能基因组学重点实验室 江苏省微生物资源产业化工程技术研究中心,南京 210023
2. Jiangsu Key Laboratory for Microbes and Functional Genomics, Jiangsu Engineering and Technology Research Center for Industrialization of Microbial Resources, College of Life Science, Nanjing Normal University, Nanjing 210023
微生物催化的腈类转化有两条途径:一条是在腈水解酶(Nitrilase,EC3.5.5.1)的催化下由腈类直接产生羧酸,例如Vaughan等[1]报道的Nocardia rhodochrous LL100-21可直接将3-氰基吡啶(3-CP)代谢为烟酸,但是在这一过程中腈水解酶的活性需要诱导。另一条代谢途径是由腈水合酶(NHase,EC4.2.1.84)和酰胺酶(Amidase,EC3.5.1.4)联合作用,将腈类先转化为酰胺,然后再由酰胺酶催化产生羧酸。目前,已报道的微生物中含腈水合酶和酰胺酶的菌株很多,例如Rhodococcus rhodococcus J1、Bacillus sp. BR449、Brevibacterium imperialis、R. eythropolis等[2-4],但在固氮菌中同时具有腈水合酶和酰胺酶的却很少见[5]。
腈水合酶和酰胺酶在生物体内是普遍存在的一类酶类,属于腈水解酶超家族中的一员[6],其中酰胺酶在化学品、生理药物、农药等的合成及污染废水的处理等方面均有广泛的应用。根据催化活性和底物特异性的不同,酰胺酶可分为很多类,有脂肪族酰胺酶、芳香族酰胺酶、α-氨基酸酰胺酶等,烟酰胺酶(NAMase)属于芳香族酰胺酶。酰胺酶的催化反应一般分为2类:水解反应和酰基转移反应,烟酰胺转化成烟酸就是典型的水解反应,产物烟酸对维持微生物细胞中烟酰胺腺嘌呤二核苷酸(NAD)和烟酰胺腺嘌呤二核苷酸磷酸(NADP)内环境稳态发挥着重要作用[7]。在原核生物体内,烟酰胺在NAMase的催化下产生的烟酸可直接进入NAD的生理代谢途径[8],由于哺乳动物体内缺乏NAMase,使NAD的生物合成途径较原核生物复杂[9-10]。NAMase除了能够调控NAD的生物合成之外,对于延长酵母,黑腹果蝇的寿命也具有一定的作用[11-13]。
草木樨剑菌Ensifer meliloti 1021是一类固氮菌,且全基因组序列已公布,基于E. meliloti 1021静息细胞的转化,探索了3-CP的代谢途径并对这一途径中酰胺酶进行了详细研究,测定了NAMase相应的酶学参数,同时NAMase转化吡嗪酰胺的功能为结核病的治疗提供了新的菌株选择。3-CP对烟酸产量的抑制为进一步研究微生物细胞内酶的调控提供了参考价值。
1 材料与方法 1.1 材料E. meliloti 1021,表达质粒pET-28a(+)和菌株E. coli Rosetta(DE3)由本实验室保存。DNA提取试剂盒、RNA提取试剂盒、反转录试剂盒(上海生工),Ni-NTA Fast Start Kit试剂盒(Sigma公司)。3-CP、烟酰胺、烟酸、苯乙酰胺、吡嗪酰胺、吡嗪酸、丙烯酰胺和乙酰胺(Sigma公司,试剂纯度在98%以上);色谱级乙腈(美国Tedia公司);其他试剂为进口或国产分析纯。
1.2 方法 1.2.1 E. meliloti 1021静息转化3-CP和烟酰胺挑取-80℃低温冰箱中保藏的E. meliloti 1021,在LB固体平板上划线,30℃倒置培养至长出单菌落。挑取单菌落接种到含3 mL液体LB培养基(含0.1 mmol/L CoCl2)的试管中,30℃,200 r/min培养24 h。按1%的接种量接种到含有200 mL LB培养基(含CoCl2)的三角瓶中,30℃,200 r/min培养18 h。将培养好的菌液(以OD600=5.0为标准)收集到50 mL离心管中,4℃,8 000 r/min离心5 min,弃去上清,用PBS清洗菌体二次,分别加入5 mL 200 mg/L和1 g/L的3-CP及烟酰胺底物,涡旋振荡混匀,取1 mL做为初始样,每种底物浓度做3个平行,设置底物对照(底物+转化液)和菌体对照(菌体+转化液),分别在1 h、3 h、5 h、7 h取样,每次取样前需补足转化过程中蒸发掉的水分。取出的样13 200 r/min离心10 min,上清液用乙腈(终止反应)稀释。样品用0.22 μm的滤膜过滤后进行HPLC分析。流动相为乙腈溶液(V(乙腈):V(水)=30:70),流速为1 mL/min,紫外线检测波长为230 nm。
1.2.2 nam克隆及其蛋白的表达与纯化从NCBI数据库中搜索“E. meliloti 1021 nicotinamidase”蛋白,下载编码NAMase的基因序列,利用Primer Premier 5软件设计引物并由上海生工生物有限公司合成,上游引物5'-ACAGCAAATGGGTCGCGGATCCGAATTCATGGCCGATGCGGCTCGGC-3',下游引物5'-ATCTCAGTGGTGGTGGTGGTGGTGCTCGAGCTAGATCAGCCGAACGCCG-3'(下划线处碱基为酶切位点序列,酶切位点前序列为pET-28a载体同源序列),PCR扩增程序95℃ 5 min,95℃ 5 s,60℃ 40 s,72℃ 1 min,30个循环。克隆的目的基因片段用EcoRI、XhoI双酶切后,用Clone ExpressⅡ重组克隆试剂盒将目的片段导入pET-28a质粒。根据本实验报道的方法化学转化至E. coli Rosetta(DE3)感受态中[14]。菌落PCR验证阳性克隆,然后送南京思普金生物有限公司测序。
挑取测序正确的阳性克隆接种到3 mL含Kan和Chl的LB液体培养基中,37℃,200 r/min培养24 h。按2%的接种量接种到含Kan和Chl抗性的100 mL LB培养基中,37℃,200 r/min,培养至OD600= 0.6-0.7,加入终浓度为0.2 mmol/L的IPTG溶液,30℃诱导6 h。4℃,8 000 r/min离心5 min,收集菌体,用PBS洗去残留的培养基。Ni-NTA Fast Start Kit试剂盒纯化蛋白,对纯化过程中收集到的流出液进行SDS-PAGE分析。纯化的蛋白用Bradford试剂测定其浓度。
1.2.3 酶学活性分析E. meliloti 1021 NAMase活力通过HPLC分析,以烟酰胺为底物测定生成的烟酸的含量。定义在37℃条件下,1 min内转化1 μmol底物所需的酶量为1个NAMase活力单位,用U表示。
1.2.3.1 pH对NAMase活性的影响配制pH分别为4、5、6、7、8、9、10的200 mg/L烟酰胺转化液,然后测定在不同pH转化液中NAMase的活性,定义pH=7时的相对酶活为100%。反应体系998 µL转化液加入2 µL纯化的NAMase,于37℃恒温孵育器上800 r/min孵育5 min,加入10%的三氯乙酸终止反应。
1.2.3.2 NAMase的pH稳定性在PCR管中加入8 µL纯化的NAMase和92 µL pH分别为4、5、6、7、8、9、10缓冲液,于4℃放置12 h,定义pH=7条件下放置的酶的相对酶活为100%。反应体系975 µL转化液,加入25 µL不同pH条件下处理的酶液。
1.2.3.3 反应温度对NAMase活性的影响转化液底物为pH=7的200 mg/L烟酰胺,反应体系998 µL转化液加入2 µL纯化的NAMase,分别在20,30,40,50,60,70℃反应5 min,定义30℃反应时的相对酶活为100%。
1.2.3.4 NAMase的热稳定性在PCR管中加入30 µL纯化的NAMase,分别置于20,30,40,50,60,70,80℃的水浴锅中孵育3 h,定义30℃条件下孵育的相对酶活为100%。998 µL的反应液加入上述不同温度条件下孵育的酶液2 µL进行反应。
1.2.3.5 金属离子对NAMase活性的影响反应体系为988 µL的转化液加入2 µL NAMase,再分别加入不同金属离子溶液,至金属离子溶液的终浓度为2 mmol/L,37℃恒温孵育器上800 r/min反应5 min,以不加金属离子的相对酶活为100%。
1.2.3.6 有机溶剂对NAMase活性的影响反应体系为978 µL的转化液加入2 µL NAMase,再分别加入不同有机溶液,至有机溶液的终浓度为2%(质量体积比),37℃恒温孵育器上800 r/min反应5 min,定义不加有机溶剂的相对酶活为100%。
1.2.3.7 底物特异性及催化动力学分析分别配制不同的酰胺类底物包括烟酰胺、吡嗪酰胺、苯甲酰胺、丙烯酰胺、苯乙酰胺和甲酰胺,PBS溶解,浓度为200 mg/L。反应体系为998 µL不同的酰胺底物加入2 µL NAMase,37℃恒温孵育器上800 r/min反应5 min。在烟酰胺浓度为0.2-4 g/L下测定NAMase动力学,Vmax及Km根据米氏方程推断。
1.2.4 野生菌株及过表达菌株的生物转化E. meliloti 1021野生菌株在含1 g/L 3-CP LB液体培养基中扩大培养,以OD600=5收集菌体;构建的含NAMase的E.coli在加入诱导剂的同时,加入终浓度为1 g/L 3-CP,诱导6 h后以OD600=3.5收集菌体;分别以1 g/L烟酰胺为底物。以诱导时不加3-CP的菌体为对照组。实验组和对照组各作3个平行。另外,做一组纯化的NAMase转化1 g/L烟酰胺,同时在转化液中加入终浓度1 g/L 3-CP。
1.2.5 3-CP对NAMase的调控机制研究将E. meliloti 1021单菌落接种到20 mL液体LB培养基中,30℃,200 r/min培养24 h,按1%的接种量分别转接到含终浓度为1 g/L 3-CP的LB(实验组)和普通LB(对照组)中,培养18 h。收集菌液,提取总RNA,反转录为cDNA,以16S rRNA基因做内参基因,反应程序为两步法扩增,第一步:预变性,循环数为1,95℃ 30 s。第二步:PCR反应,循环数40,95℃ 5 s,60℃ 30 s。溶解阶段:循环数1,95℃ 15 s,60℃ 1 min,95℃ 15 s。
1.2.6 NAMase固定化研究称取海藻酸钠0.2 g于50 mL烧杯中,加入20 mL无菌水,用微波炉缓慢加热溶解,静止冷却。将清洗好的E.coli Rosetta过表达菌株与海藻酸钠溶液按照1:2的体积混匀,用注射器制备颗粒于提前配制的2% CaCl2溶液中固定1 h。用放有尼龙膜的漏斗过滤除去CaCl2溶液,无菌水清洗颗粒两次,制备的包埋菌体可放于4℃冰箱储存。纯化后的NAMase按上述条件进行固定化。
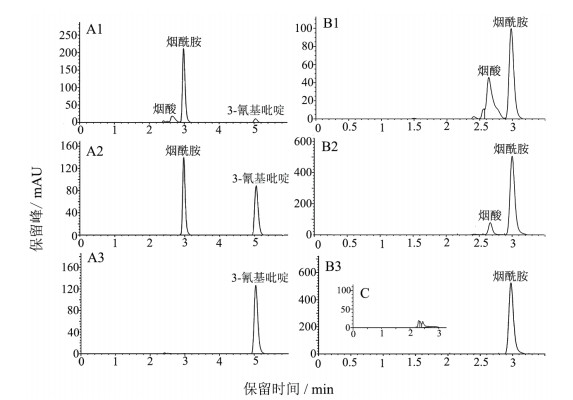
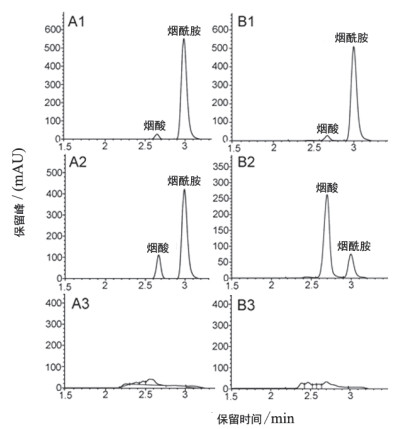
2 结果 2.1 E. meliloti 1021静息转化3-CP和烟酰胺图 1-A1显示以200 mg/L 3-CP为底物时终产物是烟酸,在3 h时烟酸的产量为0.29 mmol/L,转化率为94.2%;以1 g/L 3-CP为底物时只能产生烟酰胺,到12 h时还无烟酸保留峰的出现(图 1-A2)。E. meliloti 1021静息细胞分别以200 mg/L和1 g/L烟酰胺为底物时,均出现了烟酸的保留峰,其中以200 mg/L烟酰胺为底物时3 h烟酸的产量为0.47 mmol/L,转化率为47.5%(图 1-B1);以1 g/L烟酰胺为底物时3 h烟酸的产量为0.36 mmol/L,转化率为14%,高浓度的烟酰胺也会对酰胺酶产生一定的抑制作用。
|
| A1,A2:分别以200mg/L和1g/L 3-CP为底物进行转化;A3: 1g/L 3-CP底物对照;B1,B2:分别以200mg/L和1g/L烟酰胺为底物进行转化;B3: 1g/L烟酰胺底物对照;C:E. meliloti 1021菌体对照 图 1 E. meliloti 1021静息转化3-CP和烟酰胺 |
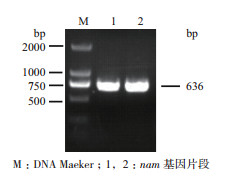
以E. meliloti 1021基因组DNA为模板,根据设计的引物扩增获得nam序列,琼脂糖凝胶电泳显示目的片段长约636 bp(图 2),导入EcoRI、XhoI双酶切的pET-28a载体,然后转入E.coli Rosetta(DE3)感受态细胞,阳性克隆片段大小符合预期。
|
| 图 2 PCR扩增nam基因的电泳结果 |
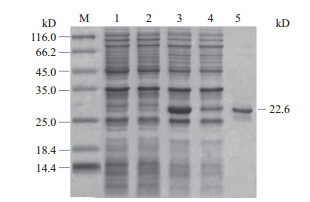
由nam编码的NAMase在30℃,0.2 mmol/L IPTG的条件下,在E.coli Rosetta-pET28a中表达情况良好,从图 3中可以看出E.coli Rosetta- pET28a空载中没有目的条带,说明pET-28a在E.coli Rosetta没有目的蛋白表达。SDS-PAGE显示NAMase蛋白分子量约22.6 kD,Bradford法测定蛋白浓度为1.13 mg/mL。
|
| M:蛋白marker;1:E. coli pET28a全蛋白;2:E. coli pET28a可溶性蛋白;3:E. coli pET28a-nam全蛋白;4:E. coli pET28a-nam可溶性蛋白;5:纯化的NAMase 图 3 NAMase的SDS-PAGE图 |
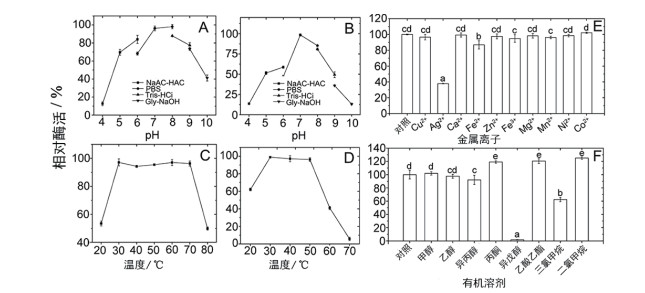
pH和温度对NAMase的影响:图 4-A显示在pH7-8时,酶的稳定性最好;该酶的最适pH是7,在pH小于6和大于8的条件下,酶的活性会迅速下降(图 4-B)。在30-70℃之间NAMase对底物烟酰胺的活性最高,该酶在30-50℃之间孵育2 h后的转化活性仍在97.1%以上(图 4-D)。
|
| A:pH稳定性;B:最适pH;C:最适温度;D:热稳定性;E:金属离子对NAMase影响;F:有机溶剂对NAMase的影响。 图 4 NAMase酶学性质分析 |
金属离子和有机溶剂对NAMase的影响:从图 4-E可知Fe对NAMase活性有抑制,其中Fe3+抑制率为5.2%,作用不明显,Fe2+抑制率为13.1%;Ag2+对NAMase抑制作用更明显,高达72.2%,而Mg2+(98.3%)、Zn2+(97.5%)和Co2+(102.2%)对该酶活性影响不大。图 4-F显示异戊醇对NAMase活性的抑制率最大,达到98.4%,三氯甲烷抑制作用为38.1%,丙酮(119.5%)、二氯甲烷(125.1%)及乙酸乙酯(120.5%)对NAMase的活性有促进作用。
底物特异性及动力学参数:E. meliloti 1021 NAMase能将烟酰胺和吡嗪酰胺分别转化为烟酸和吡嗪酸,对上文提到的其他几种酰胺底物均无代谢活性(表 1)。对纯化后的NAMase测定相关的动力学参数,Kcat / Km为0.42 mmol/L/s,Km为2.28 mmol/L,Vmax为142.86 U/mg。
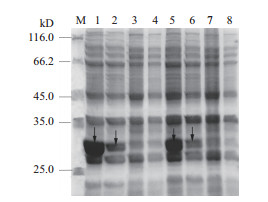
E. meliloti 1021在加入3-CP的LB中培养时,静息转化产生的烟酸0.15 mg/L(图 5-A1),用普通LB中培养的E. meliloti 1021静息转化,烟酸产量为0.45 mg/L(图 5-A2),3-CP对NAMase活性抑制率达到了66.7%。图 5-B1显示E.coli Rosetta-NAMase过表达菌株诱导时加入3-CP,收集菌体进行转化,其烟酸的产量为0.14 mg/L,而诱导时不加3-CP,E.coli Rosetta-NAMase过表达菌株转化1 g/L烟酰胺产生1.35 mg/L烟酸(图 5-B2),3-CP对E.coli Rosetta中过表达的NAMase活性抑制率达到了89.6%。纯化的NAMase在转化烟酰胺的过程中,3-CP不影响烟酸的产量。从图 6 SDS-PAGE的泳道2和6可知诱导时3-CP对NAMase可溶性蛋白表达影响不大。
|
| A1:E. meliloti 1021培养时加入1g/L 3-CP;A2:E. meliloti 1021普通LB培养;A3:E. meliloti 1021菌体对照。B1:E. coli过表达NAMase诱导时加入IPTG和1g/L 3-CP;B2:E. coli过表达NAMase诱导时只加入IPTG;B3:过表达的E. coli菌体对照。 图 5 不同条件转化1g/L烟酰胺 |
|
| M:蛋白Marker;1和3:诱导时只加入IPTG的过表达NAMase和E. coli pET-28a的全蛋白;2和4:诱导时只加入IPTG的过表达NAMase和E. coli pET-28a的可溶性蛋白;5和7:诱导时加入IPTG和1g/L 3-CP的过表达NAMase和E. coli pET-28a的全蛋白;6和8:诱导时加入IPTG和1g/L 3-CP的过表达NAMase和E. coli pET-28a的可溶性蛋白;注:箭头处为目标蛋白 图 6 不同条件下的NAMase的SDS-PAGE分析 |
提取的实验组菌体中的RNA浓度为81.3 ng/μL,OD260 / OD280=2.04;对照组菌体中的RNA浓度为103.2 ng/μL,OD260 /OD280=2.07;从转录水平分析3-CP对NAMase表达量的影响结果如下:把细菌培养时未加3-CP对照组NAMase的表达量定义为1,培养时加入3-CP的实验组NAMase表达量为1.18。
2.6 NAMase固定化对酶活的影响用最适包埋条件(2%海藻酸钠和2% CaCl2)对过表达菌株及纯化后的蛋白分别进行包埋,结果发现包埋的纯化后的酶活只有纯酶酶活的1/3,但是固定化的在E.coli Rosetta中过表达的NAMase(转化率为94%)与自由细胞(转化率为95.1%)相比活性并未下降。
3 讨论E. meliloti 1021静息细胞转化200 mg/L 3-CP终产物是羧酸,以烟酰胺为底物时也能产生烟酸。在这一代谢过程中可能存在腈水解酶或腈水合酶和酰胺酶,进一步查找E.meliloti 1021的全基因组(NC_003047.1)发现没有编码腈水解酶的基因。E. meliloti 1021催化1 g/L 3-CP产生烟酰胺,没有烟酸产生,在这一过程推测3-CP会抑制NAMase的活性,不同浓度的烟酰胺底物其转化率不同,烟酰胺对烟酸的产生也具有抑制作用。有研究称3-CP作为底物时在转化的过程中会出现底物抑制现象,酰胺水解酶也会被大量的烟酰胺类似物抑制,其中3-CP的抑制常数为2.2 μmol/L[15]。野生菌株和过表达菌株在培养及诱导时加3-CP的对比结果显示3-CP会抑制烟酸的产量,在转录水平及纯化的NAMase在转化烟酰胺时不受3-CP的干扰,推测3-CP在转化的过程中并非是底物抑制效应。
克隆并异源表达了E. meliloti 1021 NAMase的基因,诱导后分离纯化NAMase研究其酶学性质,实验发现在pH=7时活性最高。该酶在30-70℃之间酶活均维持在很高的活性,与Oceanobacillus iheyensis HTE831酰胺酶(55℃以上,酶活大幅度下降)及一些最适温度在30-40℃的酰胺酶相比在高温下更具有优势[16-18]。NAMase是一类金属离子依赖性的酶,大部分这类酶的活性中心含有Zn2+[7, 10],有研究表明Fe2+可增加NAMase的活性[19],本实验Fe2+会轻微抑制E. meliloti 1021 NAMase的活性,抑制率为13%。底物特异性与Mycobacterium tuberculosis中NAMase相似[20],这与酶本身的活性位点残基有关[16]。目前报道的吡嗪酰胺酶大都来自于M. tuberculosis[22-24],E. meliloti 1021 NAMase转化吡嗪酰胺的活性比Zhang等[20]研究中吡嗪酰胺酶的活性(81.9 U/mg)高1.8倍,为结核病治疗提供了新的目标菌株选择。固定化的E.coli过表达菌株对烟酰胺具有很高的活性,实现了酶反复利用的可能。
4 结论从E. meliloti 1021中克隆了长636 bp的NAMase基因,编码的蛋白分子量为22.6 kD,PI为5.5。利用亲和层析柱纯化异源表达的NAMase然后测定酶活,该酶的最适pH为7,30-70℃之间NAMase对底物烟酰胺的活性最高,在30-50℃之间孵育2 h后NAMase活性仍保持在97.1%以上,Ag2+和异戊醇会抑制NAMase的活性。NAMase不仅能转化烟酰胺,对吡嗪酰胺也具有很高的活性。高浓度3-CP和烟酰胺对E. meliloti 1021野生菌株代谢酰胺有抑制作用,固定化的E.coli NAMase过表达菌株对烟酰胺转化率达到94%。
| [1] |
Vaughan PA, Knowles CJ, Cheetham PSJ. Conversion of 3-cyanopyridine to nicotinic acid by Nocardia rhodochrous LL100-21[J]. Enzyme & Microbial Technology, 1989, 11(12): 815-823. |
| [2] |
Kim SH, Oriel P. Cloning and expression of the nitrile hydratase and amidase genes from Bacillus sp. BR449 into Escherichia coli[J]. Enzyme and Microbial Technology, 2000, 27: 492-501. DOI:10.1016/S0141-0229(00)00248-9 |
| [3] |
Alfani F, Cantarella M, Spera A., et al. Operational stability of Brevibacterium imperalis CBS489-74 nitrile hydratase[J]. Journal of Molecular Catalysis(B: Enzymatic), 2001, 11(4/6): 687-697. |
| [4] |
Park HJ, Park H, Rutger JF, et al. Characterization of nitrile hydrolyzing enzymes produced from Rhodococcus erythropolis[J]. Korean Society for Microbiology and Biotechnology, 2006, 34(3): 204-210. |
| [5] |
Prasad S, Bhalla TC. Nitrile hydratases(NHases): At the interface of academia and industry[J]. Biotechnology Advances, 2010, 28(6): 725-741. DOI:10.1016/j.biotechadv.2010.05.020 |
| [6] |
Pace HC, Brenner C. The nitrilase superfamily: classification, structure and function[J]. Genome Biology, 2001, 2(1): 1-9. |
| [7] |
Anderson RM, Bitterman KJ, Wood JG, et al. Nicotinamide and PNC1 govern lifespan extension by calorie restriction in Saccharomyces cerevisiae[J]. Nature, 2003, 423(6936): 181-185. DOI:10.1038/nature01578 |
| [8] |
French JB, Cen Y, Sauve AA, et al. High-resolution crystal structures of Streptococcus pneumoniae nicotinamidase with trapped intermediates provide insights into the catalytic mechanism and inhibition by aldehydes[J]. Biochemistry, 2010, 49(40): 8803-8812. DOI:10.1021/bi1012436 |
| [9] |
Rongvaux A, Shea RJ, Mulks MH, et al. Pre-B-cell colony-enhancing factor, whose expression is up-regulated in activated lymphocytes, is a nicotinamide phosphoribosyl transferase, a cytosolic enzyme involved in NAD biosynthesis[J]. European Of Immunology, 2002, 11(32): 3225-3234. |
| [10] |
Belenky P, Bogan KL, Brenner C. NAD+ metabolism in health and disease[J]. Trends in Biochemical Sciences, 2007, 32(1): 12-19. DOI:10.1016/j.tibs.2006.11.006 |
| [11] |
Smith BC, Anderson MA, Hoadley KA, et al. Structural and kinetic isotope effect studies of nicotinamidase(Pnc1)from Saccharomyces cerevisiae[J]. Biochemistry, 2012, 51(1): 243-256. DOI:10.1021/bi2015508 |
| [12] |
Balan V, Miller GS, Kaplun L, et al. Life span extension and neuronal cell protection by drosophila nicotinamidase[J]. Journal of Biological Chemistry, 2008, 283(41): 27810-27819. DOI:10.1074/jbc.M804681200 |
| [13] |
Rogina B, Helfand SL, Ames BN. Sir2 mediates longevity in the fly through a pathway related to calorie restriction[J]. Proc Natl Acad Sci USA, 2004, 101(45): 15998-16003. DOI:10.1073/pnas.0404184101 |
| [14] |
Ge F, Zhou LY, Wang Y, et al. Hydrolysis of the neonicotinoid insecticide thiacloprid by the N2-fixing bacterium Ensifer meliloti CGMCC 7333[J]. International Biodeterioration & Biodegradation, 2014, 93: 10-17. |
| [15] |
Mathew CD, Nagasawa T, Kobayashi M, et al. Nitrilase-catalyzed production of nicotinic acid from 3-cyanopyridine in Rhodococcus rhodochrous J1[J]. Applied & Environmental Microbiology, 1988, 54(4): 1030-1032. |
| [16] |
Sánchezcarrón G, Garcíagarcía MI, Zapatapérez R, et al. Biochemical and mutational analysis of a novel nicotinamidase from Oceanobacillus iheyensis HTE831[J]. PLoS One, 2013, 8(2): e56727. DOI:10.1371/journal.pone.0056727 |
| [17] |
Pardee AB, Benz EJ, Stpeter DA, et al. Hyperproduction and purification of nicotinamide deamidase, a microconstitutive enzyme of Escherichia coli[J]. Journal of Biological Chemistry, 1971, 246(22): 6792-6796. |
| [18] |
Yan C, Sloan DL. Purification and characterization of nicotinamide deamidase from yeast[J]. Journal of Biological Chemistry, 1987, 262(19): 9082-9087. |
| [19] |
Somoskovi A, Wade MM, Sun Z, et al. Iron enhances the antituberculous activity of pyrazinamide[J]. Journal of Antimicrobial Chemotherapy, 2004, 53(2): 192-196. DOI:10.1093/jac/dkh042 |
| [20] |
Zhang H, Deng JY, Bi LJ, et al. Characterization of Mycobacterium tuberculosis nicotinamidase/pyrazinamidase[J]. FEBS Journal, 2008, 275(4): 753-762. DOI:10.1111/j.1742-4658.2007.06241.x |
| [21] |
Ion BF, Kazim E, Gauld JW. A multi-scale computational study on the mechanism of streptococcus pneumoniae nicotinamidase(SpNic)[J]. Molecules, 2014, 19(10): 15735-15752. DOI:10.3390/molecules191015735 |
| [22] |
Frothingham R, Meekero'Connell WA, Talbot EA, et al. Identification, cloning, and expression of the Escherichia coli pyrazinamidase and nicotinamidase gene, pncA[J]. Antimicrobial Agents & Chemotherapy, 1996, 40(6): 1426-1431. |
| [23] |
Sheen P, Ferrer P, Gilman RH, et al. Role of metal ions on the activity of Mycobacterium tuberculosis pyrazinamidase[J]. American Journal of Tropical Medicine & Hygiene, 2012, 87(1): 153-161. |
| [24] |
Ahmady A, Poolad T, Rafee P, et al. Study of pyrazinamidase structural changes in pyrazinamide resistant and susceptible isolates of Mycobacterium tuberculosis[J]. Tuberk Toraks, 2013, 61(2): 110-114. DOI:10.5578/tt.3888 |





