乳酸菌(Lactic acid bacteria,LAB)是指一类利用可发酵糖产生大量乳酸的菌群,通常为革兰氏阳性菌,厌氧或兼性厌氧[1],主要分布在乳杆菌属、链球菌属、明串珠菌属、双歧杆菌属及片球菌属等几个属中[2-3]。目前我国卫生部已将34种微生物菌种列入《可食用菌种名单》,其中至少有28个菌种属于乳酸菌,而可用于保健食品的11种益生菌菌种全部为乳酸菌。乳酸菌可以在泌尿、生殖系统中或黏膜部位黏附存活且无病原性[4],可调节人体免疫能力,降低胆固醇、抗肿瘤,改善肠道菌群[5-6]。目前乳酸菌主要应用于食品工业生产中,在乳制品加工、果蔬发酵、酿造工业及肉制品生产领域应用广泛[7]。据统计预测,我国乳酸益生菌类产品市场规模在2020年将接近850亿元[8]。由此可见,我国乳酸菌产业发展蒸蒸日上,未来的机遇指日可待。
众多实验表明乳酸菌存在大量保健治疗功能效果,可面向不同层面人群。例如,乳酸片球菌(Pediococcusacidilactici)K15适用于免疫力低下人群,可增加唾液中的免疫球蛋白A(Immunoglobulin A,IgA),抑制病原体侵染宿主[9];芽孢杆菌(Bacillus)益生菌适用于肠道炎症人群,可清除肠道中金黄色葡萄球菌(Staphylococcus aureus),维持肠道菌群共生[10];植物乳杆菌(Lactobacillus plantarum)229v可改善重度抑郁症患者的认知功能,提高其注意力与理解力[11]。但在乳酸菌研究过程中,存在筛菌技术繁复、周期长、成功率低等难点,特别是缺少稳定繁殖特定功能乳酸菌菌株,这些都制约了乳酸菌在行业中的发展。因此,寻找行之有效的特定功能乳酸菌菌株开发方法成为了推动乳酸菌行业发展的一个重点。
合成生物学是一个新兴的研究领域,它将分子生物学、工程学、数学、化学、信息学等学科知识与实践结合在一起,为解决乳酸菌发展难题提供了一个新机遇。早在1980年,Hobom[12]和Bemer等[13]在描述转基因细菌时就提出了“合成生物学”的概念,而今已被广泛接受和应用。合成生物学可以将生物组件组装集合,以生物电路的形式来产生预测效果[14]。目前,组件主要由DNA、RNA、蛋白质等分子组成,可在电路中互换位置。通过重组、整合与鉴定,创造出全新生物模块、网络、体系和生物体细胞。合成生物学在大量科研案例中已有重大突破,如药物研发,生产胰岛素挽救糖尿病患者的生命[15-16];能源生产,使用枯草芽孢杆菌(Bacillus subtilis)有效地捕获植物生物聚合物(如纤维素)中储存的能量[17];生物修复,设计微生物来修复一些最危险的环境污染物,包括重金属和沙林等神经毒剂等[18]。乳酸菌作为当今研究热点,应用合成生物学技术对其进行改造,按照人类需求进行相应表达,对开发乳酸菌菌株有很大意义。
本文将总结乳酸菌在合成生物学领域的突出优势、构建方法以及生产应用,根据大量实验证实工程化乳酸菌的利用价值,为其后续研究进展、药物开发等方面提供相应依据[19-22]。
1 工程化乳酸菌的优势及特点合成生物学底盘即为可进行基因改造及组件引入的受体微生物细胞,可为细胞生长及组件工作提供各种组分,且足够稳定以便能在各个领域中应用[23]。目前大肠杆菌、铜绿假单胞菌等常被用作细胞底盘,其性能优良,具有广阔的设计空间、遗传部位和调控元件,繁殖能力迅速且合成蛋白质稳定,可高度适应实验室条件[24]。但存在一些安全方面的担忧:其一,这些细菌自身可能释放内毒素,存在未知的隐患问题;其二,这些细菌基因工程操作过程中主要应用抗生素抗性基因作为选择标记,具有转移到环境微生物中的风险,而工程化乳酸菌在合成生物学中具有巨大的优势。
1.1 安全性乳酸菌作为食品工业中安全系数较高的细菌,广泛应用于食品工业制造中,诸多实验证实其安全健康,不会产生致病物质[25]。在分子改造中,需要具备系统性基因编辑的工具,要求其达到食品级别,对人体无毒无害[26]。同时,一些乳酸菌载体具有在窄宿主范围内进行复制的特点,例如来自卷曲乳杆菌(Lactobacillus crispatus)的316 kb大小的质粒载体,只能在它原始的宿主菌中才能复制;分离自罗伊氏乳杆菌(Lactobacillus reuteri)的载体pLUL631,其可在罗伊氏乳杆菌和发酵乳杆菌(Lactobacillus fermentum)与及其他受试的革兰氏阳性菌中复制。这种特性使得细菌种间水平传播发生的机会较少,更具安全性[27]。
1.2 革兰氏阳性菌优势目前合成生物学菌株的开发主要以革兰氏阴性细菌为底盘细胞,其结构相对简单,细胞壁薄易操作且基因组库庞大。与革兰氏阴性菌相比,革兰氏阳性细菌的细胞壁较厚,可有效抑制机械损伤与渗透损伤,能否成功转化是普遍关心的问题。在实际应用中,革兰氏阴性菌的细胞壁结构不适用于全部合成生物学系统,但一些革兰氏阳性菌却在研究过程中发挥优势,如益生菌罗伊氏乳杆菌为革兰氏阳性菌,其细胞壁可模仿金黄色葡萄球菌表征agr(accessory gene regulatory system)群体感应的方式,来检测金黄色葡萄球菌产生的群体感应分子自诱导肽-I(Autoinducer peptide-I,AIP-I)[20]。此外,乳酸菌不含有附着于细胞膜的脂多糖(Lipopolysaccharides,LPS)。LPS是革兰氏阴性细菌外膜的组成部分,可导致细胞因子紊乱,从而引发心血管衰竭和血压不稳定,并最终导致致命性败血症综合征[21]。人体摄入的乳酸菌,则不含有这种内毒素,从而避免过敏性休克[22],可将乳酸菌广泛用于疾病治疗中。
1.3 食品发酵特殊性大肠杆菌、铜绿假单胞菌等在环境修复、污水治理等方面有较大可行性,但不适宜用于食品和医药领域。乳酸菌多用于发酵以及酿造生产,除了可以提高营养价值外,还兼备了产生乳酸、蛋白质、形成风味物质和产生抑菌物质等作用[28]。现今国内外对乳酸菌的研究越来越深入,在食品生产中可利用合成生物技术对乳酸菌进行相应改造,投入到发酵、酿造工业中,进行定位定点发酵不易合成产物。例如,Rico等[29]构建了插入乳酸脱氢酶ldh基因的LAB菌株,通过非外消旋法发酵L-乳酸,用作制药与食品生产,生产效率极高。就目前乳酸菌研究现状来看,可在其底盘上插入对抗肠胃疾病、糖尿病等疾病的特定基因,利用乳酸菌发酵原理,将药物性能体现在乳制品中,方便高效,同时增强口感,为食品与药物发展提供新思想。
1.4 肠道内定植性乳酸菌可以在宿主体内进行定植。其发挥作用的前提是可以到达肠道后进行黏附,与宿主肠上皮细胞牢固结合,避免其被胃酸、胆汁等胃肠道分泌的液体和肠道的蠕动所清除。而后乳酸菌充分适应肠道环境,进行繁殖,在黏附膜表面形成微生物膜,防止外来菌入侵,保护肠黏膜的健康。Russo等[30]使用斑马鱼模型研究了乳酸益生菌在体内定植能力。利用植物乳杆菌Lp90,植物乳杆菌B2和发酵乳杆菌PBCC11.5中的pRCR12质粒进行荧光标记,从而监测乳酸菌的黏附定能力结果发现,乳酸菌可快速定植且植物乳杆菌菌株表现出更强的黏附能力。Prado-Rebolledo等[31]研究了乳酸菌对肉鸡肠道沙门氏菌肠炎沙门氏菌肠道定植和肠道通透性的影响,将食用乳酸菌与肠炎沙门氏菌肉鸡组和食用对照与肠炎沙门氏菌肉鸡组进行比较,观察到盲肠中肠炎沙门氏菌数量的减少,且在实验的72 h周期内,乳酸菌可稳定定植。乳酸菌在肠道内进行稳定快速定植对其益生作用具有很大的意义。
2 乳酸菌合成生物学发展进程乳酸菌的应用历史十分久远,但人类能够科学地研究并利用乳酸菌却始于19世纪中叶。而对于乳酸菌合成生物学研究而言,在30多年发展期间有着重大突破。
2.1 乳酸菌基因敲除阶段20世纪末期,随着分子生物学逐渐发展,人们更加清晰地了解了乳酸菌在遗传基因水平的代谢规律。早期人们运用基因敲除技术研究乳酸菌功能作用。例如,Ferain等[32]将两个ldh基因敲除掉,研究其对植物乳杆菌中肽聚糖前体合成的影响。2001年,Bolotin等[33]完成了对乳酸乳球菌乳酸亚种IL1403的测序。这是对于乳酸菌的第一例测序,在这个过程中发现乳酸菌基因组中存在某些非必需基因,如次级代谢物基因簇、冗余序列等[34]。在这个过程中发现可将非必需基因敲除掉,促进乳酸菌基因组的简化,同时基因敲除技术可提高乳球菌染色体在遗传分析中作用的成功率[35],使得乳酸菌更容易适应丰富的营养环境[36]。在这一时期,乳酸菌基因改造中研究最多的为筛选标记基因,如红霉素[37]、氯霉素[38]等标记基因,这对鉴别受体细胞中是否含有目的基因起到识别与筛选作用。而经过基因改造过的细菌,在1987年Flickinger等[39]首次报道的乳酸菌电转化方法中,可成功转化到乳酸菌底盘中。这些研究为乳酸菌基因工程的发展奠定了坚实的基础。
2.2 工程化乳酸菌阶段随着转基因技术发展热潮,对乳酸菌的研究较多为乳酸菌基因工程技术或工程化乳酸菌技术。在这一时期,乳酸菌载体构建及应用成为该领域研究的前沿和热点。乳酸菌重组需要用到特定的重组基因载体质粒,将相应片段进行酶切,同时连接上目的基因片段,在质粒上进行基因改造。乳酸菌重组技术具有周期短、代谢快等特点。在这个过程中,其对于食品工业发展有重要作用,如高效过量产生胞外聚合物(Extracellular polymeric substances,EPS),可改善酸奶品质[40];乳球菌菌株代替添加α-酮戊二酸来改善奶酪的香气[41];增加肽酶活性,可缩短干酪成熟期,生产具有改善特性的特殊奶酪[42]。同时也发现,电转化成功率存在弊端,Rattanachaikunsopon团队[43]研究了一种利用玻璃珠震荡产生能量的方法进行转化,使其转化效率大大提高。这一时期为向合成生物学转化的过渡时期,重组乳酸菌的研究在逐渐向应用方面发展。
2.3 乳酸菌合成生物学阶段发展至今,乳酸菌合成生物学逐渐替代传统改造基因的概念,不仅能够优化乳酸菌菌种,构建遗传密码,提高对底物和能量的利用效率,同时也可以利用宿主体内自身代谢机制,通过外源物质刺激进行宿主自身调控。在这个阶段乳酸菌可以使用不同表达系统的载体质粒,还可对其进行优化,强启动子[44]的鉴别与筛选使得质粒可高效表达,提高其对外界环境的抗干扰性。除对表达元件进行了优化,载体构建开始考虑在食品生产应用中易存在的缺陷,食品级乳酸菌表达载体的构建得到研究者的重视[45-46]。这个过程中,医学与生物学方面得到乳酸菌合成生物学技术的支持,例如乳酸菌分泌胰高血糖素样肽-1(Glucagon-likepeptide1,GLP-1)使大鼠的血糖明显下降[47];重组乳酸乳球菌NZ9000预防高血糖症[48]等(图 1)。因此21世纪初期,研究人员致力于通过对乳酸菌基因组学的研究及基因工程技术的应用,改良现有的乳酸菌菌种,从而选育出集多种优势基因的对社会带来更大效益的乳酸菌新菌种。
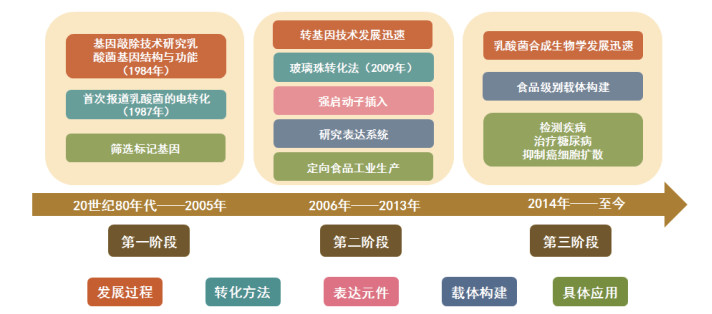
|
| 图 1 乳酸菌合成生物学发展进程 |
对于一个生物系统来说,层次化的结构是构成整体的本质。具有一定功能的DNA序列可组成最简单基因表达元件(Part);不同功能的基因表达元件按照一定的逻辑和物理顺序连接组成复杂的生物装置(Decice);而不同功能的生物装置协同运作组成更加复杂的生物系统(System),含有多种不同功能系统的生物体彼此通讯互相协调组成更复杂的多细胞或细胞群体生物系统。因此,工程化乳酸菌表达元件的设计、载体的选择,以及最终的转化、整合变得十分重要。
3.1 表达元件设计合成生物学的特点即为简便性和标准化,通过建立标准化的生物元件,形成规模体系,从而完成构建过程,达到生物合成的目的[49]。在设计细菌表达载体时,需要具备几个结构元件,如启动子(Promoter)、核糖体结合位点(Ribosome binding site)、终止子(Terminator)、操纵子(Operon)等调控元件,同时需要质粒载体骨架序列及蛋白质结构域等,实现基因电路的表达。
现如今已经从乳酸菌中分离出大量不同强度的组成型启动子和终止子在菌种中进行表达。植物乳杆菌可在工业、医学及生物技术领域中表达重组蛋白,而表达水平的控制关键取决于启动子强度、基因拷贝及翻译效率。Tauer等[44]从植物乳杆菌033和布氏乳杆菌(Lactobacillus buchneri)034中分离出强启动子(如转录延伸因子启动子),在植物乳杆菌033中都可用于表达蛋白质,且在同等条件下,高拷贝数的复制起点使表达量增加了两倍。同时使用诱变启动子的方法,可以使乳酸乳球菌(Lactococcus lactis)和植物乳杆菌产生更加庞大的组成型启动子文库。
3.2 载体的选择要形成一个完整的系统,就需要载体来承载各种元件。载体是指按照人类意愿有目的地进行改造,携带外源基因,可创建生物遗传性的运载工具。这种运载工具需要具备自主复制能力,且有高效存活率,方便操作,易于识别等特点。乳酸菌作为广泛作用的益生菌,其表达载体主要可分为非食品级乳酸菌表达载体和食品级乳酸菌表达载体两类[45-46, 50]。为了达到筛选特定菌株的目的,通常会将具有抗性筛选标记的基因连接在载体上,得到相应特定菌株[51]。这样的载体通常为非食品级乳酸菌表达载体,其中常见的抗性筛选基因为红霉素及氯霉素等基因,易转移到环境与内源微生物中。
在食品工业生产中,为了避免因食品安全引发的问题,载体必须为食品级乳酸菌表达载体,遗传特性清楚,且能稳定遗传,可为人类食用。近几年人们就食品工业生产的应用来研究乳酸菌食品级载体,将食品级选择标记基因转入载体中。例如,以乳酸乳球菌NZ3900为宿主菌的食品级乳酸菌表达载体pNZ8149[45],大小为2.5 kb,在Nco Ⅰ位点后携带有Nisin诱导的启动子PnisA,并含有lacF基因,而它则以乳糖作为选择标记,通过乳糖物质进行筛选。Takala等[46]构建的乳酸菌食品级质粒pLEB590,主要由pSH71复制子,抗性nisI基因和用于nisI表达的组成型启动子P45组成。之后将质粒pLEB590成功电转乳酸乳球菌和植物乳杆菌中,发现pLEB590这一食品级别载体在两种底盘中都可以进行成功表达。这些研究表明,食品级选择标记基因已进行了大量研究,将其插入载体上可得到安全系数较高的乳酸菌,可满足人类对食品生产的要求,同时便于识别筛选,简化合成生物学的操作。
3.3 转化方法在合成生物学中,当载体上插入相应启动子、报告基因、核糖体结合位点及终止子等元件后,要想使其进行相应表达,则需要通过转化将载体DNA导入到细菌底盘中。这一步是使得宿主细胞含有目的基因最关键的一步。合成生物学中最常用的大肠杆菌在进行转化时通常使用热激法。但由于乳酸菌有较厚的细胞壁,可阻碍DNA分子进入细菌,普通热激法无法有效地使外源DNA进入受体细胞,因此寻找行之有效的方法变得尤为重要。
目前报道的乳酸菌转化方法主要有3种,原生质体转化法、电转化法以及玻璃珠转化法。前者操作复杂且转化率低,无法在工业化生产中得到广泛使用。1987年,Flickinger等[39]首次报道了对干酪乳杆菌(Lactobacillus casei)的电转化,这是一次值得鼓舞的进步。然而乳酸菌电转化方法容易受到诸多因素的影响,例如细胞弱化剂的种类及浓度、细胞的生长状态、电击条件、质粒浓度、质粒本身构建条件等。目前已有的研究多是在细胞弱化剂、细胞生长状态、电击条件等方面进行优化与完善。如贾士芳等[52]对嗜酸乳杆菌(Lactobacillus acidophilus)JL-1的电转化条件进行了研究,指出使用氨苄青霉素处理转化受体能有效提高转化效率,而使用甘氨酸处理则无显著影响。
虽然电转化法被认为是一种流行的乳酸菌转化方法,但其设备昂贵、转化效率低。玻璃珠转化法是在DNA和聚乙二醇存在下,将细菌原生质体与玻璃珠进行搅拌,使得载体转入细菌底盘中。Rattanachaikunsopon等[43]通过使用革兰氏阳性细菌作为pGK12的受体进行玻璃珠转化(pGK12是携带红霉素抗性基因的4.4 kb大肠杆菌/乳球菌穿梭载体),将其成功转化到粪肠球菌TISTR 927、干酪乳杆菌ATCC 393、乳酸乳球菌DSM 20481、葡聚糖链球菌ATCC 19255、无害李斯特菌DSM 20649、金黄色葡萄球菌ATCC 25923和肺炎链球菌ATCC 10015中。之后,该团队再次利用pGK12质粒,运用玻璃珠转化法将其成功转化到葡聚糖明串珠菌ATCC 19255乳酸菌菌株中[53]。这一发现,使得乳酸菌转化提高了重复性与效率,通过不断优化与改进,可将其运用到工业生产中。
3.4 基因编辑技术目前,基因编辑技术逐步成为合成生物学主要技术之一。基因编辑是指对目的基因进行插入、敲除或替换等方式,来满足人类特定需要的技术。
3.4.1 同源重组早在20世纪80年代,发展起一种新型分子生物学技术手段,即为基于同源重组的基因敲除技术。同源重组依赖于基因同源序列的联会,DNA分子间或者分子内交换对等的部分,发生重新组合。运用这种技术,可将乳酸菌的代谢流向发生改变,食品工业生产中能有效降低发酵成本,提高产物的生产水平及纯度。乳酸链球菌(Streptococcus Lactis)712的乳糖质粒容易发生缺失现象,对于外源基因转化进入菌株中的方法来说,存在弊端,因此将乳酸链球菌712、乳酸链球菌C2和乳脂链球菌(Streptococcus cremoris)C3乳糖质粒的乳糖和蛋白酶基因进行转导,从而插入染色体中,构建扩大的乳糖质粒[54]。Leenhouts等[35]则进行同源重组单交换双交换进行基因敲除,并将其称之为Campbell型整合,在乳酸乳球菌中对Campbell型质粒进行基因敲除,可提高乳球菌在遗传分析中的染色体改造成功率,同时有助于稳定乳球菌中的不稳定基因。
3.4.2 单链DNA(ssDNA)重组工程同源重组技术虽然得到广泛的应用,但是其中的工具大多用于单基因操作,而在基因组突变中,如通路工程和基因电路工程突变中,则需要额外的工具[55]。一些运用于工业与医学的细菌菌株,需要高效且精准的进行编辑基因组来及时改善,增强效果。在各种乳酸菌中,可以通过ssDNA重组工程在染色体中产生突变来实现[56]。ssDNA重组工程是一种在细菌染色体上进行细微变化的技术,其中表达单链DNA结合蛋白(RecT或Bet)的细胞是通过寡核苷酸进行转化,而该寡核苷酸主要通过退火和复制进行结合。Van Pijkeren等[57]首先鉴定了乳杆菌属和乳酸乳球菌属中的ssDNA结合蛋白同源物,之后为了评估能否进一步提高罗伊氏乳杆菌ATCC PTA 6475的重组工作效率,就在该菌株中表达了几种RecT同源物。其中来自粪肠球菌(Enterococcus faecalis)19的RecT与天然RecT蛋白相比产生相当的效率,但是其他蛋白没有提高重组率。因此他们通过增加寡核苷酸浓度以及使用含有硫代磷酸酯键的寡核苷酸,成功地将乳酸乳球菌的重组工作效率提高了10倍。该研究表明通过ssDNA重组工程对乳酸菌进行定向遗传修饰可以简化菌株改良的步骤,提高其工作效率及成功率。
同时研究发现,ssDNA重组工程可诱导噬菌体衍生ssDNA结合蛋白(RecT或β)的表达[58-60]。当寡核苷酸存在于细胞中,ssDNA可结合蛋白对寡核苷酸进行保护作用,避免其被宿主核酸酶降解,有助于寡核苷酸与模板DNA形成复合物[61-62]。这对于基因编辑技术而言,又得到了进一步的提升。
3.4.3 ssDNA重组工程结合CRISPR-Cas9技术在新物种中建立ssDNA重组工作并非易事。例如,在没有选择的情况下可以产生突变的菌株很少。目前研究更多是将ssDNA重组工程与CRISPR-Cas9技术相结合,可极大程度上提高突变效率。例如,罗伊氏乳杆菌ssDNA结合蛋白(RecT)可通过寡核苷酸与DNA复制叉滞后链杂交的方式来促进诱变,而Cas9核酸酶靶向野生型基因序列进行消除,从而富集突变等位基因[63]。此外,CRISPR-Cas9系统也可以与同源重组等基因改造技术相结合,提高基因敲除或基因编辑的效率。
4 乳酸菌合成生物学的应用随着合成生物学的发展,乳酸菌作为载体传递系统,在医疗、生物等领域有广泛应用,同时可利用乳酸菌产生乳酸这一特性,发挥其在发酵、酿造等食品生产方面作用,为食品行业带来新的机遇。此外,在乳酸菌研究过程中,融入了人与自然概念,使其在环境治理、修复方面拥有重大突破,因此对其进行合成生物学技术改造,可使其在自然科学方面继续发挥作用。
4.1 传感器近年来,合成生物学广泛用来设计各种微生物,这种重组微生物具有治疗疾病、抑制癌症和治疗病原体等能力,主要作为载体运送药物。然而合成生物学最重要的一点是可以响应细菌群体感应分子,从而进行自身调控。在以前的研究中,微生物已被设计用于响应来自革兰氏阴性病原体的群体感应分子,例如铜绿假单胞菌,而关于使用革兰氏阳性菌微生物来感知群体感应的研究很少。Borrero等[64]在乳酸乳球菌中构建pCF10载体,其中插入肠球菌素A,和肠球菌素P等基因,产生并分泌抗性肽,可特异性靶向检测肠球菌且抑制多药抗性屎肠球菌菌株的生长。而David等[20]构建工程化罗伊氏乳杆菌调控系统,其设计组成型启动子slp,可表达agrC,agrA;组成型启动子P3可持续表达β-葡糖醛酸糖苷酶,这种酶可跨膜到细菌体外,使4-硝基苯基-β-D-葡糖苷酸转化成黄色色素,从而进行测定金黄色葡萄球菌产生的群体感应分子自诱导肽-I(AIP-I)。
利用乳酸菌合成生物学作为传感器检测浓度范围广,对低浓度物质十分灵敏,同时在检测过程中省时省力,避免大型仪器测样的繁杂程序,这对于疾病的诊断及定量测定具有极大意义。
4.2 疾病治疗由于生活节奏加快,导致肠胃炎症、肥胖、糖尿病等疾病已成为当今社会流行性疾病。普通治疗方法如药物治疗、手术治疗等存在花费高、风险大等隐患,存在一定副作用。乳酸菌合成生物学的发展为疾病治疗及药物传递提供了可能性[65]。
早在20世纪末,乳酸菌合成生物学在医疗方面已经创造了新方向,其利用重组乳酸菌进行基因改造,作为运输载体进入体内后,可以表达对应疫苗,使机体产生抵抗感染的免疫反应,以达到预防疾病的目的,如Robinson等[66]设计重组乳酸乳球菌MG1363表达破伤风毒素,可进行治疗破伤风。而Steidler团队[67]则对治疗结肠炎进行研究,利用乳酸乳球菌分泌白细胞介素-10治疗小鼠结肠炎。而后Xin团队[68]对于口服输送疫苗有了新发现,通过用表达HIV Env的痘苗病毒攻击小鼠腹膜,发现此组小鼠病毒载量比对照小鼠低350倍,证明该策略对HIV免疫应答过程中具有抑制功效。这些实验证明乳酸菌合成生物学在医疗方面发挥了突出作用,将口服作为另外一种运输途径,可进行与传统穿刺方式不同的免疫作用,使得疫苗具有稳定代谢途径。
利用乳酸菌合成生物学进行治疗疾病是一个循序渐进的过程。在之后研究过程中,治疗炎症性肠病成为研究重点。这种由胃肠道炎症引起的特发性疾病,会引起急性或慢性腹痛、血性腹泻、体重减轻、发烧、疲劳和食欲不振等现象[69]。目前针对炎症性肠病的治疗方法仍未确定,通常是对症治疗,旨在维持患者无症状。超氧化物歧化酶(SODs)可将超氧阴离子转化为过氧化氢(H2O2),其被认为是抵抗氧化应激的主要防御措施。然而,在炎症性肠病治疗中由于SOD短循环半衰期仅5至10 min,治疗过程中易受到阻碍,因此Han团队[70]设计了pNZ804sodA质粒,其可由nisA启动子控制,表达sodA基因从而获得MnSOD,之后将该质粒转化到乳酸乳球菌NZ9800和植物乳杆菌NCIMB8826 Int-1菌株上,发现炎症性肠病大鼠症症状减轻。而Pang团队[71]通过给予口服分泌抗TNF纳米抗体的乳酸乳球菌,在慢性结肠炎中显示出治疗功效;Motta等[72]设计表达Elafin的重组乳酸菌可防止炎症并恢复结肠稳态。将这种重组乳酸菌作为载体,其在肠道内可进行定植,安全高效,在人体内能够长时间表达,这对作用于胃肠黏膜进行治疗炎症有突出贡献。
工程化乳酸菌同时存在治疗糖尿病的潜力。糖尿病主要分为1型糖尿病与2型糖尿病,其可通过免疫功能紊乱、微生物感染等致病因子致使机体胰岛功能减退,造成代谢紊乱。其中1型糖尿病是一种自身免疫性疾病,可逐渐破坏产生胰岛素的β细胞。Ma团队[48]构建的质粒,即乳酸乳球菌菌株NZ9000工程化表达HSP65-6P277,调控β细胞分泌胰岛素,改善葡萄糖耐量,减少胰岛炎。而胰高血糖素样肽-1(GLP-1)是一种肠降血糖素,同样能够增强胰岛素分泌,成为治疗2型糖尿病主要治疗剂。Agarwal等[47]构建了LL-pUBGLP-1质粒,其上插入GLP-1基因片段,可分泌胰高血糖素样肽-1,使得大鼠的血糖明显下降,可预防高血糖症。传统口服降血糖药物容易产生过敏现象,产生耐药性,同时会对肝脏系统造成损伤,而重组乳酸菌进入胃肠道后,可进行表达相应肽,进行定点输送,提高机体自身免疫功能,这对于治疗糖尿病开辟了新的道路。
而在社会发展过程中,癌症作为三大致死疾病之一,其发病率不断上升,同时根据以往实验观察,发现其与慢性炎症息息相关。在这个过程中,乳酸菌介入治疗的方法被不断深入研究。目前已经从乳酸菌菌体细胞[73]、细胞质成分[74]等多方面证实乳酸菌存在抗癌作用。面对此现象,将乳酸菌进行合成生物学改造十分有必要,主要还是存在缺少特异性菌株、筛选繁复等问题。若将现有研究抗癌相关机理研究透彻,利用分子生物学技术、基因工程等手段对DNA进行改造,乳酸菌菌株可实现利用最大化,对医学进展也是一个极大的突破。
4.3 食品工业乳酸菌作为重要的食品微生物,可在食品生产中广泛应用,如酸奶发酵、干酪成熟、生产乳酸等。如今消费者对乳制品的产品质量要求较高,而提高质量的方法通常通过增加脂肪、糖、蛋白质或稳定剂(如果胶,淀粉,藻酸盐或明胶)的含量来实现。然而调查中发现,人类对于营养与健康有了新的见解,寻找更加高效、低脂肪、低糖、低添加剂方法成为乳制品的一个新挑战。
胞外多糖是乳酸菌生长代谢过程中分泌的一种糖类化合物,可改善发酵乳制品(例如酸奶)的流变性、质地和口感,但其存在产量低,菌株稳定性差的缺点[75-76]。Felipe等[40]发现,质粒pNZ8020插入nox -2基因,由内源性nisA启动子控制可实现NADH氧化酶的过量产生,从而产生过量胞外多糖。这些发现为工业化生产胞外多糖提供了便利条件。
干酪作为乳制品中的代表食品,对其品质的研究不在少数。为增加干酪风味,可通过质粒自我复制后,在乳酸菌中过表达肽酶基因实现干酪成熟。将pepN、pepC、pepX、pepI表达肽酶基因结合到瑞士乳杆菌pFG1中,该食品级载体转入乳酸乳球菌DN209和NM1后,发现工程化乳酸乳球菌与野生型乳酸乳球菌相比,可以增加肽酶活性,缩短干酪成熟期,生产具有改善特性的特殊奶酪(如减脂奶酪)[42]。此外,氨基酸的酶促降解在风味干酪的开发中起重要作用。编码谷氨酸脱氢酶的解淀粉芽孢杆菌的gdh基因可转入乳酸乳球菌中进行表达,使得谷氨酸高效表达,从而产生大量α-酮戊二酸[41]。实验证明可以使用产生GDH的乳球菌菌株代替添加α-酮戊二酸来改善奶酪的香气。
对于乳酸菌发酵而言,乳酸是碳水化合物快速转化后易得产物。乳酸主要可分为D-乳酸和L-乳酸。其中D-乳酸不被人代谢,而L-乳酸是食品生物技术应用中最重要的异构体,也是制药和生物聚合物工业中最重要的异构体。因此需要寻找特定方法进行定向高效生产L-乳酸。除使用合理的代谢工程方法之外,还可通过递归原生质体融合的经典菌株改良方法(亚硝基胍和紫外诱变)与全基因组改组相结合,增强乳酸的产生。这种方式可分离乳杆菌工业菌株的改组菌株,使其在pH4.0条件下产生比野生型多三倍的乳酸[77]。对于益生菌乳杆菌来说,可通过遗传操作,增强乳酸菌菌株的健康属性、开发新的益生菌活性,同时可深层次理解潜在分子机制。
4.4 生物再生能源由于环境问题日益严峻,能源可持续发展成为日益关注的问题[78-79]。近年来,利用微生物大规模生产可再生生物燃料引起了人们的极大关注,主要是由于石油燃料逐渐短缺引起的,且石油基燃料过度使用产生有害气体,严重污染了环境,导致全球气候变暖等诸多生态问题。因此代谢工程与乳酸菌合成生物学发展可以使非天然生物燃料生产清洁生物燃料。
面对石油能源不可再生问题,醇作为可燃能源已得到大力支持。丁醇已成为公认的优质生物燃料,其比乙醇蓄积能量丰富,并且可以直接用作投入燃料,无需改变发动机[80]。传统的生物燃料用粮食作物生产[81],如今丁醇可以通过革兰氏阳性梭菌属物种发酵底物来生产。然而,为了在工业规模上有利地生产丁醇,需要具有增加丁醇耐受性的新型微生物来提高效率。Liu等[82]通过对丁醇微生物分离与选择,发现卷曲乳酸杆菌NE-L 0206-47和融合乳杆菌(Weissella confusa)BR0216-18能够在3% - 4%丁醇中长期适应生长。同时木质纤维素生物质是世界上最丰富的底物类型,经济节约且可再生,但是使用微生物将木质纤维素原料转化为第二代能源产品尚未在商业上大规模使用,因此需要进行大量筛选实验,以确定菌株对来自木质纤维素生物质预处理的各种抑制剂的耐受性水平,以及它们利用各种糖底物(包括戊糖和己糖)的能力。这些实验证实乳酸菌的发酵能力在生产生物能源方面得到肯定,同时可以作为催化剂进行工业加工。
乳酸菌可进行生产生物燃料,但是其生产率仍然非常低,并且包括耐受性等因素需要进一步改进。乳酸菌本身可在酸性条件下进行存活与作用,醇存在时也可进行相应代谢,但当pH值过低、温度变化大、醇醛等物质含量过高条件下,乳酸菌生物活性受到抑制,这些因素可称为毒杀乳酸菌的外界因子,因此合成生物学技术的出现可以应对这种缺陷。在载体质粒上插入抗酸、抗醇等基因片段,对密码子进行优化,启用高拷贝数启动子等应对措施,这些因素都可在今后实验研究中得到逐步证实。
5 展望乳酸菌合成生物学作为新兴的研究学科,起步较晚,但研究颇丰,越来越受到科研人员的重视。它将科学与工程结合在一起,融汇多种领域的内容,将DNA、RNA、蛋白质等分子结合,创造出全新体系,从而满足人类需求。面对大众对于基因工程担忧,乳酸菌作为食品级别细菌在自然界分布广泛,可用于食品加工,对食品安全有保障,并且一些乳酸菌对人体健康有促进作用,因此对乳酸菌进行工程化设计,可缓解人类对于重组细菌的忧虑。目前乳酸菌合成生物学在大量的科研案例中有重大突破,可生产药物、促进食品工业生产、环境修复以及降解污染物等。
乳酸菌合成生物学与基因工程同时需要构建载体,表达目的基因,但其最大的区别在于合成生物学翻译蛋白同时,要充分调动宿主自身代谢机制,做到外源物质刺激状况下,可以进行群体感应,实现自身调控扑杀。当外源物质不存在时,其乳酸菌底盘可稳定定植于宿主肠道体内,进行普通发酵作用促进肠道健康。现如今乳酸菌合成生物学在应用中仍存在一些缺陷,表达载体构建中,表达元件的优化依旧需要持续研究。同时载体转化依旧需要寻找更加高效转化方法,形成系统性、成功效率高的方法体系。
尽管目前取得了一定进展,但乳酸菌在合成生物学方面的临床应用仍有很长的路要走。利用现有的工具和发展速度,很容易实现合成生物学对于药物发现、药物开发以及其他方面进展有巨大贡献。对于工程化乳酸菌的研究一直在持续,期待工程化乳酸菌在科研方向的其他突破。
| [1] |
杨洁彬. 乳酸菌:生物学基础及应用[M]. 北京: 中国轻工业出版社, 1996.
|
| [2] |
肖平, 吕嘉枥. 乳酸菌的分类鉴定方法的研究进展[J]. 中国酿造, 2008(12x): 8-10. |
| [3] |
凌代文, 东秀珠. 乳酸细菌分类鉴定及实验方法[M]. 北京: 中国轻工业出版社, 1999.
|
| [4] |
施安辉, 周波. 乳酸菌分类、生理特性及在食品酿造工业上的应用[J]. 中国调味品, 2001(11): 3-8. DOI:10.3969/j.issn.1000-9973.2001.11.001 |
| [5] |
焦兴弘. 乳酸菌在肉制品加工过程中的应用[J]. 畜牧兽医科技信息, 2008(2): 1. DOI:10.3969/j.issn.1671-6027.2008.02.088 |
| [6] |
岳春, 李霞, 潘勇, 等. 乳酸菌的功能及其在食品中的应用[J]. 现代农业科技, 2018(15): 237. DOI:10.3969/j.issn.1007-5739.2018.15.145 |
| [7] |
陈杰, 徐冲, 孙翠焕, 等. 乳酸菌在食品工业中的应用现状及发展前景[J]. 微生物学杂志, 2012, 32(3): 91-94. DOI:10.3969/j.issn.1005-7021.2012.03.018 |
| [8] |
中国食品科学技术学会秘书处. 青城论坛:直击益生菌行业发展新动态——第十二届益生菌与健康国际研讨会即将召开[J]. 食品与机械, 2017, 33(5): 1-2. |
| [9] |
Kawashima T, Ikari N, Kouchi T, et al. The molecular mechanism for activating IgA production by Pediococcus acidilactici K15 and the clinical impact in a randomized trial[J]. Scientific Reports, 2018, 8(1): 5065. DOI:10.1038/s41598-018-23404-4 |
| [10] |
Piewngam P, Zheng Y, Nguyen TH, et al. Pathogen elimination by probiotic Bacillus via signalling interference[J]. Nature, 2018, 562: 532-537. DOI:10.1038/s41586-018-0616-y |
| [11] |
Leszek R, Lucyna O, Dariusz P, et al. Probiotic Lactobacillus Plantarum 299v decreases kynurenine concentration and improves cognitive functions in patients with major depression:A double-blind, randomized, placebo controlled study[J]. Psychoneuroendocrinology, 2019, 100: 213-222. DOI:10.1016/j.psyneuen.2018.10.010 |
| [12] |
Hobom B. Gene surgery:on the threshold of synthetic biology[J]. Medizinische Klinik, 1980, 75(24): 834. |
| [13] |
Benner SA, Sismour AM. Synthetic biology[J]. Nature Reviews Genetics, 2005, 6(6): 533-543. |
| [14] |
Chopra P, Kamma A. Engineering life through synthetic biology[J]. Silico Biology, 2006, 6(5): 401. |
| [15] |
Ye H, Fussenegger M. A synthetic optogenetic transcription device enhances blood-glucose homeostasis in mice[J]. Science, 2011, 332(6037): 1565-1568. DOI:10.1126/science.1203535 |
| [16] |
Xue S, Yin J, Shao J, et al. A synthetic-biology-inspired therapeutic strategy for targeting and treating hepatogenous diabetes[J]. Molecular Therapy the Journal of the American Society of Gene Therapy, 2017, 25(2): 443. DOI:10.1016/j.ymthe.2016.11.008 |
| [17] |
Yu Y, You L, Liu D, et al. Development of Synechocystis sp. PCC 6803 as a phototrophic cell factory[J]. Marine Drugs, 2013, 11(8): 2894-2916. DOI:10.3390/md11082894 |
| [18] |
Costley SC, Wallis FM. Bioremediation of heavy metals in a synthetic wastewater using a rotating biological contactor[J]. Water Research, 2001, 35(15): 3715. DOI:10.1016/S0043-1354(01)00072-0 |
| [19] |
Vos WMD. Systems solutions by lactic acid bacteria:from paradigms to practice[J]. Microbial Cell Factories, 2011, 10 Suppl 1(S1): S2. |
| [20] |
David L, Chun LH, In YH, et al. Reprogramming probiotic Lactobacillus reuteri as a biosensor for Staphylococcus aureus derived AIP-I detection[J]. ACS Synth Biol, 2018, 7(5): 1229-1237. |
| [21] |
Zipfel C. A new receptor for LPS[J]. Nature Immunology, 2015, 16(4): 340-341. |
| [22] |
Rueda F, Cano-Garrido O, Mamat U, et al. Production of functional inclusion bodies in endotoxin-free Escherichia coli[J]. Applied Microbiology & Biotechnology, 2014, 98(22): 9229-9238. |
| [23] |
巫琴, 陈磊, 王江新, 等. 应用合成生物学策略优化光合蓝细菌底盘[J]. 生物工程学报, 2013, 29(8): 1086-1099. |
| [24] |
Adams BL. The next generation of synthetic biology chassis:moving synthetic biology from the laboratory to the field[J]. Acs Synthetic Biology, 2016, 5(12): 1328-1330. DOI:10.1021/acssynbio.6b00256 |
| [25] |
许本发. 酸奶和乳酸菌饮料加工[M]. 北京: 中国轻工业出版社, 1994.
|
| [26] |
王琛, 尹光辉, 余飞燕, 等. 大肠杆菌肠毒素基因突变体在乳酸菌中的表达与免疫学鉴定[J]. 中国卫生检验杂志, 2018, 28(5): 513-515, 519. |
| [27] |
易庆, 王关林, 方宏筠. 乳酸杆菌基因转化质粒载体系统及其基因工程研究进展[J]. 中国微生态学杂志, 2000, 12(1): 56-57. DOI:10.3969/j.issn.1005-376X.2000.01.029 |
| [28] |
唐鸿志. 合成生物学在环境修复中的应用[J]. 生物工程学报, 2017, 33(3): 506-515. |
| [29] |
Rico J, Yebra MJ, Pérez-Martínez G, et al. Analysis of ldh genes in Lactobacillus casei BL23:role on lactic acid production[J]. J Ind Microbiol & Biotechnol, 2008, 35(6): 579-586. |
| [30] |
Russo P, Iñaki I, Mohedano ML, et al. Zebrafish gut colonization by mCherry-labelled lactic acid bacteria[J]. Appl Microbiol Biotechnol, 2015, 99(8): 3479-3490. DOI:10.1007/s00253-014-6351-x |
| [31] |
Prado-Rebolledo OF, Delgado-Machuca JDJ, Macedo-Barragan RJ, et al. Evaluation of a selected lactic acid bacteria-based probiotic on Salmonella enterica serovar enteritidis colonization and intestinal permeability in broiler chickens[J]. Avian Pathology, 2016, 1-17. |
| [32] |
Ferain T, Hobbs JN, Richardson J, et al. Knockout of the two ldh genes has a major impact on peptidoglycan precursor synthesis in Lactobacillus plantarum[J]. Journal of Bacteriology, 1996, 178(18): 5431-5437. DOI:10.1128/jb.178.18.5431-5437.1996 |
| [33] |
Bolotin A, Wincker P, Mauger S, et al. The complete genome sequ-ence of the lactic acid bacterium[J]. Genome Research, 2001, 11(5): 731-753. DOI:10.1101/gr.GR-1697R |
| [34] |
杜胜阳, 王斌斌, 冯佳, 等. 乳酸菌基因敲除技术的研究进展[J]. 食品与发酵工业, 2016, 42(1): 244-251. |
| [35] |
Leenhouts KJ, Kok J, Venema G. Campbell-like integration of heterologous plasmid DNA into the chromosome of Lactococcus lactis subsp. lactis[J]. Appl Environ Microbiol, 1989, 55(2): 394-400. |
| [36] |
Mills DA. Mutagenesis in the post genomics era:tools for generating insertional mutations in the lactic acid bacteria[J]. Current Opinion in Biotechnology, 2001, 12(5): 503-509. DOI:10.1016/S0958-1669(00)00254-8 |
| [37] |
Van KR, Vos HR, van Swam II, et al. Functional analysis of glycosyltransferase genes from Lactococcus lactis and other gram-positive cocci:complementation, expression, and diversity[J]. Journal of Bacteriology, 1999, 181(20): 6347-6353. |
| [38] |
de Ruyter PG, Kuipers OP, de Vos WM. Controlled gene expression systems for Lactococcus lactis with the food-grade inducer nisin[J]. Appl Environ Microbiol, 1996, 62(10): 3662-3667. |
| [39] |
Flickinger JL, Chassy BM. Transformation of Lactobacillus caseiby electroporation[J]. FEMS Microbiology Letters, 2010, 44(2): 173-177. |
| [40] |
Felipe FLD, Kleerebezem M, Vos WMD, et al. Cofactor enginee-ring:a novel approach to metabolic engineering in Lactococcus lactis by controlled expression of NADH oxidase[J]. Journal of Bacteriology, 1998, 180(15): 3804-3808. |
| [41] |
Rijnen L, Courtin P, Gripon JC, et al. Expression of a heterologous glutamate dehydrogenase gene in Lactococcus lactis highly improves the conversion of amino acids to aroma compounds[J]. Appl Environ Microbiol, 2000, 66(4): 1354-1359. DOI:10.1128/AEM.66.4.1354-1359.2000 |
| [42] |
Joutsjoki V, Luoma S, Tamminen M, et al. Recombinant Lactococcus starters as a potential source of additional peptidolytic activity in cheese ripening[J]. Journal of Applied Microbiology, 2002, 92(6): 1159. DOI:10.1046/j.1365-2672.2002.01652.x |
| [43] |
Rattanachaikunsopon P, Phumkhachorn P. Glass bead transformation method for gram-positive bacteria[J]. Brazilian Journal of Microbiology, 2009, 40(4): 923-926. |
| [44] |
Tauer C, Heinl S, Egger E, et al. Tuning constitutive recombinant gene expression in Lactobacillus plantarum[J]. Microbial Cell Factories, 2014, 13(1): 1-11. DOI:10.1186/1475-2859-13-1 |
| [45] |
Ruyter PGD, Kuipers OP, Vos WMD. Controlled gene expression systems for Lactococcus lactis with the food-grade inducer nisin[J]. Appl Environ Microbiol, 1996, 62(10): 3662-3667. |
| [46] |
Takala T, Saris P. A food-grade cloning vector for lactic acid bacteria based on the nisin immunity gene nisI[J]. Appl Microbiol Biotechnol, 2002, 59(4-5): 467-471. DOI:10.1007/s00253-002-1034-4 |
| [47] |
Agarwal P, Khatri P, Billack B, et al. Oral delivery of glucagon like peptide-1 by a recombinant Lactococcus lactis[J]. Pharmaceutical Research, 2014, 31(12): 3404-3414. |
| [48] |
Ma Y, Liu J, Hou J, et al. Oral Administration of recombinant Lactococcus lactis expressing HSP65 and tandemly repeated P277 reduces the incidence of type I diabetes in non-obese diabetic mice[J]. PLoS One, 2014, 9(8): e105701. DOI:10.1371/journal.pone.0105701 |
| [49] |
王倩, 康振, 梁泉峰, 等. 合成未来:从大肠杆菌的重构看合成生物学的发展[J]. 生命科学, 2011, 23(9): 844-848. |
| [50] |
楚遵锋, 徐淑华, 王敏, 等. 乳酸杆菌基因工程载体的应用[J]. 今日畜牧兽医, 2017(1): 49-51. |
| [51] |
崔月倩, 王菁蕊, 王艳萍. 乳酸菌基因表达载体及其应用研究进展[J]. 食品科学, 2015, 36(9): 224-229. |
| [52] |
贾士芳, 王荫榆. 乳杆菌电转化条件的研究[J]. 生物工程学报, 1998, 14(4): 429-433. DOI:10.3321/j.issn:1000-3061.1998.04.014 |
| [53] |
Rattanachaikunsopon P, Phumkhachorn P. Glass bead-based transformation method for lactic acid bacteria[J]. Scienceasia, 2009, 35(3): 234-241. DOI:10.2306/scienceasia1513-1874.2009.35.234 |
| [54] |
Anderson DG, Mckay LL. In vivo cloning of lac genes in Streptococcus lactis ML3[J]. Applied & Environmental Microbiology, 1984, 47(2): 245-249. |
| [55] |
Waller MC, Bober JR, Nair NU, et al. Toward a genetic tool development pipeline for host-associated bacteria[J]. Current Opinion in Microbiology, 2017, 38: 156. DOI:10.1016/j.mib.2017.05.006 |
| [56] |
Van Pijkeren JP, Britton RA. High efficiency recombineering in lactic acid bacteria[J]. Nucleic Acids Res, 2012, 40(10): e76. DOI:10.1093/nar/gks147 |
| [57] |
Van Pijkeren JP, Neoh KM, Sirias D, et al. Exploring optimization parameters to increase ssDNA recombineering in Lactococcus lactis and Lactobacillus reuteri[J]. Bioengineered, 2012, 3(4): 209-217. DOI:10.4161/bioe.21049 |
| [58] |
Lim SI, Min BE, Jung GY. Lagging strand-biased initiation of red recombination by linear double-stranded DNAs[J]. Journal of Molecular Biology, 2008, 384(5): 1098. DOI:10.1016/j.jmb.2008.10.047 |
| [59] |
Ellis HM, Yu D, Ditizio T, et al. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides[J]. Proc Natl Acad Sci USA, 2001, 98(12): 6742. DOI:10.1073/pnas.121164898 |
| [60] |
Sawitzke JA, Costantino N, Li XT, et al. Probing cellular processes with oligo-mediated recombination and Using the knowledge gained to optimize recombineering[J]. Journal of Molecular Biology, 2011, 407(1): 45. DOI:10.1016/j.jmb.2011.01.030 |
| [61] |
Ellis HM, Yu D, Ditizio T, et al. High efficiency mutagenesis, repair, and engineering of chromosomal DNA using single-stranded oligonucleotides[J]. Proc Natl Acad Sci USA, 2001, 98(12): 6742. DOI:10.1073/pnas.121164898 |
| [62] |
Huen MSY, Li X, Lu LY, et al. The involvement of replication in single stranded oligonucleotide-mediated gene repair[J]. Nucleic Acids Research, 2006, 34(21): 6183. DOI:10.1093/nar/gkl852 |
| [63] |
Oh JH, Pijkeren JPV. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri[J]. Nucleic Acids Res, 2014, 42(17): e131. DOI:10.1093/nar/gku623 |
| [64] |
Borrero J, Chen Y, Dunny GM, et al. Modified lactic acid bacteria detect and inhibit multiresistant Enterococci[J]. ACS Synthetic Biology, 2015, 4(3): 299-306. DOI:10.1021/sb500090b |
| [65] |
颜朝阳, 张忠涛, 孟化. 手术治疗肥胖2型糖尿病的研究进展[J]. 国际外科学杂志, 2014, 41(10): 698-700. DOI:10.3760/cma.j.issn.1673-4203.2014.10.016 |
| [66] |
Robinson K, Chamberlain LM, Schofield KM, et al. Oral vaccination of mice against tetanus with recombinant Lactococcus lactis[J]. Nature Biotechnology, 1997, 15(7): 653-657. DOI:10.1038/nbt0797-653 |
| [67] |
Steidler L. Treatment of murine colitis by Lactococcus lactis secreting interleukin-10[J]. Science, 2000, 289(5483): 1352-1355. DOI:10.1126/science.289.5483.1352 |
| [68] |
Xin KQ, Hoshino Y, Toda Y, et al. Immunogenicity and protective efficacy of orally administered recombinant Lactococcus lactis expressing surface-bound HIV Env[J]. Blood, 2003, 102(1): 223-228. DOI:10.1182/blood-2003-01-0110 |
| [69] |
Kellermayer R. Epigenetics and the developmental origins of inflammatory bowel diseases[J]. Can J Gastroenterol, 2012, 26: 909-915. DOI:10.1155/2012/526408 |
| [70] |
Han W, Mercenier A, Aitbelgnaoui A, et al. Improvement of an experimental colitis in rats by lactic acid bacteria producing superoxide dismutase[J]. Inflammatory Bowel Diseases, 2010, 12(11): 1044-1052. |
| [71] |
Pang Q, Ji Y, Li Y, et al. Intragastric administration with recombinant Lactococcus lactis producing heme oxygenase-1 prevents lipopolysaccharide-induced endotoxemia in rats[J]. Fems Microbiology Letters, 2010, 283(1): 62-68. |
| [72] |
Motta JP, Humaran LB, Deraison C, et al. Food-grade bacteria expressing elafin protect against inflammation and restore colon homeostasis[J]. Science Translational Medicine, 2012, 4(158): 622-632. |
| [73] |
Wang SM, Zhang LW, Gu W, et al. Screening for antiproliferative effect of Lactobacillus strains against colon cancer HT-29 cells[J]. Advanced Materials Research, 2012, 573: 1039-1043. |
| [74] |
Kim JY, Woo HJ, Kim YS, et al. Screening for antiproliferative effects of cellular components from lactic acid bacteria against human cancer cell lines[J]. Biotechnology Letters, 2002, 24(17): 1431-1436. DOI:10.1023/A:1019875204323 |
| [75] |
Welman, Alan D, Maddox, et al. Exopolysaccharides from lactic acid bacteria:perspectives and challenges[J]. Trends in Biotechnology, 2003, 21(6): 269-274. DOI:10.1016/S0167-7799(03)00107-0 |
| [76] |
梁增澜, 李超, 王艳萍. 乳酸菌胞外多糖免疫活性的研究进展[J]. 食品与发酵工业, 2018, 44(2): 266-272. |
| [77] |
Patnaik R, Louie S, Gavrilovic V, et al. Genome shuffling of Lactobacillus for improved acidtolerance[J]. Nature Biotechnology, 2002, 20(7): 707. DOI:10.1038/nbt0702-707 |
| [78] |
Atsumi S, Cann AF, Connor MR, et al. Metabolic engineering of Escherichia coli for 1-butanol production[J]. Metabolic Engineering, 2007, 10(6): 305-311. |
| [79] |
Ezeji TC, Qureshi N, Blaschek HP. Bioproduction of butanol from biomass:from genes to bioreactors[J]. Current Opinion in Biotechnology, 2007, 18(3): 220-227. DOI:10.1016/j.copbio.2007.04.002 |
| [80] |
Antoni D, Zverlov VV, Schwarz WH. Biofuels from microbes[J]. Applied Microbiology & Biotechnology, 2007, 77(1): 23-35. |
| [81] |
Qureshi N, Hughes S, Maddox IS, et al. Energy-efficient recovery of butanol from model solutions and fermentation broth by adsorption[J]. Bioprocess Biosyst Eng, 2005, 27(4): 215-222. DOI:10.1007/s00449-005-0402-8 |
| [82] |
Liu S, Bischoff KM, Leathers TD, et al. Adaptation of lactic acid bacteria to butanol[J]. Biocatalysis & Agricultural Biotechnology, 2012, 1(1): 57-61. |




