米曲霉(Aspergillus oryzae)是我国传统酿造行业的优良菌种,在酱油酿制、豆豉制作等方面都有着广泛应用[1]。同时,米曲霉又是公认的食品安全生产菌株,发酵及后处理技术成熟,因此它除了在传统发酵工业中被用于生产酱油、豆豉产品外,还常用于生产初级、次级代谢产物及酶类产品,同时也是基因表达和蛋白分泌研究的理想对象[2-3]。
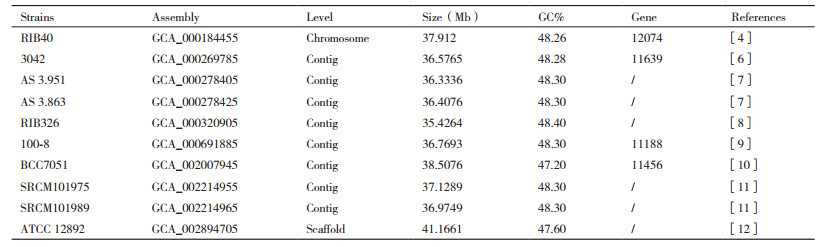
随着二代测序技术的发展,基因组测序已经变得越来越普遍,相对于植物和动物,真菌基因组小,容易被测序和注释,因此成为生物基因组研究的典型例子,为功能基因组、表达调控机理和物种进化奠定了理论基础。2005年,日本26家科研单位利用鸟枪法共同完成了世界首例米曲霉RIB40的基因组测序工作[4]。米曲霉基因组大小为37 Mb,共8条染色体,包含12 074个基因。相比构巢曲霉和烟曲霉,米曲霉基因组分别比两者大29%和34%,这些米曲霉特有的基因主要和水解酶分泌、氨基酸代谢和糖类转运相关。随后,更多的米曲霉菌株被测序,截至目前,共有10株米曲霉完成测序,基因组大小从35.43 Mb到41.16 Mb,GC含量大致为47 %-4 8%(表 1)。虽然米曲霉的基因组已经被破译,但是对米曲霉的功能基因组研究进展缓慢,米曲霉RIB40基因组中预测的编码蛋白基因是12 074个,其中仅有205个基因功能经过验证,占基因总数的1.7%[5]。究其原因,一是因为可用的筛选标记少,米曲霉对常见的抗生素具有较强的抗性,如氨苄霉素(Amp)、卡那霉素(Kana)、头孢霉素(Ca)等,同时某些合成抗生素的基因可能会向环境及其他微生物中转移,而米曲霉又常用于食品生产,因此美国FDA已禁止在食品中使用这类标记;另外一个原因是转化子的不稳定性,因为米曲霉在繁殖过程中存在多核现象,很难筛选到纯化的转化子。
有效的筛选标记是功能基因组研究的基础,它可以降低假阳性率,减少筛选工作量。目前米曲霉转化载体常用的筛选标记有抗药性标记和营养缺陷型标记基因。
1.1.1 抗药性标记抗药性筛选标记不要求受体菌具有营养缺陷型,可以作为显性筛选标记,是真菌遗传转化应用最为广泛的标记。抗药性基因转入受体菌后,可使受体菌在一定药物浓度下生长,表现出药物抗性。潮霉素B抗性基因,如hph,hygr等,是真菌遗传转化系统中应用最为广泛的抗药性标记。Fernandez等[13]将潮霉素B抗性基因hph作为筛选标记,成功构建了米曲霉的干扰载体,并抑制了米曲霉与孢子颜色相关基因wA的表达;郭继平等[14]将潮霉素B抗性基因(hygr)的置换型打靶载体pHC2,将其线性化后转化米曲霉,成功得到了米曲霉的抗潮霉素菌株。除此以外,博来霉素抗性基因,如ZeoR,也被用作米曲霉遗传转化的筛选标记。尹燕辰等[15]使用pBC-hygro作为质粒骨架,并进行了改进和优化,成功获得米曲霉的抗博来霉素菌株,并以此菌株为基础,表达米曲霉α-淀粉酶同源基因amyB,成功获得酶活提高106.27%的转化子。由于米曲霉对多种药物具有抗性,因此目前报道的抗药性筛选标记较少,主要有bar、sull、bs、km等。使用抗药性筛选标记的转化体系大多都需要使用抗生素,价格昂贵,且某些合成抗生素的基因可能会向环境及其他微生物中转移,而米曲霉又常用于食品生产,因此美国FDA已禁止在食品中使用这类标记。
1.1.2 营养缺陷型标记营养缺陷型标记可以通过转入标记基因,从而与受体细胞突变基因互补,从而使受体细胞可以和野生型一样的表型。pyrG是米曲霉转化中用的最多的一种营养缺陷型标记,其原理是pyrG编码的乳清酸核苷-5'-磷酸脱羧酶可以将5-氟乳清酸(5-FOA)催化为5-氟乳清苷酸(5-FUMP),而5-FUMP具有很强的细胞毒性,使得野生型菌株在含有5-FOA的平板上不能生长,而pyrG基因突变菌株失去了催化活性,从而可以在含有5-FOA的平板上生长。Yasuda等[16]以工业上常用的米曲霉菌株KBN出发,用pyrG作为筛选标记,成功构建了高效的基因敲除体系。Jun-Ichi等[17]以pyrG基因突变菌株为受体菌,建立了多基因缺失体系,同时敲除了2个蛋白酶表达相关的基因。Jaewoo等[18]利用该系统,实现了一个载体敲除多个基因。我国酱油、豆豉等发酵过程中大多选用米曲霉3042作为发酵菌株,该菌株尚未构建便于遗传操作的转化载体,本课题组前期基于RIB40的转化载体,成功构建了米曲霉3042菌株的pyrG 突变的转化载体,并成功进行了转化,为改良菌株性能和提高酱油品质奠定了基础[19]。除此以外,米曲霉转化常用的营养缺陷型标记还有编码硝酸盐还原酶的niaD基因,编码色氨酸生物合成酶的trp基因等。niaD编码的硝酸盐还原酶是真菌氮素代谢过程中的关键酶,可使转化子获得硝酸盐代谢能力,但niaD功能缺失的突变株能耐受高浓度的氯酸盐,而野生型则不能;trp基因是色氨酸合成的关键基因,而色氨酸是米曲霉生长的必需氨基酸之一,trp功能缺失的突变株不能正常合成色氨酸,导致色氨酸营养缺陷,即在色氨酸缺陷培养基内不能生长。营养缺陷型标记获得的转化子筛选便捷,只需要用基础培养基来筛选,无需添加抗生素等,同时可以用于不同真菌之间的转化。但是缺点在于丝状真菌大多具有多核现象,用这种方法分离纯合子比较困难。
1.2 转化方法高效的遗传转化体系是米曲霉功能基因组研究的前提,相对于其他微生物而言,如酵母和大肠杆菌,米曲霉存在着坚韧的细胞壁,所以转化效率低下。为提高遗传转化效率,现有多种转化方法,主要包括原生质体法-PEG/CaCl2、农杆菌介导法和电击转化法。
1.2.1 原生质体法-PEG/CaCl2 PEG/CaCl2介导的遗传转化是使用较早较普遍的细胞融合方法。1978年,Hinnen等[20]首先利用聚乙二醇(PEG)作为介质成功完成了酵母原生质体转化,此后PEG介导的原生质体转化法逐渐应用于粗糙链孢菌(Neurospora crassa)和构巢曲霉(Aspergillus nidulans)等,使这一技术得到广泛的应用。PEG/CaCl2是以原生质体作为感受态细胞,将所转化DNA和原生质体混合后,在含有一定浓度CaCl2和PEG等碱性环境下进行的。PEG是一种能够促进原生质体吸收外源DNA的化学诱导剂,在高pH环境可以通过电荷间的相互作用与外源DNA形成紧密复合物,原生质体通过内吞作用将这些复合物吸收至细胞内,随后再进入细胞核并整合到受体基因组中[21]。在转化过程中,可用于制备原生质体的丝状真菌出发材料有多种选择,如无性孢子、新鲜菌丝体或者担孢子[22]。PEG转化方法的优点是受体细胞数量大,容易获得纯合型的转化子,效率高、整合度和遗传性高等,因此原生质体融合在米曲霉遗传转化中应用广泛[23]。韩志双等[24]通过PEG/CaCl2介导的原生质体法选育出2株米曲霉酸性蛋白酶高产菌株。刘肖凯[25]也用该方法敲除了米曲霉4177中Rho GTPase家族中的6个成员,并获得了相应表型的突变菌株。同时,原生质体法也存在一些缺陷,如培养难度大,再生频率低和周期长,并且价格昂贵,不易获得转化体以及转化受体细胞。
1.2.2 农杆菌介导法农杆菌介导转化法(Agrobacterium tumefaciens mediated transformation,ATMT)是通过根癌农杆菌将外源DNA转入植物、细菌和真菌细胞内[26],在1995年Bundock等[27]首次成功进行了根癌农杆菌介导酵母菌的遗传转化。在此之后,科学家们相继对丝状真菌的转化方法加以重视。根癌农杆菌介导的转化技术为丝状真菌功能基因组的研究奠定了基础,遗传转化技术现在已经比较成熟,很多结果表明也都成功获得转化子[28-29],但是由于米曲霉的多耐药性,因此该方法在米曲霉中的应用目前在国内尚未见报道。而在2016年,Nguyen等[30]利用pyrG作为筛选标记首次将农杆菌介导转化法应用在米曲霉中,并与绿色荧光蛋白GFP融合,成功构建了米曲霉的农杆菌介导转化的过表达体系。随后,在2017年,该作者通过改造该载体,利用同源重组的原理,通过农杆菌介导转化成功构建了米曲霉RIB40的敲除体系[31]。农杆菌介导的米曲霉转化体系的确立,克服了原有转化体系操作繁杂、转化效率低、耗时多等缺点,将成为一种主流的转化系统。但本课题组在利用农杆菌介导法转化米曲霉时发现,通过这种方法获得的转化子不稳定,因为米曲霉大多具有多核现象,在传代过程中个别核很容易丢失,因此很难筛选到纯合子。刘雪[22]通过多轮过滤的方式,大大提高了单核孢子的比例。因此,通过农杆菌转化筛选转化子时,可以通过多轮富集和筛选的方法,将单核孢子从转化子中筛选出来,本课题组利用膜过滤的方法,将单核孢子的比例从11%提高到30%,提高了纯合子筛选概率[19, 32]。
1.2.3 电击转化法电击转化法是将原生质体或细胞质膜在高压脉冲作用下,形成瞬间通道,将外源吸收至细胞内部,由于质膜可以进行自我修复,从而修复高电压脉冲造成的穿孔。虽然电击转化步骤简单,但是转化效率较低。陈凤等[33]研究表明,在多次电击转化米曲霉niaD300时,未将基因成功转进米曲霉中。同时,电击法转化米曲霉价格昂贵,且转化前还需对电击参数进行探索,相对比较繁琐,因此电击转化法在米曲霉功能基因组研究过程中应用较少。
1.3 遗传操作有效的筛选标记和高效的转化方法是遗传操作的前提,也是功能基因组研究的基础。由于筛选标记的限制和转化体系的不成熟,传统的遗传操作(如同源重组、诱导突变等),相对于酵母和构巢曲霉,在米曲霉中报道较少。近年来,CRISPR/Cas9技术由于其高效性,已经快速应用在多个物种当中,但是在米曲霉中仅有2篇文献报道。2015年,Katayama等在米曲霉中首次建立了CRISPR/Cas9系统,并成功敲除了wA,pygG和yA基因,突变率在10%-2 0%之间[34];随后该课题组在2018年进一步改良了CRISPR/Cas9系统,在米曲霉中的敲除效率达到了50%-1 00%,并实现了多基因同步敲除[35]。
2 米曲霉功能基因组研究进展 2.1 分生孢子米曲霉作为形态结构高度复杂的微生物,发挥着越来越大的应用价值和潜力,它会在生命周期呈现出不同的生命状态,米曲霉的分生孢子是由一个特殊的发育营养结构而形成的,被称作菌丝梗[36]。菌丝梗在一定时期形成基地菌丝体上的足细胞,然后在生长发育成囊泡,囊泡出芽形成瓶梗,接着分生孢子形成在瓶梗表面。在分生孢子的形成过程中受到多个基因的调控。brlA基因的激活是曲霉分生孢子形成最基本的步骤,brlA通过控制早期的发育调控基因abaA、rodA和yA的表达调控分生孢子的表达,使其正常的分化;在中期,BrlA蛋白激活abaA基因,abaA基因与AbaA结合,控制着发育基因的表达;在后期,wetA基因受AbaA蛋白诱导激活,brlA、abaA和wetA这三个基因决定基因激活的顺序[37-38]。另外,有研究表明白光可以通过下调brlA的表达而抑制分生孢子的产生,这个现象与其他曲霉属物种相反[39]。随着生物信息学、基因组学和分子生物学等的快速发展,米曲霉产孢机制及相关基因的研究将更加深入。
2.2 蛋白分泌和表达米曲霉作为发酵工业重要的菌株,具有强大的蛋白分泌能力。Liu等[40]对米曲霉基因组通过文献总结和生物信息学分析,共鉴定米曲霉中369个分泌蛋白,与此同时还发现米曲霉相对于植物和哺乳动物具有较强的蛋白质分泌、合成能力以及快速生长,易培养等优点,与大肠杆菌和酵母相比也具有强大的翻译后修饰功能[41]。正是由于米曲霉的这些特点,米曲霉异源蛋白的表达,现今成为一个热点话题,已有很多方法提高异源蛋白的产量,如提高基因拷贝数、使用强启动子、基因融合、蛋白酶缺陷株、优化培养条件等[42]。Jin等[43]通过UV定向变异和选择标记性基因敲除技术,构建了米曲霉的多重营养缺陷型菌株,并通过构建酸性蛋白酶PepE和三肽酶TppA双基因敲除菌株来表达外源人溶菌酶(Human lysozyme,HLY)基因,使酶产量提高20倍以上。Wang等[44]利用基因重组等技术获得突变体菌株RIII-7,高果糖基转移酶(FTase)活性180 Ug-1是原始菌株的2倍,在发酵培养中,达到最大值353 U/g。虽然米曲霉具有强大的蛋白分泌能力,可作为理想的外源蛋白表达宿主,但由于米曲霉复杂的蛋白酶系和自噬系统,使得外源蛋白的表达产物难以积累和分泌,另外外源基因在转录过程中,会受多个转录因子调控,因此米曲霉蛋白分泌和表达的分子机制研究还有待加深,以实现米曲霉生产外源蛋白的工业化。
2.3 次级代谢产物米曲霉不但有较强的分泌蛋白的能力,而且还有丰富的次生代谢产物。聂丽娟[45]在米曲霉中分离得到近20余种化合物,其中包括10余种小分子药理活性物质,如抗真菌的Asperfuran,抗高血压的Mutaaspergillic acid等,也包含一些真菌毒素,如圆弧偶氮酸(Cyclopiazonic acid),曲酸(Kojic acid)等;Marui等[46]通过基因组比较发现,在米曲霉中保留有生产青霉素的基因簇,而且保持非常高度的完整性,但产量极低。通过人为调控基因的高效表达,使青霉素的产量提高了100倍。另外,虽然米曲霉在基因组上接近黄曲霉,但米曲霉不会产生黄曲霉毒素。Zhang等[47]报道了米曲霉的黄曲霉毒素生物合成基因簇的同系物,甚至在有利于黄曲霉和寄生曲霉的黄曲霉毒素表达条件下也不会表达,其原因是米曲霉缺少aflR基因,正是因为这种特性使得米曲霉可以用在食品工艺上。另外,常用于化妆品和食品加工领域的曲酸也是米曲霉的主要次级代谢产物之一。曲酸的合成依赖于米曲霉基因簇上的14个基因,从AO090113000132到AO090113000145,其中包含3个关键基因:kojR、kojA和kojT。
随着米曲霉功能基因组的迅速发展,很多研究者利用基因工程和代谢工程来提高米曲霉的次级代谢产物的产量。Li等[48]通过过表达米曲霉脂肪酸去饱和酶基因D9D,发现米曲霉在提高不饱和脂肪酸含量的同时也可以提高其耐盐性。Knuf等[49]敲除了米曲霉中乳酸脱氢酶基因以提高乳酸产量,该工程菌在以淀粉为基质的培养基中乳酸产量可达30 g/L。虽然米曲霉中被鉴定的次级代谢产物种类在逐渐增加,但是合成这些次级代谢产物的基因簇和相关基因却报道甚少。对于米曲霉发酵和蛋白分泌上的优势,是否能通过代谢工程用米曲霉大规模生产药物先导化合物或者通过基因工程手段提高有用次级代谢产物的产量?对于米曲霉的安全性,在一些特定环境中米曲霉是否会产生污染食品的真菌毒素?这些问题都值得重视并且进行深入的研究。
3 展望米曲霉作为食品工业生产的一类重要微生物,主要应用于传统酿造行业。同时,米曲霉强大的蛋白分泌能力使其在酶制剂和重组蛋白等行业也应用广泛,而其丰富的次级代谢产物使米曲霉具有药物先导化合物优质来源的潜力。随着米曲霉功能基因组学研究的不断深入,最终将从分子、进化和生产各层面阐述重要基因的生物学功能,揭示米曲霉蛋白分泌和表达、次级代谢产物合成的分子机理,最终为工业生产提高效率和质量。应用基因工程对米曲霉菌株的改造和代谢工程对米曲霉发酵的优化,还需要更深入和广泛的研究:开发更多的安全筛选标记,保证发酵食品的安全性;多种转化方法相结合,提高遗传转化效率;探讨不同条件下米曲霉的代谢物变化,提高米曲霉发酵效率和控制发酵过程中的真菌毒素;深入研究米曲霉次级代谢产物合成相关的基因簇,开发药物前体的新来源。米曲霉功能基因组的研究成果将为提高米曲霉的发酵效率、保证米曲霉发酵食品的安全性和扩大米曲霉在工业上的应用范围奠定基础。
| [1] |
赵龙, 周池虹伶, 赵谋明, 等. 解淀粉芽孢杆菌SWJS22和米曲霉混合制曲酱油发酵中的应用[J]. 食品科学, 2017, 38(22): 125-130. DOI:10.7506/spkx1002-6630-201722019 |
| [2] |
童佳, 赵国忠, 赵新建, 等. 米曲霉发酵高盐稀态酱油过程中典型挥发性风味物质的形成[J]. 中国酿造, 2017, 36(5): 22-29. |
| [3] |
Tang W, Ouyang C, Liu L, et al. Genome-wide identification of the fatty acid desaturases gene family in four Aspergillus species and their expression profile in Aspergillus oryzae[J]. AMB Express, 2018, 8(1): 169. DOI:10.1186/s13568-018-0697-x |
| [4] |
Machida M, Asai K, Sano M, et al. Genome sequen-cing and analysis of Aspergillus oryzae[J]. Nature, 2005, 438(7071): 1157-1161. DOI:10.1038/nature04300 |
| [5] |
张云峰, 邵磊. 根癌农杆菌电击转化条件的研究[J]. 淮阴师范学院学报:自然科学版, 2009, 8(3): 243-249. DOI:10.3969/j.issn.1671-6876.2009.03.016 |
| [6] |
Zhao G, Yao Y, Qi W, et al. Draft genome sequence of Aspergillus oryzae strain 3. 042[J]. Eukaryotic Cell, 2012, 11(9): 178. |
| [7] |
Lin W, Song J, Hu W, et al. Relationship between extracellular cellulase, pectinase and xylanase activity of isolated Aspergillus oryzae strains grown on Koji and the umami-tasting amino acid content of soy sauce[J]. Food Biotechnology, 2016, 30(4): 278-291. DOI:10.1080/08905436.2016.1244768 |
| [8] |
Umemura M, Koike H, Yamane N, et al. 3 Comparative genome analysis between Aspergillus oryzae strains reveals close relationship between sites of mutation localization and regions of highly divergent genes among Aspergillus species[J]. DNA Research, 2012, 19(5): 375-382. DOI:10.1093/dnares/dss019 |
| [9] |
Zhao G, Yao Y, Hou L, et al. Draft genome sequence of Aspergillus oryzae 100-8, an increased acid protease production strain[J]. Genome Announcements, 2014, 2(3): e00548-14. |
| [10] |
Thammarongtham C, Nookaew I, Vorapreeda T, et al. Genome characterization of oleaginous Aspergillus oryzae BCC7051:A potential fungal-based platform for lipid production[J]. Current Microbiology, 2017, 75(1): 57-70. |
| [11] |
Zhong Y, Lu X, Xing L, et al. Genomic and transcriptomic comparison of Aspergillus oryzae strains:a case study in soy sauce koji fermentation[J]. Journal of Industrial Microbiology & Biotechnology, 2018, 45(9): 839-853. |
| [12] |
Deng S, Pomraning KR, Bohutskyi P, et al. Draft genome sequence of Aspergillus oryzae ATCC 12892[J]. Genome Annoouncements, 2018, 6(18): e00251-18. |
| [13] |
Fernandez EQ, Moyer DL, Maiyuran S, et al. Vector-initiated transitive RNA interference in the filamentous fungus Aspergillus oryzae[J]. Fungal Genet Biol, 2012, 49(4): 294-301. DOI:10.1016/j.fgb.2012.01.011 |
| [14] |
郭继平.米曲霉碱性蛋白酶的异源表达和定向进化以及遗传改造[D].哈尔滨: 哈尔滨工业大学, 2008. http://cdmd.cnki.com.cn/article/cdmd-10213-2009224145.htm
|
| [15] |
尹燕辰.米曲霉表达系统的构建及其在产黄青霉淀粉酶表达中的应用[D].广州: 华南理工大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10251-1015559139.htm
|
| [16] |
Yasuda AY, Mori A, Ishihara N, et al. Development of a highly efficient gene replacement system for an industrial strain of Aspergillus oryzae used in the production of miso, a Japanese fermented soybean paste[J]. Food Science and Technology Research, 2011, 17(2): 161-166. DOI:10.3136/fstr.17.161 |
| [17] |
Jun-Ichi M, Katsuhiko. Multiple gene disruptions by marker recycling with highly efficient gene-targeting background(DligD)in Aspergillus oryzae[J]. Biotechnology Letters, 2008(30): 1811-1817. |
| [18] |
Jaewoo Y, Jun-Ichi M, katsuhiko K. Disruption of ten protease genes in the filamentous fungus Aspergillus oryzae highly improves production of heterologous proteins[J]. Applied Microbiology and Biotechnology, 2010, 89(3): 747-759. |
| [19] |
Sun Y, Niu Y, He B, et al. A dual selection marker transformation system using Agrobacterium tumefaciens for the industrial Aspergillus oryzae 3. 042[J]. Journal of Microbiology and Biotechnology, 2019, 29(2): 230-234. DOI:10.4014/jmb.1811.11027 |
| [20] |
Hinnen A, Hicks JB, Fink GR. Transformation of yeast[J]. Proc Natl Academic Science USA, 1978, 75: 1929-1933. DOI:10.1073/pnas.75.4.1929 |
| [21] |
刘玲玲, 王永林, 熊典广, 等. 杨树腐烂病菌(Sytospora chrysosperma)原生质体遗传转化体系的构建[J]. 微生物学通报, 2017, 44(10): 2487-2497. |
| [22] |
Li D, Tang Y, Lin J, et al. Methods for genetic transformation of filamen-tous fungi[J]. Microbial Cell Factories, 2017, 16(1): 168. DOI:10.1186/s12934-017-0785-7 |
| [23] |
Liu ZH, Frisen TL. Polyethlene glycol(PEG)-mediated transfor-mation in filamentous fungal pathogens[J]. Plant Fungal Patho-genes, 2012, 83(5): 365-275. |
| [24] |
韩志双, 刘军, 郇阿梅, 等. 应用基因组改组技术选育米曲霉酸性蛋白酶高产菌株[J]. 中国调味品, 2015, 40(1): 18-22. DOI:10.3969/j.issn.1000-9973.2015.01.005 |
| [25] |
刘肖凯.米曲霉表型相关基因的敲除及表型分析[D].广州: 华南理工大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10561-1017855547.htm
|
| [26] |
Dai Z, Deng S, Culley DE, et al. Agrobacterium tumefaciens -medi -ated transformation of oleaginous yeast Lipomyces species[J]. Applied Microbiology and Biotechnology, 2017, 101(15): 6099-6110. DOI:10.1007/s00253-017-8357-7 |
| [27] |
Bundock P, Amkeden DR, Begersbergen AGM, et al. Trans-kingdon T-DNA transfefr from Agrobacterium tumefaciens to Saccharomyces cerevisiae[J]. The EMBO Journal, 1995, 14(3): 3206-3214. |
| [28] |
Wang D, He D, Li G, et al. An efficient tool for random insertional mutagenesis:Agrobacterium tumefaciens -mediated transformation of the filamentous fungus Aspergillus terreus[J]. Journal of Microbiological Methods, 2014, 98(1): 114-118. |
| [29] |
Rodrigo ML, Judith Z, Rizk AM, et al. Development of a transformation system for Aspergillus sojae based on the Agrobacterium tumefaciens -mediated approach[J]. BMC Microbiology, 2014, 14(1): 247. DOI:10.1186/s12866-014-0247-x |
| [30] |
Nguyen K, Ho QN, Pham TH, et al. The construction and use of versatile binary vectors carrying pyrG auxotrophic marker and fluorescent reporter genes for Agrobacterium -mediated transformation of Aspergillus oryzae[J]. World Journal of Microbiology & Biotechnology, 2016, 32(12): 204. |
| [31] |
Nguyen K, Ho QN, Binh LT, et al. A new and efficient approach for construction of uridine/uracil auxotrophic mutants in the filamentous fungus Aspergillus oryzae using Agrobacterium tumefaciens -mediated transformation[J]. World Journal of Microbiology & Biotechnology, 2017, 33(6): 107. |
| [32] |
刘雪.米曲霉原生质体的制备及pyrG缺失株的构建研究[D].南昌: 南昌大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10403-1015505924.htm
|
| [33] |
陈凤.米曲霉niaD300转化系统的构建及其RNA干扰效应研究[D].广州: 华南理工大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10561-1012451047.htm
|
| [34] |
Katayama T, Tanaka Y, Okabe T, et al. Development of a genome editing technique using the CRISPR/Cas9 system in the industrial filamentous fungus Aspergillus oryzae[J]. Biotechnology Letter, 2015, 38: 637-642. |
| [35] |
Katayama T, Nakamura H, Zhang Y, et al. Forced recycling of an AMA1-based genome-editing plasmid allows for efficient multiple gene deletion/integration in the industrial filamentous fungus Aspergillus oryzae[J]. Applied and Environmental Microbiology, 2018, 85(3): e01896-18. |
| [36] |
Krijgsheld P, Bleichrodt R, Veluw GJ, et al. Development in Aspergillus[J]. Studies in Mycology, 2013, 74(74): 1-29. |
| [37] |
Ogawa M, Tokuoka M, Feng JJ, et al. Genetic analysis of conidiation regulatory pathways in koji-mold[J]. Fungal Genetics & Biology, 2010, 47(1): 10-18. |
| [38] |
Nakamura H, Kikuma T, Jin FJ, et al. AoRim15 is involved in conidial stress tolerance, conidiation and sclerotia formation in the filamentous fungus Aspergillus oryzae[J]. Journal of Bioscience & Bioengineering, 2016, 121(4): 365. |
| [39] |
Hatakeyma R, Nakahama T, Higuchi Y, et al. Light represses conidiation in koji Mold Aspergillus oryzae[J]. Bioscience, Biotechnology, and Biochemistry, 2007, 71(8): 1844-1849. DOI:10.1271/bbb.60713 |
| [40] |
Liu L, Fei ZA, Osterlund T, et al. Genome-scaleanalysis of the high-efficient protein secretion system of Aspergillus oryzae[J]. BMC Systems Biology, 2014, 8(1): 1-13. |
| [41] |
Uchima CA, Tokuda G, Watanabe H, et al. Heterologous expression and charac-terization of a glucose-stimulated β-glucosidase from the termite Neotermes koshunensis in Aspergillus oryzae[J]. Applied Microbiology and Biotechnology, 2011, 89(6): 1761-1771. DOI:10.1007/s00253-010-2963-y |
| [42] |
Yokota JI, Shiro D, Tanaka M, et al. Cellular responses to the expression of unstable secretory proteins in the filamentous fungus Aspergillus oryzae[J]. Applied Microbiology and Biotechnology, 2017, 101(6): 2437-2446. DOI:10.1007/s00253-016-8086-3 |
| [43] |
Jin FJ, Watanabe T, Juvvadi PR, et al. Double disruption of the proteinase genes, tppA, and pepE, increases the production level of human lysozyme by Aspergillus oryzae[J]. Applied Microbiology and Biotechnology, 2007, 76(5): 1059-1068. |
| [44] |
Wang S, Duan M, Liu Y, et al. Enhanced production of fructosyltr-ansferase in Aspergillus oryzae by genome shuffling[J]. Biotec-hnology Letters, 2016, 39(33): 91-396. |
| [45] |
聂丽娟.米曲霉菌次级代谢产物研究[D].武汉: 华中科技大学, 2009. http://d.wanfangdata.com.cn/Thesis/D087915
|
| [46] |
Marui J, Ohashikunihiro S, Ando T, et al. Penicillin biosynthesis in Aspergillus oryzae and its overproduction by genetic engineering[J]. Journal of Bioscience & Bioengineering, 2010, 110(11): 8-11. |
| [47] |
Zhang YQ, Wilkinson H, Keller NP, et al. Secondary metabolite gene clusters[M]. New York: Marcel Dekker, 2005: 355-386.
|
| [48] |
Li H, Ma L, Hu Z, et al. Heterologous expression of AoD9D enhances salt tolerance with increased accumulation of unsaturated fatty acid in transgenic Saccharomyces cerevisiae[J]. Journal of Industrial Microbiology & Biotechnology, 2019, 46(2): 231-239. |
| [49] |
Knuf C, Nookaew I, et al. Physiological characterization of the high malic acid-producing Aspergillus oryzae strain 2103a-68[J]. Appl Microbiol Biotechnol, 2014, 98(8): 3517-3527. DOI:10.1007/s00253-013-5465-x |