2. 中国农业科学院蔬菜花卉研究所,北京 100081;
3. 河北农业大学植物保护学院,保定 071001
2. The Institute of Vegetables and Flowers, Chinese Academy of Agricultural Sciences, Beijing 100081;
3. College of Plant Protection, Hebei Agricultural University, Baoding 071001
几丁质是昆虫表皮和围食膜的重要组成成分,几丁质代谢随昆虫不同生长发育阶段而变化,对昆虫的正常生长发育至关重要[1],参与几丁质代谢的酶是杀虫剂破坏昆虫生长机制的潜在靶标。几丁质脱乙酰酶(Chitin deacetylase,CDA)是昆虫几丁质代谢酶系中的重要组成,可催化几丁质脱乙酰化形成壳聚糖[2]。目前,对多种昆虫CDA蛋白功能研究表明,昆虫CDA类蛋白与昆虫蜕皮、羽化以及气管延伸等方面有关,为昆虫防治提供了有利依据。
昆虫CDA蛋白分为CDAⅠ、CDAⅡ、CDAⅢ、CDAⅣ及CDAⅤ五大类[3]。CDAⅠ类蛋白包含几丁质脱乙酰基酶催化区结构域(CDA)、几丁质结合结构域(ChBD)以及低密度脂蛋白结合区(LDLa),CDAⅡ类蛋白结构域与第Ⅰ类的结构域一致,但是序列差异大,研究表明,赤拟谷盗(Tribolium castaneum)TcCDA1与TcCDA2与其蜕皮和羽化有关[4];飞蝗(Locusta migratoria)LmCDA1、LmCD-A2a和LmCDA2b在昆虫细胞中成功表达,并具有不同的几丁质脱乙酰酶活性[5];CDAⅢ类蛋白和CDAⅣ类蛋白都具有CDA与ChBD结构域,不含有LDLa结构域,但是第Ⅲ类和第Ⅳ类CDA的结构域在CDA上的位置存在较大程度差异;CDAⅤ类蛋白只含有CDA结构域,不具有ChBD与LDLa结构域,CDAⅤ类蛋白研究中,粉纹夜蛾CDA研究表明,TnPM-P42蛋白具有很强的几丁质结合活性,与围食膜结合紧密[6],蓓带夜蛾CDA的研究结果推测CDA的作用可能是参与改变围食膜中几丁质的物理和化学性质,而这一生化活动对围食膜蛋白的结合程度、围食膜的完整性和孔隙率产生影响[7]。在黑腹果蝇中,克隆得到2个能编码具有典型CDA功能域的CDA,通过气管上几丁质的脱乙酰基限制气管延长[8]。赤拟谷盗第Ⅴ类CDA蛋白在中肠内特异性表达,利用RNA干扰技术对昆虫CDA表达进行调控,干扰昆虫正常生长发育和新陈代谢过程以及抵抗病原物的特性[9]。几丁质代谢随昆虫不同生长发育阶段而变化,对昆虫的正常生长发育至关重要,因此,通过破坏几丁质结构或几丁质代谢的平衡来防治害虫具有极大的发展潜力。
甜菜夜蛾Spodoptera exigua(Hübner),属鳞翅目、夜蛾科,是以危害蔬菜为主的杂食性害虫[10]。本研究克隆甜菜夜蛾几丁质脱乙酰酶secda7基因,体外表达的SeCDA7蛋白进行几丁质结合活性分析,利用实时定量PCR方法分析secda7基因转录水平在不同组织的表达,为明确深入探究昆虫几丁质脱乙酰酶的生理功能奠定理论基础,为甜菜夜蛾生物防治提供材料。
1 材料与方法 1.1 材料 1.1.1 供试昆虫与细胞培养甜菜夜蛾为北京农学院植保系农药实验室利用人工气候箱饲养;草地贪夜蛾昆虫细胞系(Sf9)为北京农学院植保系农药实验室27℃培养。
1.1.2 实验试剂、载体与菌株质粒pMD19-T、pET-30a(带有His标签)和pFastBac HT A(带有His标签)以及E.coli DH5α、BL21、JM109和E.coli DH10BacTM菌株由本实验室保存。
限制性内切酶、T4连接酶购自Promega公司;LA Taq酶、SYBR Premix Ex TaqTM购自Takara公司;RNA提取试剂盒购自QIAGEN公司;反转录试剂盒、质粒提取试剂盒购自Axygen公司;Mouse anti-6×His tag monoclonal antibody、Goat Anti-Mouse lgG(H+L)、NI-Agarose His标签蛋白纯化试剂盒购自康为试剂公司。
1.2 方法 1.2.1 甜菜夜蛾secda7基因的克隆及序列分析根据甜菜夜蛾中肠转录组数据,设计secda7全长基因引物(表 1)。提取甜菜夜蛾四龄幼虫中肠RNA,以反转录cDNA为模板,以secda7 F和secda7 R为引物进行PCR,扩增甜菜夜蛾几丁质脱乙酰酶cDNA序列片段。PCR产物经纯化后连接pMD19-T,转化DH5α感受态细胞,挑取单菌落进行双酶切鉴定后序列测定,并对secda7基因进行生物信息学分析。利用DNAMAN软件分析其开放阅读框及其氨基酸序列,NCBI Blast进行功能域分析,使用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽预测,用NetNGlyc1.0(http://www.cbs.dtu.dk/services/NetNGlyc/)预测N-联糖基化位点。
设计带有酶切位点的引物ySecda7 F和ySecda7 R进行secda7基因的克隆(表 1),将secda7与pET-30a原核表达载体使用T4连接酶进行连接反应,转化BL21感受态细胞,挑取单菌落进行双酶切鉴定。鉴定正确的阳性克隆于28℃培养时,分别加入不同浓度(0.25 mmol/L、0.5 mmol/L、0.75 mmol/L、1 mmol/L、1.25 mmol/L)的IPTG诱导蛋白表达,分别利用第一抗体小鼠抗6×His单克隆抗体(Mouse anti-6×His tag monoclonal antibody)(1:5 000)和第二抗体山羊抗小鼠IgG(Goat Anti-Mouse lgG)(1:20 000)抗体进行Western blot分析SeCDA7重组蛋白的表达。
1.2.3 secda7基因的真核表达设计带有酶切位点的引物zSecda7 F和zSecda7 F进行secda7基因的克隆(表 1),将secda7与pFastbac HTA真核表达载体进行连接,连接产物转化JM109感受态细胞,挑取单菌落进行双酶切鉴定。鉴定正确的重组质粒pFastbac HTA- secda7转化DH10 BacTM感受态细胞,利用蓝白板筛选方法筛选阳性克隆。鉴定正确重组Bacmid转染昆虫Sf9细胞,收集P3代病毒上清,分别利用第一抗体小鼠抗6×His单克隆抗体(Mouse anti-6×His tag monoclonal antibody)(1:5 000)和第二抗体山羊抗小鼠IgG(Goat Anti-Mouse lgG)(1:20 000)抗体进行Western blot分析SeCDA7重组蛋白的表达。
1.2.4 secda7基因在甜菜夜蛾各组织中的表达分析分别取甜菜夜蛾卵及预蛹,并将四龄甜菜夜蛾于冰上进行解剖,提取头、表皮、中肠、围食膜、马氏管、脂肪体和蜕皮,利用RNeasy Mini Kit试剂盒(QIAGEN)提取总RNA,测定浓度后使用RevertAid First Strand cDNA Synthesis Kit试剂盒(Thermo Scientific)进行反转录合成cDNA。设计secda7基因相关荧光定量引物qSecda7 F和qSecda7 R(表 1),以及以β-action为内参基因设计荧光定量引物(表 1),参照Takara SYBR Premin Ex TaqTM Ⅱ说明书进行实时定量PCR。利用相对定量公式2-△△Ct进行数据分析。
1.2.5 重组蛋白SeCDA7几丁质结合活性分析通过Molano等[11]及Li等[12]的方法制备再生几丁质,将10 g脱乙酰几丁质粉末溶于350 mL 10%的乙酸,室温过夜,次日,加入900 mL甲醇,利用玻璃纤维过滤后加入20 mL乙酸酐成凝胶状,静置30 min,用抹刀切片捣碎,用ddH2O洗至pH7.0。
取1 mL纯化后(利用NI-Agarose His标签蛋白纯化试剂盒纯化)的重组SeCDA7蛋白与400 mg再生几丁质在离心管中加入1 mmol/L PMSF室温温育,混匀后4℃过夜。次日,12 000 r/min离心3 min,弃上清,用1×PBS清洗2次,收集沉淀。将所得络合物分别与PBS、2%SDS、2%SDS+5%β-巯基乙醇、0.5 mol/L氯化钠、2%荧光增白剂和6 mol/L尿素室温温育15 min,离心后收集上清进行Western blot分析。
2 结果 2.1 secda7全长基因的获得及序列分析通过RT-PCR方法克隆得到编码甜菜夜蛾几丁质脱乙酰酶secda7基因。序列分析结果(图 1)显示,secda7基因(GanBank登录号为MG604929)全长1 431 bp,开放阅读框为1 134 bp,编码378个氨基酸,预测的SeCDA7蛋白的分子量和等电点约为43.156 kD和5.20。SignalP 4.1 Server进行信号肽分析显示,在17和18氨基酸位点之间存在一个信号肽切割位点;NetNGlyc 1.0 Server软件预测77和169氨基酸处存在两个N-糖基化位点;结构域分析表明SeCDA7具有一个多聚糖乙酰基转移酶催化区(氨基酸52-322位),属于第Ⅴ类CDA蛋白。其氨基酸序列与鳞翅目昆虫蓓带夜蛾几丁质脱乙酰酶(GenBank登录号HM357864)具有较高的相似性。

|
| 起始密码子ATG和终止密码子TAG加下划线加粗表示;核苷酸/氨基酸序列位置用左侧数字表示;信号肽加下划线表示;结构域加阴影标注表示 图 1 secda7基因cDNA序列及推测的氨基酸序列 |
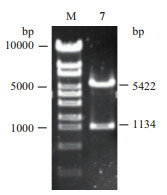
secda7基因与pET-30a载体连接转化BL21感受态细胞,成功构建重组菌株DH5α(pET-30a-secda7),经双酶切鉴定,获得5 422 bp载体和1 134 bp目的基因条带,证明成功构建目的基因的原核表达载体,结果见图 2。原核表达载体的成功构建,可使携带外源核酸序列的重组载体进入大肠杆菌中进行目的蛋白的表达。
|
| M:DL10000 marker;7:原核重组表达载体双酶切鉴定 图 2 原核重组表达载体酶切鉴定结果 |
用终浓度为1.25 mmol/L的IPTG在28℃对重组蛋白进行诱导,分别在诱导2、4、6、8 h取样,收集菌体沉淀进行检测。Western blot分析重组SeCDA7蛋白在大肠杆菌中的表达,结果见图 3,在28℃,用1.25 mmol/L IPTG诱导2 h SeCDA7蛋白表达量最高,蛋白大小约为47 kD。

|
| M:标准蛋白质;1:SeCDA7未经IPTG诱导;2-5:用1.25 mmol/L IPTG28℃分别诱导2 h、4 h、6 h、8 h表达 图 3 SeCDA7的SDS-PAGE及Western Blot表达图谱 |
将secda7基因与pFastbac HTA载体连接转化JM109感受态细胞,成功构建重组菌株JM109(pFastbac HTA-secda7),琼脂糖凝胶电泳(图 4)显示,出现4 776 bp载体大小条带和1 134 bp目的基因大小条带,证明成功构建目的基因的真核表达载体。将重组pFastbac HTA-secda7质粒转入E.coli DH10BacTM感受态细胞,蓝白斑筛选得到重组Bacmid质粒,经PCR鉴定,得到3 564 bp大小的条带,表明成功得到重组Bacmid质粒。

|
| M:DL10000 maker;1:pFastbac HTA-secda7/ EcoR Ⅰ & Hind Ⅲ;2:Bacmid DNA蓝斑对照;3:Bacmid DNA 图 4 重组杆状病毒转座载体的鉴定 |
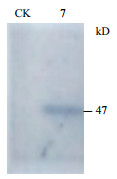
将重组Bacmid转染生长状态良好的Sf9昆虫细胞,昆虫细胞出现明显感染症状,细胞直径和细胞核增大,视野中出现颗粒体(图 5),得到P3代病毒培养72 h时,Western blot分析(图 6)显示SeCDA7在昆虫细胞中表达大小约为47 kD的目的蛋白。

|
| A:对照Sf9细胞;B:感染重组病毒72 h后的Sf9细胞 图 5 重组病毒转染前后细胞状态比较(10×) |
|
| CK:Sf9细胞;7:P3代侵染72 h后的病毒上清 图 6 SeCDA7表达产物的Western blot分析 |
实时定量PCR结果(图 7)显示,secda7基因在甜菜夜蛾的卵、预蛹、头、表皮、中肠、马氏管及脂肪体中有表达,但是表达量较低,在中肠中表达量最高,为中肠特异性表达蛋白。

|
| 图 7 secda7基因在甜菜夜蛾不同组织中的表达模式 |
外源表达的SeCDA7蛋白表现出几丁质结合活性,与鳞翅目昆虫粉纹夜蛾Tn-P42蛋白类似,在体外与几丁质可紧密结合,Western blot分析结果(图 8)显示,SeCDA7蛋白仅用强洗脱剂2%SDS+5%β-巯基乙醇和6 mol/L尿素以及2%荧光增白剂可将其洗脱,而用PBS、2%SDS、0.5 mol/L氯化钠不能将其洗脱。

|
| M:1:SeCDA7;2:2%荧光增白剂;3:2%SDS+5% β-巯基乙醇;4:6 mol/L尿素;5:PBS;6:2% SDS;7:0.5 mol/L氯化钠 图 8 SeCDA7重组蛋白几丁质结合活性Western blot分析 |
昆虫几丁质脱乙酰酶(Chitin deacetylase,CDA)是昆虫体内一类重要几丁质结合蛋白,可分布在虫体内多个含几丁质成分组织结构中,是几丁质降解酶系成员之一,能够催化几丁质中β-1,4糖苷键连接的N-乙酰基葡糖胺的乙酰胺基水解,将几丁质修饰为壳聚糖,在昆虫生长、发育和代谢中具有重要作用[13-15]。从2005年以来,研究者从不同的昆虫中开始探索CDA类蛋白的生物功能,这对昆虫生物防治具有重要生物学意义。
SeCDA7蛋白成功在大肠杆菌中表达,有利于研究蛋白纯化、定位及功能分析。利用pFastBac HT A载体使得SeCDA7蛋白成功在Sf9昆虫细胞中表达,并对其进行纯化与表达分析,并鉴定出SeCDA7蛋白具有几丁质结合活性,这与粉纹夜蛾(Trichoplusia ni)[6]、华北大黑鳃金龟(Holotrichia oblita Falder-mann)[16]第Ⅴ类CDA蛋白表现的几丁质结合活性相一致。通过荧光定量PCR技术,对secda7基因进行了转录水平的组织定位,属于中肠特异蛋白,这与第Ⅴ类CDA蛋白功能具有直接相关性。
SeCDA7蛋白属于昆虫中肠特异蛋白,围食膜作为昆虫体内抵御外源性物质的第一道天然屏障,具有保护中肠上皮细胞,阻止病原物的侵染等多种功能,围食膜主要由蛋白质、多糖和几丁质构成,其中蛋白质对于维持围食膜的致密结构至关重要,推测第Ⅴ类CDA蛋白与围食膜结构功能紧密相关,因此围食膜蛋白是进行害虫防治重要的切入点之一。对几丁质脱乙酰酶功能的研究不只局限于昆虫中,在一些真菌、细菌及蟹类、鱼类等海洋生物都有相关研究报道。在尼杜拉曲霉、苜蓿中华根瘤菌、构巢曲霉和稻瘟病菌中也分离得到不同几丁质脱乙酰酶类[17-20],参与不同的生理功能;在线虫中,也得到了几丁质脱乙酰酶类蛋白[21],在对药材甲虫的研究中,表明SpCDA1对于S. paniceum中幼虫到蛹转变是必不可少的[22],在对红雪蟹的研究中,仅在表皮中发现CDA存在,推测可能与蜕皮相关[23]。几丁质脱乙酰酶类蛋白的广泛研究,明确几丁质脱乙酰酶类蛋白在昆虫生长发育中的作用及深入探究几丁质脱乙酰酶的生理功能也显得尤为重要。
4 结论克隆了甜菜夜蛾几丁质脱乙酰酶secda7全长基因,在原核和真核中表达约47 kD的蛋白,生物信息学分析结果显示SeCDA7含有一个结构域,属于第Ⅴ类CDA蛋白,Western Blot分析结果显示SeCDA7蛋白具有几丁质结合活性,荧光定量PCR结果表明secda7基因属于中肠特异蛋白。
| [1] |
Khoushab F, Yamabhai M. Chitin research revisited[J]. Marine Drugs, 2010, 8(7): 1988-2012. DOI:10.3390/md8071988 |
| [2] |
Arakane Y, Muthukrishnan S. Insect chitinase and chitinase-like proteins[J]. Cell Mol Life Sci, 2010, 67: 201-216. DOI:10.1007/s00018-009-0161-9 |
| [3] |
Yasuyuki A, Radhika D, Khurshida B, et al. Analysis of functions of the chitin deacetylase gene family in Tribolium castaneum[J]. Insect Biochem Mol Biol, 2009, 39(5-6): 355-365. DOI:10.1016/j.ibmb.2009.02.002 |
| [4] |
Noh MY, Muthukrishnan S, Kramer KJ, et al. Group I chitin deacetylases are essential for higher order organization of chitin fibers in beetle cuticle[J]. Journal of Biological Chemistry, 2018, 293(18): 6985-6995. DOI:10.1074/jbc.RA117.001454 |
| [5] |
赵盼, 张学尧, 刘晓健, 等. 飞蝗几丁质脱乙酰基酶的真核表达、亲和纯化及酶活性[J]. 中国农业科学, 2017, 50(6): 1057-1066. |
| [6] |
Guo W, Li G, Pang Y, et al. A novel chitin-binding protein identified from the peritrophic membrane of the cabbage looper Trichoplusia ni[J]. Insect Biochem Mol Biol, 2005, 35(11): 1224-1234. DOI:10.1016/j.ibmb.2005.06.003 |
| [7] |
Toprak U, Baldwin D, Erlandson M, et al. A chitin deacetylase and putative insect intestinal lipases are components of the Mamestra configurata(Lepidoptera:Noctuidae) peritrophic matrix[J]. Insect Mol Biol, 2008, 17(5): 573-585. DOI:10.1111/j.1365-2583.2008.00829.x |
| [8] |
Liu SH, Li HF, Yang Y, et al. Genome-wide identification of chitinase and chitin deacetylase gene families in the oriental fruit fly, Bactrocera dorsalis(Hendel)[J]. Comparative Biochemistry and Physiology Part D:Genomics and Proteomics, 2018, 27: 13-22. DOI:10.1016/j.cbd.2018.04.005 |
| [9] |
Dixit R, Arakane Y, Specht CA, et al. Domain organization and phylogenetic analysis of proteins from the chitin deacetylase gene family of Tribolium castaneum and three other species of insects[J]. Insect Biochem Mol Biol, 2008, 38(4): 440-451. DOI:10.1016/j.ibmb.2007.12.002 |
| [10] |
胡科, 郑明, 汤汉华, 等. 孝感市棉田甜菜夜蛾的发生与防治[J]. 现代农业科技, 2018, 22: 98-101. |
| [11] |
Molano J, Durán A, Cabib E. A rapid and sensitive assay for chitinase using tritiated chitin[J]. Analytical Biochemistry, 1977, 83(2): 648-656. |
| [12] |
Li SY, Zhao D, Li J, et al. Response of CDA5 in Hyphantria cunea to Bt toxin ingestion and knockdown in transfected Sf9 cells[J]. Journal of Applied Entomology, 2017, 141: 308-314. DOI:10.1111/jen.12339 |
| [13] |
Zhao Y, Park RD, Muzzarelli RAA. Chitin deacetylases:properties and applications[J]. Marine Drugs, 2010, 8(1): 24-46. DOI:10.3390/md8010024 |
| [14] |
Tsigos I, Martinou A, Kafetzopoulos D, et al. Chitin deacetylases:new, versatile tools in biotechnology[J]. Trends in Biotechnology, 2000, 18(7): 305-312. DOI:10.1016/S0167-7799(00)01462-1 |
| [15] |
杨倩, 刘建辉, 蒋彤, 等. 几丁质脱乙酰酶的研究进展[J]. 食品研究与开发, 2017, 38(10): 100-103. |
| [16] |
赵丹, 闫晓平, 陆秀君, 等. 暗黑鳃金龟几丁质脱乙酰酶HpCDA5基因的细胞表达及活性分析[J]. 中国生物防治学报, 2018, 34(5): 686-692. |
| [17] |
Wang Y, Song JZ, Chen Y.The cDNA sequence of chitin deacetylase from Aspergillus nidulans and its phylogenetic analysis[C].Harbin: Study on Biological Control and Bio-technology.2009.
|
| [18] |
Chambon R, Pradeau S, Fort S, et al. High yield production of Rhizobium NodB chitin deacetylase and its use for in vitro synthesis of lipo-chitinoligosaccharide precursors[J]. Carbohydrate Research, 2017, 442: 25-30. DOI:10.1016/j.carres.2017.02.007 |
| [19] |
Liu Z, Gay LM, Tuveng TR, et al. Structure and function of a broad-specificity chitin deacetylase from Aspergillus nidulans FGSC A4[J]. Scientific Reports, 2017, 7(1): 1746. DOI:10.1038/s41598-017-02043-1 |
| [20] |
Kuroki M, Okauchi K, Yoshida S, et al. Chitin-deacetylase activity induces appressorium differentiation in the rice blast fung us Magnaporthe oryzae[J]. Scientific Reports, 2017, 7(1): 9697. DOI:10.1038/s41598-017-10322-0 |
| [21] |
Aranda-Martinez A, Grifoll-Romero L, Aragunde H, et al. Expression and specificity of a chitin deacetylase from the nematophagous fungus Pochonia chlamydosporia potentially involvedin pathogenicity[J]. Scientific Reports, 2018, 8(1): 2170. DOI:10.1038/s41598-018-19902-0 |
| [22] |
Yang WJ, Xu KK, Yan X, et al. Functional characterization of chitin deacetylase 1 gene disrupting larval-pupal transition in the drugstore beetle using RNA interference[J]. Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology, 2018, 219-220: 10-16. DOI:10.1016/j.cbpb.2018.03.001 |
| [23] |
Fujimori K, Fukushima H, Matsumiya M. Molecular cloning and phylogenetic analysis of a chitin deacetylase isolated from the epidermis of the red snow crab Chionoecetes japonicus[J]. Advances in Bioscience and Biotechnology, 2018, 9(1): 52-62. DOI:10.4236/abb.2018.91005 |





