2. 北京杂交小麦工程技术研究中心,北京 100097
2. Beijing Hybrid Wheat Engineering and Technology Research Center, Beijing 100097
小麦是我国重要的粮食作物之一,杂种优势利用是提高小麦产量和品质的主要途径,而我国杂交育种的主要途径是化杀法、二系法和三系法[1-2]。光温敏雄性不育系是二系杂交小麦的核心种质材料,其育性受外界光照条件和温度调控[3]。在低温短日照条件下,不育系的不育度达到99%以上,可用于杂交种的种子生产;在高温长日照条件下,不育系又表现为雄性可育,自交结实率达到80%左右从而实现自我繁殖[4-5]。当前对于不育系小麦的育性转换条件、育性遗传规律、不育系花粉形态及基因定位等已有研究[6],然而对杂交种育性恢复相关基因的研究相对滞后。挖掘一些对小麦杂交种具有育性恢复功能的基因,对于提高杂交小麦的制种产量具有重要意义。
PPR(Pentatricopeptide repeats)蛋白由2-30个串联重复的PPR基序组成,每个基序含有35个氨基酸残基[7-8]。由35个简并氨基酸组成的基序称为PPR基序,简称P基序。PPR长基序(L-motif)含有35或36个氨基酸残基,PPR短基序(S-motif)含有31个氨基酸残基。大多数PPR蛋白定位于线粒体或者叶绿体[9-10],也有少量定位于细胞核。研究表明,PPR家族编码的蛋白能够参与细胞质雄性不育系的育性恢复[11-12],该家族基因的沉默可能引起胚胎致死[13]。当前,作物中所鉴定的大多数育性恢复基因为PPR基因家族成员,如Koizuka等[14]发现Rfk1能够降低CMS相关转录产物ORF125;水稻Rf-1加工线粒体基因atp6恢复育性[15];水稻RF5与GRP162形成复合体,加工atp6-orf79恢复红莲型细胞质雄性不育[16];水稻RF6与OsHXK6互作共同加工atp6-orf79[17]等。然而在小麦中与育性恢复相关PPR基因的研究鲜见报道。
本研究对小麦PPR基因家族进行了全基因组鉴定,通过比较基因组学筛选了一些育性恢复相关的PPR基因,进一步利用在不育条件下减数分裂期不同杂交组合的幼穗进行荧光定量规模化筛选与杂交种育性恢复相关的PPR基因,为二系杂交小麦杂交种的育性恢复提供了重要的候选基因资源,为进一步研究PPR基因对杂交种的育性恢复机制奠定了基础。
1 材料与方法 1.1 材料 1.1.1 试验材料以北京杂交小麦工程技术研究中心提供的光温敏雄性不育系13S122和2个不同恢复系09Y花117-4、原种场亲05-4组成的杂交组合为研究材料。
1.1.2 主要试剂Trizol试剂购自Ambion公司,反转录试剂盒HiScript Ⅱ Q RT SuperMix for qPCR(+gDNA wiper)、实时荧光定量试剂盒ChamQ SYBR qPCR Master Mix均购自南京诺唯赞生物科技有限公司,其他生化试剂为进口或国产试剂。
1.2 方法 1.2.1 小麦PPR基因家族鉴定及染色体分布、进化分析从EnsemblPlants(http://plants.ensembl.org/index.html)数据库下载小麦和水稻的基因组序列、基因注释文件和蛋白质序列。在Pfam29.0[18](http://pfam.xfam.org/)下载PPR基因家族的种子序列(PF01535),使用BLAST[19]将小麦和水稻的蛋白序列与种子序列进行比对,得到本家族的候选成员。再用pfam_scan基于Pfam数据库的HMM文件进行比对,最终得到小麦及水稻PPR基因家族的成员,将这些成员重新编号。
根据EnsemblPlants中小麦PPR基因家族成员注释信息将所有成员定位到不同染色体,使用Inparanoid[20]分析小麦PPR基因家族成员的同源等位基因,再使用Circos[21]基于基因注释信息对同源关系进行可视化。
1.2.2 小麦PPR基因家族的进化分析将从EnsemblPlants数据库(http://plants.ensembl.org/index.html/)下载的小麦和水稻PPR基因家族的蛋白序列用MEGA6.0[22]中的MUSCLE进行序列比对,使用默认参数。基于序列比对结果,用MEGA6.0软件,采用邻接法(Neighbor-joining)构建进化树,Bootstrap值为1 000,Partial deletion 95。
1.2.3 育性恢复相关基因的获取从EMBL-EBI(https://www.ebi.ac.uk/)获取了7个水稻恢复PPR基因的蛋白序列,然后通过Blast搜索这7个基因在小麦中的高同源基因。
1.2.4 实时荧光定量PCR参考苑少华等[23]方法,统计不同杂交组合材料的结实率和结实小穗率。根据表型统计结果,从中选取了1个光温敏雄性不育系(13S122)和1个低恢复系(09Y花117-4)及1个高恢复系(原种场亲05-4)配置杂交组合,并对2个杂交组合在河南南阳,即不育条件下减数分裂期的小麦幼穗为材料,用镊子剥离幼穗,液氮中速冻后于-80℃保存备用。
采用Trizol试剂(Ambion,USA)提取小麦小穗总RNA,用南京诺唯赞反转录试剂盒HiScript Ⅱ Q RT SuperMix for qPCR(+gDNA wiper)进行反转录。以18S核糖体RNA作为内参基因(表 1),采用南京诺唯赞实时荧光定量试剂盒ChamQ SYBR qPCR Master Mix和实时荧光定量伯乐PCR仪(BioRad)进行实时荧光检测,反应体系与程序参考ChamQ SYBR qPCR Master Mix说明书,数据处理采用相对定量法2-△△CT法[24]。
经EMBL-EBI确认,PPR基因家族的Pfam ID为PF01535,运用HMMER程序搜索小麦和水稻的蛋白序列,去除冗余,最终分别获取1 351和495个PPR基因家族成员。将小麦和水稻PPR基因家族成员统一编号为TaRF1-TaRF1351和OsRF1-OsRF495。
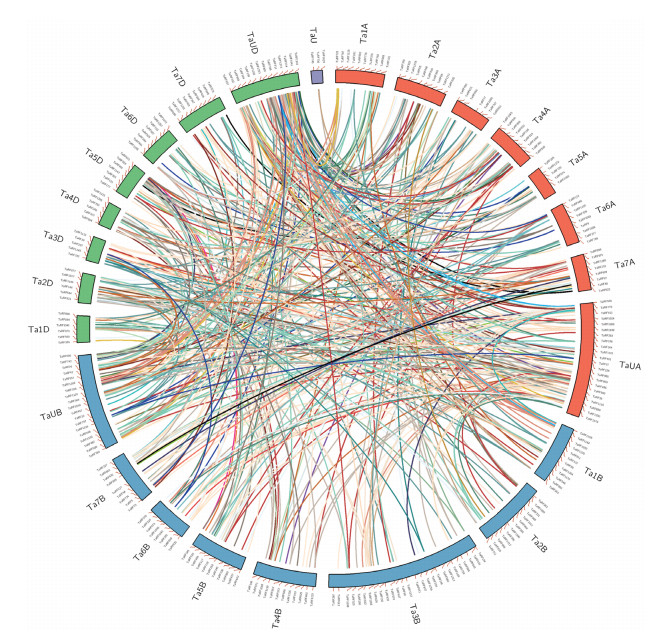
根据EnsemblPlants数据库中小麦基因组注释信息,将小麦1 351个PPR基因定位到不同染色体(图 1)。小麦PPR基因在各染色体上均有分布,A、B、D染色体分布为455、446和450个,说明B染色体在进化过程中可能发生了基因丢失事件。第1-第7染色体分别鉴定了194、219、240、167、193、154和184个成员,第6染色体成员最少,而且发现第6染色体长度也较小。同时,含有最长染色体的3B染色体分布成员最多,共鉴定到88个基因,而4B染色体含有成员最少,仅包含48个PPR基因,推测该家族成员数量可能和染色体长度有关,另外可能也与染色体结构的差异有关。
|
| 图 1 小麦PPR基因家族染色体分布与共线关系 |
从共线关系结果看出小麦PPR基因家族不仅在A、B、D 3个同源染色体间有大量同源基因,而且在A、B、D 3个同源染色体中的2个以上,以及在非同源染色体上也存在大量的同源基因,说明小麦PPR基因家族成员具有高度重复序列,基因重复事件非常复杂。
2.2 小麦和水稻PPR基因家族进化分析将鉴定的小麦与水稻PPR基因家族成员蛋白序列基于MEGA6.0的MUSCLE比对,用NJ法构建进化树,利用EvolView对进化树进行可视化(图 2)。结合Chen等[25]鉴定的水稻PPR基因家族成员,发现水稻PPR基因家族的P亚族集中分布于Ⅰ亚族,而水稻的PLS亚族集中分布于Ⅱ亚族。而E1、E2以及PLS亚族在Ⅱ亚族中分布较为分散,说明PPR基因家族蛋白序列构成较为复杂。小麦PPR家族共有760个成员与水稻的P亚族聚在一起,587个成员与PLS亚族聚在一起,有4个成员未与水稻PPR基因聚在一起。

|
| 图 2 小麦与水稻PPR基因家族系统进化树 |
从EMBL网站寻找水稻已鉴定的具有育性恢复功能的PPR基因,在EnsemblPlants网站搜索与小麦高同源的基因,将搜索到的高同源基因与水稻的育性恢复基因构建系统进化树(图 3)。发现水稻中具有育性恢复功能的7个基因间进化关系较近,猜测PPR基因家族在不同物种间分化程度较高。通过DNAMAN进行比对,发现候选基因中与水稻育性恢复基因同源性达到40%以上有7个,TaRF723和TaRF1168与水稻Rf-1A的同源性分别为45.58%和50.13%,TaRF35和TaRF1104与Rf-1B的同源性分别为44.35%和44.64%,TaRF118与Rf-4like的同源性为42.38%,TaRF1270和TaRF1164与PPR794的同源性分别为44.57%和44.08%。

|
| 图 3 小麦、水稻PPR同源基因进化树 |
采用国际法结实率统计不同杂交组合材料的穗粒数、结实小穗数、总小穗数等[国际法结实率=穗粒数/(2×总小穗数]×100%和结实小穗率=(结实小穗数/总小穗数)×100%)。结果(表 2)发现,13S122与09Y花117-4杂交后,F1结实率、结实小穗率、穗粒数分别为73.37%、72.57%和25.67%,均比父本09Y花117-4低,确定09Y花117-4为低恢复系。而13S122与原种场亲05-4杂交后,F1结实率和穗粒数分别为127.78%和49.33%,而原种场亲05-4结实率和结实小穗率分别为98.55%和36.33,F1远远大于父本材料原种场亲05-4,结实小穗率略低于父本,认定原种场亲05-4为高恢复系。
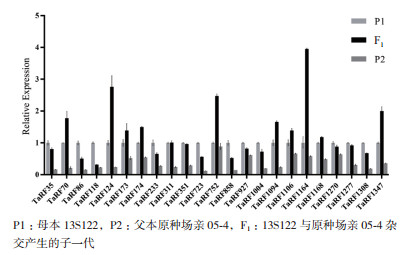
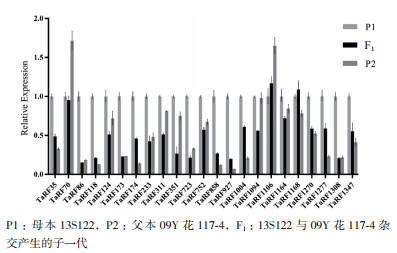
为了筛选鉴定育性恢复基因,从大量杂交组合中挑选了一个高恢复组合和一个低恢复组合,对与水稻育性恢复高度同源的23个小麦PPR基因进行荧光定量表达筛选。结果(图 4)发现,在高恢复系组合中,TaRF124、TaRF752、TaRF1164和TaRF1347表现出明显的超亲表达,差异倍数达到2倍以上。而TaRF70、TaRF173、TaRF174、TaRF752、TaRF-1094、TaRF1106和TaRF1168的表达量仅有微弱的超亲表现。同时,这些基因在低恢复系组合中除了TaRF1168有微弱的超亲表达外,其他PPR基因在F1的表达量基本处于低亲水平,这些PPR基因的表达与其杂交种的结实率表型具有高度的一致性(图 5)。因此,推测TaRF124、TaRF752、TaRF1164、TaRF1347可能对杂交种结实率起到育性恢复作用。
|
| P1:母本13S122,P2:父本原种场亲05-4,F1:13S122与原种场亲05-4杂交产生的子一代 图 4 23个候选基因在高恢复度杂交组合中的表达分析 |
|
| P1:母本13S122,P2:父本09Y花117-4,F1:13S122与09Y花117-4杂交产生的子一代 图 5 23个候选基因在低恢复度杂交组合中的表达分析 |
随着不同物种基因组测序的完成,PPR基因家族成员鉴定也有了很大进展。目前,除了模式植物拟南芥[1]、水稻[25]、番茄[26]和烟草[27],甘蓝型油菜[28]、棉花[29]、谷子[30]等物种的PPR基因家族也已被鉴定,对各物种中一些PPR基因也进行了相关研究。研究发现,PPR基因能够参与线粒体和叶绿体基因转录后加工、调控雄性不育相关基因的表达、参与胚胎形成、参与逆境防御等。本研究运用生物信息学方法对小麦全基因组进行搜索,鉴定了1 351个PPR基因,与所有已鉴定PPR基因家族成员的其他物种相比,小麦含有最多的PPR基因家族成员,这可能与小麦的庞大基因组有关。另外,与Chen等[25]鉴定的水稻PPR基因家族的染色体分布信息进行对比后,发现二者虽均分布于该物种所有染色体上,但是水稻PPR基因家族在染色体上分布相对集中,而小麦PPR基因家族成员则分布相对分散,推测小麦PPR基因家族成员的功能可能具有多样性和复杂性。
此外,本研究结合了二系杂交小麦的农艺性状,不仅在不育系与可育系之间做了表达分析,而且,在二者杂交后的子一代也做了表达分析,能充分反应PPR基因对杂交种的育性恢复是否起到作用。有趣的是,在高恢复系杂交组合中,候选基因在不育系中表达水平都比恢复系表达水平高,在低恢复系杂交组合中,TaRF70和TaRF1106在不育系表达水平比恢复系表达水平低,而其他候选基因同样是不育系表达水平比恢复系表达水平高,这与Liu等[31]在甘蓝型油菜中鉴定的育性恢复基因Rfn在不育系181A和恢复系H5中表达模式相似,这种差异表达模式有待进一步研究。本研究发现TaRF124、TaRF752、TaRF1164、TaRF1347在高恢复系杂交组合中有明显的超亲表达,而在低恢复系杂交组合中呈现了低亲的表达模式,推测这些基因可能参与调控杂交种的育性恢复,但还需要通过基因编辑技术来深入研究候选PPR基因的功能。
4 结论从小麦最新基因组数据中鉴定出1 351个PPR基因,候选出了与水稻高度同源的23个小麦PPR基因,并初步筛选了4个育性恢复相关PPR基因。
| [1] |
赵昌平. 中国杂交小麦研究现状与趋势[J]. 中国农业科技导报, 2010, 12(2): 5-8. DOI:10.3969/j.issn.1008-0864.2010.02.02 |
| [2] |
张胜全, 陈兆波, 王拯, 等. 二系杂交小麦群体结构与产量构成分析[J]. 作物杂志, 2017(6): 45-49. |
| [3] |
江红梅, 张立平. 小麦光温敏雄性不育遗传研究进展[J]. 种子, 2009, 28(5): 56-59. |
| [4] |
付志远, 秦永田, 汤继华. 主要作物光温敏核雄性不育基因的研究进展与应用[J]. 中国生物工程杂志, 2018, 38(1): 115-125. |
| [5] |
赵昌平, 张立平, 李云伏, 等. 小麦光温敏雄性不育相关基因的DDRT-PCR分析及功能预测[J]. 中国生物化学与分子生物学报, 2007, 23(1): 56-62. DOI:10.3969/j.issn.1007-7626.2007.01.010 |
| [6] |
唐忠辉.小麦温敏雄性不育系BS366不育机理探讨[D].武汉: 华中农业大学, 2011. http://cdmd.cnki.com.cn/Article/CDMD-10504-1011405212.htm
|
| [7] |
Lurin C, Andrés C, Aubourg S, et al. Genome-wide analysis of Arabidopsis pentatricopeptide repeat proteins reveals their essential role in organelle biogenesis[J]. The Plant Cell, 2004, 16(8): 2089-2103. DOI:10.1105/tpc.104.022236 |
| [8] |
黄俊然, 黄文超. 三角形五肽重复蛋白研究进展[J]. 湖北农业科学, 2018, 57(23): 19-23. |
| [9] |
陆萍, 俞嘉宁. PPR蛋白影响植物生长发育的研究进展[J]. 植物生理学报, 2013, 49(10): 989-999. |
| [10] |
Barkan A, Small I. Pentatricopeptide repeat proteins in plants[J]. Annual Review Plant Biology, 2014, 65: 415-442. DOI:10.1146/annurev-arplant-050213-040159 |
| [11] |
Schmitz-Linneweber C, Small I. Pentatricopeptide repeat proteins:a socket set for organelle gene expression[J]. Trends in Plant Science, 2008, 13(12): 663-670. DOI:10.1016/j.tplants.2008.10.001 |
| [12] |
Saha D, Prasad AM, Srinivasan R. Pentatricopeptide repeat proteins and their emerging roles in plants[J]. Plant Physiology and Biochemistry, 2007, 45(8): 521-534. DOI:10.1016/j.plaphy.2007.03.026 |
| [13] |
Verbitskiy D, Zehrmann A, Härtel B, et al. Two related RNA editing proteins target the same sites in mitochondria of Arabidopsis thaliana[J]. Journal of Biology Chemistry, 2012, 287(45): 38064-38072. DOI:10.1074/jbc.M112.397992 |
| [14] |
Koizuka N, Imai R, Fujimoto H, et al. Genetic characterization of a pentatricopeptide repeat protein gene, orf687, that restores fertility in the cytoplasmic male-sterile Kosena radish[J]. The Plant Journal, 2003, 34: 407-415. DOI:10.1046/j.1365-313X.2003.01735.x |
| [15] |
Kazama T, Toriyama K. A pentatricopeptide repeat-containing gene that promotes the processing of aberrant atp6 RNA of cytoplasmic male-sterile rice[J]. FEBS Letters, 2003, 544: 99-102. DOI:10.1016/S0014-5793(03)00480-0 |
| [16] |
Hu J, Wang K, Huang WC, et al. The rice pentatricopeptide repeat protein RF5 restores fertility in hong-lian cytoplasmic male-sterile lines via a complex with the glycine-rich protein GRP162[J]. The Plant Cell, 2012, 24(1): 109-122. DOI:10.1105/tpc.111.093211 |
| [17] |
Huang WC, Yu CC, Hu J, et al. Pentatricopeptide-repeat family protein RF6 functions with hexokinase 6 to rescue rice cytoplasmic male sterility[J]. Proc Natl Acad Sci USA, 2015, 112(48): 14984-14989. DOI:10.1073/pnas.1511748112 |
| [18] |
Finn RD, Coggill P, Eberhardt RY, et al. The Pfam protein families database:towards a more sustainable future[J]. Nucleic Acids Research, 2016, 44: 279-285. DOI:10.1093/nar/gkv1344 |
| [19] |
Kent WJ. BLAT——the BLAST-like alignment tool[J]. Genome Research, 2002, 12(4): 656-664. DOI:10.1101/gr.229202 |
| [20] |
O' Brien KP, Remm M, Sonnhammer EL. Inparanoid:a comprehensive database of eukaryotic orthologs[J]. Nucleic Acids Research, 2005, 33: 476-480. |
| [21] |
Krzywinski M, Schein J, Birol I, et al. Circos:an information aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639-1645. DOI:10.1101/gr.092759.109 |
| [22] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular evolutionary genetics analysis version 6. 0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [23] |
苑少华, 段文静, 白建芳, 等. F型小麦雄性不育系恢复性研究[J]. 麦类作物学报, 2018, 38(6): 654-660. |
| [24] |
Livark KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT Method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [25] |
Chen G, Zou Y, Hu J, et al. Genome-wide analysis of the rice PPR gene family and their expression profiles under different stress treatments[J]. BMC Genomics, 2018, 19(1): 720. DOI:10.1186/s12864-018-5088-9 |
| [26] |
丁安明, 李凌, 屈旭, 等. 番茄PPR基因家族的鉴定与生物信息学分析[J]. 遗传, 2014(1): 77-84. |
| [27] |
丁安明.番茄与烟草PPR基因家族分析及育性相关基因功能研究[D].北京: 中国农业科学院, 2014. http://cdmd.cnki.com.cn/Article/CDMD-82101-1014326586.htm
|
| [28] |
谭晖, 官春云. 甘蓝型油菜PPR家族生物信息学分析与新疆野生油菜候选育性基因克隆[J]. 作物研究, 2017, 31(3): 246-255. |
| [29] |
Zhao N, Wang YM, Hua JP. Genomewide identification of PPR gene family and prediction analysis on restorer gene in Gossypium[J]. Journal of Genetics, 2018, 97(5): 1083-1095. DOI:10.1007/s12041-018-0993-x |
| [30] |
Liu JM, Xu ZS, Lu PP, et al. Genome-wide investigation and expression analyses of the pentatricopeptide repeat protein gene family in foxtail millet[J]. BMC Genomics, 2016, 17(1): 840. DOI:10.1186/s12864-016-3184-2 |
| [31] |
Liu Z, Dong FM, Wang X, et al. A pentatricopeptide repeat protein restores nap cytoplasmic male sterility in Brassica napes[J]. Journal of Experimental Botany, 2017, 68(15): 4115-4123. DOI:10.1093/jxb/erx239 |






