microRNA(miRNA)是一类大小为20-24 nt、具有调控功能的内源性非编码RNA。众多的miRNA参与了植物对低温的感受、响应、信号转导和适应过程[1-2]。在miRNA研究中,通常需要对miRNA在植物各组织和器官中的表达量进行分析,确定miRNA表达量对于探究miRNA的生物学功能尤其重要[3-4]。实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)技术具有高灵敏性、特异性、快速简单、低样本浓度需求等优点,是研究基因表达的首选方法[1-2, 5]。然而,qRT-PCR结果中基因表达量的精确和准确性分析依赖于所选择的内参基因[6]。在qRT-PCR中,最常用的植物内参基因包括肌动蛋白(ACT)、甘油醛3-磷酸脱氢酶(GAPDH)、18S核糖体RNA(18S)和F-BOX家族蛋白(FP),但在miRNA定量检测中并不选择这些内参基因[7-8]。研究表明当选用ACT作为miRNA研究的内参时,由于目的miRNA与ACT的大小有较大的差异,在校正miRNA数据时并不理想[9]。不经筛选直接使用常见内参基因会降低试验结果的准确性[10-12]。内参基因的稳定性差导致基因表达量检测的误差,如在研究杂交兰(Cymbidium hybrid)花器官不同部位中八氢番茄红素脱氢酶基因(ChPDS)的表达模式时,以杂交兰泛素蛋白基因(ChUBQ)、杂交兰延伸因子基因(ChEF-1α)、杂交兰微管蛋白基因(ChTUB)、杂交兰肌动蛋白基因(ChACT)及杂交兰泛素蛋白基因(ChUBQ)和杂交兰延伸因子基因(ChEF-1α)组合进行校正的杂交兰ChPDS表达模式均为花瓣 > 唇瓣 > 蕊柱,而以稳定性最差的杂交兰RNA聚合酶β亚基基因(ChrpoB)进行校正时,杂交兰ChPDS相对表达量为花瓣 > 蕊柱 > 唇瓣[13]。因此,针对实际情况筛选内参基因很有必要性[1-2, 5]。
小桐子(Jatropha curcas)是一种有巨大综合开发潜力的多用途能源植物树种,原产热带及亚热带地区,抗寒性较弱,是一种喜温冷敏植物[14-16]。Maes等[17]进行的世界范围内的调查表明,小桐子生长适宜的年均温度为19.3-27.2℃,最低月均温度要高于10.5℃。而随着小桐子栽培的北移和向高海拔山区发展,低温冷害已成为限制小桐子产业的主要环境因素[15-17]。为研究miRNA在小桐子低温胁迫与适应过程中的调控作用,本课题组前期进行了小桐子小RNA-seq分析,初步获得了差异表达与稳定表达的miRNA信息[18],但要进行进一步qRT-PCR验证,需要筛选合适的内参基因用于miRNA定量分析。但目前尚未见对小桐子miRNA qRT-PCR内参基因筛选的报道。
本研究基于小桐子小RNA-seq数据[18],筛选低温处理前后表达量相对稳定的10个miRNA以及在其他物种miRNA定量分析中常用的U6作为候选内参,采用qRT-PCR方法对候选内参基因在低温胁迫下的表达稳定性进行分析,以期筛选出可用于小桐子低温胁迫下miRNA差异表达分析的稳定内参基因,为小桐子miRNA表达的定量分析提供参考。
1 材料与方法 1.1 材料 1.1.1 实验材料选取饱满的小桐子种子,参照李忠光等[19]方法,用1.5% CuSO4溶液消毒,在恒温培养箱中萌发5 d,将发芽的种子种在营养土中,在相对湿度75%、26/20℃、16/8 h光周期的培养箱中培养21 d,至第3片真叶展开[20]。
1.1.2 低温处理在小桐子长至第3片真叶展开时,转入1℃光照培养箱中低温处理5 d,每天取样1次(LT1d-LT5d,取用第2片及以上展开真叶)。以未处理的小桐子作为对照1(C0d);另在正常培养条件下第26天取样作为对照2(C5d)。依据前期结果,在1℃处理5 d时,小桐子幼苗死亡率约70%[20-21]。
1.2 方法 1.2.1 样品miRNA提取及cDNA合成参照Plant miRNA Kit(OMEGA公司)说明书提取miRNA,2%琼脂糖凝胶电泳检测miRNA的质量,NanoPhotometer-N60超微量分光光度仪(Implen,Germany)测定浓度,并通过OD260/OD280和OD260/OD230检测miRNA的纯度。采用miRcute miRNA First-Strand cDNA Synthesis Kit(Tiangen,北京)反转录cDNA。反应体系为Reaction Buffer 10 μL、Enzyme Mix 2 μL、miRNA 2 μL,加水至20 μL。42℃孵育1 h,并在95℃加热3 min失活逆转录酶(RT enzyme mix),进行miRNA加A尾反应以及cDNA第一链合成。cDNA用NanoPhotometer-N60超微量分光光度仪(Implen,Germany)测定浓度,每份样本终浓度为100 ng/µL,取1 µL用于qRT-PCR。
1.2.2 qRT-PCR各供选基因引物设计在11个候选内参基因中,U6 snRNA序列来自于Jatropha curcas Database(JCDB)和NCBI;10个来自小桐子低温胁迫下小RNA-seq[18]中表达稳定的miRNA,按照其序列设计引物,反向引物为:5'-GCTGTCAACGAT-ACGCTACGTAACG-3'。利用DNAMAN 8(Lynnon,USA)进行引物互补检测,减少引物自身延伸或两两互补延伸形成二聚体的可能性(表 1)。
根据TaKaRa公司的SYBR Premix Ex TaqTM Ⅱ(Tli RNase H PLUS)试剂盒说明书配置10 μL反应体系,cDNA模板1 µL(10 ng/µL)、正反向引物各0.4 µL(10 μmol/L)、2×TransStar Tip Green qPCR SuperMix 5 µL和ddH2O 3.2 µL。所有操作均在冰上进行,3个重复,3次生物学重复。反应程序为95℃ 30 s;95℃ 5 s,60℃ 30 s,45个循环,并在65-97℃进行熔解曲线分析。利用LightCycler® 96 System(Roche,Switzerland)进行qRT-PCR反应。
1.2.4 数据处理记录qRT-PCR分析得出的基因转录水平Ct值,每个基因在不同处理样本中的Ct值测定3个重复。qRT-PCR熔解曲线利用LightCycler 96 SW 1.1(Roche,Switzerland)软件进行作图及分析;利用geNorm(https://genorm.cmgg.be/)、NormFinder(https://www.moma.dk/normfinder-software/)和BestKeeper(https://www.gene-quantification.de/bestkeeper.html)分析11个候选内参基因的表达稳定性;利用Microsoft Excel 2016进行数据作图。
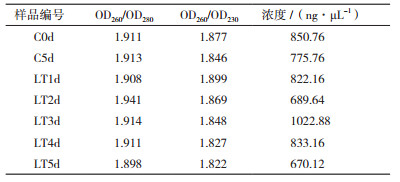
2 结果 2.1 miRNA质量检测通过2%琼脂糖凝胶电泳检测提取的miRNA样品(图 1)。可见提取的miRNA质量较好;各样品中miRNA的OD260/OD280、OD260/OD230的值均大于1.8(表 2),miRNA质量满足后续实验的需求。
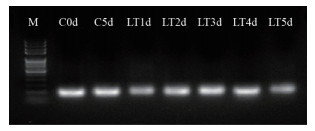
|
| M:DNA Marker 100 图 1 提取的miRNA2%的琼脂糖凝胶电泳图 |
以miRNA cDNA第一链为模板进行qRT-PCR扩增,用2%琼脂糖凝胶电泳检测(图 2)。11条内参基因条带单一,通过测序、比对等检测,确定扩增产物为引物特异性扩增产物,符合预期结果。
|
| M:DNA Marker 100 图 2 内参基因的qRT-PCR分析 |
经熔解曲线分析,11条候选内参基因的熔解曲线均呈现明显的单一峰(图 3),不存在引物二聚体,并且每个样本的重复性较好,进一步说明引物的特异性,可进行后续实验分析。

|
| -dF/dT:荧光信号强度负一次导数 图 3 十一个候选内参基因的qRT-PCR熔解曲线 |
11个候选内参基因在不同温度处理的小桐子叶片中,平均Ct值变化范围为10.53-26.03(图 4),其中U6 Ct值最小,miR2612 Ct值最大(图 5)。
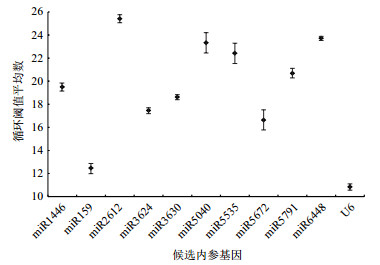
|
| 图 4 11个候选内参基因平均Ct值分布 |

|
| 图 5 各候选基因在不同处理下叶片组织中Ct值分布 |
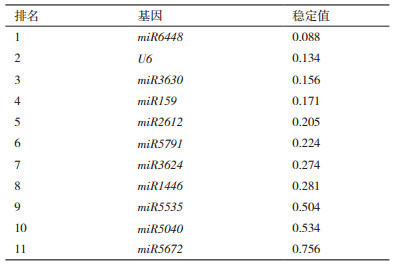
利用GeNorm软件对11个候选内参基因在不同处理下叶片组织中稳定性(M值)分析(图 6),M值由高到低为miR5672、miR5040、miR5535、miR1446、miR3624、miR5791、miR2612、miR159、miR3630、U6、miR6448,所选候选内参基因的M值均小于1.5,表明候选基因表达都较稳定,M值最低的是miR6448;其次是U6。可见miR6448在低温胁迫下表达最稳定,其次是U6。

|
| 图 6 不同处理下叶片组织中内参基因的GeNorm分析 |
通过对不同处理下叶片组织中内参基因的NormFinder分析(表 3),发现稳定值最小的是miR6448(0.088);其次是U6(0.134)。这与GeNorm分析得出的结果一致。
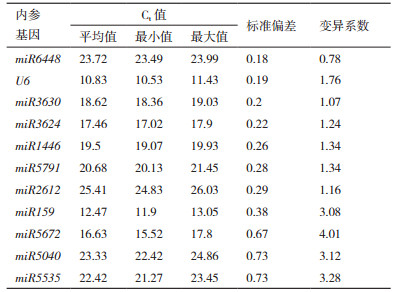
由BestKeeper分析标准偏差和变异系数,miRNA6448的标准偏差(0.18)和变异系数(0.78)都是最小的,表明miRNA6448在低温胁迫下的小桐子中表达最稳定。
3 讨论进行qRT-PCR时,对内参基因的筛选非常有必要。内参基因在qRT-PCR中可以降低或校正基因定量过程中存在的误差,选择合适的内参基因对于基因定量表达的结果有重要意义[3, 10]。而在选择内参基因时,除内参基因的稳定性,内参基因的表达丰度也会影响qRT-PCR结果[13, 22-23]。内参基因与所检测基因之间的Ct值差异越小,扩增效率对计算结果的影响也会越小,计算的结果会更加准确[23]。如果目标基因和内参基因表达量(Ct值)过大,用内参基因来衡量目标基因的表达量就不完全可靠[10, 23]。在本研究中,miR6448的Ct值为23左右,丰度适中,可以用于检测qRT-PCR试验中丰度较适中的转录本;U6的Ct值为10左右,可以用于miRNA qRT-PCR实验中丰度较高的转录本检测。
在进行miRNA研究时,提取的miRNA中往往弃除了mRNA等长链RNA,因此,相对链长较长的mRNA无法再用作miRNA定量研究的内参[24]。在qRT-PCR中,最常用的植物内参基因包括ACT、GAPDH、18S和F-BOX家族蛋白,通常在miRNA定量检测中并不选择这些内参基因[7-8]。常用于miRNA荧光定量的内参基因多为一些片段长度相对较短的基因,例如U6[3, 25-26]。在大豆[27]、小麦[28]、龙眼[29]和百合[8]等植物miRNA的qRT-PCR分析中,miRNA的表达水平比最常用的ACT、GAPDH、F-BOX等蛋白质编码基因稳定得多。
在miRNA的qRT-PCR分析时,常常需要根据不同的植物材料和处理条件来选择合适的内参。茶树低温胁迫下,miRNA(PC-3p-222)在不同低温处理以及不同茶树组织中表达最为稳定,可作为茶树低温胁迫下miRNA的qRT-PCR的内参基因[30]。甘蔗芽在低温胁迫下,miR171/18S rRNA和miR171/miR5059表达最稳定[31]。盐胁迫下,U6适合作为大豆根组织miRNA研究的内参[32]。葡萄在盐胁迫和低温胁迫下,U6是表达最稳定的基因[1]。本研究发现,小桐子叶片组织对低温胁迫处理响应的miRNA中,miR6448和U6的表达表现稳定。可见miRNA内参基因除特异性外,像U6这样的基因也表现出一定的通用性。
4 结论低温胁迫下表达最稳定的基因是miR6448和U6。miR6448适用于表达丰度适中的microRNA基因表达定量分析中;U6适用于丰度较高的microRNA的qRT-PCR分析。
| [1] |
Megha S, Basu U, Kav NNV. Regulation of low temperature stress in plants by microRNAs[J]. Plant, Cell & Environment, 2018, 41: 1-15. |
| [2] |
Bustin SA, Nolan T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction[J]. Journal of Biomolecular Techniques, 2004, 15(3): 155-166. |
| [3] |
Luo M, Gao Z, Li H, et al. Selection of reference genes for miRNA qRT-PCR under abiotic stress in grapevine[J]. Sci Rep, 2018, 8(1): 4444. DOI:10.1038/s41598-018-22743-6 |
| [4] |
Zhang FT, Luo Y, Zhang M, et al. Identification and characterization of drought stress-responsive novel microRNAs in Dongxiang wild rice[J]. Rice Science, 2018, 25(4): 175-184. DOI:10.1016/j.rsci.2018.06.001 |
| [5] |
Bae IS, Seo KS, Kim SH. Identification of endogenous microRNA references in porcine serum for quantitative real-time PCR normalization[J]. Molecular Biology Reports, 2018, 45: 943-949. DOI:10.1007/s11033-018-4242-4 |
| [6] |
马鑫瑞, 李亮, 杨梦洁, 等. 梨花芽休眠进程中microRNA实时定量PCR内参基因的筛选[J]. 分子植物育种, 2018(14): 4696-4704. |
| [7] |
赵亚运, 张玉, 等. 大黄鱼microRNA荧光定量PCR中内参基因的选择与评价[J]. 核农学报, 2017, 31(7): 1299-1309. |
| [8] |
Zhang J, Gai MZ, Xue BY, et al. The use of miRNAs as reference genes for miRNA expression normalization during Lilium somatic embryogenesis by real-time reverse transcription PCR analysis[J]. Plant Cell Tissue & Organ Culture, 2016, 129(1): 1-14. |
| [9] |
陈旭, 史菊, 蒋敏, 等. miRNA定量检测中内参基因的选择[J]. 国际检验医学杂志, 2012, 33(11): 1338-1340. DOI:10.3969/j.issn.1673-4130.2012.11.026 |
| [10] |
Yang YT, Zhang X, Chen Y, et al. Selection of reference genes for normaliz-ation of microRNA expression by RT-qPCR in sugarcane buds under cold stress[J]. Front Plant Sci, 2016, 7: 86. |
| [11] |
Peltier HJ, Latham GJ. Normalization of microRNA expression levels in quantitative RT-PCR assays:identification of suitable reference RNA targets in normal and cancerous human solid tissues[J]. RNA, 2008, 14(5): 844-852. DOI:10.1261/rna.939908 |
| [12] |
Wotschofsky Z, Meyer HA, Jung M, et al. Reference genes for the relative quantification of microRNAs in renal cell carcinomas and their metastases[J]. Anal Biochem, 2011, 417(2): 233-241. DOI:10.1016/j.ab.2011.06.009 |
| [13] |
林榕燕, 钟淮钦, 黄敏玲, 等. 杂交兰实时荧光定量PCR内参基因的筛选[J]. 中国细胞生物学学报, 2018, 40(3): 381-389. |
| [14] |
Wang HB, Gong M, et al. Genome-wide identification of Jatropha curcas MAPK, MAPKK, and MAPKKK gene families and their expression profile under cold stress[J]. Sci Rep, 2018, 8(1): 16163. DOI:10.1038/s41598-018-34614-1 |
| [15] |
陈杨玲, 王海波, 陈凯, 等. 能源植物小桐子抗逆性研究进展[J]. 中国农学通报, 2013, 29(10): 1-6. |
| [16] |
Montes JM, Melchinger AE. Domestication and breeding of Jatropha curcas L.[J]. Trends Plant Sci, 2016, 21(12): 1045-1057. DOI:10.1016/j.tplants.2016.08.008 |
| [17] |
Maes WH, Trabucco A, Achten WMJ, et al. Climatic growing conditions of Jatropha curcas L.[J]. Biomass & Bioenergy, 2009, 33(10): 1481-1485. |
| [18] |
Wang SS, Wang HB, Gong M. Identification of microRNAs involved in chilling response by deep sequencing of Jatropha curcas L. small RNAs at the global genome level. Proceedings of the 21st European Biomass Conference and Exhibition[C]. Copenhagen, 2013: 356-363.
|
| [19] |
Li ZG, Yuan LX, Wang QL, et al. Combined action of antioxidant defense system and osmolytes in chilling shock-induced chilling tolerance in Jatropha curcas seedlings[J]. Acta Physiologiae Plantarum, 2013, 35(7): 2127-2136. DOI:10.1007/s11738-013-1249-2 |
| [20] |
Ao PX, Li ZG, et al. Involvement of antioxidant defense system in chill hardening-induced chilling tolerance in Jatropha curcas seedlings[J]. Acta Physiologiae Plantarum, 2013, 35: 153-160. DOI:10.1007/s11738-012-1058-z |
| [21] |
Ao PX, Li ZG, et al. Involvement of compatible solutes in chill har-dening-induced chilling tolerance in Jatropha curcas seedlings[J]. Acta Physiologiae Plantarum, 2013, 35: 3457-3464. DOI:10.1007/s11738-013-1381-z |
| [22] |
吴建阳, 何冰, 杜玉洁, 等. 利用geNorm, NormFinder和BestKeeper软件进行内参基因稳定性分析的方法[J]. 现代农业科技, 2017(5): 278-281. DOI:10.3969/j.issn.1007-5739.2017.05.174 |
| [23] |
Czechowski T, Stitt M, Altmann T, et al. Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis[J]. Plant Physiology, 2005, 139(1): 5-17. DOI:10.1104/pp.105.063743 |
| [24] |
曾长英.木薯等大戟科植物耐寒抗旱相关miRNA的分离与鉴定[D].海口: 海南大学, 2009. http://cdmd.cnki.com.cn/Article/CDMD-10589-2010017710.htm
|
| [25] |
Lu SF, Sun YH, Shi R, et al. Novel and mechanical stress-responsive microRNAs in Populus trichocarpa that are absent from Arabidopsis W[J]. Plant Journal, 2010, 55(1): 131-151. |
| [26] |
Zanca A, Vicentini R, Ortiz F, et al. Identification and expression analysis of microRNAs and targets in the biofuel crop sugarcane[J]. BMC Plant Biol, 2010, 10(1): 260. DOI:10.1186/1471-2229-10-260 |
| [27] |
Kulcheski F, Francismar CM, Alexandre LN, et al. The use of microRNAs as reference genes for quantitative polymerase chain reaction in soybean[J]. Anal Biochem, 2010, 406(2): 185-192. DOI:10.1016/j.ab.2010.07.020 |
| [28] |
Feng H, Huang XL, Zhang Q, et al. Selection of suitable inner reference genes for relative quantification expression of microRNA in wheat[J]. Plant Physiol Biochem, 2012, 51: 116-122. DOI:10.1016/j.plaphy.2011.10.010 |
| [29] |
Lin YL, Lai ZX. Evaluation of suitable reference genes for normali-zation of microRNA expression by real-time reverse transcription PCR analysis during longan somatic embryogenesis[J]. Plant Physiol Biochem, 2013, 66: 20-25. DOI:10.1016/j.plaphy.2013.02.002 |
| [30] |
谢小芳, 添先凤, 江昌俊, 等. 茶树低温胁迫下microRNA实时定量PCR内参基因的筛选[J]. 茶叶科学, 2015, 35(6): 596-604. DOI:10.3969/j.issn.1000-369X.2015.06.014 |
| [31] |
Yang YT, Zhang X, Su YC, et al. miRNA alteration is an important mechanism in sugarcane response to low-temperature environment[J]. BMC Genomics, 2017, 18(1): 833-851. DOI:10.1186/s12864-017-4231-3 |
| [32] |
姜琼, 王幼宁, 王利祥, 等. 盐胁迫下大豆根组织定量PCR分析中内参基因的选择[J]. 植物学报, 2015, 50(6): 754-764. |