中心法则阐述了遗传信息从DNA通过转录为RNA,再经翻译合成蛋白质的基本生物学过程。信使RNA是蛋白质合成的信息蓝图,基于高通量测序的转录组测序技术(RNA-Seq)可以反映细胞中mRNA的种类和数量,并且还可以检测mRNA的序列变异和可变剪接[1-3],因此RNA-Seq被广泛应用于研究基因的表达水平,揭示动植物的生长发育和逆境胁迫应答机理;尤其对基因组序列不太清楚的物种,可根据转录组序列推测蛋白质的序列,对于开发和利用这些物种起到了很大的推动作用。蛋白质是几乎所有生理过程的实际执行者,信使RNA到蛋白质的翻译过程受到转录后、翻译水平调控的影响,翻译后修饰和降解也会影响到蛋白质的丰度。因此,转录组测序只能反映基因是否转录生成RNA,不能反映mRNA是否被翻译成相应的蛋白质以及翻译水平。Maier等[4]对多个物种的转录组和蛋白组进行比较研究后发现,mRNA水平与其编码蛋白质的量的相关系数R2的波动范围在0.01-0.5之间。因此,mRNA水平不能作为判断蛋白质水平的依据。
蛋白质组学的研究方法包括双向电泳、基于抗体的免疫学检测和质谱方法。双向电泳曾是蛋白组研究的主要技术,其利用等电点聚焦和SDS-PAGE相结合的方法将上千种不同的蛋白质依据其等电点(PI)和分子量的大小进行二维分离。该技术操作繁琐,重复性不高,难以检测到低丰度蛋白。此外,该技术对于难溶性蛋白及等电点极酸或极碱的蛋白的分离效果欠佳[5]。而基于抗体的免疫学检测由于抗体只能识别蛋白质的特殊表位,和蛋白质特定序列相结合,因此抗体免疫学检测存在一定的局限性。
随着生物质谱技术的发展和不断成熟,质谱技术是目前蛋白组研究中发展最快、也最具活力和潜力的技术。其基本原理是蛋白质经胰蛋白酶消化后变成多肽片段混合物,在质谱仪中,具有不同质量与电荷比值(即质荷比,M/Z)的肽段分子被分开,经过检测器收集进而确定肽段的M/Z值。通过与蛋白质序列数据库中蛋白质经过胰蛋白酶消化后理论上产生的一级质谱峰图和二级质谱峰图进行比对即可鉴定蛋白质的种类和含量。质谱技术具有灵敏度高、快速、能同时提供蛋白质的鉴定、定量信息等优点[6]。然而,质谱在蛋白质鉴定中也存在局限性,其解析度低,难以检测到低丰度的蛋白;蛋白质本身的物理和化学性质也会影响质谱检测,如疏水性膜蛋白、结构上对酶消化具有抗性的致密蛋白质、易于降解的蛋白质,则很难被检测到[7]。此外,质谱法只能鉴定已知的蛋白质,对氨基酸序列非常相似的基因或同一基因不同剪切体产生的蛋白无法分辨,对新的或未知蛋白更是束手无策。
众所周知,蛋白质的合成需要通过翻译进行,由mRNA到蛋白质的翻译过程包括起始、延伸和终止3个阶段。大量研究表明起始阶段是蛋白质合成的主要限速步骤[8-11]。在起始阶段,核糖体40S小亚基首先识别mRNA 5' 端甲基化帽子,当mRNA与小亚基结合后,携带有甲硫氨酸的tRNA(tRNAMet)通过反密码子与mRNA中的首个AUG起始序列结合并进入核糖体,随后核糖体大亚基(60S)则与起始复合物相结合,形成完整的80S核糖体-mRNA起始复合物,肽链合成得以启动。在肽链合成过程中,其他核糖体还可以陆续结合到该mRNA上形成多聚核糖体(Polyribosomes,polysome)。翻译过程中核糖体-新生肽链复合物(Ribosome nascent-chain complex,RNC)中的mRNA即是能翻译成蛋白的mRNA,而没有形成复合物的mRNA,则无法被翻译成蛋白质。由此可见,翻译水平调控在蛋白质合成过程中的作用明显大于转录水平的调控。
RNA定量和测序技术非常完善,通过高通量测序技术对正处于翻译状态的mRNA(翻译组)进行研究,既可以间接反应蛋白质的翻译水平,又可以检测到基因的可变剪接甚至发现新的蛋白质分子。因此,翻译组是蛋白组的最佳体现者。研究翻译组还有助于更好地阐释从mRNA到蛋白质之间翻译水平调控机制研究。
2 翻译组研究常用技术由于mRNA与核糖体的结合是一种非共价结合,多聚核糖体复合物构象非常脆弱,在细胞破碎过程中极易因为震动或环境变化而发生解离,因此翻译组研究难度较大,发展也相对滞后。多聚核糖体图谱技术(Polysome profiling)是研究翻译组的经典技术,近年来发展起来的翻译谱分析技术(RNC-RNA-Seq)、核糖体图谱技术(Ribosome profiling)、核糖体亲和纯化技术(Ribosome affinity purification,RAP)是研究翻译组的主要技术。
2.1 多聚核糖体图谱技术核糖体是细胞内部密度最大的生物大分子,结合有不同数量核糖体的mRNA其沉降速率也不同。mRNA上结合的核糖体数目越多,在蔗糖密度梯度离心时其沉降速度越快。多聚核糖体图谱技术正是利用了核糖体沉降系数较大的特性,用蔗糖密度梯度离心的方法将游离的RNA、40S核糖体小亚基、60S核糖体大亚基、经过组装的80S单核糖体以及结合有不同数目核糖体的mRNA分散到不同的蔗糖密度梯度溶液中。离心结束后从管底注入高密度的FluorinetTM FC-40,用密度梯度分离仪将不同密度的蔗糖溶液从顶部由低浓度到高浓度收集到不同试管中,同时检测其在254 nm处的吸光值(图 1-A)[12]。
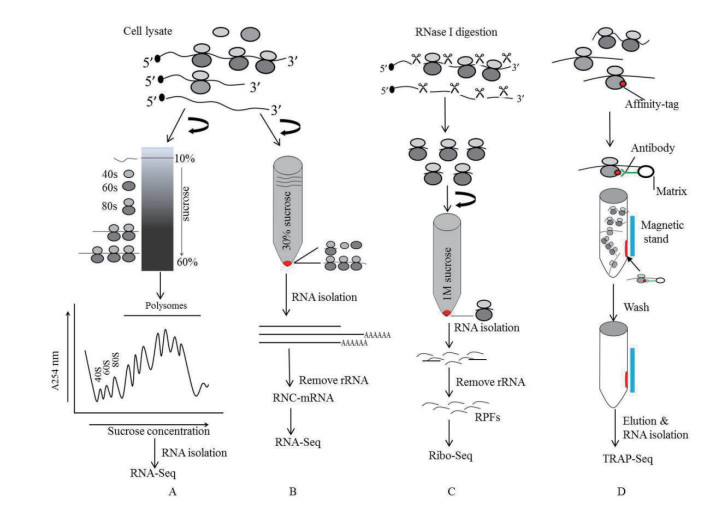
|
| A:依次为多聚核糖体图谱技术;B:翻译谱分析技术;C:核糖体图谱技术;D:核糖体亲和纯化技术 图 1 翻译组研究技术流程 |
通常认为mRNA上结合的核糖体数目越多表示该mRNA的翻译状态越活跃,因此一般将mRNA上结合≥2核糖体的蔗糖溶液合并代表多聚核糖体部分。将蔗糖溶液中的RNA进行分离纯化,通过比较mRNA在多聚核糖体部分(高浓度蔗糖溶液)和非多聚核糖体部分(低浓度蔗糖溶液)的比例,或者mRNA在多聚核糖体部分与总RNA(包括游离的RNA和与核糖体结合的RNA)的比例,则可判断待测基因在细胞内的翻译状况。多聚核糖体技术的优势还在于该方法可以根据结合的核糖体数目不同将各组分分别收集加以研究。当研究少数基因时可以采用Northern印迹或qRT-PCR技术进行检测,若针对整个翻译组水平的检测则可以采用基因芯片(Microarray),或者通过构建翻译组RNA-Seq文库、高通量测序分析[13-15]。
多聚核糖体技术被广泛用来研究动物、植物及酵母细胞的mRNA的翻译状况及在逆境胁迫下的翻译应答。例如,培养液碳源改变时酿酒酵母会迅速调整翻译的基因和翻译水平以适应碳源的改变[16];正常细胞和癌细胞随着其内部或者外在条件不同,其翻译状态有所差异[17-18];高等哺乳动物在细胞组织缺氧[19]、病毒感染[20]等胁迫下均会导致翻译组水平发生变化。紫外线照射会激发细胞的DNA损伤修复应答机制,Powley等[21]应用多聚核糖体技术发现哺乳动物细胞可通过选择性募集编码DNA修复酶的mRNA进行翻译。脊髓性肌萎缩症(SMA)是一种由于SMN蛋白不足引起的神经肌肉疾病,但是其发病机制不详。通过对SMA小鼠进行转录组和翻译组(多聚核糖体技术)的比较研究,Bernabò等[22]发现在疾病早期,一系列RNA的翻译已发生了明显的变化,揭示了SMN蛋白不足导致机体翻译水平的改变是造成SMA的主要原因。
蛋白质翻译是生物体内最消耗能量的过程,应用多聚核糖体技术,Puckette等[23]发现对臭氧敏感和不敏感的植物Medicago品种在翻译水平上对臭氧胁迫应答的差异,并发现不敏感的品种在臭氧胁迫下mRNA上结合的核糖体数目很快减少,有利于植物蓄积能量抵抗臭氧导致的氧化胁迫。拟南芥在缺氧条件下蛋白质翻译受到抑制,与之相应的是mRNA上结合的核糖体的数目明显减少,而核糖体单体数目明显增加[24]。我们对水稻耐盐品种Pokkali和不耐盐品种IR29幼苗在正常生长条件和盐胁迫下转录组和翻译组进行了比较研究[25]。发现Pokkali光合作用相关基因被盐胁迫抑制的程度明显低于IR29,抗氧化胁迫和保持细胞膜完整性能力明显优于IR29;两个品种中均有大量基因在转录水平受盐胁迫调控,但在翻译水平上无显著应答,反之,也有相当数目基因在翻译水平受盐胁迫调控,但在转录水平上无显著改变。这些数据充分说明了基因转录水平的变化并不一定会导致其编码蛋白的量发生相应的变化,从而显示了翻译组研究的重要性。
尽管多聚核糖体图谱技术是研究翻译组的经典技术,但该技术需要专用且昂贵的仪器设备(如超速冷冻离心机和密度梯度分离系统),且操作过程繁琐,不能同时处理多个样本,因此限制了该方法的广泛使用[15]。由于蔗糖密度梯度离心的体积比较大,细胞裂解液被稀释,溶液中高浓度的蔗糖对RNC-mRNA的纯化也不利,导致RNC-mRNA的回收效率低;由于该技术基于复合体的沉降系数,多聚核糖体溶液部分可能会含有其他高分子量复合物,如脂筏,假多聚核糖体等[26]。此外,该技术需要样本相对较大也限制了该技术的应用。值得我们注意的是先前许多学者认为mRNA翻译的活跃程度与其上结合的核糖体数目呈正相关性,然而近年来的研究表明这一结论并不完全成立。当蛋白质翻译到mRNA的某一位置停止或暂停时,也可导致该位置上游核糖体密集[27]。而有些mRNA上只结合有单个核糖体,但其翻译异常活跃,如在HEK293细胞和处于富营养培养基上的对数生长期的大肠杆菌中,核糖体单体占主导地位;将这些核糖体单体分离出来再放置于无细胞翻译体系中,蛋白质翻译可以继续进行[28-30]。表明mRNA的翻译状况与结合上去的核糖体数量的线性关系并不总是成立。
2.2 翻译谱分析技术mRNA翻译的活跃程度与其上结合的核糖体数目并不总呈正相关性,因此只要mRNA与核糖体结合,即表明其处于翻译状态。基于该理论2013年张弓实验室[3]开发了翻译谱分析技术(RNC-RNA-Seq)。与多聚核糖体图谱技术不同的是该技术采用单一浓度的蔗糖溶液。将细胞裂解液转移到浓度为30%的蔗糖溶液上,用超速冷冻离心机在4℃下以185 000×g超速离心5 h,核糖体组分将会与游离的mRNA分离,核糖体及其结合的mRNA沉淀于离心管底部,随后对其抽提即可得到相应的RNC-mRNA组分(图 1-B)。进一步采用高通量测序技术或基因芯片技术(Microarray)进行分析,可以得到与核糖体结合的mRNA信息。Zhong等[31]研究正常支气管上皮细胞和直肠腺癌Caco-2细胞的翻译组时发现了1 397个RefSeq数据库中被标注为非编码RNA的基因,这些非编码RNA很可能被翻译成小分子蛋白或肽段。
与多聚核糖体图谱技术相比,RNC-RNA-Seq技术比较简便,RNA回收容易,得率高。但该技术离心后的沉淀物中不仅包含单个核糖体,多聚核糖体,还包括核糖体成分(如40S、60S核糖体亚基),以及其他高分子量复合物,若要研究结合不同数量核糖体的mRNA时该方法无法实施。
2.3 核糖体图谱技术20世纪60年代人们就注意到蛋白质合成过程中核糖体保护的mRNA片段可以免受RNA酶的降解作用[32-34]。2009年Weissman课题组[35]首次将高通量测序技术应用于研究酿酒酵母核糖体保护的RNA片段(Ribosome protected fragments,RPFs),即核糖体图谱技术(Ribosomal profiling),该技术的原理是采用RNase Ⅰ对细胞裂解液进行处理,游离的、暴露的RNA片段被降解,而被核糖体保护的RNA片段则不受RNase Ⅰ的影响。酶解后的核糖体单体及其保护的RNA片段既可通过多聚核糖体图谱技术相似的方法获得,即将处理液通过10%-50%的蔗糖梯度进行冷冻超速离心,梯度分离时仅仅收集单核糖体,也可采用1 mol/L的蔗糖。
缓冲溶液作为垫层冷冻超速离心,核糖体及其保护的22-30 nt的RPFs片段位于沉淀部分。提取RNA片段,再去除其中的rRNA,即得到核糖体保护的处于翻译状态的mRNA片段(图 1-C),将其构建成cDNA文库,再经高通量测序和生物信息学分析,由此得到每个转录本上核糖体的精准分布,并用于评估该转录本翻译的活跃程度,该技术也被称为Ribo-Seq[36]。由于核糖体图谱技术的定位准确度高,能准确地反映出究竟是哪一个阅读框被翻译了,因此,可以通过该技术研究程序性框移和终止密码子通读等现象[37]。该技术自发明以来已经在酵母、动物以及植物中被广泛应用。由于核糖体图谱技术检测的是蛋白质的编码序列,应用该技术不仅揭示了许多先前没有发现的开放阅读框(ORF),甚至发现一些非编码长链RNA也可被翻译,还发现了mRNA 5' 端非编码区的异常翻译情况(uORFs)以及许多潜在的非AUG起始的编码序列[35, 37]。真核生物的基因转录有明显的昼夜节律特性,Janich等[38]利用该技术研究小鼠肝脏细胞不同时段的翻译组,发现150个基因的翻译呈现明显的生物钟节律,主要生物钟相关基因的翻译水平的变化比转录水平能更好阐释生物钟的特征。
虽然核糖体图谱技术具有诸多优点,但该技术操作相对比较复杂,对样本的需求量大,测序偏好性强,成本较高,而且影响RPF的因素众多,如RNase Ⅰ可降解单链RNA片段,而双链RNA则不受影响;此外,RNA结合蛋白也可保护其结合区域的RNA不受RNase Ⅰ的降解而存留下来,但这些片段并不是处于被翻译的状态[39];该技术检测的是蛋白质的编码区域,因此无法获得对翻译起调控作用的UTR的信息。
2.4 核糖体亲和纯化技术从生物体内分离特定组织和细胞时极易受到周围组织的污染,因此组织或细胞水平的翻译组研究报道尚非常有限,近年来发展起来的核糖体亲和纯化技术(Ribosome affinity purification RAP,或Translating RAP,TRAP)使这一难题得到有效解决。TRAP是由Gerber课题组于2002年发表[27],最初是在核糖体大亚基的L25(RPL25p)蛋白质的C端连接上亲和标签(如His、FLAG和eGFP等),后来又采用RPL16a进行C端标记,这些带有标签的核糖体蛋白基因可以被组织特异性启动子启动;通过再转化获得稳定的转化细胞系、动植物个体,再通过抗体将含有标签的核糖体进行分离,进一步得到标签核糖体上结合的mRNA(图 1-D),采用microarray或RNA-Seq技术进一步对其进行测序分析[40],该方法分离的核糖体既可以来自单个核糖体又可以来自多聚核糖体。
该技术最初在单细胞生物酵母中应用,很快被应用于动物和植物特定组织或细胞翻译组研究[41]。在植物中,TRAP最初是由Bailey-Serres实验室[42]在拟南芥中建立。他们用与发育相关的启动子启动FLAG-RPL18的表达,采用microarray技术对拟南芥幼苗中21种不同类型细胞在正常和缺氧条件下的翻译组进行分析,发现了细胞、器官、植物体不同部位在翻译水平上对缺氧胁迫应答的特异性基因[43];该课题组采用深度测序方式对拟南芥花原基和花的不同部位的转录组和翻译组进行比较研究,发现在内含子剪接以及翻译过程中广泛存在的转录后调控,同时发现了与多聚核糖体结合的一类新的非编码RNA,丰富了植物蛋白翻译过程的调控机理。
油菜素内酯(BR)既可促进又可延缓植物根部干细胞的分裂,为探究其内在机理,Vragovic等[44]利用TRAP技术研究拟南芥根部不同区域翻译组,发现BR在根顶端分生区和基部分生区的作用相反。BR诱导的基因主要在基部分生区的表皮细胞中表达,而受BR抑制的基因主要在根顶端分生区的中柱中表达。在动物中,TRAP首先在小鼠中建立[45-46]。Heintz实验室[45]使用bacTRAP转基因小鼠在不同的神经元细胞中驱动RPL10- eGFP的表达,随后用GFP抗体进行亲和纯化eGFP标记的核糖体,用microarray技术对eGFP标记核糖体上的mRNA进行分析。Heiman等[47]对24种中枢神经系统细胞的翻译组进行分析,发现了上千种细胞特异性表达的mRNA,而这些mRNA在采用组织块进行研究时很难被检测到,充分显示了TRAP在研究不同细胞类型中基因表达方面的优势。核糖体亲和纯化技术主要用于对特定组织中的翻译组进行研究,但是其应用的前提是必须建立能稳定表达含有标签核糖体蛋白的转基因细胞株或转基因动植物,因此该技术当前主要是在模式生物中应用[48]。
以上介绍了多聚核糖体图谱技术,翻译谱分析技术、核糖体图谱技术、核糖体亲和纯化技术的特点,每种方法均有各自的优势和缺点,4种方法的比较见表 1。近年来翻译组学发展较快,技术也在不断完善,但4种技术面临的共同问题是无法区分翻译活跃的核糖体和处于停滞状态的核糖体。如果在翻译的过程中核糖体暂停或停滞在mRNA某个位置时,该位置上游核糖体聚集,RPFs的丰度将会非常高,但该区域翻译活跃程度并不高。为克服这些缺点,Clamer等[49]利用嘌呤霉素——一种氨基核苷类抗生素,能够结合核糖体和新生肽链的特点,在核糖体图谱技术基础上研发出RiboLace技术。该技术特点是首先合成一种嘌呤霉素的类似物3P,该分子既具有嘌呤霉素能结合处于翻译活性状态核糖体的特点,分子表面又有生物素基团,因此可采用pull-down技术将真正处于翻译状态的核糖体与处于翻译停滞状态的核糖体进行有效分离。该技术还具有操作简便,需要材料少的优势。尽管多聚核糖体技术主要用于研究生物个体或组织中的翻译组学,Seimetz等[50]成功将该技术用于研究哺乳动物特定细胞类型中的翻译组学。
蛋白质是细胞功能的主要承担者,当前蛋白质组的主流检测方法可以对蛋白质进行鉴定和定量,但其精度和深度远不及翻译组测序,而翻译组研究不受蛋白质理化性质的影响,可作为基因翻译的证据,其覆盖率也强。更重要的是,翻译组学丰富了蛋白质的种类。越来越多的研究发现在RNC-mRNA中存在一些非编码RNA,其中一些非编码基因翻译出来的小分子蛋白质或肽段可能具有重要的调控功能。因此,翻译组学研究越来越引起广大学者的关注及应用。翻译组学也将加快蛋白组学的研究,并对蛋白组学研究进行重要的补充和完善。
翻译调控作为重要的调控机制,使得生物在面对生物胁迫或非生物胁迫时做出快速灵敏的反应以调节机体生理状态,提高自身的存活率。这一点恰恰体现了翻译调控对生物本身的重要性。从基因组到转录组的调控机理研究得较为清楚,而从转录组到蛋白组的调控机理研究还非常薄弱,而翻译组恰恰是连接转录组和蛋白组的桥梁。当前的翻译组学的研究还处于起始阶段,随着研究的深入,翻译组学将会揭示翻译过程中的奥秘,所以翻译组学研究将会丰富和完善分子生物学理论,对生物学发展具有重要的意义。
| [1] |
Nagaraj N, Wisniewski JR, Geiger T, et al. Deep proteome and transcriptome mapping of a human cancer cell line[J]. Molecular Systems Biology, 2011, 7(11): 548-554. |
| [2] |
Chen G, Gharib TG, Huang CC, et al. Discordant protein and mRNA expression in lung adenocarcinomas[J]. Molecular & Cellular Proteomics, 2002, 1(4): 304-313. |
| [3] |
Wang T, Cui Y, Jin J, et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific[J]. Nucleic Acids Research, 2013, 41(9): 4743-4754. DOI:10.1093/nar/gkt178 |
| [4] |
Maier T, Güell M, Serrano L. Correlation of mRNA and protein in complex biological samples[J]. Febs Letters, 2009, 583(24): 3966-3973. DOI:10.1016/j.febslet.2009.10.036 |
| [5] |
Ranjbar L, Foley JP, Breadmore MC. Multidimensional liquid-phase separations combining both chromatography and electrophoresis - A review[J]. Analytica Chimica Acta, 2017, 950: 7-31. DOI:10.1016/j.aca.2016.10.025 |
| [6] |
Cox J, Mann M. MaxQuant enables high peptide identification rates, individualized p. p. b. -range mass accuracies and proteome-wide protein quantification[J]. Nature Biotechnology, 2008, 26(12): 1367-1372. DOI:10.1038/nbt.1511 |
| [7] |
Cox J, Neuhauser N, Michalski A, et al. Andromeda:a peptide search engine integrated into the MaxQuant environment[J]. J Proteome Res, 2011, 10(4): 1794-1805. DOI:10.1021/pr101065j |
| [8] |
Jackson RJ, Hellen CU, Pestova TV. The mechanism of eukaryotic translation initiation and principles of its regulation[J]. Nature Reviews Molecular Cell Biology, 2010, 11(2): 113-127. DOI:10.1038/nrm2838 |
| [9] |
Ali MU, Ur Rahman MS, Jia Z, et al. Eukaryotic translation initiation factors and cancer[J]. Tumor Biology, 2017, 39(6): 101042831770980. DOI:10.1177/1010428317709805 |
| [10] |
Shirokikh NE, Archer SK, Beilharz TH, et al. Translation complex profile sequencing to study the in vivo dynamics of mRNA-ribosome interactions during translation initiation, elongation and termination[J]. Nature Protocols, 2017, 12(4): 697-731. DOI:10.1038/nprot.2016.189 |
| [11] |
Mengardi C, Limousin T, Ricci EP, et al. microRNAs stimulate translation initiation mediated by HCV-like IRESes[J]. Nucleic Acids Research, 2017, 45(8): 4810-4824. |
| [12] |
Li YF, Mahalingam R, Sunkar R. Isolation of polysomal RNA for analyzing stress-responsive genes regulated at the translational level in plants[J]. Methods Mol Biol, 2017, 1631(10): 151-161. |
| [13] |
Mašek T, Valášek L, Pospíšek M. Polysome analysis and RNA purification from sucrose gradients[J]. Methods Mol Biol, 2011, 703(10): 293-309. |
| [14] |
Spangenberg L, Shigunov P, Abud AP, et al. Polysome profiling shows extensive posttranscriptional regulation during human adipocyte stem cell differentiation into adipocytes[J]. Stem Cell Research, 2013, 11(2): 902-912. DOI:10.1016/j.scr.2013.06.002 |
| [15] |
Chassé H, Boulben S, Costache V, et al. Analysis of translation using polysome profiling[J]. Nucleic Acids Research, 2017, 45(3): e15. |
| [16] |
Kuhn KM, Derisi JL, Brown PO, et al. Global and specific translational regulation in the genomic response of Saccharomyces cerevisiae to a rapid transfer from a fermentable to a nonfermentable carbon source[J]. Molecular & Cellular Biology, 2001, 21(3): 916-927. |
| [17] |
Arava Y, Wang Y, Storey JD, et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae[J]. Proc Natl Acad Sci USA, 2003, 100(7): 3889-3894. DOI:10.1073/pnas.0635171100 |
| [18] |
Flora P, Hélène M, Pascal L, et al. Bacterial translational regulations:high diversity between all mRNAs and major role in gene expression[J]. Genomics, 2012, 13(1): 528-534. |
| [19] |
Thomas JD, Johannes GJ. Identification of mRNAs that continue to associate with polysomes during hypoxia[J]. RNA, 2007, 13(7): 1116-1131. DOI:10.1261/rna.534807 |
| [20] |
Johannes G, Carter MS, Eisen MB, et al. Identification of eukaryotic mRNAs that are translated at reduced cap binding complex eIF4F concentrations using a cDNA microarray[J]. Proc Natl Acad Sci USA, 1999, 96(23): 13118-13123. DOI:10.1073/pnas.96.23.13118 |
| [21] |
Powley IR, Kondrashov A, Young LA, et al. Translational reprogramming following UVB irradiation is mediated by DNA-PKcs and allows selective recruitment to the polysomes of mRNAs encoding DNA repair enzymes[J]. Genes & Development, 2009, 23(10): 1207-1220. |
| [22] |
Bernabò P, Tebaldi T, Groen E, et al. In Vivo translatome profiling in spinal muscular atrophy reveals a role for SMN protein in ribosome biology[J]. Cell Reports, 2017, 21(4): 953-965. DOI:10.1016/j.celrep.2017.10.010 |
| [23] |
Puckette M, Lyer NJ, Tang YJ, et al. Differential mRNA translation in medicago truncatula accessions with contrasting responses to ozone-induced oxidative stress[J]. Molecular Plant, 2012, 5(1): 187-204. DOI:10.1093/mp/ssr069 |
| [24] |
Brancoprice C, Kawaguchi R, Ferreira RB, et al. Genome-wide analysis of transcript abundance and translation in Arabidopsis seedlings subjected to oxygen deprivation[J]. Annals of Botany, 2005, 96(4): 647-660. DOI:10.1093/aob/mci217 |
| [25] |
Li Y, Zheng Y, Sunkar R, et al. Comparative transcriptome and translatome analysis in rice revealed differential mRNA translation in Pokkali compared to IR29 under salt stress[J]. BMC Genomics, 2018, 19(10): 935. |
| [26] |
Thermann R, Hentze MW. Drosophila miR2 induces pseudo-polysomes and inhibits translation initiation[J]. Nature, 2012, 447(7146): 875-882. |
| [27] |
赵晶, 张弓. 翻译组学:方法及应用[J]. 生命的化学, 2017, 37(1): 70-79. |
| [28] |
Zhong J, Xiao C, Gu W, et al. Transfer RNAs mediate the rapid adaptation of Escherichia coli to oxidative stress[J]. PLOS Genetics, 2015, 11(6): 10-19. |
| [29] |
Morello LG, Hesling C, Coltri PP, et al. The NIP7 protein is required for accurate pre-rRNA processing in human cells[J]. Nucleic Acids Research, 2011, 39(2): 648-665. DOI:10.1093/nar/gkq758 |
| [30] |
Zhang G, Hubalewska M, Ignatova Z. Transient ribosomal attenuation coordinates protein synthesis and co-translational folding[J]. Nat Struct Mol Biol, 2009, 16(3): 274-280. DOI:10.1038/nsmb.1554 |
| [31] |
Zhong J, Cui Y, Guo J, et al. Resolving chromosome-centric human proteome with translating mRNA analysis:a strategic demonstration[J]. J Proteome Res, 2014, 13(1): 50-59. DOI:10.1021/pr4007409 |
| [32] |
Lazarowitz SG, Robertson HD. Initiator regions from the small size class of reovirus messenger RNA protected by rabbit reticulocyte ribosomes[J]. J Biol Chem, 1977, 252(21): 7842-7849. |
| [33] |
Kozak M. Nucleotide sequences of 5' -terminal ribosome-protected initiation regions from two reovirus messages[J]. Nature, 1977, 269(5627): 391-394. |
| [34] |
Steitz JA. Polypeptide chain initiation:nucleotide sequences of the three ribosomal binding sites in bacteriophage R17 RNA[J]. Nature, 1969, 224(5223): 957-964. DOI:10.1038/224957a0 |
| [35] |
Ingolia NT, Ghaemmaghami S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling[J]. Science, 2009, 324(5924): 218-223. DOI:10.1126/science.1168978 |
| [36] |
Ingolia NT, Brar GA, Rouskin S, et al. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments[J]. Nature Protocols, 2012, 7(8): 1534-1550. DOI:10.1038/nprot.2012.086 |
| [37] |
Souza ND. Deep sequencing of ribosome footprints[J]. Nature Methods, 2009, 6(4): 244-244. |
| [38] |
Janich P, Arpat AB, Castelo-Szekely V, et al. Ribosome profiling reveals the rhythmic liver translatome and circadian clock regulation by upstream open reading frames[J]. Genome Research, 2015, 25(12): 1848-1859. DOI:10.1101/gr.195404.115 |
| [39] |
Terenin IM, Akulich KA, Andreev DE, et al. Sliding of a 43S ribosomal complex from the recognized AUG codon triggered by a delay in eIF2-bound GTP hydrolysis[J]. Nucleic Acids Research, 2016, 44(4): 1882-1893. DOI:10.1093/nar/gkv1514 |
| [40] |
Inada T, Winstall E, Jr TS, et al. One-step affinity purification of the yeast ribosome and its associated proteins and mRNAs[J]. RNA, 2002, 8(7): 948-958. DOI:10.1017/S1355838202026018 |
| [41] |
Zywicki M, Bakowska-Zywicka K, Polacek N. Revealing stable processing products from ribosome-associated small RNAs by deep-sequencing data analysis[J]. Nucleic Acids Research, 2012, 40(9): 4013-4024. DOI:10.1093/nar/gks020 |
| [42] |
Zanetti ME, Chang IF, Gong F, et al. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression[J]. Plant Physiology, 2005, 138(2): 624-635. DOI:10.1104/pp.105.059477 |
| [43] |
Mustroph A, Zanetti M E, Jang C J, et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis[J]. Plant Signaling & Behavior, 2009, 106(44): 18843-18848. |
| [44] |
Vragovic K, Sela A, Friedlander-Shani L, et al. Translatome analyses capture of opposing tissue-specific brassinosteroid signals orchestrating root meristem differentiation[J]. Proceedings of the National Academy of Sciences, 2015, 112(3): 923-928. DOI:10.1073/pnas.1417947112 |
| [45] |
Heiman M, Schaefer A, Gong S, et al. A translational profiling approach for the molecular characterization of CNS cell types[J]. Cell, 2008, 135(4): 738-748. DOI:10.1016/j.cell.2008.10.028 |
| [46] |
Sanz E, Yang L, Su T, et al. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues[J]. Proc Natl Acad Sci USA, 2009, 106(33): 13939-13944. DOI:10.1073/pnas.0907143106 |
| [47] |
Heiman M, Kulicke R, Fenster RJ, et al. Cell type-specific mRNA purification by translating ribosome affinity purification(TRAP)[J]. Nature Protocols, 2014, 9(6): 1282-1291. DOI:10.1038/nprot.2014.085 |
| [48] |
Wang Y, Jiao Y. Translating ribosome affinity purification(TRAP)for cell-specific translation profiling in developing flowers[J]. Methods in Molecular Biology, 2014, 1110(10): 323-328. |
| [49] |
Clamer M, Tebaldi T, Lauria F, et al. Active ribosome profiling with ribolace[J]. Cell Rep, 2018, 25(4): 1097-1108. DOI:10.1016/j.celrep.2018.09.084 |
| [50] |
Seimetz J, Arif W, Bangru S, et al. Cell-type specific polysome profiling from mammalian tissues[J]. Methods, 2018, 155: 131-139. |





