节节麦(Aegilops tauschii Coss.,DD,2n=14),又名粗山羊草,禾本科(Poaceae)、小麦族(Triticeae)、山羊草属(Aegilops L.),是普通小麦D基因组的供体种。节节麦具有较强的适应能力,在沙滩、沙漠边缘、石山、大草原、荒地、路边和潮湿的温带森林均能生长,并有进一步扩大其分布区域的趋势。节节麦在抗病性、抗虫性、抗逆性等方面具有丰富的遗传多样性,被认为是改良普通小麦的潜在基因资源[1-4]。本文对节节麦的遗传多样性及在改良普通小麦中的应用进行综述,以期为节节麦资源的收集和利用提供参考。
1 节节麦的分类及遗传多样性的研究进展从形态学上,节节麦可以分为2个亚种,分别是Ae. tauschii ssp. tauschii和Ae. tauschii ssp. strangulata[5-7]。前者又进一步分为Ae. tauschii ssp. tauschi var. typica、Ae. tauschii ssp. tauschi var. anathera和Ae. tauschii ssp. tauschi var. meyeri 3个变种。2个亚种之间主要的区别是穗部性状的不同,亚种Ae. tauschii ssp. tauschii的穗型细长,小穗呈圆柱形,整个穗型较为一致。亚种Ae. tauschii ssp. strangulata穗型粗短,穗轴扭曲,且比相邻小穗细很多,颖片的宽度通常超过其长度,整个小穗呈念珠状,籽粒圆形。同时还发现了部分具有轻微念珠形小穗特征的节节麦,归属于Ae. tauschii ssp. tauschii亚种。
除了依据形态学特征对节节麦进行分类外,利用同工酶、SSR标记、AFLP标记和基因芯片等技术对节节麦的遗传背景也进行了分析。张学勇等[8]利用醇溶蛋白电泳技术检测了多个地区的节节麦,发现中东地区的节节麦多态性最高;新疆地区次之;黄河流域最低。Dudnikov[9]对24份节节麦中的27个同工酶标记位点进行多样性分析,结果表明,Ae. tauschii ssp. strangulata亚种和Ae. tauschii ssp. tauschii亚种种内的遗传分化指数(Gst)分别为0.64和0.67,显示出较高的遗传多样性。此外,节节麦含有的高分子量谷蛋白亚基(HMW-GS)组成也具有较丰富的遗传变异类型,王亚娟等[10]对79份节节麦的HMW-GS组成进行分析,发现节节麦的HMW-GS的Glu-1Ds类型丰富,共检测到12种亚基类型和13种亚基组成。苏亚蕊等[11]对161份节节麦HMW-GS组成的遗传变异类型进行分析,并从中鉴定到了3个新发现的y-型亚基。
随着分子生物学的飞速发展,利用限制性片段长度多态性(Restriction fragment length polymorphism,RFLP)、随机扩增多态性DNA(Random Amplified Polymorphic DNA,RAPD)、简单重复序列(Simple sequence repeat,SSR)、DArT标记(Diversity arrays technology,DArT)和单核苷酸多态性(Single nucleotide polymorphism,SNP)等分子标记技术对节节麦的遗传多样性进行了更加深入地研究。Lubbers等[12]对来源于不同地区的102份节节麦的25个酶切位点进行RFLP分析,结果显示,来自里海附近的节节麦遗传变异最大;阿富汗的次之;土耳其和巴基斯坦的最小,并由此推断节节麦可能起源于里海南岸或西南岸。孔令让等[13]利用RAPD技术对29份节节麦进行了基因组DNA多态性分析,发现Ae. tauschii ssp. tauschii的基因组DNA多态性比Ae. tauschii ssp. strangulata丰富,且2个亚种之间的遗传分化大于地理分化。李玉阁等[14]利用9个ISSR标记研究了75份中国节节麦的遗传多样性,得到138个多态性位点,认为中国的节节麦具有较高的遗传变异,并筛选到5份具有独特遗传变异的黄河流域节节麦。Pestsova等[15]利用18个多态性高的SSR标记,对113个节节麦进行遗传多样性分析,发现高加索地区的节节麦多样性更丰富,中亚地区的节节麦则相反,同时,根据遗传相似系数可以将这113个节节麦分为2类,并且与其地理分布一致。苏亚蕊等[16]选用均匀分布在节节麦14条染色体上的32对SSR引物,对不同来源的147份节节麦进行遗传多样性分析,结果显示共检测到108个等位变异,平均等位基因数为4.15,且中东地区的节节麦遗传多样性高于中国黄河流域的节节麦。
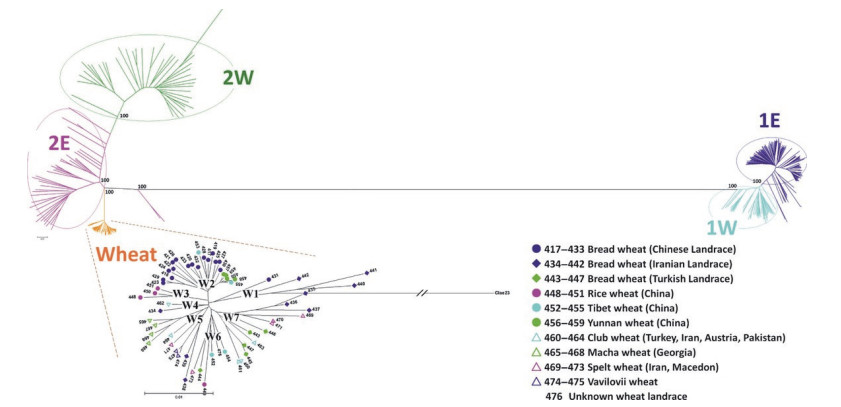
然而,基于节节麦遗传背景的系统分类与其形态学特征的分类并不完全一致,Matsuoka等[17]利用节节麦叶绿体DNA的SSR位点研究其种内变异,鉴定出4个单倍型群,分别是HG7、HG9、HG16和HG17,其中HG16单倍型群属于Ae. tauschii ssp. tauschii,HG9单倍型群属于Ae. tauschii ssp. strangulata,HG17单倍型群属于单独的“t-91s”类型,HG7单倍型群则同时含有2个亚种的节节麦。Mizuno等[18]利用AFLP标记对122份节节麦的遗传多样性进行分析,系统聚类的结果显示节节麦分为两大类群,分别命名为L1系(主要为Ae. tauschii ssp. tauschii)和L2系(主要为Ae. tauschii ssp. strangulata),差别在于经叶绿体基因组鉴定属于HG17单倍型的节节麦位于L1和L2之间。随后,Matsuoka等[19]利用169个DArT标记对206份节节麦的遗传多样性进行分析,将节节麦化分为3类,分别命名为TauL1、TauL2和TauL3,其中TauL1广泛存在于节节麦整个种的分布区域,而TauL2和TauL3则局限分布在外高加索和格鲁吉亚地区,同时认为TauL3与TauL2具有更近的亲缘关系,可作为一个类群。同时,Wang等[20]首次利用DNA芯片技术对节节麦的遗传进化及分类做了更为全面系统地分析,利用含有7 185个SNP标记的10K基因芯片,对402份节节麦进行遗传关系分析,进一步将节节麦L1系细分为L1W和L1E,L2系细分为L2W和L2E系,其中L1E和L2W之间遗传距离最远为0.84(图 1)。此外,还发现L2系包括了部分的Ae. tauschii ssp. tauschi var. typica和Ae. tauschii ssp. tauschi var. meyeri变种,并且来自黄河流域的节节麦在L1系中是一个较为独特的类型。
2 节节麦的分布及在中国的传播途径节节麦适应性较强,分布较广,横跨欧亚大陆,从叙利亚北部、土耳其到中国均有分布。已确认伊朗是节节麦的遗传多样性中心和起源中心,中东地区的土耳其、叙利亚、伊拉克、伊朗等国,亚洲中部、南部的土库曼斯坦、哈萨克斯坦、吉尔吉斯斯坦、阿富汗、巴基斯坦、克什米尔、中国的新疆和黄河流域等国家和地区,以及阿塞拜疆、格鲁吉亚、亚美尼亚等外高加索地区均有分布[21]。亚种Ae. tauschii ssp. tauschii的var. typica和var. anathera变种在整个分布区域内广泛存在,var. meyer变种只分布于伊朗境内里海的东南岸,亚种Ae. tauschii ssp. strangulala主要存在于伊朗里海西南岸和外高加索(Transcaucasia)(包括阿塞拜疆、亚美尼亚等地区)2个独立区域[21-23]。Matsuoka等[7]利用9个穗部性状指标对203个节节麦的穗型进行了分析,认为节节麦的穗型多样性是在其自西向东的扩展过程中形成的,明显受到经纬度的影响,东部和南部分布的节节麦穗型较小,且进化出较小的穗型可能是为了更好地适应当地环境。
在中国,节节麦主要分布在西部的新疆伊犁地区和中部的黄河流域(陕西、河南)。新疆节节麦最早于1976年在新疆伊犁河谷尼勒克县的蒿属荒漠中发现,之后又相继在霍城大西沟草原、巩留特克斯河渡口荒漠草原、阿吾拉勒山扎格台沟、喀什河谷等地发现,海拔高度也从最早发现的海拔600 m逐渐上升到海拔1 600 m[24]。新疆地区的节节麦有其稳定的分布区域,在荒漠草原、草原、草甸草原均有分布,并常与当地的残遗的野果林伴生,且有稳定的野生群落[24-25]。关于黄河流域节节麦的起源,一直以来是一个颇有争议的问题,黄河流域节节麦的起源可以印证古代中国与中亚通过丝绸之路进行的农业交流。节节麦在中部黄河流域最早的报道是1955年叶德娴在河南省新乡市发现节节麦,之后又在陆续在黄河流域的西安、兴平、武功、三门峡、洛阳、卢氏、偃师、辉县、温县及山西南部黄河北岸沿线等地发现[24]。黄河流域的节节麦是伴生于麦田的杂草型植物,不存在自然群落,仅在麦田中与小麦伴生或在麦田附近的地头、渠沟边缘生长。黄河流域节节麦与世界野生节节麦种群的分布中心有较大跨度的地理隔离而呈不连续分布,由于在黄河流域与新疆地区之间的区域没有发现节节麦,因而通过种子的天然传播从中亚而到达中国腹地的可能性显然不存在。而节节麦是通过什么传播途径到达中国的腹地,目前有两种观点:一是新疆地区是节节麦种群自然分布的最东缘,黄河流域的节节麦是由新疆节节麦传入的,颜济等[24]认为新疆伊犁地区的节节麦其生长区域的地形、植被分布、植被物种等特征都与中亚分布的节节麦类似。因此,认为该地区节节麦是中亚节节麦分布区的自然延伸,黄河流域的节节麦可能是由新疆地区节节麦向东传播的结果。杨武云[26]对黄河流域、新疆、伊朗、中东某地的节节麦进行了细胞学分析,结果表明,新疆和伊朗、中东某地的节节麦在染色体组水平并无明显差异,仅有细微分化。黄河流域的节节麦与新疆节节麦在染色体组水平和细微结构上均极为相似,并据此认为,黄河流域的节节麦是由新疆地区传播而来。张学勇等[8]利用醇溶蛋白电泳技术,检测了中东、苏联、新疆、黄河流域等地的节节麦材料,根据多态性的丰富程度,推测出节节麦是按照中东→苏联→中国新疆→黄河流域这样的路线传入中国的。
二是认为黄河流域的节节麦不经新疆地区,而由中东地区经古代丝绸之路直接传入的。魏会廷等[27]利用34对SSR引物分析来自伊朗、新疆和黄河流域31份节节麦的亲缘关系,结果表明,来自河南、陕西的节节麦品种与来自伊朗的节节麦聚为一类,并显示陕西节节麦和河南节节麦之间已产生一定程度的遗传分化,且陕西的节节麦与中东地区的节节麦遗传关系更近。因而,认为黄河流域的节节麦与新疆流域的节节麦无直接关系,很可能是古代经由丝绸之路从中东地区直接传入古都西安(长安),并伴随小麦的种植逐渐扩散到黄河流域的其他地区。苏亚蕊等[16]收集来自河南、陕西、新疆、中东4个区域,共147份节节麦(河南85份、陕西20份、新疆伊犁18份、中东地区24份),利用SSR标记分析其遗传关系,结果表明,黄河流域的节节麦居群、新疆伊犁的节节麦居群、中东地区节节麦居群各自分开,具有较高的遗传变异,且陕西节节麦居群与中东节节麦居群的分化程度小于陕西居群与新疆居群的分化程度,同时小于河南节节麦居群与中东节节麦居群的分化程度,即陕西节节麦与中东节节麦亲缘关系较近,且比河南节节麦与中东节节麦的亲缘关系更近。同样,张大乐等[28]通过对上述地区的214份节节麦材料的籽粒粒形和粒重进行聚类分析,结果显示,新疆的节节麦单独聚为一类,中东地区、陕西和河南的节节麦聚为一类。另外,Mizuno等[18]利用AFLP分子标记,对节节麦基因型进行分类,结果表明,黄河流域的节节麦与阿富汗、新疆地区的L1E系有明显的不同,更靠近于中东地区的L1W系。这些研究揭示了黄河流域节节麦与中东地区节节麦亲缘关系更近,且与新疆地区节节麦的亲缘关系较远,佐证了黄河流域节节麦是经由古丝绸之路直接传入中国的观点。
3 节节麦在普通小麦改良中的利用普通小麦(Triticum aestivum L.,AABBDD,2n=6x=42)是重要的粮食作物,在世界范围内超过40%的人口以小麦为主食。利用骨干亲本作为核心种质进行品种间杂交选育,已培育出很多优秀的小麦品种,如郑9023、矮抗58、郑366等。然而这种育种方式不可避免地导致小麦的遗传基础越来越窄,遗传变异度越来越低,使小麦出现了产量徘徊不前、品质不稳定、对外界适应力逐渐下降等情况。有研究表明,20世纪90年代以来推广的小麦品种的遗传距离仅为0.69左右,由于缺乏新的育种亲本,导致育成品种的多样性指数和遗传距离显著下降[29]。此外,全球性气候异常,导致白粉病、赤霉病、蚜虫、穗发芽等小麦灾害更加频发,这对小麦品种的适应性和抗性提出了新的要求。因此,为了满足日益增长的人口对粮食的需求,急需挖掘和利用新的基因资源,以丰富小麦的遗传基础。
节节麦是普通小麦D基因组的祖先种,其染色体组与小麦的D染色体组同源性高,有利于遗传物质的交换和重组,目前,在小麦育种中利用节节麦的方式主要有两种:一是间接途径,即通过四倍体小麦与节节麦杂交获得的人工合成六倍体小麦,利用该桥梁亲本与普通小麦杂交,获得含有节节麦片段的节节麦-小麦渐渗系,并从中选育含有节节麦优良性状的小麦新品种;二是直接途径,即将节节麦直接与普通小麦杂交获得杂种F1,然后与普通小麦亲本回交转移节节麦的优良基因。目前,已将包括抗性基因、加工品质基因、产量基因、形态性状控制基因在内的多种基因导入到育种材料或品种小麦中(表 1)。
目前,间接途径是将节节麦优良基因转入小麦的主要方式。通过将圆锥小麦、硬粒小麦、波斯小麦等与节节麦杂交,合成了多种具有优良性状的人工合成小麦材料,并将这些双二倍体材料运用到育种中去,已成功地将抗叶锈病和抗秆锈病基因导入到普通小麦中[2, 30-41]。廖祥政[42]利用波斯小麦-节节麦人工合成六倍体与普通小麦品种莱州953杂交构建导入系遗传群体,并检测其千粒重的QTL位点。结果表明,在高千粒重导入系群体中,检测到3个来自人工合成小麦的新QTL位点,其中QGw.caas-3D位点在3个环境中都被检测到,可解释表型变异的18.1%-31.8%,能够增加千粒重2.3-4.8g,是一个稳定的主效QTL。李文才等[43]利用人工合成小麦与普通小麦品种杂交构建的遗传群体,对其株高、单株穗数、穗长、每穗小穗数、穗粒数、单株产量和千粒重等性状的分析结果表明,人工合成小麦对上述性状具有明显正效影响,并在其D组染色体上检测到相关的主效QTL位点。Laikova等[2]利用节节麦-提莫菲维小麦合成人工小麦并与面包小麦杂交,获得了具有高谷蛋白和面包制作品质的小麦新品种,此品种还具备良好的抗叶锈病、茎锈病、白粉病能力。Pestsova等[44]构建了一套来源于人工合成六倍体小麦(四倍体小麦×节节麦)的以中国春为背景的导入系,定位了株高、花期、穗长和千粒重等17个重要性状的QTL位点。Edae等[45]利用普通小麦和人工合成六倍体小麦(四倍体小麦×节节麦)构建了一套单片段代换系,发现了一个与产量性状相关的稳定的QTL位点wpt6531,该位点位于节节麦D基因组的2DS上。利用四倍体小麦-节节麦人工合成种培育小麦新品种的显著例子是川麦42(SynCD768/SW3243//川6415),该品种是近年来西南地区育成的突破性品种,在多年的区域产量试验中比对照品种增加20%左右,其外源染色体片段主要集中在人工合成小麦的1B和3D上,并发现了一个来自节节麦4D上的多毛叶鞘QTL位点,该位点与产量、粒重、穗粒重呈显著正相关[46]。
利用直接途径将节节麦的染色体片段转移到普通小麦,可以避免A、B染色体组的干扰,更有针对性地转移节节麦D染色体组的优异基因[53, 61-62]。Gill等[63]利用节节麦作父本与普通小麦杂交,经幼胚培养得到杂种F1,该杂种自交不育,用普通小麦与之回交得到BC1F1,除部分植株不育外,再经一次回交获得较为稳定的节节麦-小麦导入系,将抗虫基因(麦二叉蚜、小麦瘿蚊)转移到了普通小麦中。Olson等[64]利用节节麦TA1662和PI 603225,分别与一个高产的小麦品种KS05HW14杂交,构建了BC1F2和BC2F2导入系,将抗秆锈基因导入到普通小麦中。然而,利用直接途径转移节节麦的有益基因的研究并不多,其主要原因有2个,一个是以节节麦作父本,小麦作母本进行杂交获得F1的难度较大,其次是获得的杂种F1与轮回亲本的回交结实率极低,造成转移到普通小麦中的节节麦片段过少。河南大学作物逆境适应与改良实验室针对上述的2个问题进行了改良,以节节麦作母本与普通小麦杂交,可以显著提高杂种F1的成胚率,达到22.9%。然后利用秋水仙素加倍技术将F1加倍获得节节麦-小麦人工八倍体,以该八倍体作父本与轮回亲本小麦杂交,可以显著提高后代的结实率,同时,也置换掉了节节麦的细胞质,利用该人工八倍体与轮回亲本周麦18杂交的结实率可以达到60%以上[56]。Zhang等[4]利用该方法获得一个含有节节麦T093染色体片段的周麦18为背景的BC3F4群体。利用该群体进行抗穗发芽试验,发现了2个抗穗发芽的主效QT位点,这为直接利用节节麦改良普通小麦提供了新的途径。
此外,解析节节麦的基因组,不仅可以为小麦的起源和进化研究提供信息,而且还为利用节节麦改良普通小麦提供了大量的分子标记和有效的高密度遗传图谱,为利用节节麦染色体片段代换系精细定位和克隆节节麦农艺性状、抗性性状基因提供数据保障。2013年,Jia等[65]以节节麦AL8/78为材料,利用Illumina高通量测序技术对测序材料进行全基因组鸟枪法测序,得到全基因组草图,其contig N50长度为4 512 bp,Scafflod N50长度为57.6 kb,基因组覆盖度为83.4%,其中65.9%是转座子。同时,注释到43 150个功能基因,并将其中30 303个锚定到遗传图谱上。同时,Luo等[66]利用SNaPshot法对节节麦AL8/78的BAC文库进行测序,并开发出SNP芯片,构建了包含7 185个标记的物理图谱,为节节麦基因组的解析奠定了基础。随着基因组测序组装技术的进步,2017年最新版的节节麦基因组测序数据又重新发布。Zhao等[67]利用新的测序组装技术,对节节麦AL8/78进行组装,得到全新的基因组数据,将92.5%的序列锚定到染色体上,Contig N50达到112.6 kb,Scafflod N50达到12.1 Mb,相比之前的版本提升了210倍,并构建了包含164 872个SNP位点的高密度遗传图谱;同时,准确描述了染色体上的基因位点、假基因、甲基化位点、重组率、TE分布等信息,并且将已知的QTL位点锚定到节节麦基因组上。Luo等[68]利用BAC文库、全基因组鸟枪法、Pacbio和BioNano光学图谱等方法相结合,对节节麦AL8/78进行测序、组装,得到了较为完整的节节麦基因组数据,并最终将92.5%的序列组装到染色体上,共注释83 117个基因,其中39 622个高可信度基因,43 495个低可信度基因,并将38 775个高可信度基因锚定到染色体上。同时,研究还发现节节麦基因组内含有大量的重复序列,TE含量达到了84.4%,这些高度相似的重复序列会导致频繁的错误重组,进而导致基因复制和染色体结构发生变化,加速基因组的进化。
4 小结与展望小麦作为我国的主要粮食作物,多年的持续增产为我国的粮食安全作出了重要贡献。自以矮秆为基础的第一次绿色革命成功以来,我国的小麦种植一直以矮秆、抗倒、密植为基础,用多施肥、高农药的管理手段,成功实现了高产、稳产的目标,并在抗虫、抗病、优质等方面取得了一定的成绩,但仍然面临遗传资源筛选利用不充分,复合抗性品种较少,作物营养利用率低,极端环境耐受性差等问题。节节麦具有丰富的遗传多样性,蕴含了大量与抗病、抗虫、抗逆、产量、品质相关的基因,可以为小麦育种提供丰富的基因资源。同时,节节麦基因组测序的完成和释放,使研究控制其优良性状的基因更为简易,使基于大量分子标记和高密度遗传图谱的分子育种更为高效。因此,充分挖掘节节麦中对小麦有益的基因资源,将是解决小麦育种资源缺乏、品种改良困难的有效途径之一。
| [1] |
Olson EL, Rouse MN, Pumphrey MO, et al. Introgression of stem rust resistance genes SrTA10187 and SrTA10171 from Aegilops tauschii to wheat[J]. Theoretical & Applied Genetics, 2013, 126(10): 2477-2484. |
| [2] |
Laikova LI, Belan IA, Badaeva ED, et al. Development and study of spring bread wheat variety Pamyati Maystrenko with introgression of genetic material from synthetic hexaploid Triticum timopheevii Zhuk.×Aegilops tauschii Coss[J]. Genetika, 2013, 49(1): 103-112. |
| [3] |
张颙, 杨武云, 谭禺. 高产、高抗条锈病小麦新品种川麦47[J]. 四川农业科技, 2008(11): 20. DOI:10.3969/j.issn.1004-1028.2008.11.009 |
| [4] |
Zhang DL, He J, Huang Lǐ, et al. An advanced backcross population through synthetic octaploid wheat as a "Bridge":Development and QTL detection for seed dormancy[J]. Front Plant Sci, 2017, 8: 2123. DOI:10.3389/fpls.2017.02123 |
| [5] |
Eig A. Monographisch-kritische ubersicht der gattung Aegilops[J]. Repert Spec nov Regni Veg, 1929, 55: 1-228. |
| [6] |
Hammer K. Preliminary work on monographs of wild plant collections:Aegilops L.[J]. Kulturpflanze, 1980, 28: 33-180. DOI:10.1007/BF02014641 |
| [7] |
Matsuoka Y, Nishioka E, Kawahara T, et al. Genealogical analysis of subspecies divergence and spikelet-shape diversification in central Eurasian wild wheat Aegilops tauschii Coss.[J]. Plant Systematics & Evolution, 2009, 279(1/4): 233-244. |
| [8] |
张学勇, 杨欣明, 董玉琛. 醇溶蛋白电泳在小麦种质资源遗传分析中的应用[J]. 中国农业科学, 1995, 28(4): 25-32. DOI:10.3321/j.issn:0578-1752.1995.04.013 |
| [9] |
Dudnikov AJ. Allozyme variation in Transcaucasian populations of Aegilops squarrosa[J]. Heredity, 1998, 80(80): 248-258. |
| [10] |
王亚娟, 王耀勇, 张德华, 等. 节节麦农艺性状及高分子量谷蛋白亚基遗传多样性研究[J]. 西北植物学报, 2007, 27(10): 1967-1972. DOI:10.3321/j.issn:1000-4025.2007.10.007 |
| [11] |
苏亚蕊, 张大乐, 张明, 等. 黄河中游粗山羊草三种y-型高分子量谷蛋白亚基的鉴定、克隆及系统进化分析[J]. 作物学报, 2009, 35(7): 1244-1252. |
| [12] |
Lubbers EL, Gill KS, Cox TS, et al. Variation of molecular markers among geographically diverse accessions[J]. Genome, 1991, 34(3): 354-361. DOI:10.1139/g91-057 |
| [13] |
孔令让, 董玉琛. 粗山羊草随机扩增多态性DNA研究[J]. 植物学报:英文版, 1998(3): 223-227. |
| [14] |
李玉阁, 苏亚中, 张大乐, 等. 中国节节麦基于ISSR标记的遗传多样性分析[J]. 麦类作物学报, 2017, 37(1): 30-39. |
| [15] |
Pestsova E, Korzun V, Goncharov NP, et al. Microsatellite analysis of Aegilops tauschii germplasm[J]. Theoretical & Applied Genetics, 2000, 101(1/2): 100-106. |
| [16] |
苏亚蕊, 张大乐, 徐守明, 等. 粗山羊草居群间遗传分化及多样性的SSR分析[J]. 生态学报, 2010, 30(4): 969-975. |
| [17] |
Matsuoka Y, Mori N, Kawahara T. Genealogical use of chloroplast DNA variation for intraspecific studies of Aegilops tauschii Coss.[J]. Theoretical & Applied Genetics, 2005, 111(2): 265-271. |
| [18] |
Mizuno N, Yamasaki M, Matsuoka Y, et al. Population structure of wild wheat D-genome progenitor Aegilops tauschii Coss. :implications for intraspecific lineage diversification and evolution of common wheat[J]. Molecular Ecology, 2010, 19(5): 999-1013. DOI:10.1111/mec.2010.19.issue-5 |
| [19] |
Matsuoka Y, Takumi S, Kawahara T. Intraspecific lineage divergence and its association with reproductive trait change during species range expansion in central Eurasian wild wheat Aegilops tauschii Coss. (Poaceae)[J]. BMC Evolutionary Biology, 2015, 15(1): 1-10. |
| [20] |
Wang J, Luo MC, Chen Z, et al. Aegilops tauschii single nucleotide polymorphisms shed light on the origins of wheat D-genome genetic diversity and pinpoint the geographic origin of hexaploid wheat[J]. New Phytologist, 2013, 198(3): 925-937. DOI:10.1111/nph.12164 |
| [21] |
Kihara H, Yamashita K, Tanaka M. Morphological, physiological, genetical and cytological studies in aegilops and triticum collected from Pakistan, Afghanistan and Iran[M]. Kyoto: Committee of the Kyoto University Scientific Expedition to the Karakoram and Hindukush, Kyoto University, 1965: 26-27.
|
| [22] |
Jaaska V. Electrophoretic survey of seedling esterases in wheats in relation to their phylogeny[J]. Theoretical & Applied Genetics, 1980, 56(6): 273-284. |
| [23] |
孔令让, 董玉琛. 粗山羊草(Aegilops tauschii)遗传多样性的研究进展[J]. 山东农业大学学报:自然科学版, 1999, 30(4): 464-470. |
| [24] |
颜济, 杨俊良, 崔乃然, 等. 新疆伊犁地区的节节麦(Aegilops tauschii Cosson)[J]. 作物学报, 1984(1): 1-8. |
| [25] |
钟骏平, 崔乃然, 林培钧. 新疆伊犁节节麦(Aegilops tauschii Cosson)的发现和分布[J]. 新疆农业大学学报, 1984(1): 35-39. |
| [26] |
杨武云. 中国节节麦的细胞遗传学研究及有关问题探讨[J]. 西南科技大学学报:哲学社会科学版, 1992(1): 16-21. |
| [27] |
魏会廷, 李俊, 彭正松, 等. 节节麦DNA指纹关系所揭示的古代中国与西方农业技术交流[J]. 自然科学进展, 2008, 18(9): 987-993. DOI:10.3321/j.issn:1002-008X.2008.09.004 |
| [28] |
张大乐, 苏亚蕊, 李玉阁, 等. 节节麦居群间的籽粒性状变异及相关性分析[J]. 麦类作物学报, 2012, 32(2): 223-228. |
| [29] |
郝晨阳, 王兰芬, 张学勇, 等. 我国育成小麦品种的遗传多样性演变[J]. 中国科学, 2005, 35(5): 408-415. |
| [30] |
许树军, 董玉琛, 陈尚安, 等. 小麦与山羊草双二倍体抗病性的研究与利用[J]. 作物学报, 1990, 16(2): 106-111. DOI:10.3321/j.issn:0496-3490.1990.02.002 |
| [31] |
Dyck PL, Kerber ER. Inheritance in hexaploid Wheat of adult-plant leaf rust resistance derived from Aegilops squarrosa[J]. Canadian Journal of Genetics & Cytology, 1970, 12(1): 175-180. |
| [32] |
Kema GHJ, Lange W. Race-specific suppression of resistance to yellow rust in synthetic hexaploids of wheat[J]. Vortraege Fuer Pflanzenzuechtung, 1992, 24: 206. |
| [33] |
Asins MJ, Carbonell EA. Distribution of genetic variability in a durum wheat world collection[J]. Theoretical & Applied Genetics, 1989, 77(2): 287-294. |
| [34] |
Kerber ER. Suppression of rust resistance in amphiploids of Triticum: International Wheat Genetics Symposium, 1983[C]. Kyoto: International Wheat Genetics Symposium, 1983: 813-817.
|
| [35] |
Martin A. Cytology and morphology of the hybrid Hordeum chilense×Aegilops squarrosa[J]. Journal of Heredity, 1983, 74(6): 487. DOI:10.1093/oxfordjournals.jhered.a109850 |
| [36] |
任强, 刘慧娟, 陈洋, 等. 人工合成小麦CI191抗条锈病基因的鉴定及分子标记定位[J]. 作物学报, 2010, 36(5): 721-727. |
| [37] |
何名召, 王丽敏, 张增艳, 等. 硬粒小麦-粗山羊草人工合成小麦CI108抗条锈病新基因的鉴定、基因推导与分子标记定位[J]. 作物学报, 2007, 33(7): 1045-1050. DOI:10.3321/j.issn:0496-3490.2007.07.001 |
| [38] |
Periyannan S, Bansal U, Bariana H, et al. Identification of a robust molecular marker for the detection of the stem rust resistance gene Sr45 in common wheat[J]. Theoretical & Applied Genetics, 2014, 127(4): 947-955. |
| [39] |
Wang T, Xu SS, Harris MO, et al. Genetic characterization and molecular mapping of Hessian fly resistance genes derived from Aegilops tauschii in synthetic wheat[J]. Theoretical Applied Genetics, 2006, 113(4): 611-618. DOI:10.1007/s00122-006-0325-z |
| [40] |
张颙, 杨武云, 彭云良, 等. 小麦新品种川麦42抗条锈病性遗传分析[J]. 植物保护学报, 2006, 33(3): 287-290. DOI:10.3321/j.issn:0577-7518.2006.03.013 |
| [41] |
兰秀锦, 郑有良, 刘登才, 等. 节节麦抗穗发芽基因的染色体定位及其抗性机理[J]. 中国农业科学, 2002, 35(1): 12-15. DOI:10.3321/j.issn:0578-1752.2002.01.003 |
| [42] |
廖祥政.发掘人工合成小麦中增加千粒重的QTL[D].雅安: 四川农业大学, 2008. http://cdmd.cnki.com.cn/article/cdmd-10626-2008197720.htm
|
| [43] |
李文才, 李涛, 赵逢涛, 等. 小麦D基因组产量性状QTL定位[J]. 华北农学报, 2005, 20(1): 27-30. DOI:10.3321/j.issn:1000-7091.2005.01.006 |
| [44] |
Pestsova EG, Andreas BR, R Der MS. Development and QTL assessment of Triticum aestivum-Aegilops tauschii introgression lines[J]. Theoretical & Applied Genetics, 2006, 112(4): 634-647. |
| [45] |
Edae EA, Byrne PF, Haley SD, et al. Genome-wide association mapping of yield and yield components of spring wheat under contrasting moisture regimes[J]. Theoretical & Applied Genetics, 2014, 127(4): 791-807. |
| [46] |
Wan H, Yang Y, Li J, et al. Mapping a major QTL for hairy leaf sheath introgressed from Aegilops tauschii and its association with enhanced grain yield in bread wheat[J]. Euphytica, 2015, 205(1): 275-285. DOI:10.1007/s10681-015-1457-5 |
| [47] |
Gill BS, Wilson DL, Raupp WJ, et al. Registration of KS89WGRC3 and KS89WGRC6 Hessian fly-resistant hard red winter wheat germplasm[J]. Crop Science, 1991, 31(1): 245. DOI:10.2135/cropsci1991.0011183X003100010079x |
| [48] |
Cox TS, Sears RG, Gill BS, et al. Registration of KS91WGRC11, KS92WGRC15, KS92WGRC23 leaf rust-resistant hard red winter wheat germplasms[J]. Crop Science, 1994, 34(2): 595. |
| [49] |
Eastwood RF, Lagudah ES, Appels R. A directed search for DNA sequences tightly linked to cereal cyst nematode resistance genes in Triticum tauschii[J]. Genome, 1994, 37(2): 311-319. DOI:10.1139/g94-043 |
| [50] |
Marais GF, Wessels WG, Horn M, et al. Association of a stem rust resistance gene(Sr45)and two Russian wheat aphid resistance genes(Dn5 and Dn7)with mapped structural loci in common wheat[J]. South African Journal of Plant & Soil, 1998, 15(2): 67-71. |
| [51] |
Cox TS, Bockus WW, Gill BS, et al. Registration of KS96WGRC40 hard red winter wheat germplasm resistant to wheat curl mite, stagnospora leaf blotch, and septoria leaf blotch[J]. Crop Science, 1999, 39(2): 597. |
| [52] |
Xu SS, Cai X, Wang T, et al. Registration of two synthetic hexaploid wheat germplasms resistant to Hessian fly[J]. Crop Science, 2006, 46(3): 1401-1402. DOI:10.2135/cropsci2005.06-0137 |
| [53] |
Olson EL, Rouse MN, Pumphrey MO, et al. Simultaneous transfer, introgression, and genomic localization of genes for resistance to stem rust race TTKSK(Ug99)from Aegilops tauschii to wheat[J]. Theoretical & Applied Genetics, 2013, 126(5): 1179-1188. |
| [54] |
Wiersma AT, Pulman JA, Brown LK, et al. Identification of Pm58 from Aegilops tauschii[J]. Theoretical & Applied Genetics, 2017, 130(1): 1-11. |
| [55] |
Nishijima R, Okamoto Y, Hatano H, et al. Quantitative trait locus analysis for spikelet shape-related traits in wild wheat progenitor Aegilops tauschii:Implications for intraspecific diversification and subspecies differentiation[J]. PLoS One, 2017, 12(3): e173210. |
| [56] |
Zhang DL, Zhou Y, Zhao XP, et al. Development and utilization of introgression lines using synthetic octaploid wheat (Aegilops tauschii× Hexaploid wheat) as donor[J]. Front Plant Sci, 2018, 9: 1113. DOI:10.3389/fpls.2018.01113 |
| [57] |
Yan L, Liang F, Xu H, et al. Identification of QTL for grain size and shape on the D genome of natural and synthetic allohexaploid wheats with near-identical AABB genomes[J]. Front Plant Sci, 2017, 8: 1705. DOI:10.3389/fpls.2017.01705 |
| [58] |
Li Y, Zhou R, Wang J, et al. Novel and favorable QTL allele clusters for end-use quality revealed by introgression lines derived from synthetic wheat[J]. Molecular Breeding, 2012, 29(3): 627-643. DOI:10.1007/s11032-011-9578-6 |
| [59] |
Jian Y, Liu Y, Pu Z, et al. Molecular characterization of high pI α-amylase and its expression QTL analysis in synthetic wheat RILs[J]. Molecular Breeding, 2014, 34(3): 1075-1085. DOI:10.1007/s11032-014-0098-z |
| [60] |
Watanabe N, Fujii Y, Takesada N, et al. Cytological and microsatellite mapping of the gene for brittle rachis in a Triticum aestivum-Aegilops tauschii introgression line[J]. Euphytica, 2006, 151(1): 63-69. DOI:10.1007/s10681-006-9128-1 |
| [61] |
Cox TS, Hatchett JH, Gill BS, et al. Agronomic performance of hexaploid wheat lines derived from direct crosses between wheat and Aegilops squarrosa[J]. Plant Breeding, 2010, 105(4): 271-277. |
| [62] |
Fritz AK, Cox TS, Gill BS, et al. Molecular marker-facilitated analysis of introgression in winter wheat×Triticum tauschii populations[J]. Crop Science, 1995, 35(6): 1691-1695. DOI:10.2135/cropsci1995.0011183X003500060030x |
| [63] |
Gill BS, Raupp WJ. Direct genetic transfers from Aegilops squarrosa L. to Hexaploid wheat[J]. Crop Science, 1987, 27(3): 445-450. DOI:10.2135/cropsci1987.0011183X002700030004x |
| [64] |
Olson EL, Rouse MN, Pumphrey MO, et al. Introgression of stem rust resistance genes SrTA10187 and SrTA10171 from Aegilops tauschii to wheat[J]. Theoretical & Applied Genetics, 2013, 126(10): 2477-2484. |
| [65] |
Jia J, Zhao S, Kong X, et al. Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation[J]. Nature, 2013, 496(7443): 91-95. DOI:10.1038/nature12028 |
| [66] |
Luo MC, Gu YQ, You FM, et al. A 4-gigabase physical map unlocks the structure and evolution of the complex genome of Aegilops tauschii, the wheat D-genome progenitor[J]. Proc Natl Acad Sci USA, 2013, 110(19): 7940-7945. DOI:10.1073/pnas.1219082110 |
| [67] |
Zhao G, Zou C, Li K, et al. The Aegilops tauschii genome reveals multiple impacts of transposons[J]. Nature Plants, 2017, 3: 946-955. DOI:10.1038/s41477-017-0067-8 |
| [68] |
Luo MC, Gu YQ, Puiu D, et al. Genome sequence of the progenitor of the wheat D genome Aegilops tauschii[J]. Nature, 2017, 551(7681): 498. DOI:10.1038/nature24486 |