2. 黑龙江中医药大学基础医学院,哈尔滨 150040;
3. 齐齐哈尔医学院医药科学研究院,齐齐哈尔 161006
2. Basic Medical College, Heilongjiang University of Chinese Medicine, Harbin 150040;
3. Research Center of Medical Science, Qiqihar Medical University, Qiqihar 161006
TOX高迁移率蛋白盒家族成员(TOX high-mobility box protein group family member3,TOX3)基因为高迁移率蛋白(High mobility group,HMG)家族成员之一。2007年,Easton等[1]通过全基因组关联分析(Genome-wide association study,GWAS)发现其与乳腺癌易感性相关。随后,在不同人群的GWAS研究中进一步证实了TOX3与乳腺癌的发展存在强烈的相关性[2-14]。研究发现其在乳腺癌组织中通常过表达,而在正常乳腺组织中低表达或表达缺失[15],且与临床TNM分期分级密切相关,与乳腺癌淋巴结转移也存在相关性[16]。但其在乳腺癌恶性病变进程中所发挥的具体生物学功能尚不明确。因此,构建稳定沉默TOX3基因的乳腺癌细胞模型对于深入研究其在乳腺癌发生发展过程中的作用及机制则尤为重要。
本研究拟构建TOX3 RNAi慢病毒表达载体,通过荧光显微镜观察转染效率,采用实时荧光定量PCR及Western blot检测转染后ZR-75-1细胞内TOX3 mRNA及蛋白表达水平,构建稳定沉默TOX3基因的乳腺癌细胞模型,并初步探讨干扰TOX3基因的表达对乳腺癌细胞增殖的影响,旨为进一步深入研究TOX3基因在乳腺癌发展进程中的生物学功能奠定实验基础。
1 材料与方法 1.1 材料人乳腺癌ZR-75-1细胞、人肾上皮293T细胞均购于中科院上海细胞库; 胎牛血清、1640培养基和DMEM培养基均购自Gibco公司; 慢病毒载体质粒、包装质粒(pGag/Pol、pRev、pVSV-G)及RNAi-Mate转染试剂均购于苏州吉玛基因股份有限公司; DNA内切酶(BamHI、EcoRI)和T4 DNA连接酶购自MBI Fermentas公司; DNA凝胶回收试剂盒购自天根生化科技有限公司; 中量抽提试剂盒购自杭州爱思进生物技术有限公司; 嘌呤霉素购自美国Sigma公司; PCR引物由生工生物工程(上海)有限公司合成,反转录试剂盒及实时荧光定量PCR试剂盒购自日本TaKaRa公司,兔抗人TOX3单克隆抗体购自Abcam公司; 抗兔二抗购自美国Cell Signaling公司。
1.2 方法 1.2.1 靶向TOX3基因shRNA慢病毒表达载体的构建GenBank搜索TOX3基因序列(NM_00108043-0.2),依据shRNA序列设计原则,设计合成3条干扰靶序列及1条阴性对照序列。退火合成双链DNA,T4 DNA连接酶连接线性化LV3-shRNA载体; 制备感受态细胞DH5α; 挑取阳性克隆,抽提质粒后进行测序鉴定。靶向TOX3基因干扰序列,如表 1所示。
将293T细胞培养于含有10%FBS、100 IU/mL青霉素和100 µg/mL链霉素的DMEM培养基中,置于37℃、5% CO2饱和湿度培养箱中常规培养。待其生长到80%左右融合时,将空质粒pGLV3/H1/GFP-Puro、重组质粒pGLV3/H1/GFP-Puro-shTOX3、和包装质粒pGag/Pol、pRev、pVSV-G按比例加入装有1.5 mL无血清培养基的离心管中,混匀。另将转染试剂RNAi-Mate加入装有1.5 mL无血清的培养基中,混匀。室温放置5 min后,将两管试剂混匀。室温放置20-25 min后,去除培养皿中的旧培养液,加入新的无血清培养液,将混合液加入细胞培养液中,于细胞培养箱中培养4-6 h。除去转染液,加入含血清DMEM培养基,继续培养72 h。吸取细胞上清液到离心管中,低速离心,0.45 μm过滤,在4℃ 20 000 r/min条件下超速离心2 h,收集浓缩液。
1.2.3 病毒滴度检测在检测病毒滴度前1 d,以3×104个/孔的细胞密度将293T细胞接种于96孔板中。常规培养24 h后,用含有10%FBS的培养基将10 μL慢病毒原液10倍稀释4个梯度。吸弃96孔板内的旧培养液,加入稀释好的病毒液,每孔100 µL,于培养箱内培养24 h后,将病毒液吸出,每孔加入完全培养基100 µL,继续培养72 h。于荧光显微镜下计数表达绿色荧光细胞的数目。并结合相应的稀释倍数计算病毒滴度。
1.2.4 转染ZR-75-1细胞并建立稳定的细胞系取生长状态良好的ZR-75-1细胞,以5×104个细胞/孔的细胞密度接种于24孔板内,加入500 µL完全培养基,过夜。将慢病毒液按照预实验得到的合适的感染参数(Multiplicity of infection,MOI)加入到含有聚凝胺的400 µL培养基中,使聚凝胺的终浓度为5 μg/mL。同时设立空白对照组(未转染的ZR-75-1细胞)、阴性对照LV3-NC组(转染空载体)、TOX3-shRNA-1组、TOX3-shRNA-2组和TOX3-shRNA-3组。12 h后弃掉含有病毒液的培养基,加入完全培养基继续培养48 h。弃掉旧培养基,加入含有嘌呤霉素2 μg/mL的筛选培养基(经预实验确定ZR-75-1细胞的最小死亡浓度为2 μg/mL),每2-3 d更换筛选培养基,连续培养7 d,随后更换为浓度为1 μg/mL的筛选培养基继续培养。获得具有抗性的转染细胞,荧光显微镜下观察表达GFP细胞的比例。筛选出稳定转染的抗性阳性克隆,扩大培养。所建立的细胞分别命名为TOX3-shRNA-1、TOX3-shRNA-2、TOX3-shRNA-3和LV3-NC。
1.2.5 实时荧光定量PCR检测ZR-75-1细胞TOX3 mRNA的表达应用Trizol提取各组细胞总RNA,将其逆转录为cDNA,应用实时荧光定量PCR仪检测TOX3 mRNA的表达。PCR反应引物如下: β-actin:上游引物: 5'-CTGGGACGACATGGAGAAAA-3',下游引物: 5'-AAGGAAGGCTGGAAGAGTGC-3'; TOX3:上游引物: 5'-TATGCCTCACACATCTCCTTCA-3',下游引物: 5'-ATGGCTCTGTTGGCTTCATC-3'。PCR反应条件为第一步: 95℃ 30 s,1个循环; 第二步:变性: 95℃ 5 s; 退火: 60℃ 31 s,40个循环。
1.2.6 Western blot检测ZR-75-1细胞TOX3蛋白的表达收集各组细胞,应用RIPA强效裂解液提取各组细胞总蛋白,采用BCA法检测蛋白浓度并定量。取20 μg蛋白进行SDS-PAGE电泳,转膜,采用BSA封闭液4℃封闭过夜。加入兔抗人TOX3单克隆抗体(1:2 000),小鼠抗人GAPDH多克隆抗体(1:3 000),室温孵育3 h,TBST洗膜,HRP标记的山羊抗兔二抗(1:3 000),马抗小鼠二抗(1:3 000)室温孵育2 h,TBST洗膜,ECL显影,曝光。
1.2.7 MTT法检测TOX3对细胞增殖能力的影响将各组细胞以3×103个/孔细胞密度接种于96孔板中,每组设5个复孔,常规培养过夜。次日弃掉旧培养基,每孔加入200 µL新鲜培养基,继续培养。于检测当日加入20 µL MTT溶液,37℃孵育4 h。终止培养,小心吸出上清液,每孔加入DMSO 150 µL,室温震荡使结晶充分溶解。于570 nm波长处,应用酶标仪检测各孔吸光度值。以时间为横坐标,测得的吸光度值为纵坐标,绘制第0天至第7天各组细胞的生长曲线。
1.2.8 平板单克隆形成实验将各组细胞接种于6孔板内,使细胞密度为3 000个/孔。视细胞生长情况每3-4 d换液一次,连续培养14 d后,除去旧培养基,PBS清洗。加入冰甲醇固定10 min。再用PBS洗2次。加入0.1%结晶紫染色10 min,PBS洗去结晶紫。计细胞数大于50个的集落数,采用凝胶成像分析系统拍照,计算集落形成率。集落形成率(%)=计数细胞集落数/细胞总数集×100%。
2 结果 2.1 重组慢病毒的测序鉴定抽提3组重组载体质粒,经测序鉴定结果(图 1)显示,各组质粒序列正确。

|
| A: TOX3-sh-1组; B: TOX3-sh-2组; C: TOX3-sh-3组 图 1 TOX3-shRNA干扰质粒测序图谱 |
病毒滴度检测结果(图 2)显示,经病毒包装重组质粒TOX3-shRNA-1,TOX3-shRNA-2,TOX3-shRNA-3的病毒滴度分别为3×108 TU/mL,4×108 TU/mL和2×108 TU/mL。
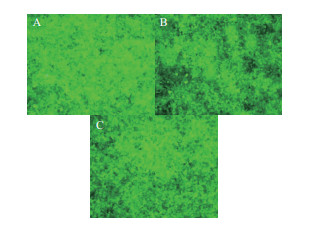
|
| A: TOX3-shRNA-1组; B: TOX3-shRNA-2组; C: TOX3-shRNA-3组 图 2 重组慢病毒转染293T细胞后GFP的表达(×40) |
经嘌呤霉素筛选后,荧光显微镜下观察表达GFP的细胞比例。结果(图 3)显示LV3-NC组、TOX3-shRNA-1组、TOX3-shRNA-2组和TOX3-shRNA-3组GFP表达均约达到95%以上。
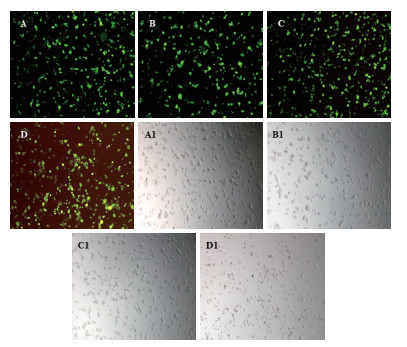
|
| A,A1: LV3-NC组; B,B1: TOX3-shRNA-1组; C,C1: TOX3-shRNA-2组; D,D1: TOX3-shRNA-3组 图 3 经嘌呤霉素筛选后ZR-75-1细胞的GFP表达(×40) |
实时荧光定量PCR检测结果(图 4)显示,与空白对照组比较,空载体LV3-NC组TOX3 mRNA的相对表达无明显差异(P > 0.05),而与空载体LV3-NC对照组比较,不同的TOX3-shRNA干扰序列均显示出明显的干扰效果(P < 0.05),其中TOX3-shRNA-3组的mRNA表达水平最低,干扰效率最高。

|
| 1:空白对照组; 2: LV3-NC组; 3: TOX3-shRNA-1组; 4: TOX3-shRNA-2组; 5: TOX3-shRNA-3组。与LV3-NC组比较,* P < 0.05,下同 图 4 转染ZR-75-1细胞后TOX3 mRNA的相对表达 |
蛋白检测结果(图 5)显示,与空白对照组比较,空载体LV3-NC组TOX3蛋白的相对表达无明显差异(P > 0.05)。与空载体LV3-NC对照组比较,不同干扰序列TOX3蛋白的表达水平均明显降低(P < 0.05),其中TOX3-shRNA-3组TOX3蛋白水平最低,与实时荧光定量PCR结果一致。因此,在后续实验中我们选取TOX3-shRNA-3组细胞作为研究TOX3基因对乳腺癌细胞增殖活性影响的细胞模型。

|
| 图 5 转染后各组细胞TOX3蛋白的相对表达 |
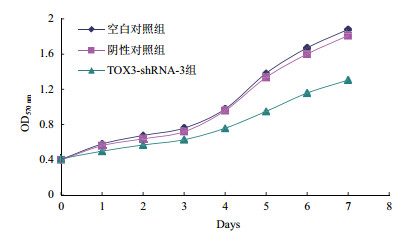
稳定干扰TOX3基因的表达后,采用MTT实验观察0-7 d干扰TOX3基因对ZR-75-1细胞增殖的影响。实验结果(图 6)显示:与空白对照组比较,阴性对照组细胞增殖活性没有显著变化。与阴性对照组比较,随着时间的推移TOX3-shRNA-3组细胞的增殖能力明显下降。平板单克隆实验结果(图 7)显示,与阴性对照组比较,干扰TOX3基因后,细胞的集落形成率明显降低,单克隆形成能力减弱。
|
| 图 6 干扰TOX3基因对ZR-75-1细胞增殖的影响 |

|
| A:各组细胞平板克隆形成图; B:各组细胞平板克隆分析图; 1:空白对照组; 2:阴性对照组; 3: TOX3-shRNA-3组; 与阴性对照组比较,*P < 0.05 图 7 干扰TOX3对ZR-75-1细胞单克隆形成的影响 |
乳腺癌疾病已成为女性死亡的重要原因,目前尚未完全阐明其发病机制。研究认为其恶性表征相关基因的异常可能为其发病机制之一。TOX3属于HMG家族成员之一,此家族蛋白在调控基因表达及细胞增殖分化等方面均起重要作用。近年研究发现TOX3与乳腺癌易感性相关[1]。随后,在不同人群的GWAS研究也中进一步证实了TOX3与乳腺癌的危险性存在强烈相关性[2-14]。此外,研究发现,与ER阴性乳腺癌相比,位于TOX3基因上的多态性位点与ER阳性乳腺癌具有更强的相关性[4, 15]。在ER阳性,HER2阴性乳腺癌患者中,TOX3基因的rs3803662多态性位点可作为绝经前和绝经后妇女乳腺癌患病风险的预测因子之一[16],但TOX3基因在乳腺癌发展中的作用机制尚不清楚,相关文献报道也较少。研究发现乳腺癌组织中TOX3蛋白的表达量明显高于正常组织,且其表达水平与临床TNM分期和分级具有明显相关性[17],在生存期较短的乳腺癌患者中,TOX3的mRNA表达水平较高,且其表达水平与乳腺癌转移相关[18]。此外,Smid等[19]研究发现TNRC9(TOX3)基因在乳腺癌患者骨中高表达,认为TOX3可能是乳腺癌骨转移相关基因。研究进一步发现过表达TOX3基因可促进乳腺癌细胞的侵袭和转移,并可通过下调BRCA1的表达增强乳腺癌细胞侵袭能力; 干扰TOX3基因后,成瘤率明显降低,且形成的肿瘤也明显减小[20]。因此,TOX3在乳腺癌的发展进程中发挥重要作用,对乳腺癌的发展可能是一个危险因素。
RNA干扰是近年用于研究特定基因功能的常用技术,其可精确沉默或敲除特定基因,对基因的抑制作用强且特异性高,为基因的功能学研究提供了有效的研究方法[21]。RNA干扰常用的表达载体主要有慢病毒、腺病毒和质粒,其中慢病毒载体能够将目的基因高效的整合到宿主细胞的基因组内,使目的基因在传代的过程中仍可长期稳定表达,其应用范围较广,转染效率高,不产生化学转染等方法引起的细胞损伤反应,是目前进行基因功能研究和基因治疗的有力工具。研究者应用慢病毒表达载体构建稳定过表达TOX3基因的乳腺癌MDA-MB-231细胞系,结果表达GFP细胞的数目达95%以上,且转染后MDA-MB-231细胞内TOX3的表达明显升高[22]。在RNAi实验中,能否抑制目的基因的表达,靶序列的设计非常重要。研究者通过设计合成3条FUT8基因的shRNA干扰靶序列,经退火,线性化连接,构建FUT8基因的RNAi慢病毒表达载体,并将其转染到乳腺癌MCF-7细胞中,转染效率达90%,各干扰组FUT8 mRNA和蛋白的表达均明显降低,其中pGC-shFUT8-2序列的干扰效率最高,且发现干扰FUT8基因后,MCF-7细胞增殖能力下降[23]。在本研究中,根据GeneBank中提供的TOX3基因序列,针对基因不同位点,按照设计原则设计合成3条shRNA靶序列,并将其连接到LV3-shRNA中,进行重组克隆,经测序验证,序列正确,成功构建了shRNA表达载体。我们将构建好的TOX3-shRNA表达载体,经病毒包装,采用慢病毒载体转染自身高表达TOX3基因的人乳腺癌细胞系ZR-75-1,通过嘌呤霉素筛选后,荧光显微镜下观察表达绿色荧光蛋白的细胞数目可达95%以上,获得稳定转染TOX3基因的细胞系ZR-75-1。通过实时荧光定量PCR和Western blot进一步验证干扰效果,筛选出干扰效果最好的靶序列TOX3-shRNA-3,成功获得了稳定沉默TOX3基因的乳腺癌细胞系ZR-75-1,并将其确定为后续实验研究细胞模型。MTT实验和平板克隆实验初步探讨干扰TOX3基因的表达对乳腺癌细胞增殖的影响。研究发现干扰TOX3可使ZR-75-1细胞的增殖能力下降,单克隆形成能力减弱,而TOX3基因的其他生物学功能及调控机制仍需进一步研究。
4 结论本研究成功构建TOX3基因的RNAi慢病毒载体,并获得稳定沉默TOX3基因的乳腺癌ZR-75-1细胞系,初步发现沉默TOX3后ZR-75-1细胞的增殖能力下降。
| [1] |
Easton DF, Pooley KA, Dunning AM, et al. Genome-wide association study identifies novel breast cancer susceptibility loci[J]. Nature, 2007, 447(7148): 1087-1093. DOI:10.1038/nature05887 |
| [2] |
Stacey SN, Manolescu A, Sulem P, et al. Common variants on chromosomes 2q35 and 16q12 confer susceptibility to estrogen receptor-positive breast cancer[J]. Nat Genet, 2007, 39(7): 865-869. DOI:10.1038/ng2064 |
| [3] |
Ruiz-Narváez EA, Rosenberg L, Cozier YC, et al. Polymorphisms in the TOX3/LOC643714 locus and risk of breast cancer in African-American women[J]. Cancer Epidemiol Biomarkers Prev, 2010, 19(5): 1320-1327. DOI:10.1158/1055-9965.EPI-09-1250 |
| [4] |
Liang J, Chen P, Hu Z, et al. Genetic variants in trinucleotide repeat-containing 9(TNRC9)are associated with risk of estrogen receptor positive breast cancer in a Chinese population[J]. Breast Cancer Res Treat, 2010, 124(1): 237-241. |
| [5] |
Chen MB, Wu XY, Shen W, et al. Association between polymorphisms of trinucleotide repeat containing 9 gene and breast cancer risk: evidence from 62, 005 subjects[J]. Breast Cancer Res Treat, 2011, 126(1): 177-183. |
| [6] |
Han W, Woo JH, Yu JH, et al. Common genetic variants associated with breast cancer in Korean women and differential susceptibility according to intrinsic subtype[J]. Cancer Epidemiol Biomarkers Prev, 2011, 20(5): 793-798. DOI:10.1158/1055-9965.EPI-10-1282 |
| [7] |
Elematore I, Gonzalez-Hormazabal P, Reyes JM, et al. Association of genetic variants at TOX3, 2q35 and 8q24 with the risk of familial and early-onset breast cancer in a South-American population[J]. Mol Biol Rep, 2014, 41(6): 3715-3722. DOI:10.1007/s11033-014-3236-0 |
| [8] |
Long J, Cai Q, S hu, et al. Identification of a functional genetic variant at 16q12. 1 for breast cancer risk: Results from the Asia Breast Cancer Consortium[J]. PLoS Genet, 2010, 6(6): e1001002. DOI:10.1371/journal.pgen.1001002 |
| [9] |
He X, Yao G, Li F, et al. Risk-association of five SNPs in TOX3/LOC643714 with breast cancer in southern China[J]. Int J Mol Sci, 2014, 15(2): 2130-2141. DOI:10.3390/ijms15022130 |
| [10] |
Liao J, Chen Y, Zhu J, et al. Polymorphisms in the TOX3/LOC643714 and risk of breast cancer in south China[J]. Int J Biol Markers, 2018, 4(1): 1-8. |
| [11] |
Zhang B, Chen MY, Shen YJ, et al. A large-scale, exome-wide association study of han chinese women identifies three novel loci predisposing to breast cancer[J]. Cancer Res, 2018, 78(11): 3087-3097. DOI:10.1158/0008-5472.CAN-17-1721 |
| [12] |
Hsieh YC, Tu SH, Su CT, et al. A polygenic risk score for breast cancer risk in a Taiwanese population[J]. Breast Cancer Res Treat, 2017, 163(1): 131-138. |
| [13] |
Al-Eitan, Laith N, Jamous, et al. Candidate gene analysis of breast cancer in the jordanian population of arab descent: A case-control study[J]. Cancer Invest, 2017, 35(4): 256-270. DOI:10.1080/07357907.2017.1289217 |
| [14] |
Palomba G, Loi A, Porcu E, et al. Genome-wide association study of susceptibility loci for breast cancer in Sardinian population[J]. BMC Cancer, 2015, 15: 383-392. DOI:10.1186/s12885-015-1392-9 |
| [15] |
Garcia-Closas M, Chanock S. Genetic susceptibility loci for breast cancer by estrogen receptor status[J]. Clin Cancer Res, 2008, 14(24): 8000-8009. DOI:10.1158/1078-0432.CCR-08-0975 |
| [16] |
Guo JZ, Sueta A, Nakamura K, et al. Genetic and environmental factors and serum hormones, and risk of estrogen receptor-positive breast cancer in pre-and postmenopausal Japanese women[J]. Oncotarget, 2017, 8(39): 65759-65769. |
| [17] |
Han CC, Yue LL, Yang Y, et al. TOX3 protein expression is correlated with pathological characteristics in breast cancer[J]. Oncology Letters, 2016, 11: 1762-1768. DOI:10.3892/ol.2016.4117 |
| [18] |
Gudmundsdottir ET, Barkardottir RB, Arason A, et al. The risk allele of SNP rs3803662 and the mRNA level of its closest genes TOX3 and LOC643714 predict adverse outcome for breast cancer patients[J]. BMC Cancer, 2012, 12: 621-630. DOI:10.1186/1471-2407-12-621 |
| [19] |
Smid M, Wang Y, Klijn JG, et al. Genes associated with breast cancer metastatic to bone[J]. J Clin Oncol, 2006, 24(15): 2261-2267. DOI:10.1200/JCO.2005.03.8802 |
| [20] |
Shan J, Dsouza SP, Bakhru S, et al. TNRC9 down-regulates BRCA1 expression and promotes breast cancer aggressiveness[J]. Cancer Res, 2013, 73(9): 2840-2849. DOI:10.1158/0008-5472.CAN-12-4313 |
| [21] |
Easton DF, Pooley KA, Dunning AM, et al. Genome-wide association study identifies novel breast cancer susceptibility loci[J]. Nature, 2007, 447(7148): 1087-1093. DOI:10.1038/nature05887 |
| [22] |
韩翠翠, 岳丽玲, 杨莹, 等. 稳定过表达TOX3的乳腺癌MDA-MB-231细胞系的建立[J]. 细胞与分子免疫学杂志, 2014, 30(11): 1154-1158. |
| [23] |
温宪春, 韩翠翠, 赵月生, 等. FUT8基因RNAi慢病毒载体的构建及对MCF-7细胞增殖的影响[J]. 生物技术通报, 2015, 31(5): 231-236. |





