2. 兰州大学生命科学学院细胞活动与逆境适应教育部重点实验室,兰州730000;
3. 四川大学华西临床医学院,成都610041
2. Ministry of Education Key Laboratory of Cell Activities and Stress Adaptations, School of Life Sciences, Lanzhou University, Lanzhou 730000;
3. West China School of Medicine, Sichuan University, Chengdu 610041
荧光假单胞菌(Pseudomonas fluorescens,Pf)是一种革兰氏阴性可动杆菌,广泛分布于土壤、水等自然环境中[1];其最适生长温度和最适生长pH分别为4-32℃和4-8,具有广泛适应性、营养多样性和代谢复杂性的特点[2]。一方面,Pf是生防菌株的重要一员并参与生物降解与工业生物转化[3],其产生的丰富次级代谢产物,如2,4-二乙酰基间三苯酚、吩嗪和藤黄绿菌素等[4-5],及多样的代谢机制已被广泛研究。另一方面,Pf对环境中多种有害化合物的响应机制也备受关注,尤其是多样的抗生素抗性基因,很可能成为基因水平转移的储库[6-7]。
研究表明,Pf对多种抗生素的耐受性常由外排系统介导。外排泵是一种膜转运蛋白,广泛存在于许多微生物基因组中,它不仅参与正常物质运输和代谢,还能转运抗生素、重金属、有机溶剂、去污剂等毒性分子,调节微生物对不同环境的适应能力[8]。EmhABC是荧光假单胞菌2P24中最主要的RND(Resistance nodulation division)家族外排泵,其基因缺失菌株对氨苄西林、卡那霉素、庆大霉素等多种毒物的耐受性大幅下降[9],可见EmhABC能够显著影响细菌对外界有毒物质的抵抗能力,但目前对EmhABC的调控机制并不清楚。本研究中,我们鉴定了荧光假单胞菌2P24中一个新的OmpR家族转录因子RstA,证实RstA能够直接调控外排泵基因emhABC的表达并影响细菌多重耐药性。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒野生型荧光假单胞菌2P24菌株由中国农业大学张立群教授惠赠;大肠杆菌DH5α、Wm3064及BL31(DE3)感受态细胞购自美国Novagen;表达载体pET28b、敲除载体pK18mobsacB及报告基因载体pRG970由本实验室保存;本研究中所使用的引物见表 1。
细菌基因组提取试剂盒、质粒小提试剂盒、胶回收试剂盒购自北京天根生化科技有限公司,Taq MIX购自北京佳兰生物科技有限公司,T4连接酶、限制性内切酶NdeI和XhoI、定量PCR相关制品购自大连宝生物工程有限公司,氯霉素、卡那霉素、庆大霉素、洛美沙星、氨苄霉素、四环素、强力霉素、IPTG购自百灵威科技有限公司,美罗培南、BSA购自索来宝生物,o-nitrophenol、咪唑购自美国Sigma-Aldrich。DU530 UV/Vis分光光度计购自美国Beckman Coulter,NanoDrop2000购自美国Thermo Fisher,凝胶成像系统购自Bio-Rad公司。
1.2 方法 1.2.1 共适应性分析Cofitness Browser Database(http://fit.genomics.lbl.gov/cgi-bin/myFrontPage.cgi)由美国加州大学伯克利分校研究组建立,通过构建随机转座子插入突变体库并联合全基因组测序(Random Bar Code Transposon-site Sequencing,RB-TnSeq),评估了32株来自不同种属菌株突变体对多种生长环境的适应度。每个菌株内都涉及上千种基因缺失突变;生长条件设置包括94种碳源、45种氮源及34-55种抗生素或金属离子。具体评估过程如下:将某菌种所有转座子突变株的混合体在某一生长条件下培养4-8代后,采用DNA Barcode Sequencing技术对比所有突变体培养前后各自标记基因的丰度,并以培养前后该基因突变体丰度比值的对数表征菌株适应度(Strain fitness);基因适应度Gene fitness为不同菌株适应度的加权平均值,并通过计算t值评估每个基因的菌株适应度值的一致性(|t| < 4时无参考意义),即:

在Cofitness Database中,参数Fitness即指上文中的Gene fitness,Fitness < 0表示某基因对某特定生长条件的适应很重要,Fitness > 0则表示某基因对增殖有害,突变体具有生长优势。一般当Fitness < -2或 > 2时表示对生长影响明显。而数据库中的Cofitness值则指同一细菌中一对基因的所有适应度值具有Pearson相关性,即具有相似的适应度模式。通常,Cofitness值> 0.75,提示两基因可能在相同的通路中发挥作用;Cofitness值> 0.6,且它们的直系同源物Cofitness值也 > 0.6,即保守共适应性(Conserved Cofitness),则亦证实紧密功能关系[10-11]。本研究中分别BLAST EmhA、EmhB、EmhC蛋白序列后分析其共适应蛋白及共适应条件。荧光假单胞菌2P24基因组数据取自NCBI,同源序列比对采用软件MEGA7.0完成。
1.2.2 rstA基因缺失菌株的构建本研究中采用两步同源重组法构建基因缺失菌株[9],即以荧光假单胞菌2P24基因组为模板,分别用引物rstAkoU-F/rstAkoU-R和rstAkoD-F/rstAkoD-R扩增出rstA上下游同源臂,并利用重叠延伸PCR技术融合[12],随后采用同源重组的方法将该片段构入pK18mobsacB质粒。重组质粒先转化入大肠杆菌Wm3064感受态细胞,过夜培养后经接合转入荧光假单胞菌2P24中,经Kan、Amp各50 µg/mL的双抗King’s B(KB)培养基[13](蛋白胨20 g/L,MgSO4·7H2O 1.5 g/L,K2HPO4 1.97 g/L,丙三醇10 mL/L)28℃过夜培养后,筛选出载体rstA删除序列与基因组序列发生第一次同源重组成功的菌株并采用引物RstA-F/RstA-R加以验证,随后该菌株于无抗KB培养基28℃培养8 h后利用10%蔗糖无抗KB平板筛选,获得第二次同源重组成功的菌株,经引物RstA-F/RstA-R验证得到rstA基因缺失菌株。
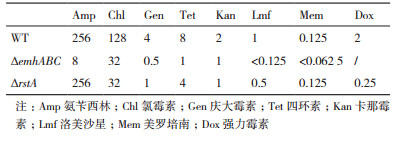
1.2.3 抗生素最小抑制浓度测定将荧光假单胞菌2P24野生型及ΔrstA菌株以1:100接种至新鲜KB无抗培养基,28℃过夜培养后测定OD600,以1 OD600 ≈ 109 CFU/mL计数并稀释。准备无菌96孔板,采用2倍稀释法准备不同浓度的抗生素培养基,取100 μL分别加入96孔板第1至第12孔,之后于每孔中加入100 μL稀释后菌液,确保每孔约104-105个细胞,每种抗生素设置三次平行试验,每板设置一个阴性对照组,即以双蒸水代替抗生素,其余操作步骤均相同。将平板置于28℃摇床培养16-18 h后用酶标仪测定每孔的吸光值OD570,获得抗生素对细菌的最小抑菌浓度(Minimum inhibitory concentration,MIC)[14]。
1.2.4 emhA及emhC转录水平检测荧光假单胞菌2P24野生型及ΔrstA菌株以1:100接种至新鲜无抗KB培养基,于28℃摇至OD600 = 0.3。按RNA提取试剂盒说明提取总RNA,确定RNA浓度及纯度后按反转录试剂盒说明获得cDNA并于-20℃保存。设计靶基因emhA(引物AQ-F/ AQ-R)、emhC(引物CQ-F/CQ-R)及参照基因16S rRNA(引物16s-F/16s-R)的qRT-PCR引物,以cDNA为模板,采用Real Master Mix(SYBR Green)试剂盒完成qRT-PCR。最后以ΔΔCT法进行目的基因表达量分析。
1.2.5 β-半乳糖苷酶实验在pRG970半乳糖苷酶表达基因上游融合emhABC基因的调控序列(引物emhAlacZr-F/ emhAlacZr-R)[15],将构建的pRG970-pemhABC报告质粒经供体菌(大肠杆菌WM3064感受态细胞)接合转入荧光假单胞菌2P24野生型及ΔrstA菌株。上述质粒转入成功的菌株以1:100接种至新鲜无抗KB培养基,28℃培养至OD600 = 0.6后检测β-半乳糖苷酶活性[16]。
1.2.6 rstA基因的克隆以荧光假单胞菌2P24基因组为模板,采用引物RstA-F/RstA-R扩增出rstA全基因序列,将该扩增产物与表达载体pET28b分别使用限制性内切酶NdeI和XhoI进行同步双酶切,凝胶回收后T4连接酶连接并转化至大肠杆菌DH5α,挑选双酶切验证成功的重组表达载体pET28b-rstA送至金唯智测序。
1.2.7 RstA蛋白的表达与纯化将重组质粒转化到表达宿主BL21(DE3),并转接于50 mL含Kan(50 µg/mL)的LB液体培养基中37℃过夜培养。过夜培养物按1:50转接到1 L含Kan(50 µg/mL)的LB培养基中,37℃培养至OD600为0.6-0.8时,加入终浓度为0.2 mmol/L的IPTG,16℃诱导表达20 h后通过离心收集菌体(8000×g,10 min),而后用30 mL悬浮液(20 mmol/L Tris-HCl,10 mmol/L咪唑,400 mmol/L NaCl,pH 8.0)重悬菌体并超声破碎,经4℃,12 000×g,30 min离心得上清液并采用Ni-NTA亲和柱纯化目标蛋白,挂载的His-RstA蛋白由洗脱液(20 mmol/L Tris-HCl,250 mmol/L咪唑,400 mmol/L NaCl,pH 8.0)洗脱后采用超滤法进行溶液置换,使咪唑浓度低于5 mM,最终将蛋白浓度浓缩至4 mg/mL冻存于-80℃待用。
1.2.8 凝胶阻滞实验利用引物FAM-emhAp-F/FAM-emhAp-R扩增emhA基因启动子区域,并用NanoDrop2000测定其浓度,将纯化后的DNA片段(0.225 pmol/L)与不同浓度纯化后的His-RstA蛋白在EMSA结合缓冲液中混合,反应的体系为20 μL,于常温下孵育30 min后加入5 μL上样缓冲液,通过6%的非变性PAGE电泳检测结合活性[17]。
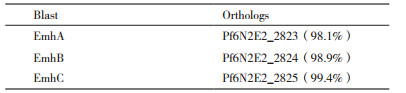
2 结果 2.1 转录因子RstA与外排泵EmhABC共适应性分析采用Cofitness Browser Database数据平台对Em-hABC的共适应蛋白进行预测分析。分别对荧光假单胞菌外排泵EmhABC不同组件EmhA、EmhB、Em-hC在该数据库中BLAST后,选择匹配度高且注释显示同为RND外排泵家族的Pf6N2E2_2823(98.1% identity)、Pf6N2E2_2824(98.9%)、Pf6N2E2_2825(99.4%)作为emhABC的功能同源蛋白[11](表 2),分别预测其共适应蛋白后结果显示:Pf6N2E2_2823、Pf6N2E2_2824、Pf6N2E2_2825三者之间互为最强共适应关系,Cofitness值及Conserved值均大于0.9;同时,三者共适应物列表中均可见Pf6N2E2_463(注释为转录因子蛋白RstA),共适应强度排名分别为9#、9#、11#(图 1-A),Cofitness值分别为0.5、0.53、0.53,且Conserved值均大于0.6。同时在Pf6N2E2_463共适应物列表中Pf6N2E2_2823、Pf6N2E2_2824、Pf6N2E2_2825分别位于列表的11#、7#、8#位,Cofitness值及Conserved值同上。对比Pf6N2E2_463与Pf6N2E2_2823、Pf6N2E2_2824、Pf6N2E2_2825在多种生长条件的适应度(Fitness)数据,发现它们的基因缺失突变株对多种有害物质胁迫环境的适应度下降,其中夫西地酸、四环素、洛美沙星、壮观霉素及氯霉素等是它们有着共同适应度改变的胁迫条件,Fitness值均在-1 - -6之间(图 1-B)。

|
| A. EmhA、EmhB、EmhC共适应列表中RstA的排序(9#,9#,11#);B. emhA、emhB、emhC及rstA突变株对不同培养条件的适应分数。 图 1 外排泵EmhABC与转录因子RstA的共适应分析 |
以荧光假单胞菌2P24基因组为检索对象,将Pf6N2E2_463基因序列BLAST后得到序列C0J56_12295,经序列比对明确C0J56_12295(rstA)是Pf6N2E2_463在荧光假单胞菌2P24中的同源序列,序列匹配度(Identity)大于90%。rstA编码序列全长702 bp,编码多肽含有233个氨基酸。RstA蛋白预测分子量为26.30 kD,理论等电点为5.53。
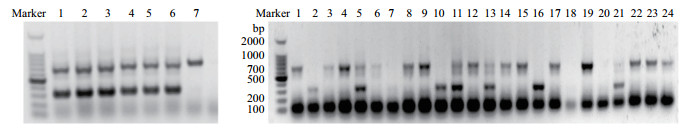
2.3 rstA基因缺失菌株的鉴定经编码框内删除的方式构建rstA基因缺失菌株(ΔrstA),框内删除后剩余序列约250 bp。如图 2(左)示单交换菌落筛选结果,即相比野生型荧光假单胞菌2P24菌株,以引物RstA-F/RstA-R扩增后分别在700 bp和250 bp显示条带,说明pK18mobsacB载体上包含的rstA编码框内删除后序列已同荧光假单胞菌2P24基因组发生第一次同源重组。随后,经10%蔗糖固体培养基筛选后得到发生第二次同源重组的菌株,如图 2(右)示双交换菌落筛选结果,即由引物RstA-F/RstA-R扩增得到两种长度的单一片段,其中若第二次同源重组后回复为野生型菌株,则条带位于700 bp;若位于250 bp附近,则表明rstA编码框内删除后序列已在基因组水平完全取代原序列,ΔrstA菌株构建成功。
|
| 图左为基因敲除时第一次同源重组成功菌株筛选,1-6均成功,7为野生型菌株;图右为基因敲除时第二次同源重组成功菌株筛选,第2,10,16,21为构建成功的△rstA菌株,其余为回复野生型菌株,第7,18,20为验证失败的样品。 图 2 rstA基因缺失菌株的构建 |
抗生素最小抑制浓度(MIC)测定显示,相较于荧光假单胞菌2P24野生型菌株,ΔrstA菌株对多种抗生素敏感性增加,其中氯霉素、卡那霉素及庆大霉素对ΔrstA的MIC值均降低4倍,强力霉素降低8倍;而氨苄霉素、四环素、洛美沙星和美罗培南对ΔrstA的MIC值下降不显著,降低2倍或不变(表 3)。可见RstA能够直接影响荧光假单胞菌2P24对多种抗生素胁迫环境的耐受。
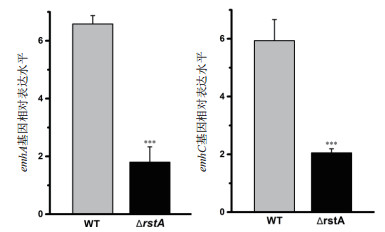
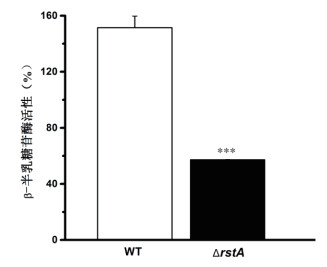
ΔrstA菌株中emhA与emhC基因的mRNA水平较野生型下降超过3倍(图 3);同样,β-半乳糖苷酶报告基因实验也显示,ΔrstA菌株中β-半乳糖苷酶的活性较野生型下降3倍(图 4)。该结果表明RstA对emhABC有正向调控作用。
|
| 图 3 qRT-PCR检测emhA、emhC基因转录水平 |
|
| 图 4 β-半乳糖苷酶实验检测emhABC表达水平 |
构建pET28b-rstA重组质粒并转化至表达宿主菌BL21中,经IPTG诱导,如图 5可见27 kD处有一明显条带,说明目标蛋白His-RstA已成功表达。经固定金属离子亲和色谱的方法对His标签蛋白纯化后得到如图所示高纯度His-RstA蛋白。

|
| 1. marker,2. 500 mM咪唑洗脱,3. 250 mM咪唑洗脱,4. 100 mM咪唑洗脱,5. 50 mM咪唑洗脱,6.过Ni柱后流穿,7.破碎后沉淀 图 5 His-RstA蛋白纯化SDS-PAGE电泳图 |
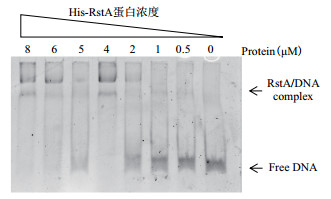
凝胶阻滞实验结果(图 6)显示:随着RstA蛋白浓度的升高,emhABC基因上游调控区内长度为0.225 pmol/L DNA片段的迁移速率逐渐降低,当蛋白质终浓度达到4 μmol/L时,反应体系中大部分DNA的迁移被阻滞,表明RstA能够特异性地结合在emhABC启动子区域。
|
| DNA:emhABC启动子序列 图 6 重组蛋白His-RstA与启动子区域结合活性 |
荧光假单胞菌2P24最早分离自山东省小麦根际周围的土壤中[18],目前大多研究主要针对其次级代谢产物2,4-二乙酰基间三苯酚的产生与调节[16, 19]、群体感应效应对其在植物根际定植的影响[20]、抗生素合成调节[21]及三型分泌系统结构功能[22]等方面展开。EmhABC是目前报道荧光假单胞菌2P24中最重要的RND家族外排泵,张立群课题组研究发现,EmhABC不仅参与调控次级代谢产物2,4-二乙酰基间三苯酚的产生,还显著影响细菌对多种毒性小分子的耐受性[9]。为了进一步明确荧光假单胞菌2P24对该重要外排泵的调控机制,我们借助Cofitness Browser数据库及序列比对,发现OmpR家族转录因子RstA同EmhABC存在共适应性,二者对多种抗生素胁迫环境的适应贡献相似。因此,我们推测RstA很可能同EmhABC位于同一调控通路。为了证实这一猜想,我们首先构建了rstA基因缺失菌株,明确RstA直接影响荧光假单胞菌2P24对多种抗生素的耐药性。之后,通过对emhA、emhC转录水平及emhABC表达水平的检测,证实RstA能够正向调控emhABC的表达,这也同共适应性预测结果及抗生素敏感性测定实验的结果相一致。
OmpR家族成员均为DNA结合蛋白,属于翼状螺旋转录因子,其蛋白折叠模式很保守,即包含有与DNA大沟相互作用的识别螺旋,及与DNA小沟接触的侧环或“侧翼”,常结合于靶基因启动子区域发挥转录调控作用[23]。OmpR家族成员几乎均为双组分系统中的反应调节蛋白(Response regulator,RR)组分,常与同源的组氨酸激酶蛋白(Histidine kinase,HK)组分一起参与微生物对外界环境的感受和应答[24]。RR常经磷酸化后发生构象改变而获得功能活性,并进一步发挥调控作用[25]。据报道,OmpR家族RR常通过发生二聚体化结合在DNA启动子区的串联重复序列,进而发挥转录激活或抑制功能[26]。
根据NCBI中的功能预测注释,RstA属于RR,为了进一步明确RstA对emhABC的调控机制,我们表达纯化出RstA蛋白,并通过凝胶阻滞实验证实RstA能够特异性地结合在emhABC上游启动子区域,表明RstA能够直接与emhABC启动子区发生相互作用,但其中更为深入的作用机制,如二者的互作位点、RstA同源HK在其中扮演的角色及RstA的效应模式是否符合OmpR家族RR的常规工作模式等仍需深入研究。当然,RstA是否还参与调控其他生理功能也需要进一步探索。观察ΔrstA与ΔemhABC相较于野生型对各抗生素的敏感性变化不难发现,二者的抗性变化谱存在一定差异,因此我们推断RstA很可能还通过调控其他外排泵影响细菌的多重耐药性。
4 结论本研究以荧光假单胞菌2P24为研究对象,发现了一个新的OmpR家族转录因子RstA并对其进行功能鉴定。结果表明,RstA通过结合在emhABC上游启动子区域正向调控emhABC的表达,并可能经多个途径影响荧光假单胞菌2P24的多重耐药性。
| [1] |
Peix A, Ramírez-Bahena MH, Velázquez E. The current status on the taxonomy of Pseudomonas revisited:an update[J]. Infection, Genetics and Evolution, 2018, 57: 106-116. DOI:10.1016/j.meegid.2017.10.026 |
| [2] |
Scales BS, Dickson RP, LiPuma JJ, et al. Microbiology, genomics, and clinical significance of the Pseudomonas fluorescens species complex, an unappreciated colonizer of humans[J]. Clinical Microbiology Reviews, 2014, 27(4): 927-948. |
| [3] |
Goldberg JB, Hancock RE W, Parales RE, et al. Pseudomonas 2007[J]. Journal of Bacteriology, 2008, 190(8): 2649-2662. DOI:10.1128/JB.01950-07 |
| [4] |
Haas D, Défago G. Biological control of soil-borne pathogens by fluorescent pseudomonads[J]. Nature Reviews Microbiology, 2005, 3(4): 307-319. DOI:10.1038/nrmicro1129 |
| [5] |
Weller DM, Landa BB, Mavrodi OV, et al. Role of 2, 4-diacetylphlo-roglucinol-producing fluorescent pseudomonas spp. in the defense of plant roots[J]. Plant Biology, 2007, 9(1): 4-20. DOI:10.1055/s-2006-924473 |
| [6] |
Kittinger C, Lipp M, Baumert R, et al. Antibiotic resistance patterns of Pseudomonas spp. isolated from the River Danube[J]. Frontiers in Microbiology, 2016, 7: 586. |
| [7] |
Zhou Y, Xu YB, Xu JX, et al. Combined toxic effects of heavy metals and antibiotics on a Pseudomonas fluorescens strain Zy2 isolated from Swine wastewater[J]. International Journal of Molecular Sciences, 2015, 16(2): 2839-2850. DOI:10.3390/ijms16022839 |
| [8] |
Martinez JL, Sánchez MB, Martínez-Solano L, et al. Functional role of bacterial multidrug efflux pumps in microbial natural ecosystems[J]. FEMS Microbiology Reviews, 2009, 33(2): 430-449. DOI:10.1111/j.1574-6976.2008.00157.x |
| [9] |
Tian T, Wu XG, Duan HM, et al. The resistance-nodulation-division efflux pump EmhABC influences the production of 2, 4-diacetylphloroglucinol in Pseudomonas fluorescens 2P24[J]. Microbiology, 2010, 156(1): 39-48. |
| [10] |
Wetmore KM, Price MN, Waters RJ, et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons[J]. MBio, 2015, 6(3): e00306-15. |
| [11] |
Price MN, Wetmore KM, Waters RJ, et al. Mutant phenotypes for thousands of bacterial genes of unknown function[J]. Nature, 2018, 557(7706): 503-509. DOI:10.1038/s41586-018-0124-0 |
| [12] |
Zhao C, Zhao J, Wang W, et al. Expression of MLAA34-HSP70 fusion gene constructed by SOE-PCR[J]. Pakistan Journal of Pharmaceutical Sciences, 2017, 30: 1125-1127. |
| [13] |
King EO, Ward MK, Raney DE. Two simple media for the demonstration of pyocyanin and fluorescin[J]. The Journal of Laboratory and Clinical Medicine, 1954, 44(2): 301-307. |
| [14] |
Schwalbe R, Steele-Moore L, Goodwin AC, . Antimicrobial susceptibility testing protocols[M]. Boca Raton: CRC Press, 2007.
|
| [15] |
Yan Q, Wu XG, Wei HL, et al. Differential control of the PcoI/PcoR quorum-sensing system in Pseudomonas fluorescens 2P24 by sigma factor RpoS and the GacS/GacA two-component regulatory system[J]. Microbiological Research, 2009, 164(1): 18-26. DOI:10.1016/j.micres.2008.02.001 |
| [16] |
Yan X, Yang R, Zhao RX, et al. Transcriptional regulator PhlH modulates 2, 4-diacetylphloroglucinol biosynthesis in response to the biosynthetic intermediate and end product[J]. Applied and Environmental Microbiology, 2017, 83(21): e01419-17. |
| [17] |
Hellman LM, Fried MG. Electrophoretic mobility shift assay(EMSA)for detecting protein-nucleic acid interactions[J]. Nature Protocols, 2007, 2(8): 1849-1861. DOI:10.1038/nprot.2007.249 |
| [18] |
魏海雷, 张力群. 荧光假单胞杆菌2p24中生防相关调控基因gacS的克隆和功能分析[J]. 微生物学报, 2005, 45(3): 368-272. DOI:10.3321/j.issn:0001-6209.2005.03.011 |
| [19] |
Zhang W, Zhao Z, Zhang B, et al. Posttranscriptional regulation of 2, 4-diacetylphloroglucinol production by GidA and TrmE in Pseudomonas fluorescens 2P24[J]. Applied and Environ Microbiology, 2014, 80(13): 3972-3981. DOI:10.1128/AEM.00455-14 |
| [20] |
Wei HL, Zhang LQ. Quorum-sensing system influences root colonization and biological control ability in Pseudomonas fluorescens 2P24[J]. Antonie Van Leeuwenhoek, 2006, 89(2): 267-280. DOI:10.1007/s10482-005-9028-8 |
| [21] |
Li X, Gu GQ, Chen W, et al. The outer membrane protein OprF and the sigma factor SigX regulate antibiotic production in Pseudomonas fluorescens 2P24[J]. Microbiological Research, 2018, 206: 159-167. DOI:10.1016/j.micres.2017.10.006 |
| [22] |
Liu P, Zhang W, Zhang LQ, et al. Supramolecular structure and functional analysis of the type Ⅲ secretion system in Pseudomonas fluorescens 2P24[J]. Frontiers in Plant Science, 2016, 6: 1190. |
| [23] |
Martinez-Hackert E, Stock AM. The DNA-binding domain of OmpR:crystal structures of a winged helix transcription factor[J]. Structure, 1997, 5(1): 109-124. |
| [24] |
Stock AM, Robinson VL, Goudreau PN. Two-component signal transduction[J]. Annual Review of Biochemistry, 2000, 69(1): 183-215. |
| [25] |
Groisman EA. Feedback control of two-component regulatory systems[J]. Annual Review of Microbiology, 2016, 70(1): 103-124. |
| [26] |
Li YC, Chang CK, Chang CF, et al. Structural dynamics of the two-component response regulator RstA in recognition of promoter DNA element[J]. Nucleic Acids Research, 2014, 42(13): 8777-8788. DOI:10.1093/nar/gku572 |