2. 曲靖师范学院 生物资源与食品工程学院, 曲靖 655011
2. College of Biological Resource and Food Engineering, Qujing Normal University, Qujing 655011
能源植物小桐子是目前世界上最主要的生物柴油植物之一, 我国在“十五”、“十一五”、“十二五”能源发展纲要中都把发展小桐子产业列为可再生能源中长期发展规划的重点[1]。小桐子(Jatropha curcas L.)属大戟科(Euphorbiaceae)麻疯树属(Jatropha)多年生落叶半肉质小乔木或大灌木[2]。原产中南美洲地区, 目前在我国滇、黔、川、桂、闽、粤及琼等省份有较多野生资源分布[3]。其种子含油量高, 且流动性与石化油掺和性好, 在闪点、凝固点、含硫量等关键技术指标上达到了欧四标准, 成为未来最有可能替代化石能源的潜力树种, 具有广阔的开发与利用前景。
蔗糖非发酵-1型相关蛋白激酶家族(Sucrose non-fermenting-1 related protein kinase, SnRK)属丝氨酸/苏氨酸(Ser/Thr)类蛋白激酶超家族(Protein kinase Superfamily), 通过共价修饰调节(Covalent modification)广泛参与植物细胞分裂、物质代谢、基因表达调控及生物与非生物抗逆响应等过程[4]。根据蛋白质序列聚类结果将SnRK家族分为SnRK1、SnRK2、SnRK3三个亚家族[5]。其中, SnRK1与酵母SNF1(Sucose non-fermenting 1)和哺乳动物AMPK(AMP-activated protein kinase)在结构与功能上同源性较高, 共同组成SNF1蛋白激酶家族[6], 负责感知并响应胞内低糖/低能量状态(Energy-sensing protein kinase family)。根据氨基酸序列的相似性与表达模式, SnRK1可进一步分为SnRK1a与SnRK1b两类[7], 其中, SnRK1a在所有植物中均有表达, 而SnRK1b仅存在于单子叶植物中, 且具有种子高表达特异性[5]。在植物中, SnRK1存在于信号转导的中间部位, 本身受到上游因子磷酸化调节, 又可以通过磷酸化调节下游诸多关键蛋白, 如α-淀粉酶(α-amylase, α-AMS)[8]、磷酸蔗糖合成酶(Sucrose phosphate synthase, SPS)[5]、6-磷酸海藻糖合成酶(Trehalose-6-phosphate synthase, TPS)[9]、3-羟基-3-甲基-戊二酸单酰CoA还原酶(3-hydroxy-3-methylglutaryl coenzyme-A reductase, HMGR)[10]、硝酸还原酶(Nitrate reductase, NR)[11]、天冬酰胺合成酶(Asparagine synthetase1, ASN1/DIN6)、脯氨酸脱氢酶(Proline dehydrogenase, ProDH)[12]等, 从而广泛参与糖代谢及糖信号应答反应(响应高蔗糖/低葡萄糖信号)[13]、蔗糖合成、淀粉合成与降解[14]、生长发育[5]及逆境应答[15-16]等生理生化过程。
SnRK1蛋白激酶是由α、β、γ三个亚基组成的异源三聚体, 其中催化亚基α具有保守的激酶结构域及核心序列。第一个被克隆的SnRK1 α亚基基因来自黑麦胚乳cDNA文库, 其编码57.7kD的蛋白质[17], 而对于小桐子SnRK1蛋白激酶α亚基基因的鉴定、克隆及表达研究还未见报道。本研究基于小桐子基因组信息鉴定到1个小桐子SnRK1蛋白激酶α亚基基因, 与拟南芥AKIN10同源[18-22], 克隆到了该基因的全长cDNA序列, 并对其进行了生物信息学分析、器官与低温胁迫差异表达及原核表达分析, 为研究小桐子SnRK1蛋白激酶在糖信号转导中的功能及抗逆性形成的机制提供参考。
1 材料与方法 1.1 材料实验所用小桐子种子采自云南省元谋县干热河谷地区。选取饱满的小桐子种子, 用1.0%CuSO4消毒20 min, 无菌水漂洗5次, 置于三角瓶中于26℃培养箱中吸涨24 h, 之后无菌水中漂洗3次, 播于用无菌水湿润滤纸的托盘中, 于相对湿度75%、温度26℃、16/8 h光周期的恒温培养箱中萌发5 d。将发芽的种子种植于消毒的培养土中并于同上条件的恒温培养箱中生长15 d至第2片真叶展开, 每天用无菌水浇灌培养土。将生长15 d的小桐子幼苗置于相对湿度75%、温度12℃、16/8 h光周期的低温培养箱中进行低温处理, 分别取低温胁迫0.5、3、12 h与对照(CK, 正常培养)的第二片真叶、茎及根, 液氮速冻后保存于-80℃冰箱中。
1.2 方法 1.2.1 小桐子JcSnRK1α基因的克隆SnRK1α与酵母SNF1和哺乳动物AMPK同源。从GenBank下载酵母SNF1(AAA35058.1)、人类AMPK(AAB32732.1)、植物中同源基因拟南芥AKIN1.3(NP_198760.1)、AKIN10(AAA32736.1)、AKIN11(BAB01993.1)、水稻OSK1(BAA36298.1)、OSK24(BACA56589.1)、黑麦(AAA33921.1)及烟草(BAA05649.1)的SnRK1α蛋白序列, 以上述序列为种子序列, 利用NCBI Blast程序对小桐子蛋白质数据库进行BlastP相似性检索(阈值E < 1e-40, 序列相似性 > 50%), 得到候选的小桐子SnRK1α蛋白质序列XP_012084751.1, 同时, 下载其对应基因序列(NW_012124717.1)与mRNA序列(XM_01222936.1)。以mRNA序列为基础设计全长扩增引物(JcSnRK1α_C), 上游:5′-CTCTTTGAGCTCGATTGTGTCCATC-3′; 下游:5′-GAAGGACAGTTATTTACACCGTG-3′, 菌落PCR验证引物(JcSnRK1α_T), 上游:5′-CCTGGTCATCATGAAGG-3′; 下游:5′-CCGACGTCGACAAGGACTCGGAGTTGTGCAAGAAAAG-3′, 并送深圳华大基因公司合成。
利用TriZol法提取小桐子对照与不同低温处理下的叶片、茎及根的总RNA, 并利用DNase I(TaKaRa公司)消化RNA中的残余基因组DNA, 得到纯化的总RNA。分别取1.5 μg总RNA, 以Random primer为逆转录引物, 利用PrimeScriptTM RT reagent Kit with gDNA Eraser(TaKaRa公司)合成第一链cDNA。以茎cDNA为模板, 使用高保真DNA聚合酶KOD FX Neo DNA Polymerase(TOYOBO公司)及JcSnRK1α_C引物进行PCR扩增, 扩增条件为:98℃预变性5 min; 98℃变性10 s, 60℃退火30 s, 68℃延伸2 min, 30个循环, 68℃后延伸5 min。电泳检测正确的PCR扩增产物20 μL, 加1 μL Taq DNA Polymerase(TaKaRa公司), 72℃反应25 min进行加A处理, 利用Gel Extraction Kit(OMEGA公司)回收目的基因条带。将回收基因片段与克隆载体pMD18-T连接, 命名为pMD18-T-JcSnRK1α, 转化大肠杆菌DH5α感受态细胞, 涂LB抗性平板(包含50 mg/L Amp), 过夜生长, 进行蓝白斑筛选。利用引物JcSnRK1α_T进行菌落PCR验证的阳性克隆, 送深圳华大基因公司进行双向测序。
1.2.2 小桐子JcSnRK1α基因的序列分析利用ExPaSy提供的在线工具ProtParam(http://web.expasy.org/protparam/)对得到的小桐子JcSnRK1α蛋白序列进行氨基酸数目、理论分子量、等电点等基本参数的分析。将获得的小桐子JcSnRK1α cDNA与基因序列利用软件Splign进行比对以确定基因内含子与外显子的结构, 并利用GSDS(Gene Structure Display Server, http://gsds.cbi.pku.edu.cn/)绘制基因结构图。利用GenBank CDD(Conserved domains)在线分析工具对JcSnRK1α进行结构域分析。根据JcSnRK1α的编码区序列(Coding sequence, CDS)对小桐子基因组进行Blastn相似性检索, 得到JcSnRK1α基因起始密码子ATG上游1 500 bp的调控序列, 并通过PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对其顺式作用元件进行鉴定。将克隆到的小桐子JcSnRK1α基因的蛋白序列与其他物种SnRK1α同源序列利用ClustalX 2.0进行序列相似性比对, 然后用MEGA 6.0软件通过邻接法(NJ)构建系统进化树。
1.2.3 小桐子JcSnRK1α基因的表达分析利用小桐子各组织及低温处理材料的cDNA为模板, 以18S rRNA为内参基因进行JcSnRK1α基因的实时荧光定量PCR(Quantitative real-time PCR, qRT-PCR)表达分析。引物序列为:18S rRNA上游:5′-AGAAACG-GCTACCACATC-3′、下游:5′-CCAAGGTCCAACTA-CGAG-3′, JcSnRK1α(JcSnRK1α_Q)上游:5′-GAC-AACCGATTCCGTGTT-3′、下游:5′-GAAGTCCAAG-TGCCCATT-3′。20 μL反应体系, 每个样品重复3次, 仪器为LightCycler96(Roche公司)。扩增条件为:94℃预变性30 s; 94℃变性10 s, 53℃退火15 s, 72℃延伸15 s, 45个循环, 之后增加溶解曲线程序:95℃ 10 s, 65℃ 60 s, 97℃ 1 s, 连续检测信号。采用2-ΔΔCt方法进行基因差异表达分析。器官差异表达分析以叶片的表达量为基准, 低温差异表达分析以对照(CK)的表达量为基准。
1.2.4 原核表达载体的构建与目的蛋白的诱导表达设计带酶切位点的引物JcSnRK1α_E, 上游:5′- CGGGATCCATGGATGGGTCAAATCACCGAAGCAG -3′(下划线表示BamH I酶切位点)、下游:5′- CCGCTCGAGCTAAAGGACTCGGAGTTGTGC -3′(下划线表示Xho I酶切位点), 扩增小桐子JcSnRK1α基因的全长编码框序列, 扩增产物与原核表达载体pGEX-4T-1经BamH I与Xho I双酶切, 切胶回收后利用T4 DNA连接酶16℃过夜连接, 获得重组质粒pGEX-4T-1-JcSnRK1α, 将其转化大肠杆菌DH5α感受态细胞, 涂布LB抗性平板(含50 mg/L的Amp), 37℃过夜生长。经BamH I与Xho I双酶切验证的阳性克隆, 送深圳华大基因公司进行测序。
将构建的小桐子原核表达载体pGEX-4T-1-JcSnRK1α转化大肠杆菌Rosetta菌株, 同时转化pGEX-4T-1空载体作为对照, 挑取阳性单克隆接种至5 mL LB液体培养基中(含50 mg/L的Amp), 37℃ 200 r/min过夜振荡培养。之后按体积比1:100接种于100 mL LB液体培养基中(含50 mg/L的Amp), 37℃ 200 r/min振荡培养至OD600为0.4-0.6, 取1 mL菌液作为诱导前对照, 4℃保存。加入IPTG至终浓度为1 mmol/L, 37℃诱导表达, 分别取诱导1、3、6、12 h的菌液于4℃保存备用。将未诱导与诱导不同时间的菌液12 000 r/min离心5 min, 弃上清, 在菌体沉淀中加入200 μL PBS缓冲液(pH值7.4)重悬浮, 加入50 μL 5×SDS上样缓冲液, 100℃煮沸10 min, 冷却至室温, 12 000 r/min离心5 min, 取20 μL上清蛋白样品上样, 进行SDS-PAGE电泳(5%浓缩胶, 12%分离胶), 考马斯亮蓝染色检测蛋白质的表达情况。
2 结果 2.1 小桐子JcSnRK1α基因的克隆以小桐子茎cDNA为模板, 扩增JcSnRK1α基因cDNA全长序列。测序结果(图 1)表明, 克隆的小桐子JcSnRK1α基因全长1 700 bp。ORF finder分析表明其包含完整的开放阅读框1 545 bp, 编码514个氨基酸残基, 结合基因序列分析显示, 在5′与3′端分别包含294 bp与407 bp的5′-UTR与3′-UTR非编码区。

|
| 注:起始(ATG)与终止(TAG)密码子用下划线标注 图 1 小桐子JcSnRK1α基因的cDNA与推导的氨基酸序列 |
将cDNA序列与基因序列(NW_012124717.1)通过Splign进行比对显示(图 2-A), 小桐子JcSnRK1α基因包含11个外显子与10个内含子, 且3′端最后一个外显子与3′-UTR区域重合。ProtParam计算结果表明, 小桐子JcSnRK1α蛋白分子量为58.8 kD, 理论等电点为8.57, 包含较多的非极性氨基酸, 其中Leu、Ile、Val的比例占24.5%。

|
| A:小桐子JcSnRK1α基因内含子/外显子结构, 黑色框代表外显子, 线条表示内含子, 灰色框代表非编码UTR区域; B:小桐子JcSnRK1α蛋白保守结构域分析; C、小桐子JcSnRK1α基因的顺式作用元件鉴定 图 2 小桐子JcSnRK1α的基因结构、结构域及顺式作用元件分析 |
进一步通过GenBank CDD程序对小桐子JcSnRK1α氨基酸序列进行功能结构域分析, 确定其属于小桐子SnRK1蛋白的α亚基, 从N端到C端依次鉴定到激酶结构域(Kinase domain)、自抑制调控结构域(Ubiquitin-associated domain, UBA)及激酶相关结构域1(Kinase associated domain1, KA1)/β-亚基结合结构域(β-subunit interaction action, β-SID)(图 2-B)。
将其激酶结构域与其他物种进行比对发现, 除酵母(Saccharomyces cerevisiae)以外, 都包含约250 aa氨基酸残基, 序列相似性达到60%以上, 其中, 在激酶结构域内部, 还鉴定到ATP结合基序(分布于30-160 aa之间)与保守的磷酸化T-loop区域(分布于160-190 aa之间)(图 3)。克隆的小桐子SnRK1α激酶结构域位于16-271位(长度256 bp), T-loop区域位于160-186位(核心Thr175残基), 同时, ATP结合基序中的保守Lys残基对于SnRK1α结合ATP也是必需的, 本研究鉴定的小桐子SnRK1α中保守的Lys残基位于48位。另外, UBA结构域与KA1结构域分别位于293-333位与390-511位, 与拟南芥KIN10的各结构域位置基本一致。

|
| 三角形表示ATP结合基序(30-160 aa)的保守Lys残基, 虚线表示T-loop区域(160-190 aa), 箭头表示磷酸化的保守Thr残基 图 3 小桐子JcSnRK1α及同源蛋白激酶结构域序列比对 |
SnRK1α与酵母SNF1、哺乳动物AMPK同属于SNF1家族激酶, 进一步将小桐子SnRK1的T-loop区域与其他物种进行序列比对并构建系统进化树, 结果(图 4)显示, 同属双子叶植物的拟南芥、烟草及小桐子其T-loop区域序列完全相同, 而单子叶植物的水稻与黑麦存在2个氨基酸残基的差异, SNF1家族的人类AMPK与酵母SNF1单独聚类为一个分支, 酵母SNF1在进化上更为古老, 且与其他物种的序列差异性最大, 但所有物种T-loop区域中的保守Thr残基及其两侧氨基酸完全一致, 是SnRK1α家族蛋白受上游信号调控所必需的。

|
| 图 4 小桐子JcSnRK1α及同源蛋白T-loop区域序列比对, 箭头表示保守的Thr残基 |
SnRK1α是与低能量逆境相关的基因, 对小桐子JcSnRK1α基因上游1 500 bp的调控序列通过PlantCARE在线程序进行顺式作用元件的分析, 发现了基因启动子、增强子区域常见的作用元件, 如辅助RNA聚合酶Ⅱ结合的位点TATA-box, 以及控制转录起始频率并增强转录活性的位点CAAT-box。另外, 还鉴定到不同的防御与非生物胁迫逆境响应元件如创伤(WUN-motif, AAATTTCCT)、高温(HSE, AAAAAATTC)、低温(LTR, CCGAAA)及干旱MYB元件(MBS, TAACTG)等, 以及赤霉素应答元件(GARE-motif, AAACAGA)、类黄酮合成基因(Flavonoid biosynthetic gene)响应元件(MBSI, AAAAAACG/CGTTA)(图 2-C), 说明小桐子JcSnRK1α基因的表达受多重信号系统的调节。
2.3 小桐子JcSnRK1α基因的差异表达分析在小桐子JcSnRK1α基因的启动子序列中鉴定到了低温、高温、干旱及创伤等非生物胁迫逆境响应元件(图 2-C), 通过qRT-PCR技术对JcSnRK1α基因在小桐子不同器官与低温胁迫处理下的表达特性进行了研究, 结果(图 5-A)表明, JcSnRK1α基因在小桐子各器官中都有表达, 但存在表达特异性, 其中, 在茎中表达量较高(较叶片中高31.05倍), 其次为根, 而在叶片中表达量较低。

|
| A:JcSnRK1α基因的器官差异表达特性; B与C分别表示:JcSnRK1α基因在茎与根中的低温胁迫表达特性, ##与#表示相对于叶片表达量在P < 0.01、P < 0.05水平的差异显著性, **与*表示相对于对照表达量在P < 0.01、P < 0.05水平的差异显著性 图 5 小桐子JcSnRK1α基因的器官与低温胁迫差异表达分析 |
低温胁迫条件下, 小桐子JcSnRK1α基因在茎与根中表现出相似的表达变化趋势, 与对照相比, 在茎中, 在低温胁迫3 h达到最大表达量, 较对照提高2.04倍(P < 0.01), 之后表达量迅速下降, 且低于对照的表达水平(图 5-B); 而在根中, 在低温胁迫0.5 h出现表达峰值, 较对照提高3.16倍(P < 0.01), 之后表达量逐渐下调, 且在低温胁迫12 h时回到对照表达水平(图 5-C), 说明JcSnRK1α基因在根中更快速响应低温胁迫。
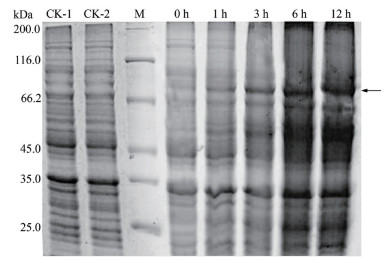
2.4 小桐子JcSnRK1α原核表达载体的构建与蛋白表达将大肠杆菌Rosetta菌株、转pGEX-4T-1空载体与pGEX-4T-1-JcSnRK1α重组载体的菌液裂解后进行SDS-PAGE表达蛋白的电泳分析。结果(图 6)表明, 大肠杆菌Rosetta菌株(CK-1)、转pGEX-4T-1空载体(CK-2)及转pGEX-4T-1-JcSnRK1α重组载体未诱导(0 h)的菌株都未出现相应分子量的条带。而经过IPTG诱导1、3、6、12 h的菌株都出现1条约84.8 kD的蛋白条带(pGEX-4T-1载体的表达标签为26.0 kD, 小桐子JcSnRK1α蛋白预测分子量为58.8 kD), 与理论融合蛋白的分子量一致, 且随着诱导时间的延长, 蛋白表达量逐渐增加, 表明JcSnRK1α基因已经在大肠杆菌中成功表达。
|
| CK-1表示大肠杆菌Rosetta菌株, CK-2表示转空pGEX-4T-1表达载体, 0h表示转重组表达载体未诱导, 1 h、3 h、6 h、12 h表示转重组表达载体分别诱导1、3、6、12 h, M表示蛋白质Marker 图 6 小桐子JcSnRK1α基因表达蛋白的SDS-PAGE电泳 |
SnRK1的活性主要受到细胞能量水平的调节, 但其是否像AMPK一样受到AMP的激活, 还不太清楚[11]。而6-磷酸海藻糖(Trehalose-6-phosphate, T6P)是SnRK1的抑制剂, 海藻糖的代谢与SnRK1感应低糖信号之间有重要的联系[23], 并且T6P还需要其他介导因子参与完成SnRK1的抑制过程[24], 另外, 其他报道显示, SnRK1的活性还受到Ca2+[25]、6-磷酸葡萄糖(Glucose-6-phosphate)[26], 以及GRIK1/2(Geminivirus Rap Interacting kinase 1/2)的调节[27], 其中GRIKs被认为是磷酸化SnRK1α T-loop中保守Thr残基的核心蛋白激酶[19]。而在小桐子的SnRK1α基因启动子中还鉴定到类黄酮合成基因响应相关顺式作用元件(图 2C), 表明其还受到类黄酮类化合物的调节。
植物中SnRK1是糖代谢网络、依赖或不依赖miRNA的能量代谢[28-29]、糖信号转导、ABA信号转导的关键因子, 能够调节编码糖代谢关键基因的时空表达特性, 同时也可通过磷酸化特定SAMS序列(-HMRSAMSGLHLVKR-)的Ser残基调节一系列酶的活性[30]。本研究对小桐子全蛋白质数据库进行相似性检索, 也鉴定到包含SAMS序列的糖代谢相关酶, 如D-3-磷酸甘油酸脱氢酶(D-3-phosphoglycerate dehydrogenase, XP_012068288.1)、焦磷酸:果糖-6-磷酸1-磷酸转移酶(Pyrophosphate:fructose 6-phosphate 1-phosphotransferase, XP_012073552.1)、木糖葡聚糖葡基转移酶(Xyloglucan glycosyltransferase, XP_012085176.1), 也证明SnRK1与小桐子的糖代谢信号网络密切相关。
另外, SnRK1对植物发育与抗逆相关基因表达也有影响。反义表达SnRK1α的豌豆植株表现出了多种成熟缺陷, 如淀粉含量降低、花粉粒变小以及败育等[30-31], Halford等推测是由于转化酶(Inverase)不能被激活, 导致不能利用摄入的蔗糖, 最终花粉粒因不能响应“高蔗糖/低葡萄糖”能量状态信号, 从而在含有蔗糖的培养基上饥饿而败育[5, 7, 32-33], 同时, 在马铃薯中反义表达GAI83(SnRK1复合体的β亚基)的同源基因StubGAI83, 导致转基因植株对高盐敏感, 并且影响主根的生长[15], 相反, 在烟草中过表达SnRK1α基因可明显提高烟草对病毒的抗性[16]。本研究在小桐子SnRK1α基因的启动子中也鉴定到大量的逆境响应元件如低温响应元件, qRT-PCR实验也验证了该基因参与小桐子的低温胁迫响应过程, 与小桐子抗冷性的形成直接相关, 但该基因在根中响应低温胁迫(0.5 h)显著快于茎(3 h)中, 且上调表达量也高于茎中, 暗示SnRK1α基因在根中响应低温胁迫更加敏感, 同时, 在茎中, 随着低温胁迫的持续, SnRK1α基因的表达量迅速下调, 且显著低于对照表达水平, 推测该基因在茎中受到更多上游信号分子的表达调节作用。以上结果表明SnRK1处于调控网络的核心位置, 受到上游信号分子如激酶的调节, 同时通过磷酸化/去磷酸化共价修饰调节参与感知细胞能量状态转录因子的信号放大过程、碳氮代谢关键基因的表达与活性调节过程以及抗逆信号转导过程等, 为小桐子SnRK1α基因在其他生物与非生物逆境胁迫下的差异表达分析与异源转化功能验证等后续研究工作奠定了基础。
4 结论本研究克隆到小桐子JcSnRK1α基因1 700 bp cDNA, 包含完整开放阅读框1 545 bp, 编码514个氨基酸。从N端到C端具备激酶结构域、自抑制调控结构域及激酶相关结构域。该基因启动子区域鉴定到低温等非生物胁迫应答元件, 参与小桐子抗冷性过程。表达分析显示, JcSnRK1α在小桐子茎与根中表达量较高, 且都属于低温诱导上调表达基因。另外, 构建该基因的原核表达载体并成功诱导表达, 得到84.8 kDa的融合蛋白条带。
| [1] |
Makkar HPS, Becker K. Jatropha curcas, a promising crop for the generation of biodiesel and value-added coproducts[J]. European Journal of Lipid Science and Technology, 2009, 111(8): 773-787. DOI:10.1002/ejlt.v111:8 |
| [2] |
Yang CY, Fang Z, Li B, et al. Review and prospects of Jatropha biodiesel industry in China[J]. Renewable & Sustainable Energy Reviews, 2012, 16(4): 2178-2190. |
| [3] |
林娟, 周选围, 唐克轩, 等. 麻疯树植物资源研究概况[J]. 热带亚热带植物学报, 2004, 12(3): 285-290. DOI:10.3969/j.issn.1005-3395.2004.03.018 |
| [4] |
Bohnert HJ, Nelson DE, Jensen RG. Adaptation to environmental stresses[J]. The Plant Cell, 1995, 7(7): 1099-1111. DOI:10.2307/3870060 |
| [5] |
Halford NG, Hey R, et al. Metabolic signalling and carbon partitioning:role of Snf1-related(SnRK1)protein kinase[J]. Journal of Experimental Botany, 2003, 54(382): 467-475. DOI:10.1093/jxb/erg038 |
| [6] |
Honigberg SM, Lee RH. Snf1 kinase connects nutritional pathways controlling meiosis in Saccharomyces cerevisiae[J]. Molecular Cell Biology, 1998, 18(8): 4548-4555. DOI:10.1128/MCB.18.8.4548 |
| [7] |
Halford NG, Hey S, et al. Highly conserved protein kinases involved in the regulation of carbon and amino acid metabolism[J]. Journal of Experimental Botany, 2004, 55(394): 35-42. |
| [8] |
Laurie S, McKibbin RS, Halford NG. Antisense SNF1-related(SnRK1)protein kinase gene represses transient activity of an a-amylase(α-AMY2)gene promoter in cultured wheat embryos[J]. Journal of Experimental Botany, 2003, 54(383): 739-747. DOI:10.1093/jxb/erg085 |
| [9] |
Harthill JE, Meek SE, Morrice N, et al. Phosphorylation and 14-3-3 binding of Arabidopsis trehalose-phosphate synthase 5 in response to 2-deoxyglucose[J]. The Plant Journal, 2006, 47(2): 211-223. DOI:10.1111/tpj.2006.47.issue-2 |
| [10] |
Dale S, Arro M, Becerra B, et al. Bacterial expression of the catalytic domain of 3-hydroxy-3-methylglutaryl-CoA reductase(isoform HMGR1)from Arabidopsis thaliana, and its inactivation by phosphorylation at Ser577 by Brassica oleracea 3-hydroxy-3-methylglutaryl-CoA reductase kinase[J]. European Journal of Biochemistry, 1995, 233(2): 506-513. DOI:10.1111/ejb.1995.233.issue-2 |
| [11] |
Sugden C, Donaghy PG, Halford NG, et al. Two SNF1-related protein kinases from spinach leaf phosphorylate and inactivate 3-hydroxy-3-methylglutaryl-coenzyme A reductase, nitrate reductase, and sucrose phosphate synthase in vitro[J]. Plant Physiology, 1999, 120(1): 257-274. DOI:10.1104/pp.120.1.257 |
| [12] |
Hanson J, Hanssen M, Wiese A, et al. The sucrose regulated transcription factor bZIP11 affects amino acid metabolism by regulating the expression of asparagine synthetase 1 and proline dehydrogenase 2[J]. Plant Journal, 2008, 53(6): 935-949. |
| [13] |
Holappa LD, Walker-Simmons MK, Ho THD, et al. A Triticum tauschii protein kinase related to wheat PKABA1 is associated with ABA signaling and is distributed between the nucleus and cytosol[J]. Journal of Cereal Science, 2005, 41(3): 333-346. DOI:10.1016/j.jcs.2004.11.002 |
| [14] |
Geigenberger P. Regulation of sucrose to starch conversion in growing potato tubers[J]. Journal of Experimental Botany, 2003, 54(328): 457-465. |
| [15] |
Lovas A, Bimbo A, Szabo L, et al. Antisense repression of StubGAL83 affects root and tuber development in potato[J]. The Plant Journal, 2003, 33(1): 139-147. |
| [16] |
Hao L, Wang H, Sunter G, et al. Geminivirus AL2 and L2 proteins interact with and inactivate SNF1 kinase[J]. The Plant Cell, 2003, 15(4): 1034-1048. DOI:10.1105/tpc.009530 |
| [17] |
Alderson A, Sabelli PA, Dickinson JR, et al. Complementation of snf1, a mutation affecting global regulation of carbon metabolism in yeast, by a plant protein kinase cDNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 1991, 88(19): 8602-8605. DOI:10.1073/pnas.88.19.8602 |
| [18] |
Tome F, Nagele T, Adamo M, et al. The low energy signaling network[J]. Frontiers in Plant Science, 2014, 5(5): 353. |
| [19] |
Crozet P, Margalha L, Confraria A, et al. Mechanisms of regulation of SNF1/AMPK/SnRK1 protein kinases[J]. Frontiers in Plant Science, 2014, 5(2): 190. |
| [20] |
Ghillebert R, Swinnen E, Wen J, et al. The AMPK/SNF1/SnRK1 fuel gauge and energy regulator:structure, function and regulation[J]. The FEBS Journal, 2011, 278(21): 3978-3990. DOI:10.1111/j.1742-4658.2011.08315.x |
| [21] |
Jiang R, Carlson M. The Snf1 protein kinsae and its activating subunit Snf4 interact with distinct domains of the Sip1/Sip2/Gal83 component in the kinase complex[J]. Molecular Cell Biolology, 1997, 17(4): 2099-2106. DOI:10.1128/MCB.17.4.2099 |
| [22] |
Baena-Gonzalez E, Rolland F, Thevelein JM, et al. A central integrator of transcription networks in plant stress and energy signaling[J]. Nature, 2007, 448(7156): 938-942. DOI:10.1038/nature06069 |
| [23] |
Tsai AYL, Gazzarrini S. Trehalose-6-phosphate and SnRK1 kinases in plant development and signaling:the emerging picture[J]. Frontiers in Plant Science, 2014, 5(5): 119. |
| [24] |
Lunn JE, Delorge I, Figueroa C, et al. Trehalose metabolism in plants[J]. Plant Journal for Cell & Molecular Biology, 2014, 79(4): 544-567. |
| [25] |
Lu CA, Lin C, Wei K, et al. The SnRK1A protein kinase plays a key role in sugar signaling during germination and seedling growth of rice[J]. Plant Cell, 2007, 19(8): 2484-2499. DOI:10.1105/tpc.105.037887 |
| [26] |
Nunes C, Primavesi LF, Patel MK, et al. Inhibition of SnRK1 by metabolites:tissue-dependent effects and cooperative inhibition by glucose 1-phosphate in combination with trehalose 6-phosphate[J]. Plant Physiology and Biochemistry, 2013, 63(4): 89-98. |
| [27] |
Shen W, Reyes MI, et al. Arabidopsis protein kinases GRIK1 and GRIK2 specifically activate SnRK1 by phosphorylating its activa-tion Loop[J]. Plant Physiology, 2009, 150(2): 996-1005. DOI:10.1104/pp.108.132787 |
| [28] |
Tsai AYL, Gazzarrini S. AKIN10 and FUSCA3 interact to control lateral organ development and phase transitions in Arabidopsis[J]. Plant Journal for Cell & Molecular Biology, 2012, 69(5): 809-821. |
| [29] |
Confraria A, Martinho C, Elias A, et al. miRNAs mediate SnRK1-dependent energy signalling in Arabidopsis[J]. Frontiers in Plant Science, 2013, 4(1): 197. |
| [30] |
Machintosh RW, Davies SP, Clarike PR, et al. Evidence for a protein kinase cascade in higher plants 3-hydroxy-3-methylglutaryl-CoA reductase kinase[J]. European Journal of Biochemistry, 1992, 209(3): 923-931. DOI:10.1111/ejb.1992.209.issue-3 |
| [31] |
Radchuk R, Radchuk V, Weschke W, et al. Repressing the expression of the sucrose non fermenting-1-related protein kinase gene in pea embryo causes pleiotropic defects of maturation similar to an abscisic acid-insensitive phenotype[J]. Plant Physiology, 2006, 140(1): 263-278. DOI:10.1104/pp.105.071167 |
| [32] |
Zhang Y, Shewry PR, et al. Expression of antisense SnRK1 protein kinase sequence causes abnormal pollen development and male sterility in transgenic barley[J]. Plant Journal, 2001, 28(4): 431-441. |
| [33] |
Kleinow T, Himbert S, Krenz B, et al. NAC domain transcription factor ATAF1 interacts with SNF1-related kinases and silencing of its subfamily causes severe developmental defects in Arabidopsis[J]. Plant Science, 2009, 177(4): 360-370. DOI:10.1016/j.plantsci.2009.06.011 |




