2. 贵州医科大学基础医学院,贵阳 550025;
3. 贵州省普通高等学校病原生物学特色重点实验室,贵阳 550025;
4. 贵州医科大学分子生物学重点实验室,贵阳 550004;
5. 贵州医科大学附属医院,贵阳 550004
2. School of Basic Medical Sciences, Guizhou Medical University, Guiyang 550025;
3. Key Laboratory of Medical Microbiology and Parasitology in Higher Education Department of Guizhou, Guiyang 550025;
4. Key Laboratory of Molecular Biology, Guizhou Medical University, Guiyang 550004;5. Affiliated Hospital of Guizhou Medical University, Guiyang 550004;
Ⅱ型内含子(Group Ⅱ Intron)是在细菌、古细菌及高等植物细胞器(线粒体、叶绿体)中发现的一类核酶,由内含子RNA和内含子编码蛋白(Intron Encoded Protein,IEP)组成[1-4]。其中,内含子RNA具有核酶活性,通过碱基互补配对原则识别双链DNA;IEP具有反转录酶和DNA核酸内切酶活性,与内含子RNA组装为核糖核蛋白复合体,维持内含子RNA的空间结构,辅助内含子RNA识别靶位点,并发挥核酸内切酶活性切割靶位点,而且IEP能以内含子RNA为模板合成cDNA,从而将内含子RNA以DNA形式插入到染色体靶位点,实现内含子RNA的“归巢”[5-9](图 1)。

|
| a:靶位点识别:Ⅱ型内含子经过诱导表达后,内含子RNA与IEP结合形成核糖核蛋白体复合物,内含子RNA在IEP辅助下通过碱基互补配对识别目标DNA上的靶位点;b:反向剪接:内含子RNA通过反向剪接插入到DNA靶位点的一条链中;c:切割DNA链:IEP具有的DNA核酸内切酶活性切割DNA互补链;d:反转录:DNA链断裂后,IEP具有的反转录酶以断裂处3' -OH端为引物,以内含子RNA为模板合成cDNA;e:DNA修复:机体以新合成的cDNA为模板合成互补DNA,从而将内含子RNA以DNA的形式插入到目标DNA序列 图 1 Ⅱ型内含子的“归巢”示意图 |
来源于乳酸乳球菌(Lactococcus lactis)的Ll.LtrB内含子因其较高的“归巢”效率,已被开发为高效Targetron基因打靶系统,并在革兰阳性细菌(如丙酮丁醇梭菌、解纤维梭菌、艰难梭菌、金黄色葡萄球菌)[10-16]和革兰阴性细菌(如大肠杆菌、绿脓杆菌和根癌农杆菌)[17]等多个细菌中获得广泛应用,尤其适用于难改造的革兰氏阳性厚壁细菌,如肉毒梭菌、艰难梭菌和解纤维梭菌等。该技术在梭菌中也称为Clos tron技术[6]。在前期研究中发现,Ⅱ型内含子的表达方式对Targetron系统的打靶效率及打靶精度具有很大的影响[18]。如来源于pSY6的Targetron系统,因其Ⅱ型内含子的表达是受ptb组成型启动子调控,Ⅱ型内含子在细胞内长期大量存在,使其在解纤维梭菌中存在一定的脱靶现象。当把组成型启动子更换为阿拉伯糖诱导型启动子后,因其表达量和表达模式可以根据需要随时受到调控,其脱靶现象大幅度降低[18]。因此,构建不同的诱导型Targetron系统可以提高其打靶效果。
本研究将来源于Ll.LtrB的Ⅱ型内含子置于T7-lac操纵子控制之下,构建了IPTG诱导型Targetron质粒,并通过优化诱导剂浓度和诱导时间,在大肠杆菌中建立了诱导型Targetron体系,旨为Ⅱ型内含子的机理研究和应用奠定基础。
1 材料与方法 1.1 材料 1.1.1 培养基及培养方法大肠杆菌(Escherichia coli)DH5α、BL21(DE3)均用37℃在LB培养基中进行培养,LB液体培养基的配制:1% TRYPTONE、0.5% YEAST EXTRACT、1% NaCl,在LB液体培养基中加入1.5%的Agar Powder成为LB固体培养基。质粒构建及Ⅱ型内含子诱导表达所用氨苄青霉素的筛选浓度为100 μg/mL,蓝白斑筛选时,在LB固体培养基中加入氨苄青霉素(100 μg/mL)、X-gal(40 μg/mL)和IPTG(0.1 mmol/L)。
1.1.2 主要试剂高保真DNA聚合酶、DNA marker、T4 DNA连接酶购自北京全式金生物技术有限公司,Taq DNA聚合酶购自南京诺唯赞生物科技有限公司,限制性内切酶购自Thermo Fisher Scientific,Gibson组装试剂盒购自NEB,琼脂糖凝胶DNA回收试剂盒、PCR产物纯化试剂盒、质粒提取试剂盒购自北京天根公司,IPTG、X-gal、氨苄青霉素购自北京索莱宝公司,引物合成及测序由上海生工完成。
1.1.3 引物本研究所用到的引物详见表 1。
本研究中用的菌株及质粒详见表 2。
以大肠杆菌lacZ基因为靶基因,利用在线网址(www.clostron.com),结合评分高低,选择lacZ基因正义链第635位点(lacZ-635s)和反义链第1063位点(lacZ-1063a)作为打靶位点,每个打靶位点软件会给出4种引物(IBS、EBS1d、EBS2、EBS Universal),设计获得的引物序列详见表 1。
1.2.2 IPTG诱导的Targetron打靶质粒构建以pET28a载体为模板,lacI-T7-F和lacI-T7-R为引物,PCR扩增lacI-T7片段。利用Gibson组装试剂盒将lacI-T7扩增产物与XmaⅠ/XhoⅠ双酶切的pSY6载体组装,获得ⅠPTG诱导型Targetron载体pSY7(图 2)。
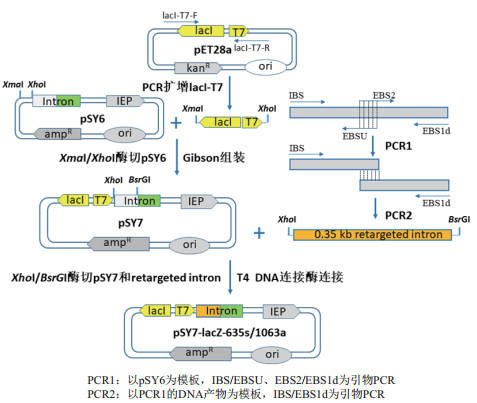
|
| 利用lacI-T7-F/ lacI-T7-R为引物扩增质粒pET28a上的T7-lac操纵子,以XmaⅠ/XhoⅠ双酶切质粒pSY6,利用Gibson组装试剂盒组装线性质粒pSY6和lacI-T7片段,获得IPTG诱导型Targetron质粒pSY7。以lacZ-635s和lacZ-1063a位点为例,将特异性打靶位点设计在引物IBS、EBS1d、EBS2上,以pSY6为模板,通过重叠延伸PCR得到0.35 kb的特异性内含子RNA打靶序列。利用XhoⅠ/BsrGI双酶切质粒pSY7和特异性内含子RNA打靶序列,T4 DNA连接酶连接得到特异性Targetron基因打靶载体pSY7-lacZ-635s/1063a 图 2 打靶载体构建流程图 |
通过重叠延伸PCR方法构建内含子RNA打靶识别片段,用XhoⅠ和BsrGI双酶切PCR产物和pSY7质粒,T4 DNA连接酶连接得到两个位点的特异性打靶质粒pSY7-lacZ-635s和pSY7-lacZ-1063a,详细构建流程如图 2所示。
1.2.3 大肠杆菌BL21(DE3)的转化利用热激法将质粒pSY7-lacZ-635s和pSY7-lacZ-1063a转入BL21(DE3)感受态细胞,置于含100 μg/mL氨苄青霉素的LB液体培养基中,37℃、180 r/min过夜培养。
1.2.4 IPTG浓度对Targetron打靶效率的影响取40 μL过夜培养的菌液加入到4 mL的LB液体培养基中(含100 μg/mL氨苄青霉素),37℃、200 r/min培养1 h,加入不同终浓度的IPTG置于37℃、200 r/min培养45 min,离心收集细胞沉淀,稀释后涂布LB固体培养基(含氨苄青霉素100 μg/mL、X-gal 40 μg/mL、IPTG 0.1 mmol/L),37℃过夜培养,通过蓝白斑计数计算其打靶效率。
1.2.5 诱导时间对Targetron打靶效率的影响加入终浓度0.5 mmol/L的IPTG进行不同时间的诱导,通过蓝白斑计数计算其打靶效率,其余操作同1.2.4。
2 结果 2.1 IPTG诱导的Targetron质粒构建IPTG诱导的pSY7质粒详细构建流程如图 1所示。pSY7质粒经XmaⅠ/XhoⅠ双酶切后琼脂糖凝胶电泳检测,可见清晰的约1.7 kb大小的电泳条带(图 3),与预期结果一致,表明诱导型Targetron质粒构建成功。
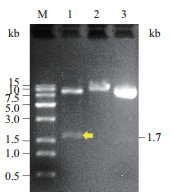
|
| M:15k marker;1:XmaⅠ/XhoⅠ双酶切质粒pSY7;2:XhoⅠ单酶切质粒pSY7;3:质粒pSY7 图 3 质粒pSY7酶切验证及琼脂糖凝胶电泳检测 |
利用重叠延伸PCR方法,以设计的lacZ-635s和lacZ-1063a两个位点的特异引物为引物,以Targetron载体指导RNA序列为模板,扩增lacZ-635s和lacZ-1063a两个位点的特异性打靶片段。图 4-A表示第一轮PCR扩增结果,琼脂糖凝胶电泳显示,分别获得打靶序列上游约0.25 kb和下游约0.1 kb的片段;图 4-B表示第二轮PCR扩增结果,琼脂糖凝胶电泳显示获得0.35 kb特异性条带。然后,分别用XhoⅠ/BsrGI双酶切第二轮PCR产物和pSY7载体,T4 DNA连接酶连接后获得特异性打靶载体pSY7-lacZ-635s和pSY7-lacZ-1063a。双酶切验证与预期结果一致(图 4-C);最后,通过测序进一步证实两个位点的打靶载体均构建成功。

|
| A:构建Targetron载体第一轮PCR(M:8000 marker;1:lacZ-635s-IBS/EBS Universal为引物;2:lacZ-635s-EBS2/lacZ-635s-EBS1d为引物;3:lacZ-1063a-IBS/EBS Universal为引物;4:lacZ-1063a-EBS2/lacZ-1063a-EBS1d为引物);B:构建Targetron载体第二轮PCR(M:8000 marker;1:lacZ-635s-IBS/lacZ-635s-EBS1d为引物;2:lacZ-1063a-IBS/lacZ-1063a-EBS1d为引物);C:质粒pSY7-lacZ-635s和pSY7-lacZ-1063a的酶切验证(1-3:质粒pSY7-lacZ-635s的酶切验证;4-6:质粒pSY7-lacZ-1063a的酶切验证;1和4:XhoⅠ/BsrGI双酶切;2和5:XhoⅠ单酶切;3和6:质粒pSY7-lacZ-635s、pSY7-lacZ-1063a) 图 4 Targetron载体构建及酶切验证 |
将打靶质粒pSY7-lacZ-635s和pSY7-lacZ-1063a分别转化大肠杆菌BL21(DE3),复苏后直接涂布LB平板,过夜培养后分别挑取单菌落进行菌落PCR验证。结果表明,未经诱导的Targetron质粒涂平板后获得的菌落均是野生型,表明本研究构建的Targetron系统具有较好的严谨性。
同时,进行平行试验,将转化后的大肠杆菌首先经0.2 mmol/L IPTG诱导1 h,稀释后涂布LB固体培养基(含氨苄青霉素100 μg/mL、X-gal 40 μg/mL、IPTG 0.1 mmol/L),过夜培养后,分别挑取平板上的白色菌落和蓝色菌落进行菌落PCR验证。结果如图 5所示:当用打靶位点上下游引物进行PCR扩增验证时,突变株得到约1.9 kb的PCR扩增条带,而野生型对照菌株得到约1.0 kb的PCR扩增条带(图 5-B)。当利用插入序列的内侧引物和外侧引物进一步进行PCR验证时,两种突变株均得到0.65 kb和1.6 kb的PCR扩增条带(图 5-C)。总之,上述结果表明,本研究成功构建IPTG诱导型Targetron打靶系统。
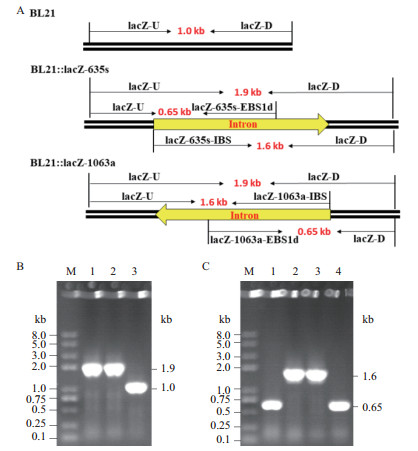
|
| A:Targetron系统功能验证示意图;B:插入位点外侧引物检测突变株(M:8000 marker;1:用lacZ-U和lacZ-D引物检测BL21::lacZ-635s;2:用lacZ-U和lacZ-D引物检测BL21::lacZ-1063a;3:用lacZ-U和lacZ-D引物检测BL21(DE3));C:特异性引物检测突变株(M:8000 marker;1:用lacZ-U和lacZ-635s-EBS1d引物检测BL21::lacZ-635s;2:用lacZ-635s-IBS和lacZ-D引物检测BL21::lacZ-635s;3:用lacZ-U和lacZ-1063a-IBS引物检测BL21::lacZ-1063a;4:用lacZ-1063a-EBS1d和lacZ-D引物检测BL21::lacZ-1063a) 图 5 Targetron基因打靶系统的功能验证 |
质粒pSY7-lacZ-635s和pSY7-lacZ-1063a分别转化大肠杆菌BL21(DE3),使用不同浓度的IPTG分别诱导45 min。结果(图 6)显示,在没有IPTG诱导时,两种质粒的打靶效率均为0,随着IPTG浓度的提高,其归巢效率也逐步提高,当IPTG浓度达到0.5 mmol/L时,两种质粒的打靶效率达到最高值,达到90%以上。

|
| 以大肠杆菌BL21(DE3)的lacZ基因正义链635位点(lacZ-635s)和反义链1063位点(lacZ-1063a)为靶位点,分别用终浓度为0.1、0.2、0.5、1.0、1.5、2.0 mmol/L的IPTG诱导45 min,在0-0.5 mmol/L的IPTG浓度范围内,Targetron的“归巢”效率随着IPTG浓度增高而增高,IPTG浓度大于0.5 mmol/L后,Targetron的“归巢”效率趋于平稳 图 6 IPTG浓度对归巢效率的影响 |
质粒pSY7-lacZ-635s和pSY7-lacZ-1063a分别转化大肠杆菌BL21(DE3),用0.5 mmol/L的IPTG分别诱导15 min、30 min、45 min、60 min。如图 7所示,两种质粒的打靶效率随着诱导时间的增加而增加,当诱导时间达到45 min时,两种质粒的打靶效率达到最大值。
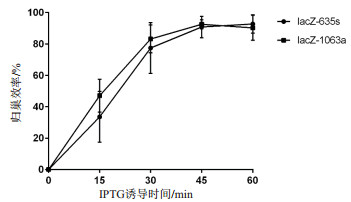
|
| 以大肠杆菌BL21(DE3)的lacZ基因正义链635位点(lacZ-635s)和反义链1063位点(lacZ-1063a)为靶位点,用终浓度为0.5 mmol/L的IPTG分别诱导15 min、30 min、45 min、60 min,在0-45 min内,Targetron的“归巢”效率随着IPTG诱导时间增加而增高,IPTG诱导时间大于45 min后,Targetron的“归巢”效率趋于平稳 图 7 诱导时间对归巢效率的影响 |
来源于乳酸乳球菌的LtrB内含子,在内含子编码蛋白的辅助下,成为高效可移动的元件。由于Ⅱ型内含子通过IEP及内含子RNA共同识别目标DNA序列,利用Targetron在线设计网址找到潜在内含子识别位点,结合PCR技术对内含子RNA修饰改造,从而实现内含子RNA的特异性“归巢”,基于此建立的基因打靶技术被广泛应用于原核生物的遗传改造,展现了可观的应用前景。有研究人员借助Targetron技术得以在包含毒性因子[19-21]、潜在药物靶点[22-23]等方面深入研究。选择Targetron设计网址得到的较高得分的潜在插入位点,可防止Ⅱ型内含子的脱靶,并且不需要遗传筛选标记,通过克隆PCR能轻易地筛选到突变菌株。
然而,无论在何种微生物中应用,Ⅱ型内含子的长期表达和大量积累均会降低其打靶精度,不利于其后续的遗传筛选。本研究建立的IPTG诱导型基因打靶系统,利用T7-lac操纵子与内含子组装,实现了Targetron系统在大肠杆菌中的可控诱导表达,而且可以根据需要,调控Targetron系统的表达模式,如在何时需要Ⅱ型内含子表达、Ⅱ型内含子的表达的强度是多少等,从而实现对靶基因的精确调控。
目前常用的基因打靶技术如基于同源重组原理的RecA系统和Red系统,其重组效率较低,需要进行多次转化和筛选方可获得突变株,实验周期较长[24]。基于DNA断裂损伤、激发机体修复机制的基因编辑方法如锌指核酸酶(Zinc finger nucleases,ZFNs)技术和类转录激活因子效应物核酸酶(Transcription activator-like effector nucleases,TALENs)技术,该技术具有靶向精确、细胞毒性低等优势,已在多个物种中获得广泛应用。然而,该技术均以蛋白作为向导,需要构建复杂的载体系统,技术难度较大,成本较高[25]。此外,CRISPR-Cas9(Clustered regularly interspaced short palindromic repeats/CRISPR-associated 9,CRISPR/Cas9 system)技术是目前应用最广泛的基因编辑技术,该技术以RNA为向导,仅需改变约20 bp的RNA序列即可实现不同基因的打靶,具有效率高、成本低、操作方便等优势,但仍存在一定的脱靶风险[25]。本研究中采用的Targetron基因打靶技术是基于细菌Ⅱ型内含子的“归巢”原理而设计,该技术以内含子RNA为向导,通过改变内含子RNA的序列实现不同基因的打靶,具有效率高,操作方便等优势,尤其适用于微生物的遗传改造。本研究将T7-lac操纵子与Targetron基因打靶系统联合,成功构建诱导型Targetron系统,极大地缩短了实验周期,提高了工作效率。
本研究中建立的方法由于采用的是T7-lac操纵子启动内含子RNA及内含子编码蛋白的表达,而T7启动子需要和T7 RNA聚合酶相互识别才能发挥功能,并且在不同的微生物中,需要不同的复制元件,故本研究建立的方法限于编码T7 RNA聚合酶的大肠杆菌系统。但是通过在质粒上构建T7 RNA聚合酶的序列及更换合适的复制元件,便可用于其他微生物的遗传改造。总之,本研究建立的诱导型Targetron技术,为Ⅱ型内含子的功能应用与机理研究奠定了基础。
4 结论本研究将T7-lac操纵子与Ⅱ型内含子组装得到了大肠杆菌IPTG诱导型Targetron基因打靶系统,利用克隆PCR,验证了其具有严谨表达的优点,并通过优化诱导条件,提高了基因打靶的效率。
致谢: 感谢中国科学院上海生命科学院植物生理生态研究所姜卫红课题组提供pSY6质粒。
| [1] |
Lambowitz AM, Zimmerly S. Mobile group Ⅱ introns[J]. Annu Rev Genet, 2004, 38: 1-35. DOI:10.1146/annurev.genet.38.072902.091600 |
| [2] |
Toro N, Molina-Sanchez MD, Nisa-Martinez R, et al. Bacterial group Ⅱ introns: identification and mobility assay[J]. Methods in Molecular Biology, 2016, 1400: 21-32. DOI:10.1007/978-1-4939-3372-3 |
| [3] |
Pyle AM. Group Ⅱ intron self-splicing[J]. Annu Rev Biophys, 2016, 45: 183-205. DOI:10.1146/annurev-biophys-062215-011149 |
| [4] |
Zhao C, Pyle AM. The maturase: a reverse transcriptase and splicing factor go hand in hand[J]. Curr Opin Struct Biol, 2017, 47: 30-39. DOI:10.1016/j.sbi.2017.05.002 |
| [5] |
Lambowitz AM, Zimmerly S. Group Ⅱ introns: mobile ribozymes that invade DNA[J]. Cold Spring Harb Perspect Biol, 2011, 3(8): a003616. |
| [6] |
Enyeart PJ, Mohr G, Ellington AD, et al. Biotechnological applications of mobile group Ⅱ introns and their reverse transcriptases: gene targeting, RNA-seq, and non-coding RNA analysis[J]. Mobile DNA, 2014, 5(1): 2. DOI:10.1186/1759-8753-5-2 |
| [7] |
Lambowitz AM, Belfort M. Mobile Bacterial Group Ⅱ introns at the crux of eukaryotic evolution[J]. Microbiology Spectrum, 2015, 3(1): MDNA3-0050-2014. |
| [8] |
McNeil BA, Semper C, Zimmerly S. Group Ⅱ introns: versatile ribozymes and retroelements[J]. Wiley interdisciplinary reviews RNA, 2016, 7(3): 341-355. DOI:10.1002/wrna.1339 |
| [9] |
Qu G, Kaushal PS, Wang J, et al. Structure of a group Ⅱ intron in complex with its reverse transcriptase[J]. Nat Struct Mol Biol, 2016, 23(6): 549-557. DOI:10.1038/nsmb.3220 |
| [10] |
刘亚君, 崔古贞, 洪伟, 等. 典型产纤维小体梭菌的遗传改造及其在纤维素乙醇中的应用研究进展[J]. 生物加工过程, 2014, 12(1): 55-62. |
| [11] |
Cui GZ, Zhang J, Hong W, et al. Improvement of ClosTron for successive gene disruption in Clostridium cellulolyticum using a pyrF-based screening system[J]. Appl Microbiol Biotechnol, 2014, 98(1): 313-323. DOI:10.1007/s00253-013-5330-y |
| [12] |
Cui GZ, Hong W, Zhang J, et al. Targeted gene engineering in Clostridium cellulolyticum H10 without methylation[J]. Journal of Microbiological Methods, 2012, 89(3): 201-208. DOI:10.1016/j.mimet.2012.02.015 |
| [13] |
Heap JT, Pennington OJ, Cartman ST, et al. A modular system for Clostridiumshuttle plasmids[J]. Journal of Microbiological Methods, 2009, 78(1): 79-85. DOI:10.1016/j.mimet.2009.05.004 |
| [14] |
Shao L, Hu S, Yang Y, et al. Targeted gene disruption by use of a group Ⅱ intron(targetron)vector in Clostridium acetobutylicum[J]. Cell Research, 2007, 17(11): 963-965. DOI:10.1038/cr.2007.91 |
| [15] |
Heap JT, Pennington OJ, Cartman ST, et al. The ClosTron: a universal gene knock-out system for the genus Clostridium[J]. Journal of Microbiological Methods, 2007, 70(3): 452-464. DOI:10.1016/j.mimet.2007.05.021 |
| [16] |
Yao J, Zhong J, Fang Y, et al. Use of targetrons to disrupt essential and nonessential genes inStaphylococcus aureus reveals temperature sensitivity of Ll. LtrB group Ⅱ intron splicing[J]. RNA, 2006, 12(7): 1271-1281. DOI:10.1261/rna.68706 |
| [17] |
Yao J, Lambowitz AM. Gene targeting in gram-negative bacteria by use of a mobile group Ⅱ intron("Targetron")expressed from a broad-host-range vector[J]. Applied and Environmental Microbiology, 2007, 73(8): 2735-2743. DOI:10.1128/AEM.02829-06 |
| [18] |
Zhang J, Liu YJ, Cui GZ, et al. A novel arabinose-inducible genetic operation system developed for Clostridium cellulolyticum[J]. Biotechnology for Biofuels, 2015, 8: 36. DOI:10.1186/s13068-015-0214-2 |
| [19] |
Alonzo F, 3 rd, Port GC, Cao M, et al. The posttranslocation chaperone PrsA2 contributes to multiple facets of Listeria monocytogenes pathogenesis[J]. Infection and Immunity, 2009, 77(7): 2612-2623. DOI:10.1128/IAI.00280-09 |
| [20] |
Sayeed S, Uzal FA, Fisher DJ, et al. Beta toxin is essential for the intestinal virulence of Clostridium perfringens type C disease isolate CN3685 in a rabbit ileal loop model[J]. Molecular Microbiology, 2008, 67(1): 15-30. |
| [21] |
Carter GP, Awad MM, Hao Y, et al. TcsL is an essential virulence factor in ATCC 9714[J]. Infection and Immunity, 2011, 79(3): 1025-1032. DOI:10.1128/IAI.00968-10 |
| [22] |
Zoraghi R, See RH, Gong H, et al. Functional analysis, overexpression, and kinetic characterization of pyruvate kinase from methicillin-resistant Staphylococcus aureus[J]. Biochemistry, 2010, 49(35): 7733-7747. DOI:10.1021/bi100780t |
| [23] |
Zoraghi R, Worrall L, See RH, et al. Methicillin-resistant Staphylococcus aureus(MRSA)pyruvate kinase as a target for bis-indole alkaloids with antibacterial activities[J]. The Journal of Biological Chemistry, 2011, 286(52): 44716-44725. DOI:10.1074/jbc.M111.289033 |
| [24] |
张雪, 温廷益. Red重组系统用于大肠杆菌基因修饰研究进展[J]. 中国生物工程杂志, 2008, 28(12): 89-93. |
| [25] |
吴璐, 王磊, 任远, 等. 基因组编辑技术研究进展[J]. 生物技术通报, 2014(11): 84-90. |






