桃(Prunus persica)是世界上栽培广泛的落叶果树之一, 世界桃产量一直呈上升趋势, 其中中国占一半以上。迄今为止, 桃树在中国已有3 000多年的栽培历史, 全国各地的栽培品种1 000多个[1], 具有重要的商业价值。桃作为重要的经济果树之一, 其果实品质的好坏与成熟期的早晚直接影响果农的经济效益。
NAC基因家族是植物基因组中含量极其丰富且植物特有的最大转录因子家族之一。最早是在矮牵牛NAM、拟南芥AATF1/2和CUC2蛋白的N端被发现的一段高度保守的氨基酸序列, Aida等[2]将其命名为NAC结构域, 并将包含NAC结构域的蛋白称为NAC转录因子。迄今为止, 已经预测或鉴定出许多来自不同植物的NAC基因, 主要有玉米(Zea mays)、拟南芥(Arabidopsis thaliana)、高粱(Sorghum bicolor)、番木瓜(Carica papaya)[3]以及大豆(Soybean)[4]等。同时还有研究表明NAC家族具有非常重要的生物学功能, 如广泛参与花的成熟[5]、木纤维的形成[6]、侧根的发育[7-8]、植物的抽枝[9]、植物的衰老[10-11]以及影响激素信号的传导[12-14]等, 在各种发育过程和逆境应答中起非常重要的调节作用。
桃果实成熟受多因素影响, 包括环境、生长素及基因型等。Bailey等[15]推测桃果实成熟期共受3个位点的6个累加基因的控制, 其中每个贡献基因控制大概15 d的成熟期, 例如基因型Sm1Sm1Sm2Sm2Sm3Sm3比Sm1Sm1Sm2Sm2Sm3sm3早成熟15 d, 基因型sm1sm1sm2sm2sm3sm3比sm1sm1sm2sm2Sm3sm3晚成熟15 d, 但景士西等[16]认为此说法尚缺乏试验证据进行支持。Eduardo等[17]研究发现, 编码NAC家族的转录因子候选基因ppa008301m(PpNAC72)是植物中NAC基因家族中的一员, MD基因座的精细定位[18]鉴定出ppa008301m(PpNAC72)作为可能的至熟基因在转录水平上调节基因表达, 在调节植物生长发育过程中起着非常重要的作用。
近年来, 对桃NAC基因家族的研究取得了一定进展, 但对于桃成熟相关分子的作用机制有待于进一步研究。本研究基于前期对影响桃成熟期基因的筛选与研究, 以‘津柳早红’为材料, 通过RT-PCR技术克隆获得PpNAC72, 并对其进行生物信息学分析。利用实时荧光定量PCR检测该基因在不同器官和不同时期的表达变化, 为更好地研究桃果实成熟的机制提供理论与实验支持。
1 材料与方法 1.1 材料实验材料为‘津柳早红’, 成熟期在6月上旬, 属于极早熟桃品种。于2018年4月至6月, 采取‘津柳早红’的花蕾、叶片、茎、根及果实; 于2018年4月下旬至6月中旬, 每隔8 d取‘津柳早红’各时期的果实。试验材料均采自天津市大柳滩桃园基地, 材料经液氮中速冻后于-80℃保存备用。
1.2 方法 1.2.1 引物设计与合成根据桃参考基因组(https://www.rosaceae.org/)中的ppa008301m序列, 使用Primer 5.0设计引物, NAC-F1:5′-GGAATTCCATATGGCTCTCTTTCTTTCTCTC-3′, NAC-R1:5′-TAACCCGGGACTACTCGATTTCTCCAC-3′(下划线处分别为NdeⅠ和SmaⅠ酶切位点), 均由生工生物工程(上海)股份有限公司合成。
1.2.2 桃总RNA提取及cDNA的合成根据植物总RNA快速提取试剂盒EASYspin Plus(Aidlab, 北京)说明书提取叶片、花蕾、果实、根、茎的RNA, 并使用Revert-Aid First Strand cDNA Synthesis Kit(TaKaRa, 中国大连)合成cDNA。
1.2.3 PpNAC72的克隆以‘津柳早红’cDNA为模板进行PCR扩增, 反应体系为cDNA模板1.0 μL、5×PCR Buffer 2.5 μL、2.5 mmol/L dNTPs 2.0 μL、5 U/μL rTaq酶0.2 μL、10 μmol/L正、反向引物各1.0 μL和dd H2O 17.3 μL。扩增程序为94℃ 3 min; 94℃ 30 s, 57℃ 30 s, 72℃ 1 min, 30个循环; 72℃ 8 min, 4℃保存, PCR产物于1%琼脂糖凝胶电泳检测, 回收, 并与pEASY-Blunt载体连接, 转入大肠杆菌感受态细胞。筛选阳性克隆, 送至生工生物工程(上海)股份有限公司测序。
1.2.4 PpNAC72的qRT-PCR分析提取‘津柳早红’根、茎、花、叶以及不同时期果实的RNA, 并反转录成cDNA。使用Primer 5.0引物设计软件, 根据序列非保守区设计引物, qPCR-F1:5′-GTCGTCTTCCTGCTCGTCTCA-3′和qPCR-R1:5′-TCACATTCATTTGCCCTTGG-3′, 产物长度为245 bp。内参基因RPⅡ(RNA polymeraseⅡ)上游引物F2:5′-TGAAGCATACACCTATGATGATGAAG-3′, 下游引物R2:5′-CTTTGACAGCACC-AGTAGATTCC-3′[19]。根据iTaq Un-iversal SYBR Green Supermix操作说明, 以RPⅡ为内参基因, 利用BIO-RAD CFX96荧光定量PCR仪(伯乐, 美国)进行qRT-PCR扩增。实时荧光定量PCR反应体系为:iTaq Universal SYBR Green Supermix 10 μL、上游引物(qPCR-F1/RP Ⅱ-F2)1 μL、下游引物(qPCR-R1/RP Ⅱ-R2)1 μL、cDNA模板1 μL、ddH2O补至20 μL。qRT-PCR反应程序为95℃ 3 min; 95℃ 5 s, 60℃ 30 s, 72℃ 30 s, 40个循环, 每个样品3个重复。采用2-ΔΔCt法进行数据分析, 计算不同器官和不同时期PpNAC72的表达量。
1.2.5 PpNAC72的生物信息学分析利用NCBI-BLAST对获得的目的片段进行同源性比对, 利用Bioedit进行氨基酸序列分析, 并利用MEGA 6.0构建PpNAC72系统发育进化树。进一步利用在线工具expasy(http://web.expasy.org/compute_pi/)推测该蛋白的等电点, 并利用expasy上的ProtParam工具对PpNAC72所编码的氨基酸序列进行亲水性分析。利用在线工具SignalP4.1(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽分析, 利用在线工具TMHMM v2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构域分析, 利用在线工具PredictProtein(https://www.predictprotein.org/)进行蛋白质的二级结构分析, 利用Swissmodel(http://swissmodel.expasy.org/)预测蛋白质的三级结构。通过PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行启动子元件分析。
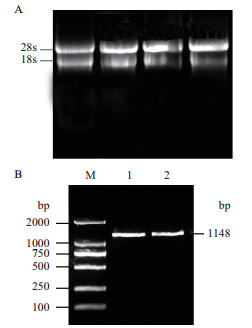
2 结果 2.1 PpNAC72的克隆从‘津柳早红’果实中提取RNA, 反转录得到cDNA后使用特异性引物PCR扩增得到约1 100 bp的片段(图 1)。结果表明, 该基因全长编码序列为1 050 bp, 编码349个氨基酸, BLAST结果(图 2)显示, 该基因含有个1个NAM结构域, 从N端第15-139氨基酸。此基因命名为PpNAC72。经序列对比, 发现目的基因比桃参考基因组中的该基因多出一段长度为36 bp的片段(图 3)。
|
| M:2000 DNA分子标记;1、2:PpNAC72 PCR产物 图 1 桃RNA检测(A)和PpNAC72的扩增(B) |

|
| 图 2 PpNAC72蛋白中NAM结构域分析 |

|
| 图 3 PpNAC72的同源性分析 |
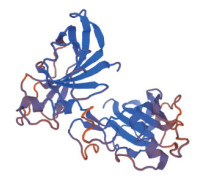
PpNAC72所编码的蛋白质分子量为39.2 kD, 等电点为8.34, 不稳定系数为39.14, 为稳定蛋白。亲水性分析表明, 亲水性氨基酸为153个, 占43.9%;疏水性氨基酸为142个, 占40.6%, 平均疏水系数为-0.623, 为亲水性蛋白, 其中, 亲水性最高的氨基酸是第78位的精氨酸(Arg, R), 其亲水指数位-2.800。氨基酸组成中带有负电荷的氨基酸(Asp+Glu)共有37个, 占10.6%;带有正电荷的氨基酸(Arg+Lys)共有40个, 占11.5%。该蛋白没有跨膜区域, 非膜蛋白。该蛋白的二级结构预测结果表明, 桃树PpNAC72蛋白的二级结构中α-螺旋结构占3.44%, β-折叠结构占14.04%, 环肽链占82.52%。PpNAC72蛋白的三级结构见图 4。
|
| 图 4 PpNAC72蛋白的三级结构预测图 |
启动子是基因的一个重要组成部分。选取PpNAC72起始密码子上游2 000 bp进行启动子元件分析, 发现启动子和增强子区域中常见的顺式作用元件——CAAT-box、参与脱落酸反应作用的顺式作用元件(如ABRE)、参与光响应的元件(如G-Box、Box 4和ATCT-motif)、与生长素相关的响应元件(如TGA-element)。结果表明, PpNAC72可能参与多重激素调控果实成熟的响应。
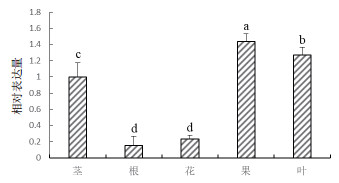
2.4 PpNAC72的组织特异性分析‘津柳早红’不同器官的荧光定量PCR检测结果(图 5)表明, PpNAC72在所有被检测的器官(包括营养器官以及生殖器官)中都有不同程度的表达。在果实中表达量最高, 在根部、花朵中相对较少, 在根部表达量最低。
|
| 图 5 PpNAC72的组织特异性分析 |
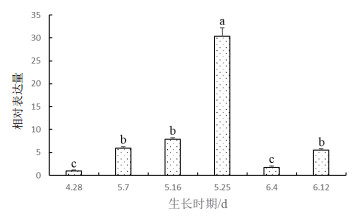
该基因在不同时期果实中的表达量随着果实的成熟不断增加, 在果实生长阶段中后期表达量达到最高, 其表达量是前一个时期的3倍, 并且远远高于其他时期; 随后表达量降低, 但在果实接近成熟时基因表达量增加(图 6)。
|
| 图 6 PpNAC72在不同时期果实中的表达量分析 |
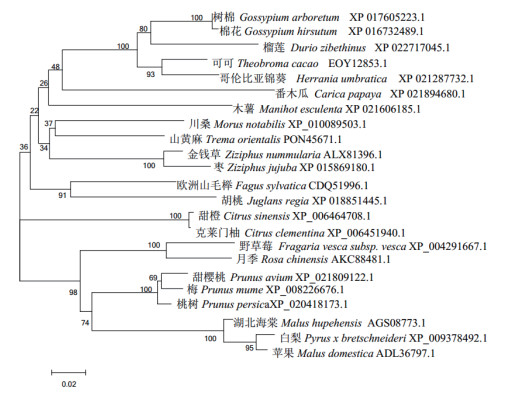
将桃的转录因子PpNAC72与甜樱桃(Prunus avium, XP_021809122.1)、梅(Prunus mume, XP_008226676.1)、野草莓(Fragaria vesca subsp. Vesca, XP_004291667.1)、月季(Rosa chinensis, AKC88481.1)、白梨(Pyrus x bretschneideri, XP_009378492.1)、苹果(Malus domestica, ADL36797.1、川桑(Morusnotabilis, XP_010089503.1)、湖北海棠(Ma-lus hupehensis, AGS08773.1)、山黄麻(Tremaorien-talis, PON45671.1)、可可(Theobroma cacao, EOY12853.1)、胡桃(Juglans regia, XP_018851445.1)、甜橙(Citrus sinensis, XP_006464708.1)等物种的NAC家族氨基酸序列进行多重序列比对, 发现桃树的PpNAC72与甜樱桃同源性最高, 达96%, 与树棉(Gossypium arboreum, XP_017605223.1)最低(图 7)。利用Neighbor-Joining法构建系统进化树, 结果(图 8)表明, 桃的转录因子PpNAC72与甜樱桃和梅的NAC转录因子位于同一进化分支, 亲缘关系最近, 与其他植物的NAC转录因子亲缘关系较远。

|
| 黑色表示相同残基;深灰表示保守替换;浅灰表示半保守替换 图 7 桃树PpNAC72蛋白与其他物种中NAC蛋白的氨基酸序列比对 |
|
| 图 8 桃树PpNAC72蛋白与其他物种NAC蛋白的聚类分析 |
NAC类转录因子是一类植物中特有的转录因子, 该类转录因子具有自身的结构特点:其N端含有约150个氨基酸组成的高度保守NAM结构域, 并且在该结构域中含有A、B、C、D和E 5个亚结构域[20]。而且, NAC类转录因子C端一般特异性很高, 具有转录激活功能[21-22], 此外C端还富含丝氨酸、谷氨酸、苏氨酸、脯氨酸等。另有研究表明, 含有60个残基的NAM结构域, 是由包围着一些螺旋状元件的一个扭曲的β-折叠组成[23]。绝大多数NAC基因仅有1个NAM结构域, 少数含有2个NAM结构域, 且大多数NAM结构域两端基本都具有保守基序[24]。
本实验通过RT-PCR技术, 获得了PpNAC72的开放阅读框编码序列, 利用生物信息学对所获得的PpNAC72氨基酸序列分析显示:该蛋白具有一个NAM结构域, 位置从N端第15-139氨基酸, 其二级结构中α-螺旋结构占3.44%, β-折叠结构占14.04%。该基因的蛋白C端富含丝氨酸、谷氨酸、苏氨酸、脯氨酸等, 与NAC转录因子编码的蛋白质特点一致。使用Bioedit对桃PpNAC72的氨基酸序列比对, 发现该转录因子与其中与甜樱桃NAC同源性最高, 与其他物种的NAC蛋白同源性比对均在96%-70%, 说明桃PpNAC72相对保守。
经多项研究发现, 在拟南芥、番茄等模式作物上调控果实成熟的NAC转录因子较多, 拟南芥中与果实成熟相关的NAC基因已有报道, 乙烯信号转导途径中的下游转录因子基因AtNAC2可以被乙烯合成前体ACC诱导, 推测该基因是乙烯和生长素信号传导途径中的一个下游基因, 该基因超表达促进转基因植株的侧根生长, 同时具有明显的耐盐性[8]。在拟南芥中发现的NAP转录因子在调控植物生长发育、叶片衰老、响应生物和非生物胁迫等方面有重要作用, NAP转录因子不仅与叶片衰老有关, 也与果实衰老紧密相关[25]。Liu等[26]研究发现甜橙的CitNAC在其成熟期和衰老期的果实中均有相应的表达, 预示着CitNAC可能调控甜橙果实的成熟衰老。拟南芥的3个NAC基因RD26/ANAC072、ANAC019和ANAC055均受干旱、高盐或低温的诱导表达, 证实ANAC072参与胁迫响应和ABA信号传导途径[27]。马铃薯StNAC72可能参与干旱及水分刺激信号传导过程[28]。表明该基因在不同植物中的功能有所不同, 在植物发育过程和逆境应答中均发挥作用, 是一个多功能转录因子。
本研究通过从桃树‘津柳早红’中分离克隆转录因子PpNAC72, 对其结构和功能进行初步分析, 并且对桃各个器官的PpNAC72进行定量表达分析, 结果表明, 该基因在桃树的各个部位均有表达, 且在果实中的表达量达到最高, 同时该基因的表达量在其生长期的不同时期有明显变化, 并呈现增加-减少-增加的趋势。桃树的果实发育分为3个时期:一是第一个迅速生长期, 此期幼果纵径生长迅速; 二是缓慢生长期, 即硬核期, 此期果实生长较慢, 主要是果核的硬化和胚胎的形成; 三是第二个迅速生长期, 此期细胞体积迅速膨大。PpNAC72的表达量随着果实的生长不断增加, 在其果实生长阶段的中后期表达量达到最高, 此时果实可能处于迅速生长期; 随后表达量降低; 但在果实接近成熟是时基因表达量增加, 此时果实可能处于第二个迅速生长期。该基因的表达量的变化趋势符合桃树的生长规律, 说明该基因极有可能参与果树的生长发育, 并有可能影响桃树的成熟, 为进一步研究该基因的功能奠定基础。
4 结论从桃树中克隆获得PpNAC72, 其开放阅读框1 050 bp, 编码349个氨基酸, 具有一个NAM结构域, PpNAC72蛋白为亲水性蛋白, 非膜蛋白。PpNAC72与甜樱桃同源性最高, 亲缘性最近。PpNAC72可能参与果树的生长发育, 并有可能影响桃树的成熟。
| [1] |
汪祖华, 庄恩及. 中国果树志—桃卷[M]. 北京: 中国林业出版社, 2001.
|
| [2] |
Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation in Arabidopsis:an analysis of the cup-shaped cotyledon mutant[J]. The Plant Cell, 1997, 9(6): 841-857. DOI:10.1105/tpc.9.6.841 |
| [3] |
Pérezrodríguez P, Corrêa LGG, Rensing SA, et al. PlnTFDB:updated content and new features of the plant transcription factor database[J]. Nucleic Acids Research, 2010, 38: 822-827. DOI:10.1093/nar/gkp1056 |
| [4] |
Pinheiro GL, Marques CS, Costa MDBL, et al. Complete inventory of soybean NAC transcription factors:sequence conservation and expression analysis uncover their distinct roles in stress response[J]. Gene, 2009, 444(1): 10-23. |
| [5] |
Olsen AN, Ernst HA, Lehhio LL, et al. NAC transcription factors:structurally distinct, functionally diverse[J]. Trends in Plant Science, 2005, 10(2): 79-87. |
| [6] |
Ko JH, Yang SH, Park AH, et al. ANAC012, a member of the plant-specific NAC transcription factor family, negatively regulates xylary fiber development in Arabidopsis thaliana[J]. The Plant Journal, 2007, 50(6): 1035-1048. DOI:10.1111/j.1365-313X.2007.03109.x |
| [7] |
Xie Q, Frugis G, Colgan D, et al. Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J]. Genes & Development, 2000, 14(23): 3024-3036. |
| [8] |
He XJ, Mu RL, Cao WH, et al. AtNAC2, a transcription factor downstream of ethylene and auxin signaling pathways, is involved in salt stress response and lateral root development[J]. The Plant Journal, 2010, 44(6): 903-916. |
| [9] |
Mao C, Ding W, Wu Y, et al. Overexpression of a NAC-domain protein promotes shoot branching in rice[J]. New Phytologist, 2007, 176(2): 288-298. DOI:10.1111/j.1469-8137.2007.02177.x |
| [10] |
Uauy C, Distelfeld A, Fahima T, et al. A NAC gene regulating senescence improves grain protein, zinc, and iron content in wheat[J]. Science, 2006, 314(5803): 1298-1301. DOI:10.1126/science.1133649 |
| [11] |
Guo Y, Gan S. AtNAP, a NAC family transcription factor, has an important role in leaf senescence[J]. The Plant Journal, 2010, 46(4): 601-612. |
| [12] |
Jensen MK, Hagedorn PH, De TM, et al. Transcriptional regulation by an NAC(NAM-ATAF1, 2-CUC2)transcription factor attenuates ABA signalling for efficient basal defence towards Blumeria graminis f. sp. hordei in Arabidopsis[J]. The Plant Journal, 2010, 56(6): 867-880. |
| [13] |
Bu Q, Jiang H, Li CB, et al. Role of the Arabidopsis thaliana NAC transcription factors ANAC019 and ANAC055 in regulating jasmonic acid-signaled defense responses[J]. Plant Signaling & Behavior, 2009, 18(5): 756-767. |
| [14] |
Kim SG, Lee AK, Yoon HK, et al. A membrane-bound NAC transcription factor NTL8 regulates gibberellic acid-mediated salt signaling in Arabidopsis seed germination[J]. The Plant Journal, 2008, 55(1): 77-88. DOI:10.1111/tpj.2008.55.issue-1 |
| [15] |
Bailey CH, Hough LF. A hypothesis for the inheritance of season of ripening in progenies from certain early ripening peach varieties and selections[J]. Proc Amer Soc Hort Sci, 1959, 73: 125-133. |
| [16] |
景士西, 吴禄平. 园艺学年评[M]. 北京: 科学出版社, 1996.
|
| [17] |
Eduardo I, Picañol R, Rojas E, et al. Mapping of a major gene for the slow ripening character in peach:co-location with the maturity date gene and development of a candidate gene-based diagnostic marker for its selection[J]. Euphytica, 2015, 205(2): 627-636. DOI:10.1007/s10681-015-1445-9 |
| [18] |
Pirona R, Eduardo I, Pacheco I, et al. Fine mapping and identification of a candidate gene for a major locus controlling maturity date in peach[J]. BMC Plant Biology, 2013, 13(1): 166. DOI:10.1186/1471-2229-13-166 |
| [19] |
Tong ZG, Gao ZH, Wang F, et al. Selection of reliable reference genes for gene expression studies in peach using real-time PCR[J]. BMC Molecular Biology, 2009, 10(1): 1-13. |
| [20] |
Puranik S, Sahu PP, Srivastava PS, et al. NAC proteins:regulation and role in stress tolerance[J]. Trends in Plant Science, 2012, 17(6): 369-381. DOI:10.1016/j.tplants.2012.02.004 |
| [21] |
Duval M, Hsieh TF, Kim SY, et al. Molecular characterization of AtNAM:A member of the Arabidopsis NAC domain superfamily[J]. Plant Molecular Biology, 2002, 50(2): 237-248. DOI:10.1023/A:1016028530943 |
| [22] |
Hao YJ, Wei W, Song QX, et al. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants[J]. The Plant Journal, 2011, 68(2): 302-313. DOI:10.1111/j.1365-313X.2011.04687.x |
| [23] |
Wang Z, Dane F. NAC(NAM/ATAF/CUC)transcription factors in different stresses and their signaling pathway[J]. Acta Physiologiae Plantarum, 2013, 35(5): 1397-1408. DOI:10.1007/s11738-012-1195-4 |
| [24] |
邹嘉欣, 吕楠, 朱梦丽, 等. NAC家族生物信息学分析[J]. 生物技术通讯, 2016, 14(8): 1948-1964. |
| [25] |
Kou X, Watkins CB, Gan SS. Arabidopsis AtNAP regulates fruit senescence[J]. Journal of Experimental Botany, 2012, 63(17): 6139-6147. DOI:10.1093/jxb/ers266 |
| [26] |
Liu YZ, Baig MNR, Fan R, et al. Identification and expression pattern of a novel NAM, ATAF, and CUC-like gene from Citrus sinensis Osbeck[J]. Plant Molecular Biology Reporter, 2009, 27(3): 292-297. DOI:10.1007/s11105-008-0082-z |
| [27] |
Fujita M, Fujita Y, Maruyama K, et al. A dehydration-induced NAC protein RD26 is involved in a novel ABA-dependent stress-signaling pathway[J]. The Plant Journal, 2004, 39(6): 863-876. DOI:10.1111/tpj.2004.39.issue-6 |
| [28] |
林菁.辣椒转录因子CaNAC72的克隆及其功能鉴定[D].福建: 福建农林大学, 2012.
|




