2. 开江县动物疫病预防控制中心,达州 636250;
3. 达州市动物疫病预防控制中心,达州 635099
2. Animal Disease Prevention and Control Center of Kaijiang County, Dazhou 636250;
3. Animal Disease Prevention and Control Center of Dazhou, Dazhou 635099
非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒(African swine fever virus,ASFV)引起的一种急性、烈性、出血性猪传染病,急性感染死亡率为100%[1]。急性和亚急性ASF常在多种器官上表现出严重的血管性病变,如肾脏出血、淋巴结弥漫性出血、肺水肿、弥散性血管内凝血和血小板减少等。其主要临床症状是高热、皮肤发绀、有出血点、呕吐和便血等[2]。因此,ASF被世界动物卫生组织(OIE)列为必须报告的法定动物疫病之一,我国将其列为一类动物疫病[1]。1921年,在非洲(肯尼亚)首次发现ASF。1957年,欧洲(葡萄牙)首次暴发ASF疫情,随后蔓延到全球多个国家和地区,如俄罗斯、乌克兰、爱沙尼亚、拉脱维亚、波兰等东欧国家和高加索地区[3-8]。2018年以来,全球有波兰、俄罗斯、拉脱维亚、捷克、罗马尼亚、摩尔多瓦、乌克兰、匈牙利、保加利亚、比利时、科特迪瓦、南非、赞比亚和中国等14个国家发生ASF疫情,国际疫情形势十分严峻[9]。2018年8月,辽宁省出现我国首例ASF疫情[10]。截至目前,在全国23个省(市、自治区)先后发生100起疫情,给我国养猪业造成严重的经济损失。
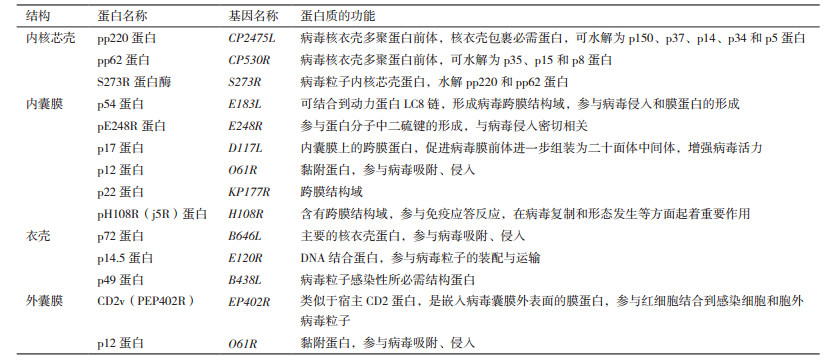
ASFV是一种有囊膜的双链DNA病毒,属于非洲猪瘟病毒科、非洲猪瘟病毒属的唯一成员,也是唯一虫媒DNA病毒[11]。家猪、野猪和软蜱均为ASFV的宿主,猪单核细胞-巨噬细胞为ASFV主要的靶细胞[12-13]。病毒基因组全长约为170-193 kb,基因组中间是一段长度约为125 kb的保守区[14-15]。基因组拥有150-167个开放阅读框(ORFs),编码150-200种蛋白质,其中约50种为结构蛋白[16],主要结构蛋白有pp220、pp62、p72、p54、p22和CD2v蛋白等。ASFV粒子由病毒类核、内核芯壳、内囊膜、衣壳和外囊膜中由内向外组装而成[16]。结构蛋白作为病毒粒子的主要成分,在ASFV吸附、侵入和复制等感染过程中起作用[16-17]。因此,本文综述了ASFV结构蛋白在病毒感染中的作用,以期为ASFV结构蛋白的进一步研究提供参考。
1 内核芯壳蛋白 1.1 pp220和pp62蛋白ASFV粒子直径为170-190 nm,是由病毒类核、内核芯壳、内囊膜、衣壳和外囊膜组成复杂的多层结构,核心结构直径为70-100 nm,包括病毒基因组、编码酶和被组装成病毒颗粒的蛋白质[16]。pp220和pp62蛋白为ASFV多聚蛋白前体,分别由CP2475L和CP530R基因编码,属于病毒感染过程中的晚期蛋白质。pp220蛋白含有多个蛋白酶水解靶基序,在Gly-Gly-X(任意)氨基酸序列水解位点,病毒编码的SUMO样S273R蛋白酶将pp220水解为成熟的病毒粒子蛋白p150、p37、p14和p34。因此,成熟的病毒粒子中不含pp220蛋白。按照相似的机理,pp62被水解为p35和p15蛋白[17],S273R蛋白酶作为ASFV编码蛋白,也是病毒粒子内核芯壳蛋白之一[18-19]。多聚蛋白水解产物主要位于病毒粒子内核芯壳,约占病毒蛋白总量的30%,是内核芯壳的主要成分,pp220、pp62的水解与病毒粒子的组装是同时进行,该类蛋白的水解产物在病毒衣壳的组装过程中起关键作用[20-21]。有学者研究发现,病毒颗粒主要聚集在靠近细胞核的离散细胞质区域,该区域又被称为“病毒加工厂”,pp220蛋白则位于“病毒加工厂”之中[22]。有学者进一步研究发现,多聚蛋白pp220和pp62的加工需要病毒衣壳蛋白p72的表达,当p72表达受阻时,未加工的多聚蛋白pp220和pp62组装成异常的拉链样结构,该结构由细长的膜结合蛋白组成。此外,pp220和pp62在COS细胞中共表达时,该两种蛋白相互作用形成拉链样结构,pp220的表达则为pp62的加工奠定了基础[19]。因此,pp220和pp62水解为p150、p37、p14、p34、p35和p15蛋白成为病毒颗粒成熟的重要标志[23-24],当pp220和pp62蛋白的水解受阻,组装的病毒颗粒则缺少内核芯壳,或者组装的病毒粒子不具有感染性[21-22]。
1.2 pp220和pp62蛋白水解产物p37蛋白作为病毒内核芯壳的组分之一,是ASFV编码的第一个细胞核-细胞质骨架穿梭蛋白,具有输出和输入活性[25]。p37是pp220的水解产物,并与SUMO-1家族的蛋白酶具有较高的氨基酸序列相似性。通过对ASFV感染的细胞中p37进行定位,发现在病毒感染的早期,p37存在于细胞核的多个不同区域;而在感染后期,其仅存在于细胞质[26]。有学者研究发现,p37蛋白N-末端和C-末端区域主动从细胞核转运到细胞质,CRM1依赖性和CRM1非依赖性核转运途径介导p37核酸转运途径,p37蛋白中疏水性氨基酸对上述3种核转运信号功能至关重要[27]。这些研究表明,p37核酸转运途径对整个ASFV复制周期起着重要作用[26]。
p34蛋白作为重要的结构蛋白,有学者研究发现,p34蛋白受胰蛋白酶的保护。同时,可从胞质溶胶和膜组分中回收被错误加工的p34蛋白[21]。p34和p150蛋白膜相关组分可组装成病毒基质结构,大多数被错误加工的p150亦可从胞质溶胶之中回收,而p150的正确加工产物则被选择性地聚集到内囊膜[23]。p35和p15蛋白主要存在于内核芯壳,其基质样结构域位于含有DNA的类核和内囊膜之间,p35、p15蛋白和pp220水解产物在病毒粒子中以等分子量形式存在[18]。p14蛋白作为pp220的又一水解产物,参与抑制病毒DNA的复制,属于早期病毒蛋白。采用免疫荧光技术发现,该蛋白存在于被ASFV感染细胞的胞质膜,p14特异性抗体介导ASFV感染细胞的补体依赖性细胞溶解和抗体依赖性细胞毒性[28]。
有学者采用蛋白质组学分析技术对pp220和pp62蛋白成熟产物进行预测,发现两种从未检测到的小蛋白p8和p5[16]。p8蛋白是含有67个氨基酸序列的成熟蛋白,该蛋白由病毒pS273R蛋白酶在pp62蛋白Gly-Gly-X(任意)序列处切割水解而成。在第158-159氨基酸位点(GGG位点),pp62蛋白被水解切割为p15蛋白和中间前体(pp46蛋白);在第463-464氨基酸位点(GGR位点),进一步水解切割为成熟的p35和p8蛋白。p35和p15蛋白易被其特异性抗体检测,但是通过免疫印迹技术发现,pp46中间前体蛋白诱导产生的p8抗体不能检测p8蛋白,其原理尚不清楚[16, 29-30]。p5蛋白为含有43个氨基酸序列的成熟蛋白,在pp220蛋白第44-45氨基酸位点(GGG位点)水解切割而成。p5蛋白与p8蛋白相比,其蛋白小,免疫原性低,可快速降解,不易被检测[16, 29-30]。有学者采用质谱(MS)分析技术进一步证实了p5和p8蛋白是病毒颗粒的结构组分[16]。总之,pp220、pp62及其水解加工产物在病毒的组装和感染过程中具有重要的功能。
2 内囊膜蛋白 2.1 p54蛋白ASFV通过受体介导的胞吞机制进入靶细胞,利用囊膜和内吞泡膜的融合机制将病毒释放到细胞浆。p54蛋白由E183L基因编码,含有跨膜结构域和Gly-Gly-X氨基酸基序,以及多种ASFV结构蛋白的加工识别序列,位于病毒颗粒的内囊膜,是ASFV非常重要的抗原蛋白[31]。有学者制备了一种具有感染性的重组ASFV,确定感染细胞内病毒的运动轨迹和速度,从而将“病毒加工厂”的形成动态实现可视化[31]。采用转染细胞瞬时表达p54蛋白,发现该蛋白主要存在于转染细胞内质网膜。当p54蛋白合成受阻,则病毒粒子的形成受抑制。这表明,在病毒蛋白经内质网膜转化成病毒包膜时,p54蛋白扮演重要角色[32]。在ASFV感染的早期阶段,p54可以激活caspase 3,进而诱导细胞凋亡,这是首次报道ASFV蛋白可以诱导细胞凋亡[33]。
在病毒感染期间,p54直接结合到动力蛋白C-末端的轻链8(LC8),进而与微管动力蛋白相互作用[34]。采用核磁共振(NMR)技术发现,p54和突触后支架蛋白是动力蛋白轻链(DYNLL1)的两个靶标,这两种蛋白与DYNLL1直接相互作用,同时谷氨酰胺对其结合起着重要作用,并且这些蛋白序列都含有GIQVD和KXTQT基序[35-36]。采用减毒毒株免疫接种动物,发现p54在诱导特异性抗体中起重要作用,p54抗体可以抑制ASFV的吸附[31, 44]。因此,在杆状病毒和大肠杆菌系统中表达的p54常被用于ASFV的血清学诊断[37-38]。曹琛福等[39]采用单克隆抗体对p54蛋白抗原表位进行鉴定,发现第23-29位、第36-45位、第72-94位、第114-120位和第137-150位氨基酸为p54蛋白抗原位点。p54和p30蛋白作为与ASFV侵入宿主细胞密切相关的结构蛋白,其在病毒感染过程中具有相似的作用。与此同时,p54和p30作为抗原蛋白,可联合用于ASFV酶联免疫吸附试验(ELISA)方法的研发,提高检测方法的灵敏度[38-40]。
2.2 pE248R蛋白及其他内囊膜蛋白pE248R蛋白是由E248R基因编码的ASFV晚期结构蛋白,是重要的内囊膜蛋白,参与蛋白分子中二硫键的形成。在病毒感染过程中,该蛋白被酰基化处理,并与病毒粒子内囊膜相连[41]。有学者发现,pE248R蛋白的缺失不影响病毒的吸附和内化,但抑制病毒粒子在宿主细胞中复制,以及病毒基因的早期和晚期表达。因此,pE248R蛋白缺陷的诱导型重组ASFV内囊膜与内体膜不发生融合作用,抑制其向细胞质核心区域转运[42]。pE248R和pE199L蛋白与痘病毒侵入/融合复合体具有很高的序列相似性,这表明该蛋白与ASFV融合机制有着密切联系[42-43]。采用免疫电子显微镜发现,pE248R、p17、p12和pH108R蛋白位于病毒内囊膜前体和“病毒加工厂”内二十面体颗粒,而非细胞表面。p17和pE183L蛋白是病毒粒子组装必需蛋白,而pE248R、p12和pE199L蛋白则与病毒侵入密切相关[20, 41, 44-45]。总之,在ASFV侵入宿主细胞后,pE248R蛋白对病毒基因的表达起重要作用。
p17蛋白作为ASFV重要的内囊膜蛋白,是病毒内囊膜上的跨膜蛋白,该蛋白可促进病毒膜前体进一步组装为二十面体中间体,以及增强病毒活力。当p17表达受阻,将抑制pp220和pp62蛋白的水解[45]。p12蛋白作为又一内囊膜蛋白,由O61R基因编码,参与病毒吸附,但是其吸附作用机制仍需进一步研究[46]。p22蛋白由KP177L基因编码,位于病毒颗粒外部,可被非离子去污剂从病毒颗粒表面上溶解处理。有学者采用冷冻切片免疫金标记技术,确定p22蛋白的亚病毒定位[47]。J5R蛋白,又名pH108R蛋白,由H108R基因编码,111个氨基酸组成,属于ASFV感染晚期表达蛋白[44]。该蛋白N-末端含有跨膜结构域,参与免疫应答反应,在病毒复制和形态发生等方面起重要作用[48]。
3 衣壳蛋白 3.1 p72蛋白p72蛋白由B646L(VP72)基因编码,是ASFV主要衣壳蛋白,是病毒二十面体衣壳的主要成分,具有高度抗原性和免疫原性。在病毒感染的晚期阶段,p72与其他相关蛋白的表达,以及形成病毒衣壳都紧密相关[16]。在病毒颗粒形成初期,p72在细胞质中富集,并与内质网膜结合,在内质网膜凸出的表面上形成病毒颗粒衣壳,在凹进的表面上形成内核芯壳[16, 49]。p72参与ASFV的吸附和侵入过程,该蛋白的表达常作为病毒复制的标志[49-51]。p72抗体可阻断病毒与巨噬细胞的结合,但是其在抗体介导的免疫保护中不起决定性作用,这或许是目前尚无有效ASF疫苗的重要原因之一[40]。有学者采用针对p72和A151R基因的小干扰RNA(siRNA)技术来控制ASFV的体外复制,研究结果发现,靶向基因p72的siRNA可以降低病毒的复制,以及信使RNA(mRNA)水平[52]。将来自不同地区的ASFV毒株p72编码基因序列进行分析发现,p72基因在不同分离株中具有高度的保守性[53]。这表明,p72蛋白有较稳定的抗原性,为ASFV抗原研究提供了良好的靶蛋白[54-57]。有学者研究发现p72蛋白N端第11-18位、第26-48位、第73-82位、第136-150位、第159-174位、第181-189位、第191-210位、第247-276位、第279-295位、第313-323位和第382-392位氨基酸为p72蛋白的抗原位点[58]。
3.2 其他衣壳蛋白p14.5蛋白,又称pE120R蛋白,是病毒衣壳蛋白之一,是DNA结合蛋白,参与抑制病毒DNA复制,是病毒粒子从“病毒加工厂”转移到胞质膜过程中的必需蛋白质。该蛋白由E120R基因编码,参与病毒粒子的胞内运输,其在病毒内核芯壳和衣壳的形成过程中起着重要作用[59]。有学者采用二维凝胶电泳(2DE)和MS技术发现,在病毒感染期间,p14.5蛋白N-末端丙氨酸(Ala)残基被乙酰化处理。与正常表达条件下比较,当pE120R蛋白表达受阻时,胞内病毒滴度未受影响,但胞外病毒滴度比对照组低100倍[59-60]。p49蛋白由B438L基因编码,属于ASFV感染晚期表达的病毒衣壳蛋白,是形成具有感染性病毒粒子的必需结构蛋白,由438个氨基酸组成,存在于“病毒加工厂”[61]。该蛋白缺乏时,组装的病毒粒子不呈二十面体对称结构,而呈异常的管状结构。在病毒感染期间,pB438L蛋白与膜结合,表现为完整的膜蛋白[61-62]。
4 外囊膜蛋白CD2v蛋白,又称pEP402R蛋白,是一种糖蛋白,类似于T淋巴细胞表面的黏附受体CD2,由信号肽、跨膜域以及147个氨基酸的胞质尾组成。在ASFV感染期间,调节性反式高尔基体网络(TGN)蛋白复合物AP-1作为CD2v蛋白的靶标。CD2v蛋白主要在T细胞和NK细胞上表达,采用共聚焦显微技术发现,大部分表达的CD2v蛋白主要位于细胞质“病毒加工厂”周围区域,而非细胞表面[63]。在缺乏细胞外配体时,绝大多数的CD2v蛋白主要位于细胞核周膜腔区域[64]。在感染后期,红细胞吸附现象表明感染细胞中CD2v蛋白得到表达。在内质网或高尔基体中,CD2v蛋白可被切割成N-末端糖基化和C-末端非糖基化两种形式。与此同时,在感染细胞中,这两种切割形式可与CD2v完整蛋白同时存在[63]。CD2v蛋白参与细胞间黏附、毒力增强和免疫应答调节过程,对ASFV的组织趋向性、免疫逃避等一系列致病机理起着重要作用[65]。CD2v蛋白可破坏淋巴细胞功能,并且该蛋白的表达与ASFV在家猪之间的传播有着密切联系。有学者采用酵母双杂交系统发现,CD2v蛋白的细胞质尾部可与细胞质接头蛋白SH3P7相互作用,而SH3P7蛋白质参与多种细胞功能调节,如囊泡转运和信号转导[64]。有学者提出,将CD2v蛋白的细胞质区域作为新的遗传标记,这一特性可用于分析来自不同区域的ASFV毒株,并追踪病毒的传播轨迹,最终建立基于蛋白质细胞外部分的全球毒株血清学分类体系[66-67],研究ASFV毒株在自然界中的多样性,并为疫苗研发奠定坚实的基础[66-68]。p12蛋白C-末端区域富含半胱氨酸的结构域,参与病毒的吸附,在病毒感染的晚期,由O61R基因表达,有学者通过免疫电子显微镜发现,该蛋白位于病毒粒子层,细胞表面的膜蛋白是ASFV的受体。在机体外,p12蛋白可阻断ASFV侵入宿主细胞,但p12蛋白抗体被动接种机体内,其不能中和ASFV,进而达到被动保护机体的作用[69]。
5 总结与展望ASF作为一种急性、烈性、出血性的猪传染性疫病,给疫情发生国家(地区)的养猪业带来严重的经济损失。我国作为对外贸易强国和养猪业大国,一旦ASF在我国完全定殖,将会给我国的养猪业带来严重损失,ASF防控形势不容乐观[70-71]。ASFV基因型较多,种类庞大,免疫逃逸机制复杂多样。ASFV编码蛋白的数量众多,其中结构蛋白作为病毒颗粒的主要组分,参与病毒的吸附、进入和复制等感染阶段。虽然相关蛋白的研究不断得到深入,但仍有大量编码蛋白的功能,以及与宿主细胞的作用机制尚不清楚,这系列问题必将成为ASFV研究的热点。国内学者先后对ASFV的致病机理、分子流行病学、诊断方法、疫苗研究等方面进行了综述[72-75],对ASFV的致病机制有了一定的认识,但仍有不足之处。因此,本文通过对参与ASFV感染的结构蛋白,以及这些蛋白和宿主细胞之间相互作用的机制进行综述,进一步阐释ASFV结构蛋白在感染过程中的作用,为我国的ASF防控提供理论参考。
| [1] |
Costard S, Mur L, Lubroth J, et al. Epidemiology of African swine fever virus[J]. Virus Res, 2013, 173(1): 191-197. |
| [2] |
Galindo-Cardiel I, Ballester M, Solanes D, et al. Standardization of pathological investigations in the framework of experimental ASFV infections[J]. Virus Res, 2013, 173(1): 180-190. |
| [3] |
Lubisi BA, Bastos ADS, Dwarka RM, et al. Molecular epidemiology of African swine fever in east Africa[J]. Arch Virol, 2005, 150(12): 2439-2452. |
| [4] |
Woźniakowski G, Kozak E, Kowalczyk A, et al. Current status of African swine fever virus in a population of wild boar in eastern Poland(2014-2015)[J]. Arch Virol, 2016, 161(1): 189-195. |
| [5] |
Misinzo G, Kwavi DE, Sikombe CD, et al. Molecular characterization of African swine fever virus from domestic pigs in northern Tanzania during an outbreak in 2013[J]. Trop Anim Health Pro, 2014, 46(7): 1199-1207. |
| [6] |
Penrith ML, Vosloo W, Jori F, et al. African swine fever virus eradication in Africa[J]. Virus Res, 2013, 173(1): 228-246. |
| [7] |
José M, Sánchez V, Lina M. African swine fever(ASF): five years around Europe[J]. Vet Microbiol, 2013, 165(1-2): 45-50. DOI:10.1016/j.vetmic.2012.11.030 |
| [8] |
Śmietanka K, Woźniakowski G, Kozak E, et al. African swine fever epidemic, Poland, 2014-2015[J]. Emerg Infect Dis, 2016, 22(7): 1201-1207. DOI:10.3201/eid2207.151708 |
| [9] |
African Swine Fever(ASF)report N°1-6[R]. OIE's World Animal Health Information Department, 2018.
|
| [10] |
Ge S, Li J, Fan X, et al. Molecular characterization of African swine fever virus, China[J]. Emerg Infect Dis, 2018, 24(11): 2131-2133. DOI:10.3201/eid2411.181274 |
| [11] |
Chapman DAG, Darby AC, Silva MD, et al. Genomic analysis of highly virulent Georgia 2007/1 isolate of African swine fever virus[J]. Emerg Infect Dis, 2011, 17(4): 599-605. |
| [12] |
Muñoz MR, Galindo I, Cuesta-Geijo Má, et al. Host cell targets for African swine fever virus[J]. Virus Res, 2015, 209: 118-127. DOI:10.1016/j.virusres.2015.05.026 |
| [13] |
Galindo I, Cuesta-Geijo MA, Hlavova K, et al. African swine fever virus infects macrophages, the natural host cells, via clathrin- and cholesterol-dependent endocytosis[J]. Virus Res, 2015, 200: 45-55. DOI:10.1016/j.virusres.2015.01.022 |
| [14] |
Linda KD, David AG, Chapman L, et al. African swine fever virus replication and genomics[J]. Virus Res, 2013, 173(1): 3-14. |
| [15] |
de Villiers EP, Gallardo C, Arias M, et al. Phylogenomic analysis of 11 complete African swine fever virus genome sequences[J]. Virology, 2010, 400(1): 128-136. |
| [16] |
Alejo A, Matamoros T, Guerra M, et al. A proteomic atlas of the African swine fever virus particle[J]. J Virol, 2018, 92(3): 1681-1690. |
| [17] |
Catharina K, Jan HF, Günther MK, et al. The intracellular proteome of African swine fever virus[J]. Sci Rep, 2018, 8(1): 1-9. DOI:10.1038/s41598-017-17765-5 |
| [18] |
Andrés G, Alejo A, Simon MC, et al. African swine fever virus protease, a new viral member of the SUMO-1-specific protease family[J]. J Bio Chem, 2001, 276(1): 780-787. DOI:10.1074/jbc.M006844200 |
| [19] |
Alí A, Germán A, María LS. African swine fever virus proteinase is essential for core maturation and infectivity[J]. J Virol, 2003, 77(10): 5571-5577. DOI:10.1128/JVI.77.10.5571-5577.2003 |
| [20] |
Salas ML, Andrés G. African swine fever virus morphogenesis[J]. Virus Res, 2013, 173(1): 29-41. |
| [21] |
Andrés G, Alejo A, Salas J, et al. African swine fever virus polyproteins pp220 and pp62 assemble into the core shell[J]. J Virol, 2002, 76(24): 12473-12482. DOI:10.1128/JVI.76.24.12473-12482.2002 |
| [22] |
Gallardo C, Blanco E, Rodríguez JM, et al. Antigenic properties and diagnostic potential of African swine fever virus protein pp62 expressed in insect cells[J]. J Clin Microbiol, 2006, 44(3): 950-956. |
| [23] |
Heath CM, Windsor M, Wileman T. Membrane association facilitates the correct processing of pp220 during production of the major matrix proteins of African swine fever virus[J]. J Virol, 2003, 77(3): 1682-1690. DOI:10.1128/JVI.77.3.1682-1690.2003 |
| [24] |
Suárez C, Salas ML, Rodríguez JM. African swine fever virus polyprotein pp62 is essential for viral core development[J]. J Virol, 2010, 84(1): 176-187. DOI:10.1128/JVI.01858-09 |
| [25] |
Eulálio A, Nunes CI, Carvalho AL, et al. Two African swine fever virus proteins derived from a common precursor exhibit different nucleocytoplasmic transport activities[J]. J Virol, 2004, 78(18): 9731-9739. DOI:10.1128/JVI.78.18.9731-9739.2004 |
| [26] |
Eulálio A, Nunes CI, Salas J, et al. African swine fever virus p37 structural protein is localized in nuclear foci containing the viral DNA at early post-infection times[J]. Virus Res, 2007, 130(1): 18-27. |
| [27] |
Eulálio A, Nunes CI, Carvalho AL, et al. Nuclear export of African swine fever virus p37 protein occurs through two distinct pathways and is mediated by three independent signals[J]. J Virol, 2006, 80(3): 1393-404. DOI:10.1128/JVI.80.3.1393-1404.2006 |
| [28] |
Kolontsov AA, Ustin AV, Shubina NG, et al. Polypeptides p14 and p31 of the African swine fever virus-early proteins located on the membrane of the infected cell[J]. Vopr Virusoli, 1992, 37(3): 165-168. |
| [29] |
Simón MC, Andrés G, Viñuela E. Polyprotein processing in African swine fever virus: a novel gene expression strategy for a DNA virus[J]. Embo Journal, 1993, 12(7): 2977-2987. DOI:10.1002/embj.1993.12.issue-7 |
| [30] |
Simón MC, Andrés G, Almazán F, et al. Proteolytic processing in African swine fever virus: evidence for a new structural polyprotein, pp62[J]. J Virol, 1997, 71(8): 5799-5804. |
| [31] |
Hernáez B, Escribano JM, Alonso C. Visualization of the African swine fever virus infection in living cells by incorporation into the virus particle of green fluorescent protein-p54 membrane protein chimera[J]. Virology, 2006, 350(1): 1-14. |
| [32] |
Rodriguez JM, Garcia ER, Salas ML, et al. African swine fever virus structural protein p54 is essential for the recruitment of envelope precursors to assembly sites[J]. J Virol, 2004, 78(8): 4299-4313. DOI:10.1128/JVI.78.8.4299-4313.2004 |
| [33] |
Hernáez B, Gema DG, Mónica GG, et al. The African swine fever virus dynein-binding protein p54 induces infected cell apoptosis[J]. FEBS Lett, 2004, 569: 224-228. DOI:10.1016/j.febslet.2004.06.001 |
| [34] |
Alonso C, Miskin J, Hernáez B, et al. African swine fever virus protein p54 interacts with the microtubular motor complex through direct binding to light-chain dynein[J]. J Virol, 2001, 75(20): 9819-9827. DOI:10.1128/JVI.75.20.9819-9827.2001 |
| [35] |
Gallardo C, Mwaengo DM, Macharia JM, et al. Enhanced discrimination of African swine fever virus isolates through nucleotide sequencing of the p54, p72, and pB602L(CVR)genes[J]. Virus Gen, 2009, 38(1): 85-95. DOI:10.1007/s11262-008-0293-2 |
| [36] |
Garcíamayoral MF, Rodríguezcrespo I, Bruix M. Structural models of DYNLL1 with interacting partners: African swine fever virus protein p54 and postsynaptic scaffolding protein gephyrin[J]. FEBS Lett, 2011, 585(1): 53-57. |
| [37] |
Cubillos C, Gómez SS, Moreno N, et al. African swine fever virus serodiagnosis: a general review with a focus on the analyses of African serum samples[J]. Virus Res, 2013, 173(1): 159-167. |
| [38] |
Barderas MG, Rodríguez F, Gómez PP, et al. Antigenic and immunogenic properties of a chimera of two immunodominant African swine fever virus proteins[J]. Arch Virol, 2001, 146(9): 1681-1691. DOI:10.1007/s007050170056 |
| [39] |
曹琛福, 梁云浩, 花群俊, 等. 非洲猪瘟病毒p54蛋白抗原表位预测及鉴定[J]. 中国预防兽医学报, 2014, 36(11): 848-851. DOI:10.3969/j.issn.1008-0589.2014.11.05 |
| [40] |
Neilan JG, Zsak L, Lu Z, et al. Neutralizing antibodies to African swine fever virus proteins p30, p54, and p72 are not sufficient for antibody-mediated protection[J]. Virology, 2004, 319(2): 337-342. |
| [41] |
Irene R, María LN, Modesto RR, et al. The African swine fever virus virion membrane protein pE248R is required for virus infectivity and an early postentry event[J]. J Virol, 2009, 83(23): 12290-300. DOI:10.1128/JVI.01333-09 |
| [42] |
Bruno H, Guerra M, María LS, et al. African swine fever virus undergoes outer envelope disruption, capsid disassembly and inner envelope fusion before core release from multivesicular endosomes[J]. Plos Pathog, 2016, 12(4): 1-32. |
| [43] |
Senkevich TG, Ojeda S, Townsley A, et al. , Poxvirus multiprotein entry-fusion complex[J]. Proc Natl Acad Sci USA, 2005, 102(51): 18572-18577. DOI:10.1073/pnas.0509239102 |
| [44] |
Brookes SM, Sun H, Dixon LK, et al. Characterization of African swine fever virion proteins j5R and j13L: immuno-localization in virus particles and assembly sites[J]. J Gen Virol, 1998, 79: 1179-1188. DOI:10.1099/0022-1317-79-5-1179 |
| [45] |
Suárez C, Gutiérrez BJ, Andrés G, et al. African swine fever virus protein p17 is essential for the progression of viral membrane precursors toward icosahedral intermediates[J]. J Virol, 2010, 84(15): 7484-7499. DOI:10.1128/JVI.00600-10 |
| [46] |
Angulo A, Viñuela E, Alcamí A. Inhibition of African swine fever virus binding and infectivity by purified recombinant virus attachment protein p12[J]. J Virol, 1993, 67(9): 5463-5469. |
| [47] |
Camacho A, Viñuela E. Protein p22 of African swine fever virus: an early structural protein that is incorporated into the membrane of infected cells[J]. Virology, 1991, 181(1): 251-257. DOI:10.1016/0042-6822(91)90490-3 |
| [48] |
孙怀昌, Dixon LK, Arkhouse RME. 非洲猪瘟病毒j5R膜蛋白的电脑预测和实验证实[J]. 中国病毒学, 1999, 14(3): 236-243. |
| [49] |
Lithgow P, Takamatsu H, Werling D, et al. Correlation of cell surface marker expression with African swine fever virus infection[J]. Vet Microbiol, 2014, 168(2-4): 413-419. DOI:10.1016/j.vetmic.2013.12.001 |
| [50] |
Sánchez EG, Quintas A, Nogal M, et al. African swine fever virus controls the host transcription and cellular machinery of protein synthesis[J]. Virus Res, 2013, 173(1): 58-75. |
| [51] |
Alexandre L, Malur A, Cornelis P, et al. Identification of a 25-aminoacid sequence from the major African swine fever virus structural protein VP72 recognised by porcine cytotoxic T lymphocytes using a lipoprotein based expression system[J]. J Virol Methods, 1998, 75(1): 113-120. DOI:10.1016/S0166-0934(98)00105-0 |
| [52] |
Keita D, Heath L, Albina E. Control of African swine fever virus replication by small interfering RNA targeting the A151R and VP72 genes[J]. Antivir Ther, 2010, 15(5): 727-736. DOI:10.3851/IMP1593 |
| [53] |
Muangkram Y, Sukmak M, Wajjwalku W. Phylogeographic analysis of African swine fever virus based on the p72 gene sequence[J]. Genet Mol Res, 2015, 14(2): 4566-4574. |
| [54] |
Chen X, Yang J, Ji Y, et al. Recombinant Newcastle disease virus expressing African swine fever virus protein 72 is safe and immunogenic in mice[J]. Virol Sin, 2016, 31(2): 1-10. |
| [55] |
李秋霞, 滕达, 童德文, 等. 非洲猪瘟病毒VP73基因主要抗原表位区的融合原核表达[J]. 中国生物工程杂志, 2010, 30(2): 60-65. |
| [56] |
靳雯雯, 杨晓红, 朱碧波, 等. 非洲猪瘟病毒VP73基因在哺乳动物细胞中的表达[J]. 中国预防兽医学报, 2014, 36(7): 574-576. DOI:10.3969/j.issn.1008-0589.2014.07.18 |
| [57] |
Sastre P, Pérez T, Costa S, et al. Development of a duplex lateral flow assay for simultaneous detection of antibodies against African and Classical swine fever viruses[J]. J Vet Diagn Invest, 2016, 28(5): 543-549. DOI:10.1177/1040638716654942 |
| [58] |
李倩, 姚淑霞. 非洲猪瘟病毒VP73蛋白的B细胞表位预测[J]. 农业科学与技术, 2008, 36(1): 7680-7682. |
| [59] |
Andrés G, Garcíaescudero R, Viñuela E, et al. African swine fever virus structural protein pE120R is essential for virus transport from assembly sites to plasma membrane but not for infectivity[J]. J Virol, 2001, 75(15): 6758-6768. DOI:10.1128/JVI.75.15.6758-6768.2001 |
| [60] |
Alfonso P, José I, Quetglas, et al. Protein pE120R of African swine fever virus is post-translationally acetylated as revealed by post-source decay MALDI mass spectrometry[J]. Virus Gen, 2007, 35(1): 81-85. DOI:10.1007/s11262-006-0015-6 |
| [61] |
Epifano C, Krijnse LJ, Salas ML, et al. Generation of filamentous instead of icosahedral particles by repression of African swine fever virus structural protein pB438L[J]. J Virol, 2006, 80(23): 11456-11466. DOI:10.1128/JVI.01468-06 |
| [62] |
Galindo I, Uela EV, Carrascosa AL. Characterization of the African swine fever virus protein p49: a new late structural polypeptide[J]. J Gen Virol, 2000, 81(1): 59-65. DOI:10.1099/0022-1317-81-1-59 |
| [63] |
Goatley LC, Dixon LK. Processing and localization of the African swine fever virus CD2v transmembrane protein[J]. J Virol, 2011, 85(7): 3294-3305. DOI:10.1128/JVI.01994-10 |
| [64] |
Jackson PC, Goatley LC, Cox L, et al. The CD2v protein of African swine fever virus interacts with the actin-binding adaptor protein SH3P7[J]. J Gen Virol, 2004, 85(1): 119-130. DOI:10.1099/vir.0.19435-0 |
| [65] |
Rowlands RJ, Duarte MM, Boinas F, et al. The CD2v protein enhances African swine fever virus replication in the tick vector, Ornithodoros erraticus[J]. Virology, 2009, 393(2): 319-328. |
| [66] |
Burmakina G, Malogolovkin A, Tulman ER, et al. African swine fever virus serotype-specific proteins are significant protective antigens for African swine fever[J]. J Gen Virol, 2016, 97(7): 1670-1675. DOI:10.1099/jgv.0.000490 |
| [67] |
Sanna G, Dei GS, Bacciu D, et al. Improved strategy for molecular characterization of African swine fever viruses from Sardinia, based on analysis of p30, CD2V and I73R/I329L variable regions[J]. Trans Emerg Dis, 2017, 64(4): 1280-1286. DOI:10.1111/tbed.2017.64.issue-4 |
| [68] |
Malogolovkin A, Burmakina G, Tulman ER, et al. African swine fever virus CD2v and C-type lectin gene loci mediate serological specificity[J]. J Gen Virol, 2015, 96(4): 866-873. |
| [69] |
Galindo I, Viñuela E, Carrascosa AL. Protein cell receptors mediate the saturable interaction of African swine fever virus attachment protein p12 with the surface of permissive cells[J]. Virus Res, 1997, 49(2): 193-204. DOI:10.1016/S0168-1702(97)00037-3 |
| [70] |
陈腾, 张守峰, 周鑫韬, 等. 我国首次非洲猪瘟疫情的发现和流行分析[J]. 中国兽医学报, 2018(9): 1831-1832. |
| [71] |
Dennis N. Arrival of deadly pig disease could spell disaster for China[J]. Science, 2018, 361(6404): 741-741. DOI:10.1126/science.361.6404.741 |
| [72] |
欧云文, 马小元, 王俊, 等. 非洲猪瘟分子病原学及分子流行病学研究进展[J]. 中国兽医学报, 2018(2): 416-420. |
| [73] |
欧云文, 阎传忠, 张杰, 等. 非洲猪瘟病毒的分子病原学及致病机理研究进展[J]. 中国畜牧兽医, 2017, 44(7): 2139-2146. |
| [74] |
王西西, 陈青, 陈鸿军, 等. 非洲猪瘟病毒免疫逃逸相关蛋白研究进展[J]. 病毒学报, 2018(6): 929-935. |
| [75] |
戈胜强, 吴晓东, 张志诚, 等. 非洲猪瘟疫苗研究进展[J]. 畜牧兽医学报, 2016, 47(1): 10-15. |