2. 中国科学院遗传与发育生物学研究所资源研究中心,北京 100101;
3. 中国科学院西北高原生物研究所,西宁 810008
2. Genetic Research Centre, Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101;
3. Northwest Institute of Plateau Biology, Chinese Academy of Sciences, Xining 810001
滩羊是蒙古羊经游牧迁徙至宁夏境内,于黄河两岸草滩世代繁育形成的优良裘皮与肉用羊[1],是我国特有的地方绵羊品种,属国家重点保护羊种之一。现主要分布于宁夏以及毗邻的甘肃、内蒙古、陕西等部分地区[2]。滩羊肉富含人体所需的脂肪酸、维生素、氨基酸等营养成分以及铁、磷、钾等矿物质[3-4],风味氨基酸含量高,肉质细嫩鲜美。与我国地方羊品种类似,滩羊存在生长发育缓慢、繁殖率低(1年平均胎产羔率102%)、尾脂和内脂沉积较多等缺点,导致养殖经济效益较低[1, 3, 5]。因此,需要利用各类先进的技术手段保持滩羊的特色性状,同时克服其自身缺点。开展对基础母羊群体的深入了解则是开展有计划改良的前提。
在众多对群体遗传结构进行分析的技术手段中,广布于基因组的微卫星标记(Microsatellite或Simple sequence repeats,SSR)能提供丰富的多态位点,易于检测、分析方便[6],是快速了解群体遗传背景的有效而经济的手段[7-9]。例如,陈仁金等通过10个高多态性微卫星位点分析宁夏白色滩羊与黑色滩羊群体,发现它们之间存在一定的遗传分化[10],但汤晓良通过10个微卫星位点分析滩羊群体内部遗传变异和亲缘关系时却发现宁夏白色和黑色滩羊群体间分化程度较低[11]。究其原因主要与取样时群体的状态和样本量、使用位点的不统一及其位点信息的多寡有关。闫路娜等[12]在种群微卫星DNA分析中发现微卫星分析群体所需样本量最低应大于30才不会对各种遗传多样性度量指标有影响;李鸥等[13]研究认为在分析群体遗传多样性时样本含量应大于40,较高多态性的微卫星位点数量应大于25。显然,针对同一品种的基因组信息使用一套数量较多和多态信息丰富的标记位点将是比较的前提和基础。
此外,微卫星标记还用来检测与性状间的关联而作为分离功能基因的手段之一,被广泛用于构建遗传连锁图谱、定位数量性状基因座、评估遗传多样性、反应品种间的遗传关系等。Pei等[14]从71个微卫星标记中筛选出17个可对牦牛进行亲子鉴定;孙业良等[15]分析微卫星标记与中国美利奴羊肉用品系体重的相关关系时,发现紧密连锁的基因座OARHH35和BMS648与体重间存在显著差异(P < 0.05),找到一个影响体重的数量性状座位(Quantitative trait locus,QTL),同时检测出6个微卫星位点与体重呈正相关。
本研究旨使用筛选的一套适用于滩羊检测的29个微卫星标记位点,检测了宁夏滩羊核心产区部分基础母羊的群体遗传多样性,分析了上述微卫星位点与滩羊8项体尺指标间的关联性,旨为滩羊的种质资源保护、群体遗传改良的辅助育种提供依据。
1 材料与方法 1.1 材料实验动物为从宁夏回族自治区盐池县滩羊种羊场群体中随机选择年龄在1-3岁范围的滩羊母羊96只。主要扩增试剂为南京诺维赞生物科技公司生产的2×Taq Master Mix预混酶,荧光引物于北京擎科生物科技有限公司合成。
1.2 方法 1.2.1 引物筛选通过文献查阅和搜寻绵羊相关基因组数据库汇集可用的绵羊微卫星标记位点,以20只左右随机选取的小群体滩羊DNA为模版,进行PCR扩增筛选。反应体系为15 μL,使用2×Taq Master Mix预混酶,采用TP-M13-SSR技术[16],通过巢式PCR扩增反应添加荧光标记,反应条件为:94℃预变性3 min,15个循环的PCR扩增(94℃变性40 s,56℃退火30 s,72℃延伸1 min),之后25个循环的荧光标记PCR(94℃变性40 s,53℃退火30 s,72℃延伸1 min)最后72℃延伸10 min,反应完成后4℃保存。对PCR结果进行3%琼脂糖凝胶电泳检测,根据扩增条带状态和多态性确定是否入选。
1.2.2 体尺测量、组织样本采集与DNA提取逐一测量每个个体的体重、体尺性状(体斜长、体高、荐高、胸围、胸深、胸宽、腰角宽和管围),同时剪取其耳组织样品,置无水乙醇中,存放于-20℃冰箱。使用酚-氯仿法提取滩羊每个个体的基因组DNA,1%琼脂糖凝胶检测纯度及浓度,原液置于-20℃冰箱内长期保存,灭菌去离子水稀释为20 ng/μL工作液,4℃冰箱保存备用。
1.2.3 SSR位点扩增与数据分析对96个滩羊个体DNA样品按照1.2.1步骤进行PCR扩增,电泳检测,带型合格的样品稀释纯化后,送北京天一辉远生物科技有限公司用ABI3730 DNA Analyzer分析获取电泳荧光数据。之后用GeneMarker V2.2.0[17]软件读取,使用基于Excel的MStools[18]进行数据格式转换及统计,并计算多态信息含量(Polymorphism information content,PIC)。分别用GENEPOP 1.32[19]分析计算上述SSR标记在群体中的期望杂合度(He)、观测杂合度(Ho)、等位基因数(Na)和有效等位基因数(Ne);用GenAlex 6.5[20]软件估算群体的遗传距离并生成主坐标分析图;STRUCTURE 2.3[21]软件和在线软件Structure Harvester(http://taylor0.biology.ucla.edu/structureHarvester/#)进行群体结构分析;TASSEL 5.0[21]软件进行性状的关联分析。
2 结果 2.1 滩羊体重、体尺测定测定的96只滩羊基础母羊的体重与体尺数据,见表 1。
利用TP-M13-SSR系统和琼脂糖电泳检测,获得29个条带清晰明亮、具有较好多态性的SSR标记,可用于滩羊群体遗传分析。用此套标记检测96个滩羊基础母羊的群体遗传背景,结果(表 2)显示,该群体的等位基因数为4-22,平均等位基因数9.5;有效等位基因数1.7-9.6,平均有效等位基因数4.5;期望杂合度0.41-0.90,平均期望杂合度0.72;观测杂合度为0.22-0.93,平均观测杂合度0.64;多态信息含量0.38-0.89,平均多态信息含量0.69,属于多样性较高的群体。
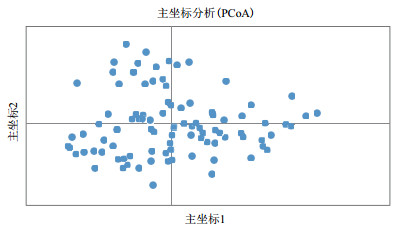
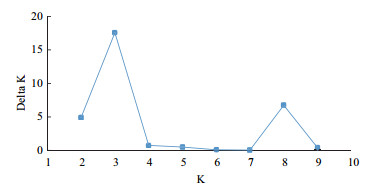
用遗传距离矩阵进行96个个体的主坐标分析(PCoA)结果见图 1。图中显示该基础母羊群个体间遗传距离分布较为均匀,不存在明显的分化。利用STRUCTURE软件基于群体基因型分布频率进行的群体结构估算显示,本实验所抽样的群体内遗传背景来源丰富,具有一定的多样性,并根据在线软件Structure Harvester计算出:当K=3时,ΔK值最大,滩羊基础母羊群体结构最佳(图 1和图 2)。
|
| 图 1 滩羊基础母羊遗传距离的主坐标(PCoA)分析结果 |
|
| 图 2 滩羊基础母群群体ΔK值折线结果 |
滩羊微卫星标记与基础母羊体尺性状的关联分析结果见表 3。结果显示,MAF33与体高、胸深存在显著关联(P < 0.05);标记BMS1788与荐高、胸围显著或极显著关联(P < 0.05,或P < 0.01);而管围与5个标记MAF33、BMS500、BL41、BMS835和BOVILS56均极显著关联(P < 0.01)。

|
| 不同颜色代表不同的K类群,X轴每个柱形代表一个滩羊个体,Y轴代表该个体遗传组成上分别属于每一个K的可能性 图 3 滩羊基础母羊群体STRUCTURE结果图 |
本研究对96只1-3岁成年滩羊种羊场母羊群体的体重和体尺进行了测定,成年母羊的体重为49.20±5.27 kg,与《中国羊品种志》中滩羊的测定数据[22]和吉帅[23]2013年对12月龄成年母羊测定的数据(41.58±4.11 kg)相比,体重较大。《中国羊品种志》数据显示,母滩羊的成年体重一般春季为35 kg,秋季为45 kg,本次测定的体重数据呈现增加态势,一方面说明随着宁夏禁牧舍饲措施的实施,舍饲状态下羊只体重比放牧状态有所提高;另一方面也说明持续实施的保种选育措施在体重性状的改良上呈现出良好的选择效应,这一结论也得到钱文熙等和周静静等[24-26]研究结果的支持。
Barker等[27]的研究表明微卫星位点最少应该有四个等位基因才可进一步利用遗传参数进行多样性评估,本研究所筛选的29个滩羊微卫星位点均具有超过4个等位基因,平均有效等位基因数4.41。从衡量基因片段多态性的多态信息含量(PIC)来看,若微卫星位点的PIC > 0.5,该微卫星位点为高度多态性位点;若0.25 < PIC < 0.5,则该位点是中度多态性;若PIC < 0.25,则属于低度多态性位点[28]。本研究筛选出的位点中,除DU264615、HUJ625、BM2504属中度多态性位点外,其他26个微卫星位点均是高度多态性位点。说明该29个滩羊微卫星位点适宜用于滩羊群体的遗传多样性和群体遗传结构的正确评估以及生产性状的相关性分析。
从对96个抽样个体的群体检测结果来看,29个位点的等位基因数与有效等位基因数相差较大,群体的平均多态信息含量值为0.691 8,说明该群体遗传多样性较为丰富。从广泛用于度量群体遗传变异的参数杂合度来看,若群体杂合度大于0.5,说明该群体未受到较高强度的选择,有丰富的多样性;反之,小于0.5,说明该群体遗传多样性较低[29]。本抽样群体的平均期望杂合度为0.722 4,平均观测杂合度0.643 4,表明该滩羊群体的遗传变异丰富。汤晓良[11]利用10个微卫星标记分析宁夏滩羊群体时测定的平均期望杂合度是0.801 4-0.814 2,而平均观测杂合度是0.269 3-0.300 0,两者差值相差较大,可能由于各个种羊场配种种公羊较少或为获得某一性状而近交导致,他依据当时滩羊繁育面临的困难局面,由此得出滩羊品质严重退化的结论,但这也可能与使用的微卫星位点数量不足和多态程度不高有关。2011年,娄渊根[30]用10个微卫星标记测定的宁夏滩羊平均期望杂合度是0.671 1,平均观测杂合度0.492 5。本研究测定的相应值高于汤、娄的结果,无论从使用的微卫星数量还是从各位点的多态程度来看,应更具客观性。
通常,由于饲养环境长期相对隔离,对于小群体羊场或农户则会造成群体内个体间的遗传距离过小,甚至群体的后代可能出自同一个家系而长期近交造成种群衰退[31]。据统计,2016年宁夏盐池县建成饲养量大于1 000只的规模化养殖场317个,大型养殖园区23个[32],区内每年以补贴机制低价调配供给各养殖场种公羊,避免了各羊场母羊群体内家系狭窄而出现的近交现象。本研究基于个体间遗传距离得出的主坐标分析图显示,滩羊基础母羊群体内不存在明显的遗传分化;从推断群体遗传结构的STRUCTURE直观分析结果来看,当K = 3时,ΔK最大,群体均一但遗传背景丰富。说明该滩羊群体既保持了丰富的遗传变异,又在很大程度上保持了品种的同质性,不会产生群体性分化而可能导致新的基因群出现[33]。
对家畜测量的体尺是生长性状的外在表现。本次用微卫星进行的性状关联分析共发现6个位点MAF33、BMS1788、BMS500、BL41、BMS835和BOVILS56与生长性状间有显著或极显著关联。通过与数据库对比发现,这些关联位点中有些在其他绵羊品种或山羊中也存在与经济性状的关联,如BMS1788与新疆细绒山羊的绒毛细度、长度和产量相关[34];BMS835与中国美利奴羊羊毛的长度及细度相关[35];而MAF33标记与眼肌面积、净肉重相关联[36]。说明这些标记可能与控制性状的QTL或主基因连锁[12]。另外,MB116、MB076和BM7109的期望杂合度分别是0.664 1、0.720 0、0.753 2,观测杂合度分别是0.276 6、0.234 0、0.215 9,期望杂合度与观测杂合度相差较大,说明这些位点的等位基因在群体内的分布并不均匀,这可能与位点受到选择作用相关,提示其与某些性状的关联性。本研究中,与标记位点的关联中既有一个标记同几个性状相关联,也有几个标记同一个性状相关联的,看似是位点存在一因多效或多因一效的现象[37]。显然,无论是连锁还是多效关联,均应是位点所在的基因片段与实际控制性状的基因相关,或者控制该性状的基因本身就位于该区域,直接或参与了该性状呈现表型的生理过程,值得后期在大规模基因组关联分析和功能基因的分离中予以重点关注。
4 结论本研究中,微卫星位点DU264615、HUJ625和BM2504是中度多态性位点,其他26个微卫星位点是高度多态性位点,而位点MB116、MB076和BM7109呈现出观测杂合度显著偏离期望杂合度的现象,说明这几个位点可能与某些性状相关联;位点MAF33、BMS1788、BMS500、BL41、BMS835和BOVILS56与生长性状间有显著或极显著关联,值得进一步研究。综合多个指标的群体遗传背景分析来看,本试验所抽样的滩羊群体其遗传变异丰富,存在较好的多样性,可供该品种内选择的范围较广;保种选育加环境改善导致基础母羊体重显著增大的实践也表明,对该滩羊群体实施品种内提纯复壮的遗传改良前景良好。所以,建立完善的谱系档案和交配记录,定期开展群体遗传距离和遗传结构的监测有利于监控种群在遗传水平上的健康情况。
| [1] |
赵有璋. 羊生产学[M]. 第2版. 北京: 中国农业出版社, 2003: 117.
|
| [2] |
杨秀芳. 宁夏盐池滩羊品种资源的保护现状及发展建议[J]. 中国草食动物, 2006(6): 36-37. DOI:10.3969/j.issn.2095-3887.2006.06.015 |
| [3] |
毛宁, 文志林, 王菊花. 宁夏盐池县滩羊产业发展面临的问题及对策[J]. 畜牧与饲料科学, 2011, 32(5): 55-56. DOI:10.3969/j.issn.1672-5190.2011.05.023 |
| [4] |
云华, 李颖康. 宁夏滩羊及其品种选育[J]. 当代畜牧, 2008(3): 41-43. |
| [5] |
侯鹏霞.滩羊羔羊早期补饲以及不同体重阶段羊肉品质的研究[D].银川: 宁夏大学, 2014. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2552354
|
| [6] |
Siju S, Dhanya K, Bhaskara, et al. Methods for development of microsatellite markers: An overview[J]. Not Sci Biol, 2014, 6(1): 1-13. |
| [7] |
David C, Quirino C, Vega W, et al. Diversity of indigenous sheep of an isolated population[J]. BMC Vet Res, 2018, 14(1): 350. DOI:10.1186/s12917-018-1682-y |
| [8] |
Tapio M, Ozerov M, Tapio I, et al. Microsatellite-based genetic diversity and population structure of domestic sheep in northern Eurasia[J]. BMC Genetics, 2010, 11(1): 1-11. DOI:10.1186/1471-2156-11-1 |
| [9] |
Erwin J, Vargas K, Blais B, et al. Genetic assessment of a big horn sheep population expansion in the Silver Bell Mountains, Arizona[J]. Peer J, 2018, 6: e5978. DOI:10.7717/peerj.5978 |
| [10] |
陈仁金, 杨章平, 汤晓良, 等. 利用微卫星标记分析滩羊群体的遗传多样性及遗传分化[J]. 扬州大学学报:农业与生命科学版, 2010, 31(4): 66-72. |
| [11] |
汤晓良.利用微卫星标记分析滩羊群体的遗传多样性与遗传分化[D].扬州: 扬州大学, 2008. http://cdmd.cnki.com.cn/article/cdmd-11117-2008086758.htm
|
| [12] |
闫路娜, 张德兴. 种群微卫星DNA分析中样本量对各种遗传多样性度量指标的影响[J]. 动物学报, 2004(2): 279-290. |
| [13] |
李鸥, 赵莹莹, 郭娜, 等. 草鱼种群SSR分析中样本量及标记数量对遗传多度的影响[J]. 动物学研究, 2009, 30(2): 121-130. |
| [14] |
Pei J, Bao P, Chu M, et al. Evaluation of 17 microsatellite markers for parentage testing and individual identification of domestic yak(Bos grunniens)[J]. Peer J, 2018, 6(11): 5946-5963. |
| [15] |
孙业良, 刘国庆, 王刚, 等. 绵羊微卫星标记与体重的相关分析[J]. 中国农业科学, 2006(10): 2095-2100. DOI:10.3321/j.issn:0578-1752.2006.10.022 |
| [16] |
牛俊巍, 李红然, 段恋, 等. 利用TP-M13-SSR技术分析我国两个地方猪种的遗传多样性[J]. 中国动物分类学报, 2013, 38(2): 413-420. |
| [17] |
Hulce D, Li X, Snyderleiby T, et al. GeneMarker genotyping software: Tools to increase the statistical power of DNA fragment analysis[J]. J Biomol Tech, 2011, 22(Suppl): 35-36. |
| [18] |
Dias P, Julier B, et al. Genetic diversity in red clover(Trifolium pratense L.)revealed by morphological and microsatellite(SSR)markers[J]. Euphytica, 2008, 160(2): 189-205. DOI:10.1007/s10681-007-9534-z |
| [19] |
Fossøy F, Johnsen A, Lifjeld JT. Multiple genetic benefits of female promiscuity in a socially monogamous passerine[J]. Evolution, 2010, 62(1): 145-156. |
| [20] |
李艳香.中国岩羊(Pseudois nayaur)遗传多样性与遗传结构研究[D].哈尔滨: 东北林业大学, 2014. http://cdmd.cnki.com.cn/article/cdmd-10225-1014402154.htm
|
| [21] |
Falush D, Stephens M, Pritchard JK. Inference of population structure using multilocus genotype data: dominant markers and null alleles[J]. Mol Ecol Notes, 2007, 7(4): 574-578. DOI:10.1111/j.1471-8286.2007.01758.x |
| [22] |
《中国羊品种志》编写组. 中国羊品种志[M]. 上海: 上海科学技术出版社, 1989.
|
| [23] |
吉帅.舍饲滩羊生长发育与肉品质变化规律的研究[D].银川: 宁夏大学, 2013. http://d.wanfangdata.com.cn/Thesis/Y2385976
|
| [24] |
钱文熙, 阎宏, 张苏江, 等. 放牧、舍饲滩羊肉质理化特性研究[J]. 黑龙江畜牧兽医, 2007(3): 37-40. DOI:10.3969/j.issn.1004-7034.2007.03.018 |
| [25] |
周静静, 马红彬, 周瑶, 等. 轮牧方式对荒漠草原滩羊牧食特征、体重及繁殖性能的影响[J]. 中国农业科学, 2017, 50(8): 1525-1534. |
| [26] |
Arthy V, Venkataramanan R, Sivaselvam SN, et al. Genetic evaluation of growth in farmers' flocks of Madras Red sheep under long-term selection in a group breeding[J]. Trop Anim Health Prod, 2018, 50(7): 1463-1471. DOI:10.1007/s11250-018-1581-z |
| [27] |
Barker J, Moore S, Hetzel D, et al. Genetic diversity of Asian water buffalo(Bubalus bubalis): microsatellite variation and a comparison with protein coding loci[J]. Animal Genetics, 2015, 28(2): 103-115. |
| [28] |
Botstein D. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32(3): 314-331. |
| [29] |
Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 1978, 89(3): 583-590. |
| [30] |
娄渊根.运用微卫星标记方法对中国绵羊遗传多样性研究[D].洛阳: 河南科技大学, 2011. http://cdmd.cnki.com.cn/article/cdmd-10464-1011284837.htm
|
| [31] |
张相伦, 游伟, 靳青, 等. 4个肉牛品种微卫星多态性分析[J]. 中国畜牧兽医, 2018, 45(1): 162-170. |
| [32] |
朱雯静, 张吉清. 宁夏盐池滩羊产业发展现状分析[J]. 现代畜牧科技, 2016(9): 3-4. |
| [33] |
李向阳, 胡成华, 赵玉民, 等. 5个群体微卫星DNA多态性的研究[J]. 吉林农业科学, 2008, 33(3): 37-40. DOI:10.3969/j.issn.1003-8701.2008.03.012 |
| [34] |
古丽尼沙·吐拉甫.新疆地方山羊品种微卫星标记遗传多态性与部分经济性状的关联性分析[D].乌鲁木齐: 新疆农业大学, 2013. http://d.wanfangdata.com.cn/Thesis_D368803.aspx
|
| [35] |
许汉峰, 赵宗胜, 李广录, 等. 绵羊13个微卫星基因座对毛用性状的相关性研究[J]. 石河子大学学报:自然科学版, 2008(4): 459-464. |
| [36] |
Margawati E, Subandriyo, Fullard K, et al. Detection of quantitative trait loci(QTL)affecting carcass traits in backcross family of indonesian thin tail sheep[J]. Genet Sel Evol, 2009, 41(1): 34-51. |
| [37] |
乌仁套迪, 斯琴图雅, 白俊艳. 阿拉善沙漠双峰驼的微卫星多态性与体尺性状的关联分析[J]. 畜牧与兽医, 2018, 50(9): 4-9. |