细胞内物质的定向运输由分子马达完成。驱动蛋白是一类非常重要的分子马达,主要以二聚体形式存在,包括马达头部、茎部、连接头部与茎部的颈链以及与“货物”相结合的尾部4个部分[1]。已知的驱动蛋白种类繁多[2-4]功能多样,不仅参与细胞内多种成分的定向运输,对大脑发育、记忆功能提升及神经元活性等也非常重要[5-6]。驱动蛋白的诸多功能均与其卷曲螺旋结构密切相关。
卷曲螺旋结构(Coiled-coil,CC)存在于多种天然蛋白质中,典型的CC由两股右手α螺旋链以左手螺旋形式相互缠绕而成,在该结构中氨基酸残基(aa)每旋转两圈形成一个七肽单元重复序列,7个aa依次表示为a、b、c、d、e、f、g,其中a、d位多为非极性疏水氨基酸常特异性地位于卷曲螺旋的内侧,e、g位多为极性带电荷氨基酸常处在疏水核心的外侧。这4个位置的氨基酸残基对于整个卷曲螺旋结构的稳定性和特异性的维持非常重要[7-8]。
NtTkr是烟草驱动蛋白超家族新成员,异于绝大多数泛表达的植物驱动蛋白,仅在幼叶、发育各时期胚及其他分裂旺盛的组织中优势表达,参与细胞周期调控,与胚、种子大小及萌发有关[9]。NtTkr尾部共有3个CC结构,CC1、CC2参与茎秆的形成,CC3识别并结合底物。本文通过卷曲螺旋结构分析和双杂交检测,探讨了NtTkr尾部CC3中T1084缺失和替换突变对CC3结构和靶蛋白相互作用的影响。
1 材料与方法 1.1 材料AH109、Y187菌株、质粒:pBI121-NtTkr、pGBKT7-NtTkrT大肠杆菌DH5α、pGBKT7、pGADT7等。
SD及缺陷培养基、X-α-Gal(Clontech公司,美国);KOD-plus高保真酶、dNTPs(2 mmol/L each)、10×KOD-plus buffer、25 mmol/L MgSO4(ToYoBo);DNA markerλ-Eco T14、6×Loading buffer、限制性内切酶、T4DNA连接酶、DNA凝胶回收试剂盒(Takara,日本);质粒提取试剂盒(TIANGEN,北京);鲑鱼精DNA(上海浩然生物技术有限公司)。
1.2 方法 1.2.1 目的基因片段的扩增与检测缺失和替换突变采用两对引物的3轮PCR来完成,两端引物为:P1:5' -cccggggatggtccctgtggaagg-3' 、P2:5' -ggatcctatgcgttcctggtaaatggc-3' ;含突变位点的中间互补引物分别为:T1084A P3:5' -ctcaacttaaagacgctgctgaagctgttc-3' 、P4:5' -gaacagcttcagcagcgtctttaagttgag-3' ;T1084d P3:5' -ctcaacttaaagacgctgaagctgttc-3' 、P4:5' -gaacagcttcagcgtctttaagttgag-3' ;P1和P2两端分别引入Sma I和Bam HI酶切位点,以pBI121NtTkr为模板,分别以P1、P3;P2、P4及P1、P3;P2、P4为引物分别进行PCR扩增,得到的产物凝胶回收后等摩尔混合,取适量作为模板,以P1、P2为引物扩增完整尾部片段。
1.2.2 诱饵表达质粒的构建和鉴定凝胶电泳回收Sma I-Bam HI线性化的pUC19和PCR产物进行连接并转化DH5α感受态细胞,涂AMP+和x-gal选择平板。Sma I-Bam HI双酶切鉴定正确重组子并送去基因测序。将pGBKT7和测序正确的pUC19T1084d,pUC19T1084A分别进行Sma I- Bam HI双酶切,回收7.3 kb的pGBKT7载体和各PCR片段,连接并转化DH5α感受态细胞,涂Kan+选择平板,Sma I-Bam HI双酶切鉴定。
1.2.3 酵母感受态细胞的转化采用乙酸锂转化法[10-11]将8 μL pGBKT7-NtTkrT/T1084d/T1084A和pGADT7-H2A/Sulfer/NT3分别加入50 μL制备的吸光度值为0.4-0.5的AH109及Y187感受态细胞,SD/-Trp或SD/-Leu平板筛选转化子。
1.2.4 重组质粒对酵母宿主的毒性作用与自激活验证按照Clontech操作说明,将重组质粒pGBKT7-NtTkrT/T1084d/T1084A和空载体pGBKT7、pGADT7-H2A/Sulfer/NT3、pGADT7分别转化感受态AH109和Y187,在SD/-Trp或SD/-Leu选择平板上分别挑取直径大于2 mm的各转化子单克隆接种至缺陷培养基:SD/-Trp、SD/-Trp/-His、SD/-Trp/-Ade、SD/-Trp-Ade/-His或SD/-Leu、SD/-Leu/-His、SD/-Leu/-Ade以及SD/-Leu/-Ade/-His,观察生长情况并检测自激活作用;同法选取上述转化子接种于50mL的SD/-Trp/+Kan或SD/-Leu/+Kan(20 μg/mL)液体培养基中,30℃,250 r/min培养16 h,测定A600,检测毒性作用。
1.2.5 酵母双杂交鉴定分别挑取直径约2 mm的含pGBKT7-NtTkrT/T1084d/T1084A的AH109和含pGADT7-H2A/Sulfer/NT3的Y187克隆,共同接种于含0.5 mL YPDA培养液的1.5 mL离心管中,涡旋1min彻底打散混匀,30℃、30-50 r/min振荡培养20-24 h后接种于SD/-Trp/-Leu平板,30℃倒置培养3-5d后挑取直径约2 mm的二倍体单克隆于100 μL灭菌双蒸水中混匀,取5 μL点样于SD/-Trp/-Leu/-His/-Ade/+X-α-Gal平板,30℃倒置5d,观察结果。
1.2.6 T1084d、T1084A点突变对蛋白互作影响的检测挑取转入T1084d、T1084A和野生(T)单菌落于适量培养基中,30℃、200 r/min过夜培养后,计数测其浓度,梯度稀释后接种适量细胞数(1 000,2 000,400个)于配制好的SD-Ade/-Trp/-His/-Leu+x-α-gal平板上,30℃培养3-5 d。
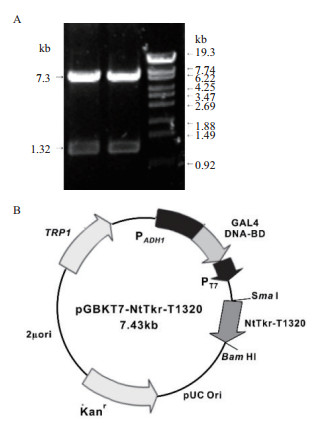
2 结果 2.1 T1084d和T1084A的获得及克隆入pUC19后的鉴定以pBI121NtTkr为模板PCR扩增1 kb和350 bp的T1083上下游片段(图 1-A)及两片段搭接为模板PCR后获得的T1084d、T1084A片段(图 1-B),与预期长度相符。将T1084d、T1084A克隆入pUC19经蓝白斑筛选获重组子进行Sma I-Bam HI双酶切鉴定得到了约1.32 kb的插入片段(图 1-C),与NtTkr尾部1 320 bp编码区符合。测序结果显示,编码第1 084位苏氨酸已经缺失或为丙氨酸替代。

|
| A:缺失和替换突变短片段PCR产物;1、2:克隆T1084d和T1084A的350 bp短片段;3:DNA标准分子量λEcoT14;4、5:克隆T1084d和T1084A的1 kb长片段;B:缺失和替换突变完整片段PCR产物;1、2:1 320 bp的T1084d和T1084A片段;3:DNA标准分子量λEcoT14;C:缺失和替换突变尾部克隆入pUC19后的酶切鉴定;1、2:SmaI-BamHI双酶切后的pUC19载体和1 320 bp的T1084d和T1084A片段;3:DNA标准分子量λEcoT14 图 1 含T1084d和T1084A的获得及克隆入pUC19后的鉴定 |
将测序正确的pUC19-T1084d、pUC19-T1084A和pGBKT7质粒均用SmaI-Bam HI双酶切,回收1.32 kb的目的片段和7.3 kb的载体片段进行连接。转化并筛选得到重组子再进行Sma I-Bam HI的双酶切鉴定,电泳结果(图 2-A)显示得到了预期的片段,表明获得了所需的T1084d和T1084A与BD融合诱饵表达质粒。
|
| A:诱饵表达质粒的酶切鉴定1、2:SmaI-Bam HI双酶切后的pGBKT7载体和1 320 bp的T1084d和T1084A片段;3:DNA标准分子量λEcoT14;B:pMXB10-NtTkr-T1320图谱,示1 320 bp的尾部插入pGBKT7的酶切位点 图 2 诱饵表达质粒pGBKT7-T1084d、pGBKT7-T1084A的酶切鉴定 |
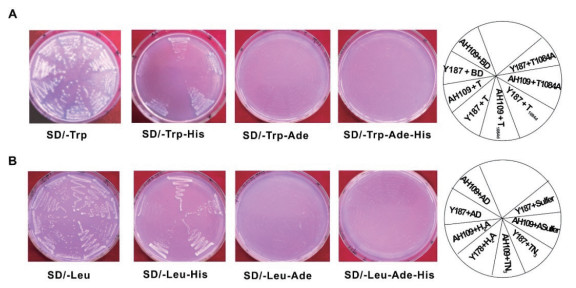
分别转化pGBKT7-NtTkrT/T1084d/T1084A和pGBKT7的AH109在SD/-Trp平板长出的单克隆(直径>1 mm)数量与分别转化pGADT7-H2A/Sulfer/NT3和pGADT7的Y187在SD/-Leu平板长出的单克隆(直径>1 mm)相似,均在45-50个之间;且含pGBKT7-NtTkrT/T1084d/T1084A的AH109(图 3-A)和含pGADT7-H2A/Sulfer/NT3的Y187(图 3-B)分别只可在单缺培养基SD/-Trp和SD/-Leu上生长,在不含His的二缺培养基上AH109菌株有轻微的自激活现象,但在加入强营养缺陷筛选标记Ade的二缺、三缺培养基上均不能生长,表明在AH109菌株中各质粒虽有His报告基因的泄漏表达,但不能激活强选择性标记基因ADE表达。表明上述诱饵和供试靶蛋白可以在SD/-Trp/-Ade/-His/-Leu+X-α-gal筛选条件下用于酵母双杂交分析。上述各转化株在含20 μg/mL Kan的液体培养基中,30℃、250 r/min培养16 h的A600均大于0.8,提示其对酵母生长无毒性,可用于酵母双杂交检测。
|
| A:BD融合的诱饵表达质粒分别转入AH109和Y187在SD/-Trp、SD/-Trp/-His、SD/-Trp/-Ade及SD/-Trp/-Ade/-His平板的生长情况;B:AD融合的靶蛋白表达质粒分别转入AH109和Y187在SD/-Leu、SD/-Leu/-His、SD/-Leu/-Ade及SD/-Leu/-Ade/-His平板的生长情况 图 3 诱饵和供试靶蛋白表达质粒转入AH109和Y187菌株的自激活实验 |
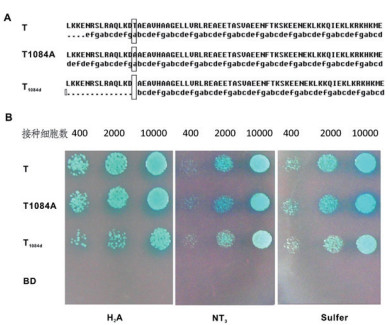
Coiled-Coiled软件分析表明NtTkr的T1084极性亲水氨基酸苏氨酸处于卷曲螺旋CC3的a位,将其替换为非极性的疏水氨基酸丙氨酸不仅增强HR结构域的疏水性,还造成了卷曲螺旋结构的增长,而T1084缺失则造成了卷曲螺旋结构的缩短(图 4-A)。上述两突变诱饵与3个候选蛋白进行蛋白互作的双杂交检测(图 4-B)显示T1084A与野生T的情况接近,但是先于T显示蓝色并深于T,T1084d则明显不如T的情况,与H2A的接合株表现尤为明显。表明将1 084位T替换为A,增强HR结构域的疏水性和七肽重复顺序的长度造成NtTKrp与候选蛋白互作的增强,而缺失T1084位缩短卷曲螺旋的长度则造成其相互作用的减弱。且对于不同的靶蛋白上述突变对其相互作用影响程度不同。
|
| A:Coiled-Coiled软件分析T、T1084A和T1084d的七肽重复顺序,显示T1084A引起卷曲螺旋结构的延长,T1084d导致其缩短;B:T、T1084A和T1084d与候选蛋白互作的酵母双杂交检测,含T、T1084A和T1084d及pGBKT7的AH109分别与含H2A、NT3和Sulfer的Y187结合后的二倍体菌株以10 000、2 000、400个细胞的接种量接种于SD/-Trp/-Leu/-His/-Ade+X-α-gal培养基上的生长情况 图 4 T、T1084A和T1084d卷曲螺旋结构分析及其与3个候选蛋白间相互作用检测 |
NtTkr尾部的CC3(1049-1143 aa)结构域中有个保守性基序LIG-FHA-1[12]。FHA(Forkhead-associated domain)结构域通过介导蛋白与蛋白之间的相互作用,直接参与信号转导、细胞周期调控和DNA损伤修复等过程[13-16],LIG-FHA-1特定苏氨酸残基的磷酸化修饰对于蛋白结合是必需的,苏氨酸残基替换则会削弱蛋白间的相互作用[17]。磷酸化途径目前已经被证实是体内驱动蛋白活性调控的一种重要手段[18-20]。NtTkr尾部1084位保守的苏氨酸的磷酸化如果也调节其与靶底物的结合,那么T1084的替换或缺失突变,必然也能引起NtKrp与候选蛋白相互作用的削弱。因此我们将野生、T1084缺失或被丙氨酸替代的尾部分别与筛选到的三个候选蛋白H2A、NT3和Sufer进行酵母细胞内互作分析,结果替换突变的互作明显强于野生,而缺失突变T1084d互作减弱,表明NtTkr与上述3个蛋白的相互作用与FHA结构域中保守苏氨酸的磷酸化修饰无关。
NtTkr卷曲螺旋结构T1084是极性亲水氨基酸苏氨酸位于七肽重复序列的a位。Lau等[21]将天然卷曲螺旋a、d位全部替换为疏水氨基酸,得到的蛋白卷曲螺旋结构比天然的稳定性更强。Monera等[22-23]研究发现丙氨酸在疏水核心中位置不同也可导致卷曲螺旋结构的改变。由此可知同Lau等一样,将处于a位的极性亲水苏氨酸替换为非极性疏水丙氨酸增强了卷曲螺旋结构的疏水性和稳定性,这可能就是T1084A最先显现蓝色,颜色深而且菌株生长最好的原因。螺旋链的长度也影响卷曲螺旋结构的稳定性[24],由图 4-A可知T1084的缺失突变使七肽重复序列缩短,因而会降低卷曲螺旋结构的稳定性,这也是T1084d生长最差,显色最浅的原因。此前本室曾将EGFP融合的NtTkr全长和尾部转入洋葱表皮,二者均分布于胞质,但转入洋葱表皮的T1084d、T1084A却在胞质和核中均有分布,而NtTkr本身并无核定位信号,其分布的改变显然受与其结合的蛋白底物的影响。
4 结论酵母双杂交证实T1084缺失或替换可影响其与靶蛋白的结合强度,NtTkr与3个候选蛋白之间的相互作用与其尾部卷曲螺旋结构有关,故它们在细胞中分布的改变很可能是NtTkr与底物相互作用的改变引起的。
| [1] |
Vale RD, Reese TS, Sheetz MP. Identification of a novel force-generating protein, kinesin, involved in microtubule-based motility[J]. Cell, 1985, 42(1): 39-50. DOI:10.1016/S0092-8674(85)80099-4 |
| [2] |
Sablin EP, Kull FJ, Cooke R, et al. Crystal structure of the motor domain of the kinesin-related motor ncd[J]. Nature, 1996, 380(6574): 555-559. DOI:10.1038/380555a0 |
| [3] |
Asbury CL, Fehr AN, Block SM. Kinesin moves by an asymmetric hand-over-hand mechanism[J]. Science, 2003, 302(5653): 2130-2134. DOI:10.1126/science.1092985 |
| [4] |
Yildiz A, Tomishige M, Vale RD, et al. Kinesin walks hand-over-hand[J]. Science, 2004, 303(5658): 676-678. DOI:10.1126/science.1093753 |
| [5] |
Vale RD, Fletterick RJ. The design plan of kinesin motors[J]. Annu Rev Cell Dev Biol, 1997, 13: 745-777. DOI:10.1146/annurev.cellbio.13.1.745 |
| [6] |
Lawrence CJ, Dawe RK, Christie KR, et al. A standardized kinesin nomenclature[J]. Journal of Cell Boilogy, 2004, 167(1): 19-22. DOI:10.1083/jcb.200408113 |
| [7] |
Yu YB. Coiled-coils:stability, specificity, and delivery potential[J]. Adv Drug Deliv Rev, 2002, 54(8): 1113-1129. DOI:10.1016/S0169-409X(02)00058-3 |
| [8] |
Mason JM, Arndt KM. Coiled coil domains:stability, specificity, and biological implications[J]. Chembiochem, 2004, 5(2): 170-176. DOI:10.1002/cbic.v5:2 |
| [9] |
Tian S, Wu J, Li F, et al. NtKRP, a kinesin-12 protein, regulates embryo/seed size and seed germination via involving in cell cycle progression at the G2/M transition[J]. Scientific Reports, 2016, 6: 35641. DOI:10.1038/srep35641 |
| [10] |
Li F, Ma JX, Ma Y, et al. hElp3 Directly modulates the expression of HSP70 gene in HeLa cells via HAT activity[J]. PLoS One, 2011, 6(12): e29303. DOI:10.1371/journal.pone.0029303 |
| [11] |
Li F, Han QJ, Lu J, et al. The Elp3 subunit of human elongator complex is functionally similar to its counterpart in yeast[J]. Mol Genet Genomics, 2005, 273(3): 264-272. DOI:10.1007/s00438-005-1120-2 |
| [12] |
李芬, 吴秀丽, 郭君丽. 新基因NtTKR及其同源物LeTKR的生物信息学分析[J]. 河南师范大学学报:自然科学版, 2014, 42(5): 110-115. |
| [13] |
Matthews LA, Selvaratnam R, Jones DR, et al. A novel non-canonical forkhead-associated (FHA) domain-binding interface mediates the interaction between Rad53 and Dbf4 proteins[J]. J Biol Chem, 2014, 31;289(5): 2589-2599. |
| [14] |
Li C, Chai J, Li H, et al. Pellino protein from pacific white shrimp Litopenaeus vannamei positively regulates NF-κB activation[J]. Dev Comp Immunol, 2014, 44(2): 341-350. DOI:10.1016/j.dci.2014.01.012 |
| [15] |
Chuang CN, Cheng YH, Wang TF. Mek1 stabilizes Hop1-Thr318 phosphorylation to promote interhomolog recombination and checkpoint responses during yeast meiosis[J]. Nucleic Acids Res, 2012, Dec; 40(22): 11416-11427. |
| [16] |
Li M, Lu LY, Yang CY, et al. The FHA and BRCT domains recognize ADP-ribosylation during DNA damage response[J]. Genes Dev, 2013, 15;27(16): 1752-1768. |
| [17] |
Machida S, Yuan YA. Crystal structure of Arabidopsis thaliana Dawdle forkhead-associated domain reveals a conserved phospho-threonine recognition cleft for dicer-like 1 binding[J]. Mol Plant, 2013, 6(4): 1290-1300. DOI:10.1093/mp/sst007 |
| [18] |
DeBerg HA, Blehm BH, Sheung J, et al. Motor domain phosphorylation modulates kinesin-1 transport[J]. J Biol Chem, 2013, 288(45): 32612-32621. DOI:10.1074/jbc.M113.515510 |
| [19] |
Phang HQ, Hoon JL, Lai SK, et al. POPX2 phosphatase regulates the KIF3 kinesin motor complex[J]. J Cell Sci, 2014, 127(Pt 4): 727-739. |
| [20] |
Ems-McClung SC, Hainline SG, Devare J, et al. Aurora B inhibits MCAK activity through a phosphoconformational switch that reduces microtubule association[J]. Curr Biol, 2013, 23(24): 2491-2499. DOI:10.1016/j.cub.2013.10.054 |
| [21] |
Lau SYM, Taneja AK, Hodge RS. Synthesis of a model protein of defined secondary structure and quaternary structure[J]. J Biol Chem, 1984, 259: 13253-13261. |
| [22] |
Monera OD, Sonnichsen FD, Hicks L, et al. The relative positions of alanine residues in the hydrophobic core control the formation of two-stranded or four-stranded α-helical coiled-coils[J]. Protein Eng, 1996, 9: 353-363. DOI:10.1093/protein/9.4.353 |
| [23] |
Monera OD, Zhou NE, Lavigne P, et al. Formation of parallel and antiparallel coiled-2coils controlled by the relative positions of alanine residues in the hydrophobic core[J]. J Biol Chem, 1996, 271: 3995-4001. DOI:10.1074/jbc.271.8.3995 |
| [24] |
Litowski JR, Hodges RS. Designing heterodimeric two-stranded alpha-helical coiled-coils:the effect of chain length on protein folding, stability and specificity[J]. Journal of Peptide Research Official Journal of the American Peptide Society, 2010, 58(6): 477-492. |




