2. 中国农业科学院生物技术研究所,北京 100081
2. Biotechnology Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081
土地盐渍化严重制约农业生产,导致世界范围内作物产量受损。据统计,全球有50%浇灌地和20%农业用地遭受不同程度盐害[1]。我国盐渍土分布广泛,大约占全球盐渍土面积的1/10,严重影响我国农业的发展[2]。玉米是世界三大粮食作物之一,在中国种植面积居世界第二位,在国民经济中占有重要地位[3]。玉米是中度盐敏感植物,耐盐能力比较低,因此限制了玉米种植面积和产量[4]。随着生物技术的不断发展,采用转基因技术培育抗逆转基因玉米已经成为提高玉米抗逆性的有效途径之一[5]。
植物中bZIP类转录因子参与多种生物过程和对干旱、盐渍和极端温度等逆境胁迫的反应[6-7]。中国农业科学院生物技术研究所课题组前期利用酵母单杂交方法从玉米中克隆了一个可与过氧化氢酶基因Cat1[8]上游顺式作用元件ABRE2相互作用的bZIP类转录因子基因ABP9[9]。通过对诸多转ABP9植物研究表明,该基因可以大幅度提高植物对盐渍等多种非生物胁迫的耐性[10-14]。
ABP9过表达可以提高拟南芥、水稻等植物的抗逆性,但在玉米中的研究还知之甚少。为了改良玉米品种的耐盐性和进一步研究ABP9过表达提高植物耐盐性的调控机制,中国农业科学院生物技术研究所课题组创制了郑58背景由逆境诱导型启动子Pabp9[15]驱动ABP9表达的转基因玉米株系。旨在通过研究其在NaCl胁迫下的生理和基因表达变化,分析其耐盐性和初步探讨其耐盐分子调控机制,为后续研究工作提供依据。
1 材料与方法 1.1 材料试验材料为郑58背景由逆境诱导型启动子Pabp9驱动ABP9表达的转基因玉米株系,对照为同一遗传背景的转基因阴性株系,种子均系中国农业科学院实验室保藏。
1.2 方法 1.2.1 转基因株系分子鉴定采用盆栽法(蛭石:营养土=1:1)于人工气候室内种植,温度27℃(±3℃),光周期16 h/8 h,光照强度500-1 000 μmol/m2·s1,相对湿度为40%-50%。三叶期取叶片保存于-80℃备用。
(1)基因组DNA提取及PCR检测玉米叶片基因组DNA提取使用新型植物基因组DNA提取试剂盒(天根生化科技有限公司)。
使用特异引物ABP9-Fw:5'-CGAAGAAAAAG-AAGCTGGTGGAG-3',Tnos-Rv:5'-TGCGGGACTCT-AATCATAAAAACC-3',对转基因ABP9株系进行PCR检测。
PCR反应程序为95℃ 5 min;95℃ 30 s,60℃ 40 s,72℃ 40 s,32个循环;72℃ 10 min。经电泳检测PCR产物长度为582 bp的即为阳性植株。
(2)Southern blot分析采用CTAB法大量提取玉米基因组DNA,探针合成及杂交试剂盒均购自罗氏。具体方法参照试剂盒说明及《分子克隆实验指南·第三版》
(3)植物总RNA提取及qRT-PCR分析采用RNAprep Pure植物总RNA提取试剂盒(天根生化科技有限公司)提取植物总RNA,参照TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix试剂盒(北京全式金生物技术有限公司)反转录成cDNA,用7500荧光定量PCR仪检测相关基因的表达,反应程序为95℃ 30 s;95℃ 15 s,60℃ 15 s,72℃ 30 s,40个循环;采用2-∆∆Ct方法分析基因的表达水平。引物如下:
ABP9-Fw:5'-AGGAAGCAGGCGTATACGGT-3',
ABP9-Rv:5'-TCACCACATGGCGCTGC-3'。
内源参照基因Actin引物为:
Actin-Fw:5'-ACCTCACCGACCACCTAATG-3',
Actin-Rv:5'-CTGAACCTTTCTGACCCAAT-3'。
1.2.2 生理指标测定挑选籽粒饱满程度一致的种子用5%次氯酸钠溶液消毒20 min,去离子水冲洗干净,种于蛭石中。生长至一叶期时在温室中用1/2 Hoagland营养液进行水培,光周期、温度及湿度与1.2相同。两叶期时逐渐增加营养液浓度至全营养液培养,每天气泵增氧2 h,每隔2 d更换一次营养液。三叶期开始盐胁迫处理,为了防止盐应激,每隔12 h增加50 mmol/L NaCl,直至终浓度150 mmol/L NaCl,对照组全营养液培养。处理7 d,期间观察表型及测量各项生理指标。
(1)叶绿素含量测定采用乙醇丙酮法测定叶绿素含量[16]。
(2)叶绿素荧光参数的测定挑选长势一致的幼苗,选取相同叶位功能叶,固定叶夹,避光20 min,使用便携式植物荧光检测仪(Handy PEA,Hansatech Instrument Ltd,UK)测PSII的最大光化学效率(The maximal efficiency of PSII photochemistry,Fv/Fm),测定光源为波长650 nm、光强3 000 μmol/m2·s1的红光,荧光信号的记录时长为2 s。计算公式为Fv/Fm=(Fm-Fo)/Fm,具体生理意义参照王立丰等的研究结果[17]。
(3)叶片相对含水量测定三叶期水培苗直接用150 mmol/L NaCl处理12 h,恢复生长3 h后,剪取第二片全展叶1-2 cm称取鲜重(Fresh Weight,FW),将叶片放入去离子水中浸泡至恒重(Total Weigh,TW)。再将叶片放入烘箱105℃杀青10 min,80℃烘干至恒重(Dry Weight,DW)。叶片相对含水量(%)=(FW-DW)/(TW-DW)×100%。
(4)游离脯氨酸及可溶性糖含量测定脯氨酸含量测定参照脯氨酸(PRO)测试盒(南京建成生物工程研究所),可溶性糖测定参照植物可溶性糖含量检测试剂盒提供的方法(北京索莱宝生物科技有限公司)。
(5)丙二醛(Malondialdehyde,MDA)含量测定测定方法参照植物MDA试剂盒提供的方法(南京建成生物工程研究所)。
(6)相对电导率测定三叶期水培苗用150 mmol/L NaCl处理3 d时,将幼苗叶片用超纯水中冲洗3次,使用打孔器剪取叶片,置于装有10 mL超纯水的离心管中,抽真空10 min,让叶片全部沉入管底,室温静置1 h,摇匀后测初始电导值(E1)。沸水浴10 min以杀死植物组织,流水冷却至室温,摇匀后次测定终电导值(E2)。相对电导率计算公式为相对电导率(RE)=E1 /E2。
(7)抗氧化酶活性测定超氧化物歧化酶(Superoxide dismutase,SOD)、过氧化物酶(Peroxidase,POD)及过氧化氢酶(Catalase,CAT)活性测定参照试剂盒提供的方法(南京建成生物工程研究所)。粗酶液制备方法为称取0.5 g叶片,加2 mL磷酸盐缓冲液(0.1 mol/L,pH=7.4),冰水浴条件下加入石英砂研磨,离心取上清。
1.2.3 转录组测序用150 mol/L NaCl处理三叶期玉米幼苗,取处理0 h、15 min、30 min、1 h、2 h、3 h、6 h、9 h和12 h的玉米叶片,液氮速冻后-80℃保存。用RNAprep Pure植物总RNA提取试剂盒(天根生化科技有限公司)提取RNA,由诺禾致源测序公司完成测序。利用Tophat2软件分析测序后的序列信息,并且只保留比对到基因组上单一位点的序列进行后续分析。利用GenomicFeatures和GenomicAlignments软件包统计比对到参考基因上的序列数目。然后用DESeq2软件包进行样本间的归一化,并且计算基因的表达量(FPKM值)。样本之间的差异基因也是利用DESeq2软件包完成。
基因的GO富集分析利用在线分析软件agriGO完成。每一个条目中允许的最少基因数为5,Fisher精确检验用来检测输入的基因列表与背景基因(AGPv3,Ensembl release 30)之间的差异。采用Benjamini-Yekutieli方法校正P值,最终取FDR < 0.05作为最终显著性检验的标准。对筛选的部分差异表达基因进行qRT-PCR验证,方法同1.2.3,引物见表 1。
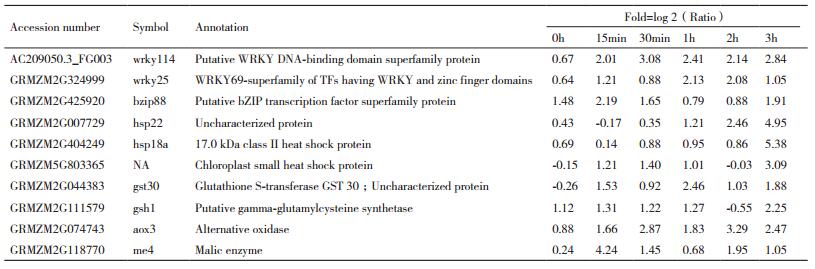
利用特异性引物进行PCR检测,植株均能扩增出特异性条带(图 1-A)。根据T-DNA片段所在区域,选用HindⅢ和EcoRI酶切后,进行southern blot分析,结果(图 1-C)显示为单个拷贝插入。
|
| A:PCR;B:Southern blot;C:qRT-PCR;Plasmid:ABP9表达载体质粒DNA;Neg:转基因阴性植株;ABP9:转ABP9基因阳性植株 图 1 转基因植株分子鉴定 |
利用qRT-PCR检测ABP9在转基因及非转基因植株中的表达水平,转基因植株中ABP9表达量高于阴性对照(图 1-B)。结果表明,ABP9以单拷贝形式整合到转基因玉米基因组中,并得到较高水平表达。
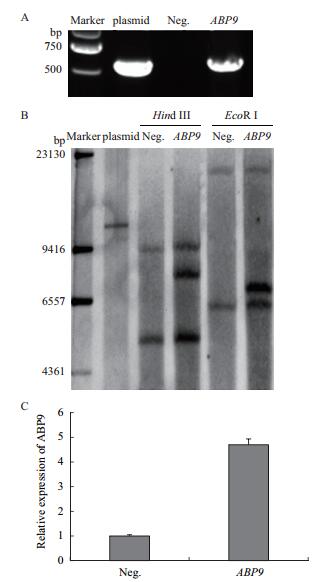
2.2 表型观察及叶绿素含量测定盐处理至第3 d时,阴性植株第一片真叶失绿枯萎,而转基因植株仍能保持较鲜绿状态(图 2-A)。分别测定处理3 d的第一片真叶(图 2-B)和处理7 d第二片全展叶(图 2-C)的叶绿素含量,结果显示,与对照组相比,处理组叶绿素含量均有下降,但转基因植株叶绿素含量均极显著高于阴性植株(P < 0.01)。
|
| A:盐处理3 d的叶片表型;B:盐处理3 d的叶片叶绿素含量;C:盐处理7 d的叶片叶绿素含量。Control:Hoagland营养液正常培养。数值代表 3个重复的平均值±标准差(t-test,*:P < 0.05;**:P < 0.01)。下同 图 2 NaCl胁迫对玉米幼苗叶绿素含量的影响 |
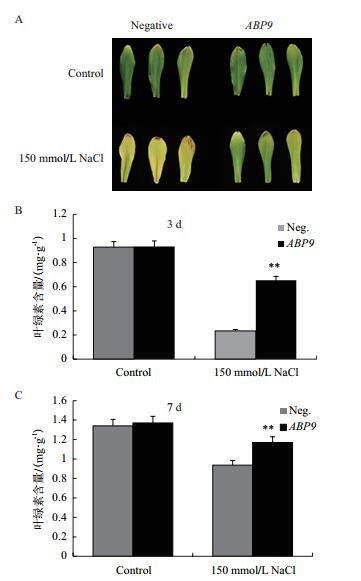
叶绿素荧光参数是植物耐盐性分析中一个重要指标,测定NaCl胁迫下玉米幼苗PSII最大光能转化效率(Fv/Fm)的变化。随着胁迫时间延长,玉米幼苗Fv/Fm逐渐下降。当处理至第5天和第7天时,转基因植株Fv/Fm显著高于阴性植株(图 3)。
|
| 图 3 NaCl胁迫对玉米幼苗Fv/Fm的影响 |
在150 mmol/L NaCl胁迫下,转基因与阴性幼苗叶片逐渐失水萎蔫,但两者间无显著差异。恢复生长3 h后叶片含水量逐渐上升,但转基因植株叶片含水量极显著高于阴性植株(P < 0.01)(图 4)。
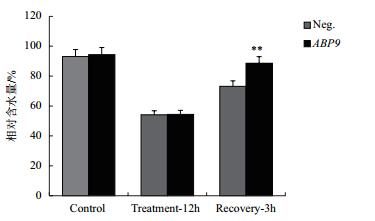
|
| Control:Hoagland营养液正常培养;Treatment-12h:150 mmol/L NaCl处理12 h;Recovery-3h:盐处理后正常培养3 h 图 4 NaCl胁迫对玉米幼苗叶片相对含水量的影响 |
盐胁迫条件下,植物常通过有机物质的积累来维持较高的细胞质渗透压。通过测定脯氨酸和可溶性糖含量的变化,发现两种渗透调节物质随处理时间增长,呈先上升后下降趋势,但转基因植株的脯氨酸(图 5-A)和可溶性糖(图 5-B)含量显著高于阴性植株。
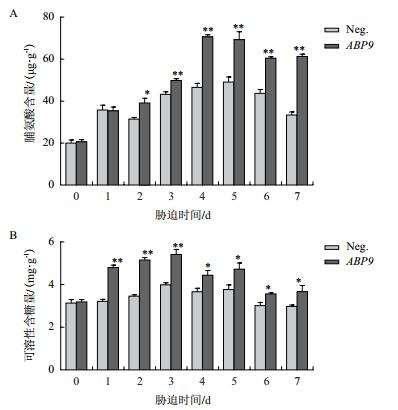
|
| 图 5 NaCl胁迫对玉米幼苗脯氨酸(A)和可溶性糖(B)含量的影响 |
盐胁迫导致植物细胞受到伤害发生膜脂过氧化作用,细胞膜透性增加,MDA含量和电导率上升。如图 6所示,随胁迫时间增加,丙二醛含量呈先上升后下降趋势,但阴性植株丙二醛含量要高于转基因植株(图 6-A),在多个时间点达到显著水平。处理至3 d时,阴性植株叶片相对电导率显著高于转基因植株(图 6-B)。
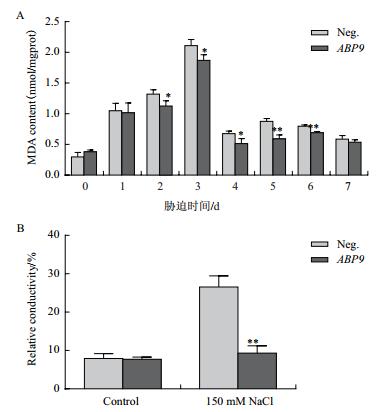
|
| 图 6 NaCl胁迫对玉米幼苗丙二醛含量(A)和电导率(B)的影响 |
逆境可使植物体内产生大量活性氧,而SOD、POD和CAT是清除除活性氧的重要抗氧化酶,其活性上升是提高玉米抗逆能力的重要因素。如图 7所示,受到盐胁迫后,转基因与阴性植株叶片中的SOD、POD和CAT均呈先上升后下降的趋势,但转基因植株中抗氧化酶活性显著高于阴性植株。
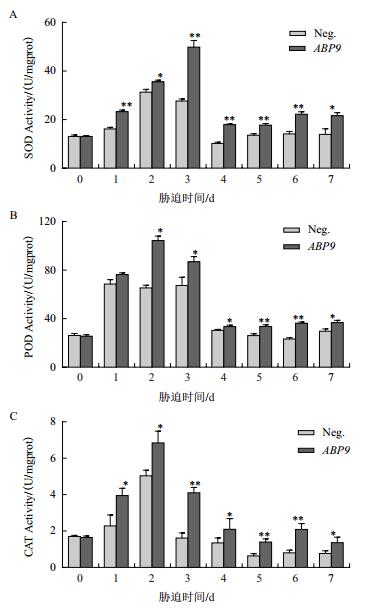
|
| 图 7 NaCl胁迫对玉米幼苗SOD(A)、POD(B)和CAT(C)活性的影响 |
对转基因和阴性玉米的三叶期幼苗盐胁迫处理0 h、15 min、30 min、1 h、2 h、3 h、6 h、9 h和12 h的叶片进行RNA-seq分析。首先比较各个时间点中转基因和阴性玉米的基因表达差异,将各时间点的所有差异基因合并并去除重复基因,共得到2 139个差异表达基因,这些基因是受ABP9调控的基因。
同时,利用转基因阴性植株的RNA-seq数据挖掘其中响应盐胁迫的基因,将盐处理各个时间点的转录组数据分别与0 h的数据进行比较,合并所有差异表达的基因之后共得到2 453个基因。将两组差异基因作交集后得到1 075个基因,这些基因为受ABP9调控的且响应盐胁迫的基因。将这些基因进行GO功能富集分析,发现这些基因显著富集在响应非生物胁迫及活性氧清除等生物学过程中。
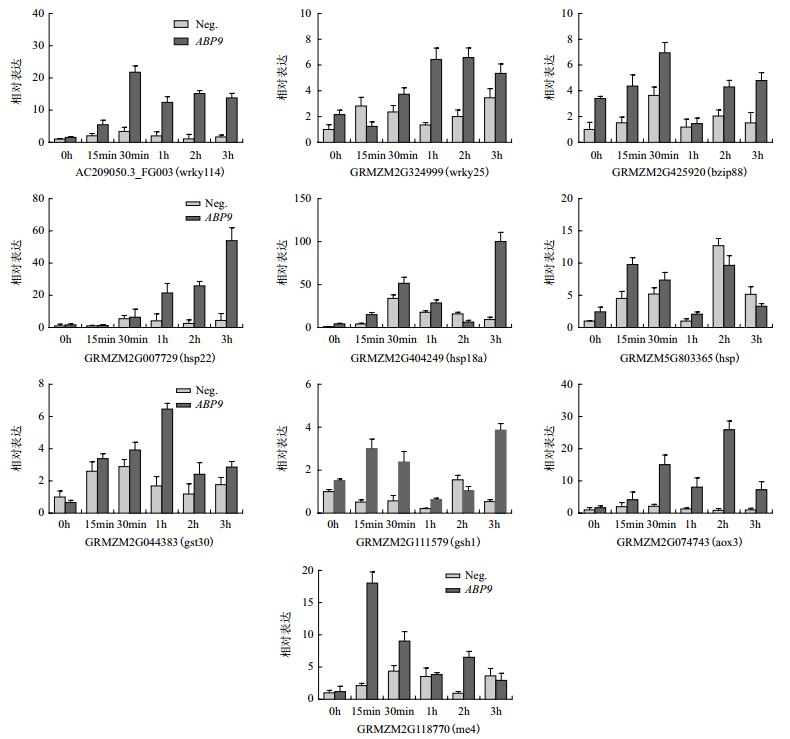
选取其中一些响应盐胁迫上调表达且在转基因和对照植株中表达差异倍数≥2的基因(表 2)进行qTR-PCR验证(图 8),结果显示,与转录组测序结果基本一致,这些基因均是盐诱导基因,且其胁迫过程中的表达在转基因植株中相比阴性对照明显提高。
|
| 图 8 盐胁迫下转基因植株中差异表达基因qRT-PCR鉴定 |
近年来,随着转基因技术的不断发展,应用该技术对农作物改良已成为培育具有优良性状新品种的有效途径[18]。已有诸多报道成功获得转基因植株,并在提高其耐盐性等方面有显著成效[19-20]。在前期工作的基础上,创制了郑58背景的转ABP9玉米株系,通过PCR检测和Southern blot分析证明ABP9已成功整合到玉米基因组中,且为单拷贝形式插入。qRT-PCR分析结果显示ABP9在转基因株系中表达量较高。
当植物遭受盐胁迫时,可在生理及分子水平快速响应,通过一系列的生理生化反应适应和抵御盐害。叶绿素是对盐胁迫敏感的光合色素,盐胁迫会加速植物叶绿素降解,降低叶绿体对光的吸收,从而导致光合速率下降[21]。本研究结果显示,盐胁迫后转基因和阴性对照玉米叶片的叶绿素含量下降,说明长时间高浓度的盐胁迫导致叶绿素大量分解。但转基因植株叶绿素含量显著(P < 0.01)高于阴性植株,说明前者具有较强的抵御叶绿素降解的能力,进而保持了较高的光合能力。通过叶绿素荧光参数的变化可以了解植物光系统对光能的吸收、耗散等一系列反应,能够反映植物受逆境影响的情况[22]。研究表明,盐胁迫会破坏PSII、抑制光反应活性,进而导致植物光合能力降低[23]。本研究Fv/Fm测定结果显示,随盐胁迫时间增加,Fv/Fm逐渐下降,但在胁迫第5天、第7天时转基因植株显著(P < 0.05)高于阴性植株,说明虽然转基因和阴性对照玉米植株光反应活性均受到抑制,但转基因植株的PSII活性中心受损程度较小,且具有较高光反应活性,从而保持了较高的光能捕获能力与转化效率。进一步说明盐胁迫下,转基因玉米植株的光合能力要高于阴性植株。
盐胁迫对植物的危害来源之一就是渗透胁迫。高盐导致环境渗透势降低,抑制植物水分吸收或致使植物失水,造成植物生理干旱。本研究中,盐胁迫后玉米叶片相对含水量下降,转基因植株与阴性株之间无显著差异,但恢复生长后,转基因植株能够快速吸收水分,叶片相对含水量高于阴性植株(图 4),说明其具有较高的恢复能力,推测这可能与渗透调节物质积累有关。研究表明,盐胁迫后植物细胞常通过积累一些有机小分子物质(如脯氨酸、可溶性糖等)来维持较高的细胞质渗透压[24]。本研究中玉米叶片中脯氨酸和可溶性糖含量受到胁迫后上升,且转基因植株始终高于阴性。由此可见,遭受盐胁迫后转基因植株可以积累更多的有机调节物质,从而提高自身对高盐胁迫的耐受性。
盐胁迫导致植物体内产生大量活性氧,造成膜脂过氧化,破坏细胞膜完整性[25]。MDA是膜脂过氧化作用的产物之一,而相对电导率能够代表细胞膜透性大小,二者可以反映植物细胞膜的损伤程度[26]。本研究的结果显示,受到盐胁迫后,玉米叶片中MDA含量上升,在第3天时达到最高,但转基因植株低于阴性;测定3 d时叶片相对电导率发现转基因植株膜透性也低于阴性株,说明在盐胁迫条件下转基因植株细胞膜损伤程度较小。
逆境胁迫下,植物细胞的存活与活性氧清除能力密切相关,而抗氧化酶的存在是清除活性氧的重要机制。Vijayalakshmi等[27]和Farhangi-Abriz等[28]研究表明,盐胁迫下植物通过提高抗氧化酶活性,能够保护植物细胞免受氧化伤害,提高了植物的耐盐能力。本研究测定了SOD、POD和CAT活性,在遭受盐胁迫后抗氧化酶活性上升,且转基因植株高于阴性植株,说明前者具有更高的活性氧清除和降低膜脂过氧化的能力。此外,有研究表明,脯氨酸等一些渗透调节物质也和植物抗氧化有关,不仅能够维持细胞质渗透压,还起到保护抗氧化酶、增强植物蛋白稳定性的作用[29-30],因而本研究中转基因植株中脯氨酸含量的提高也使转基因植株具有更高的抗氧化能力。
本研究通过RNA-seq和qRT-PCR分析发现,与阴性植株相比,转基因植株中包括转录因子、热激蛋白,抗氧化酶等大量盐诱导基因的表达显著增强。例如,WRKY转录因子,大量研究表明,其参与植物多种生理生化过程,包括应对多种非生物胁迫、参与叶片衰老及次生代谢等[31-34]。研究中发现,在受到盐胁迫后,转基因株系中WRKY基因AC209050.3_FG003的表达能在短时间内迅速升高,而在阴性材料中表达量变化幅度较小。热激蛋白作为分子伴侣,能够防止蛋白质降解,保护细胞免受伤害,相关研究表明其在植物抗逆方面发挥着重要作用[35-37]。结果显示,热激蛋白基因GRMZM2G007729在转基因株系中高表达,且上升幅度较大。此外,还有一些与抗氧化相关基因,如GRMZM2G044383、GRMZM2G074743等,在转基因植株中的表达显著高于阴性植株。研究表明,这些基因在活性氧清除方面起重要作用[38-39]。因此,上述抗逆基因的增强表达很可能是导致转基因玉米植株耐盐性提高的关键。
4 结论ABP9的增强表达提高了大量抗逆相关基因的表达,导致盐胁迫下其抗氧化能力、渗透调解能力、膜完整性和光合能力的提高。
| [1] |
Zorb C, Herbst R, Forreiter CEA. Short-term effects of salt exposure on the maize chloroplast protein pattern[J]. Proteomics, 2009, 9(17): 4209-4220. DOI:10.1002/pmic.v9:17 |
| [2] |
张金林, 李惠茹, 郭姝媛, 等. 高等植物适应盐逆境研究进展[J]. 草业学报, 2015(12): 220-236. DOI:10.11686/cyxb2015233 |
| [3] |
李倩, 王帅, 隋颂扬, 等. 农杆菌介导的ZmRLK基因转化玉米的分析[J]. 分子植物育种, 2017(1): 150-154. |
| [4] |
Soares A, Geilfus CM, Carpentier SC. Genotype-specific growth and proteomic responses of maize toward salt stress[J]. Front Plant Sci, 2018, 9: 661. DOI:10.3389/fpls.2018.00661 |
| [5] |
Yamaguchi T, Blumwald E. Developing salt-tolerant crop plants:challenges and opportunities[J]. Trends Plant Sci, 2005, 10(12): 615-620. DOI:10.1016/j.tplants.2005.10.002 |
| [6] |
Lindemose S, O'Shea C, Jensen MK, et al. Structure, function and networks of transcription factors involved in abiotic stress responses[J]. Int J Mol Sci, 2013, 14(3): 5842-5878. DOI:10.3390/ijms14035842 |
| [7] |
Sornaraj P, Sukanya LSLM. Basic leucine zipper (bZIP) transcription factors involved in abiotic stresses A molecular model of a wheat bZIP factor and implications of its structure in function[J]. Biochimica et Biophysica Acta (BBA) -General Subjects 2016, 1860(1): 46-56. |
| [8] |
John G, Guan LQ, Polidoros, et al. Catalases in plants:Gene structure, properties, regulation, and expression[J]. Oxidative Stress and the Molecular Biology of Antioxidant Defenses, 1997, 343-406. |
| [9] |
Wang L, Zhao J, Fang YL. Gene cloning and function analysis of ABP9 protein which specifically binds to ABRE2 motif of maize Cat1 gene[J]. Chinese Science Bulletin, 2002, 47(22): 1871-1875. DOI:10.1360/02tb9409 |
| [10] |
王昌陵, 赵军, 李英慧, 等. 转录因子ABP9转化大豆(Glycine max L.)及遗传转化条件优化[J]. 中国农业科学, 2008, 41(7): 1908-1916. DOI:10.3864/j.issn.0578-1752.2008.07.005 |
| [11] |
Zhang X, Wollenweber B, Jiang D, et al. Water deficits and heat shock effects on photosynthesis of a transgenic Arabidopsis thaliana constitutively expressing ABP9, a bZIP transcription factor[J]. J Exp Bot, 2008, 59(4): 839-848. DOI:10.1093/jxb/erm364 |
| [12] |
张磊, 吴金霞, 董芳, 等. 抗逆转ABP9基因黑麦草和高羊茅植株的鉴定[J]. 草业科学, 2010, 27(7): 72-77. |
| [13] |
Zhang X, Wang L, Meng H, et al. Maize ABP9 enhances tolerance to multiple stresses in transgenic Arabidopsis by modulating ABA signaling and cellular levels of reactive oxygen species[J]. Plant Mol Biol, 2011, 75(4/5): 365-378. |
| [14] |
Wang C, Lu G, Hao Y, et al. ABP9, a maize bZIP transcription factor, enhances tolerance to salt and drought in transgenic cotton[J]. Planta, 2017, 246(3): 453-469. DOI:10.1007/s00425-017-2704-x |
| [15] |
邹维华. ABP9启动子克隆、功能鉴定和编码区序列转录后6个核苷酸插入修饰分析[D].北京: 中国农业科学院研究生院, 2008.
|
| [16] |
张宪政. 植物叶绿素含量测定——丙酮乙醇混合液法[J]. 辽宁农业科学, 1986, 3(3): 26-28. |
| [17] |
王立丰, 王纪坤. 叶绿素荧光动力学原理及在热带作物研究中的应用[J]. 热带农业科学, 2013, 33(11): 16-23. DOI:10.3969/j.issn.1009-2196.2013.11.005 |
| [18] |
Yadava P, Abhishek A, Singh R, et al. Advances in maize transformation technologies and development of transgenic maize[J]. Front Plant Sci, 2016, 7(421): 1949. |
| [19] |
Kumar M, Choi J, An G, et al. Ectopic expression of OsSta2 enhances salt stress tolerance in rice[J]. Front Plant Sci, 2017, 8: 316. |
| [20] |
Lescano CI, Martini C, Gonzalez CA, et al. Allantoin accumulation mediated by allantoinase downregulation and transport by Ureide Permease 5 confers salt stress tolerance to Arabidopsis plants[J]. Plant Mol Biol, 2016, 91(4/5): 581-595. |
| [21] |
Mittal S, Kumari N, Sharma V. Differential response of salt stress on Brassica juncea:photosynthetic performance, pigment, proline, D1 and antioxidant enzymes[J]. Plant Physiol Biochem, 2012, 54: 17-26. DOI:10.1016/j.plaphy.2012.02.003 |
| [22] |
Morant-Manceau A, Pradier E, Tremblin G. Osmotic adjustment, gas exchanges and chlorophyll fluorescence of a hexaploid triticale and its parental species under salt stress[J]. J Plant Physiol, 2004, 161(1): 25-33. DOI:10.1078/0176-1617-00963 |
| [23] |
Mehta P, Jajoo A, Mathur S, et al. Chlorophyll a fluorescence study revealing effects of high salt stress on Photosystem Ⅱ in wheat leaves[J]. Plant Physiol Biochem, 2010, 48(1): 16-20. DOI:10.1016/j.plaphy.2009.10.006 |
| [24] |
Hajlaoui H, Ayeb N, Garrec JP, et al. Differential effects of salt stress on osmotic adjustment and solutes allocation on the basis of root and leaf tissue senescence of two silage maize (Zea mays L.) varieties[J]. Industrial Crops and Products, 2010, 31: 122-130. DOI:10.1016/j.indcrop.2009.09.007 |
| [25] |
Horie T, Karahara I, Katsuhara M. Salinity tolerance mechanisms in glycophytes:An overview with the central focus on rice plants[J]. Rice, 2012. |
| [26] |
Kandoi D, Mohanty S, Tripathy BC. Overexpression of plastidic maize NADP-malate dehydrogenase (ZmNADP-MDH) in Arabidopsis thaliana confers tolerance to salt stress[J]. Protoplasma, 2018, 255(2): 547-563. DOI:10.1007/s00709-017-1168-y |
| [27] |
Vijayalakshmi T, Vijayakumar AS, Kiranmai K, et al. Salt stress induced modulations in growth, compatible solutes and antioxidant enzymes response in two cultivars of safflower (Carthamus tinctorius L. Cultivar TSF1 and Cultivar SM) differing in salt tolerance[J]. American Journal of Plant Sciences, 2016, 1802-1819. |
| [28] |
Farhangi-Abriz S, Torabian S. Antioxidant enzyme and osmotic adjustment changes in bean seedlings as affected by biochar under salt stress[J]. Ecotoxicol Environ Saf, 2017, 137: 64-70. DOI:10.1016/j.ecoenv.2016.11.029 |
| [29] |
Ashraf M, Foolad MR. Roles of glycine betaine and proline in improving plant abiotic stress resistance[J]. Environmental and Experimental Botany, 2007, 59: 206-216. DOI:10.1016/j.envexpbot.2005.12.006 |
| [30] |
Silveira G, Viégas R, Rocha M, et al. Proline accumulation and glutamine synthetase activity are increased bysalt-induced proteolysis in cashew leaves[J]. Plant Physiol, 2003, 160: 115-123. DOI:10.1078/0176-1617-00890 |
| [31] |
Tripathi P, Rabara RC, Rushton PJ. A systems biology perspective on the role of WRKY transcription factors in drought responses in plants[J]. Planta, 2014, 239(2): 255-266. DOI:10.1007/s00425-013-1985-y |
| [32] |
Ning P, Liu C, Kang J, et al. Genome-wide analysis of WRKY transcription factors in wheat (Triticum aestivum L.) and differential expression under water deficit condition[J]. Peer J, 2017, 5: e3232. DOI:10.7717/peerj.3232 |
| [33] |
Chen H, Lai Z, Shi J, et al. Roles of arabidopsis WRKY18, WRKY40 and WRKY60 transcription factors in plant responses to abscisic acid and abiotic stress[J]. BMC Plant Biol, 2010, 10: 281. DOI:10.1186/1471-2229-10-281 |
| [34] |
Pandey SP, Somssich IE. The role of WRKY transcription factors in plant immunity[J]. Plant Physiol, 2009, 150(4): 1648-1655. DOI:10.1104/pp.109.138990 |
| [35] |
Wang W, Vinocur B, Shoseyov O, et al. Role of plant heat-shock proteins and molecular chaperones in the abiotic stress response[J]. Trends Plant Sci, 2004, 9(5): 244-252. DOI:10.1016/j.tplants.2004.03.006 |
| [36] |
Waters ER. The molecular evolution of the small heat-shock proteins in plants[J]. Genetics, 1995, 141(2): 785-795. |
| [37] |
Swindell WR, Huebner M, Weber AP. Transcriptional profiling of Arabidopsis heat shock proteins and transcription factors reveals extensive overlap between heat and non-heat stress response pathways[J]. BMC Genomics, 2007, 8: 125. DOI:10.1186/1471-2164-8-125 |
| [38] |
Chen JH, Jiang HW, Hsieh EJ, et al. Drought and salt stress tolerance of an Arabidopsis glutathione S-transferase U17 knockout mutant are attributed to the combined effect of glutathione and abscisic acid[J]. Plant Physiol, 2012, 158(1): 340-351. DOI:10.1104/pp.111.181875 |
| [39] |
Maxwell DP, Wang Y, McIntosh L. The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells[J]. Proc Natl Acad Sci USA, 1999, 96(14): 8271-8276. DOI:10.1073/pnas.96.14.8271 |