2. 国际热带半干旱地区作物研究所,帕坦奇鲁 印度 502324
2. International Crops Research Institute for the Semi-Arid Tropics, Patancheru India 502324
细胞质雄性不育系(Cytoplasmic male sterility,CMS)的发现和利用,在作物遗传育种研究和杂种优势利用等方面发挥了至关重要的作用,为作物杂交育种和杂交种子的生产提供了简便、高效的途径。
在主要粮食作物中,水稻细胞质雄性不育系的选育、应用及研究是最深入的[1]。目前,水稻主要有9种CMS不育系,包括野败(WA)型、K型、矮败(DA)型、D型、冈(GA)型、印尼水田(ID)型、红莲(HL)型、包台(BT)型和滇(DT)型,其中,WA型、HL型和BT型是生产上应用较多的CMS,对于其细胞质雄性不育和育性恢复的分子机制基本阐明[2-4]。CMS-WA不育系中线粒体不育基因WA352在特定时期的花药绒毡层细胞中大量积累,与核基因编码的线粒体蛋白COX11相互作用,抑制COX11在过氧化物代谢中的功能,促进绒毡层细胞的过早程序性死亡和降解,引起花粉败育[2]。不同的细胞质雄性不育表型是由于不同的线粒体基因组重排产生嵌合基因,扰乱了正常基因的功能,导致雄性配子体异常发育。通过比较分析不育系、保持系和恢复系中线粒体基因组特异的嵌合基因以及其转录产物的差异,鉴定出细胞质雄性不育的线粒体基因,如水稻orf79[5-6]、orfH79[7]、WA352[2]和orf182[8]、甜菜orf129[9]、辣椒orf456[10]、芥菜orf220[11]和油菜orf288[12]等。
Stephens于1929年在甜苏丹草中发现了第一个高粱雄性不育材料,但由于是细胞核控制的雄性不育系,没能大面积应用于杂交高粱的商业化生产,经过多年的努力,直到1954年,育成了生产上可以利用的第一个高粱细胞质雄性不育系Tx3197A,拉开了高粱杂交育种的帷幕,高粱选育常规品种转入优良“三系”及其杂交种,极大地提高了高粱的单产[13-14]。根据细胞质类型的不同,目前将高粱核质互作雄性不育系主要分为8种,包括A1、A2、A3、A4、A5、A6、9E和KS[15]。张福耀等[16]和侯荷亭等[17]开展了不同类型细胞质不育系育性测交鉴定试验,结果表明,7种(A1、A2、A3、A4、A5、A6、9E)核质互作不育系杂交的F1育性反应各不相同。
关于高粱线粒体基因组研究以及高粱细胞质雄性不育系中线粒体基因组中不育基因的报道比较少,只在高粱CMS-A3不育系中发现了一个导致细胞质雄性不育的嵌合基因orf107,是由atp9的5'末端和水稻orf79的3'末端共同组成的,能够转录一个18.8 kD的多肽[18]。目前,在生产中应用较多的是CMS-A1和CMS-A2型不育系,但是这两种不育系细胞质和细胞核基因如何互作以及分别由几对基因调控都未知[19-20],其相关的分子遗传调控机制至今还不清楚。
Tx623A是辽宁省农业科学院1979年从美国得克萨斯农业和机械大学引进的细胞质雄性不育系,具有A1型细胞质,以它作为母本选育出了一批高产、优质、高抗丝黑穗病的杂交种。保持系Tx623B是高粱参考基因组测序的品种,在NCBI数据库中只有Tx623B的线粒体基因组序列(https://www.ncbi.nlm.nih.gov/search/all/?term=DQ984518),而没有Tx623A线粒体基因组序列。
本研究以CMS-A1型不育系Tx623A和其保持系Tx623B为材料,利用二代结合三代测序技术组装不育系和保持系线粒体基因组,进一步分析不育系和保持系线粒体基因组的差异以及基因组结构的变异。旨在为A1型细胞质雄性不育基因克隆奠定基础,也为与其他类型细胞质雄性不育系的比较提供了基因组信息。
1 材料与方法 1.1 材料Tx623A为CMS-A1型,Tx623B是其对应的保持系。Tx623A和Tx623B均来自辽宁省农业科学院高粱研究所。
1.2 方法 1.2.1 Tx623A和Tx623B全基因组提取选取籽粒饱满、均匀一致的种子约100粒,放在垫有三层滤纸的发芽盒中,加入一定量的无菌水,置于人工气候箱(国产MGC-350HP-2型),16 h光照/8 h黑暗,25℃培养,待两片子叶完全展开后用于基因组的提取。取大约100 mg新鲜叶片,采用TIANGEN公司植物基因组DNA提取试剂盒(目录号:DP305)提取总DNA,详细步骤参考试剂盒操作说明书。
1.2.2 基因组测序及数据质控使用Illumina Hiseq X测序平台对样品进行PE150测序,产生的原始数据(Raw Data)存在一定比例低质量数据,为了使得后续分析的结果更加准确可靠,会对原始的测序数据进行如下处理:(1)去除reads中的adapter序列;(2)去除5'端含有非AGCT的碱基;(3)去除测序质量较低的reads末端(测序质量值小于Q20);(4)去除含N的比例达到10%的reads;(5)舍弃adapter及质量修剪后长度小于75 bp的小片段。
1.2.3 线粒体基因组组装(1)利用blasR将有效数据(Clean data)比对Pacbio三代数据,根据比对结果对单分子测序数据进行一次矫正与纠错,目的在于减少单分子长序列中单碱基插入缺失的错误;最后利用纠正过的单分子测序数据与二代数据进行混合组装,使用的软件是SPAdes-3.10.1;挑选覆盖深度足够高且组装长度较长的序列作为候选序列,比对NCBI确认线粒体基因组的Contig序列。(2)将clean reads比对回组装获得的Contig,根据reads的paired-end和overlap关系,对组装结果进行局部组装和优化,并确定Contig的顺序和方向。然后使用Gap Closer(v1.12,http://soap.genomics.org.cn/soapdenovo.htm)软件对组装结果进行内洞修补,得到最终的线粒体基因组序列。
1.2.4 线粒体基因组注释使用tRNAscan-SE(http://lowelab.ucsc.edu/tRNAscan-SE)和Artemis(http://www.sanger.ac.uk/Software/Artemis)识别线粒体的tRNA和ORFs。以高粱线粒体基因组(https://www.ncbi.nlm.nih.gov/search/all/?term=DQ984518)为参考基因组,对蛋白编码基因和rRNA基因进行同源比对预测,使用的软件是BLAST+2.7.1,参数默认。然后对同源比对结果去冗余,人工校正基因的首尾及外显子/内含子边界,获得高准确性的基因集。
1.2.5 基因比对数据库线粒体的基因蛋白序列,与已知的蛋白数据库进行blastp比对。由于每一条序列比对结果可能超过一条,为保证其生物学意义,只保留一条最优比对结果作为该基因的数据库比对信息。常用的数据库包括:NR(http://www.ncbi.nlm.nih.gov/)、Swiss-Prot(http://www.ebi.ac.uk/uniprot/)、eggNOG(http://eggnogdb.embl.de/)、KEGG(http://www.genome.jp/kegg/)和GO(http://geneontology.org/)。
1.2.6 全基因组共线性分析使用MUMmerv3.23(http://mummer.sourceforge.net/)软件对目标基因组和参考基因组进行比对,确定基因组间的大范围共线性关系。然后使用LASTZ v1.03.54(http://www.bx.psu.edu/miller_lab/dist/README.lastz-1.02.00/)对区域间进行比对,确认局部位置排列关系,并从中查找易位(Translocation/Trans),倒置(Inversion/Inv)和易位+倒置(Trans+Inv)的区域。
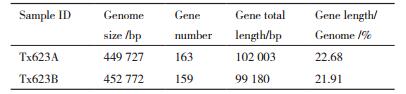
2 结果 2.1 核基因组测序法组装Tx623A和Tx623B线粒体基因组为了获得更加完整、准确的线粒体基因组序列,采用二代和三代测序结合的方法对Tx623A和Tx623B核基因组进行测序,将2次测序数据进行混合组装,完成对2个样品线粒体基因组的高质量拼接(表 1),Tx623A和Tx623B线粒体基因组大小分别为449 727和452 772 bp,预测到的线粒体基因分别为163个和159个。根据预测Tx623A和Tx623B线粒体编码基因总长度分别为102 003 bp和99 180 bp,分别占Tx623A和Tx623B线粒体基因组的22.68%和21.91%。
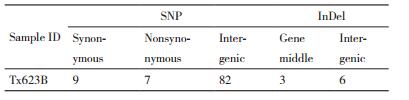
为了探明2个样品中线粒体基因组的区别,对Tx623A和Tx623B样品线粒体基因组之间存在的SNP(Single nucleotide polymorphism)和InDel标记(Insertion and deletion marker)进行比较(表 2),在基因内造成同义突变的SNP有9个,非同义突变的SNP有7个;在非基因区间内有差异的SNP共有82个。Tx623B线粒体基因组与Tx623A线粒体基因组相比,在CDS区内,存在InDel差异的有3个;在非基因区域内,存在InDel差异的有6个。
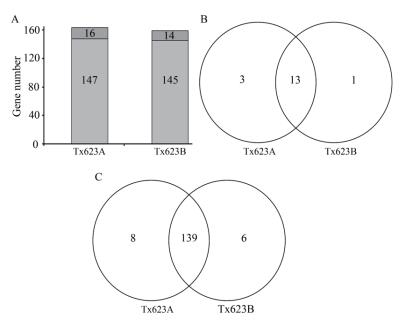
同时也对2个样品的预测基因进行了详细的分析比较。在Tx623A中预测的163个线粒体基因中,其中16个基因是两个拷贝的重复基因,由于在染色体的重复位置不同,故而计为2个基因。除去重复基因,Tx623A线粒体基因组共编码147个ORFs。Tx623B线粒体基因组中有14个重复基因,因此,Tx623B线粒体基因组共编码145个ORFs(图 1-A)。通过分析比较Tx623A和Tx623B线粒体基因组中的重复基因,发现其中13个基因是两者共有的,3个重复基因是Tx623A特有的,1个重复基因是Tx623B特有的(图 1-B)。通过比较分析Tx623A和Tx623B线粒体基因组编码的147个和145个ORFs,发现139个ORFs是两者共有的,8个ORFs是Tx623A特有的,6个ORFs是Tx623B特有的(图 1-C)。
|
| A:编码基因;B:重复基因;C:特异基因 图 1 Tx623A和Tx623B线粒体基因组比较 |
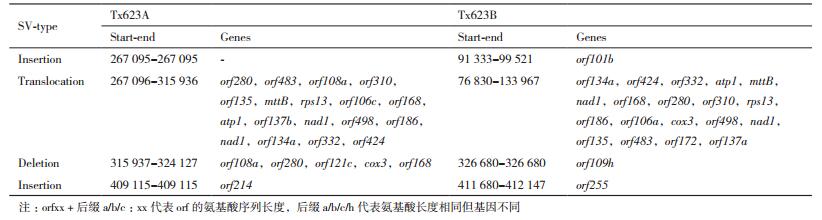
根据前人研究报道,细胞质雄性不育与线粒体基因组易位有关,为此,使用MUMmer v3.23软件对Tx623A和Tx623B线粒体基因组进行共线性分析,发现Tx623A和Tx623B线粒体基因组中存在基因组结构变异(SV)区间(图 2)。其中267 095-324 127(57 032 bp)为Tx623A和Tx623B线粒体基因组中存在的易位区间,该区间共含有22个ORFs(表 3)。
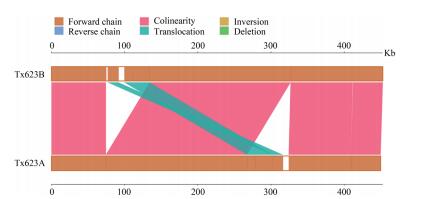
|
| 图 2 Tx623A和Tx623B线粒体基因组共线性分析 |
植物线粒体基因组大部分由非编码的DNA序列组成,包含大量重复序列和非编码RNA插入,研究一直受测序技术发展水平桎梏。近年来,随着第三代单分子实时测序技术的兴起并不断改进,超长读长和无偏好性等优势给植物线粒体基因组组装提供了新技术支撑。本研究没有提取高粱线粒体基因组直接测序组装,而是利用已有的高粱线粒体基因组序列,通过二代和三代测序相结合的方法测核基因组序列来组装线粒体基因组,为植物线粒体基因组测序组装提供了新方法。高粱线粒体NCBI上组装大小为468 628 bp(DQ984518),而利用核基因组测序组装的线粒体基因组大小为450 kb左右,它们之间相差19 kb左右,这些大小差异主要是非编码区的差异,更多的是重复序列组装造成的,编码区都很保守,而且2个基因组的组装均成环。水稻线粒体参考基因组在NCBI上公布的由于品种不同,线粒体基因组序列有20多个,大小从400-600 kb均可见,可能都是重复序列造成的差异。
从完成测序的植物线粒体基因组特征来看,高粱与玉米、水稻等线粒体基因组大小较为相近,但高粱含有的基因数与玉米和水稻差别较大,高粱线粒体基因数目为150个左右,而玉米和水稻等植物线粒体基因数目为52-60个[21-22],而差别的基因多体现在ORFs基因类型上,其他基因类型保守,即使在不育系Tx623A和其保持系Tx623B线粒体基因组也存在ORFs基因类型的差异。高等植物线粒体基因组结构变化很快,重排率比叶绿体基因组高得多,甚至不同亚种之间的线粒体基因组结构差别也很大,存在明显的分子间和分子内重组。已有研究表明,植物细胞质雄性不育主要是线粒体易位造成的,因此本研究鉴定的易位区间将为高粱A1型细胞质雄性不育基因克隆提供线粒体基因组信息。
对于水稻CMS-WA型不育系如何与核编码的线粒体蛋白相互作用,导致不育以及如何与CMS的恢复基因相互作用恢复育性等分子机制已经清晰[23-24]。高粱细胞质雄性不育的理论研究远远落后于生产应用,雄性不育早在1954年就在高粱杂交育种生产中得到了广泛应用,目前,关于细胞质雄性不育的研究仍不够深入,具体不育机理的研究仍很滞后。高粱不同类型CMS的不育机理以及恢复育性机理是否相同,还需要进行深入的研究。本研究高粱A1型细胞质雄性不育系Tx623A及其保持系Tx623B的线粒体基因组比较,为将来解析高粱细胞质雄性不育分子机制奠定了良好的基础。
4 结论通过对高粱CMS-A1型的不育系Tx623A和其保持系Tx623B线粒体基因组比较分析,鉴定出与高粱细胞质雄性不育有关的易位结构区间,具体结论如下。
测序数据组装拼接后Tx623A和Tx623B线粒体基因组大小分别为449 727 bp和452 772 bp。
Tx623A和Tx623B线粒体基因组分别预测到147个和145个ORFs,其中2个样品中分别含有特异基因8个和6个。
以Tx623A线粒体基因组为参考序列,Tx623B线粒体基因组中,在基因内造成同义突变的SNP有9个,非同义突变的SNP有7个;82个SNP位于非基因区间内。3个InDel位于CDS区内,6个InDel位于非基因区域内。
Tx623A和Tx623B线粒体基因组中存在57 kb大小的易位结构变异区间,这些结构变异可能是导致Tx623A细胞质雄性不育性的原因。
| [1] |
Tang HW, Xie YY, Liu YG, et al. Advances in understanding the molecular mechanisms of cytoplasmic male sterility and restoration in rice[J]. Plant Report, 2017, 30(4): 179-184. DOI:10.1007/s00497-017-0308-z |
| [2] |
Luo DP, Xu H, Liu ZL, et al. A detrimental mitochondrial-nuclear interaction causes cytoplasmic male sterility in rice[J]. Nat Genet, 2013, 45(5): 573-577. DOI:10.1038/ng.2570 |
| [3] |
Charlesworth D. Origins of rice cytoplasmic male sterility genes[J]. Cell Res, 2017, 27(1): 3-4. DOI:10.1038/cr.2016.158 |
| [4] |
Wang X, Guan Z, Gong Z, et al. Crystal structure of WA352 provides insight into cytoplasmic male sterility in rice[J]. Biochem Biophys Res Commun, 2018, 501(4): 898-904. DOI:10.1016/j.bbrc.2018.05.079 |
| [5] |
Wang ZH, Zou YJ, Li XY, et al. Cytoplasmic male sterility of rice with boro Ⅱ cytoplasm is caused by a cytotoxic peptide and is restored by two related PPR motif genes via distinct modes of mRNA silencing[J]. The Plant Cell, 2006, 18(3): 676-687. DOI:10.1105/tpc.105.038240 |
| [6] |
Kazama T, Itabashi E, Fujii S, et al. Mitochondrial ORF79 levels determine pollen abortion in cytoplasmic male sterile rice[J]. Plant J, 2016, 85(6): 707-716. DOI:10.1111/tpj.13135 |
| [7] |
Wang K, Gao F, Ji YX, et al. ORFH79 impairs mitochondrial function via interaction with a subunit of electron transport chain complex Ⅲ in Honglian cytoplasmic male sterile rice[J]. New Phytol, 2013, 198(2): 408-418. DOI:10.1111/nph.12180 |
| [8] |
Xie HW, Peng XJ, Qian MJ, et al. The chimeric mitochondrial gene orf182 causes non-pollen-type abortion in Dongxiang cytoplasmic male-sterile rice[J]. Plant J, 2018, 95(4): 715-726. DOI:10.1111/tpj.2018.95.issue-4 |
| [9] |
Yamamoto MP, Shinada H, Onodera Y, et al. A male sterility-associated mitochondrial protein in wild beets causes pollen disruption in transgenic plants[J]. Plant J, 2008, 54: 1027-1036. DOI:10.1111/j.1365-313X.2008.03473.x |
| [10] |
Kim DH, Kang JG, Kim BD. Isolation and characterization of the cytoplasmic male sterility associated orf456 gene of chili pepper (Capsicum annuum L.)[J]. Plant Mol Biol, 2007, 63: 519-532. DOI:10.1007/s11103-006-9106-y |
| [11] |
Yang J, Liu X, Yang X, et al. Mitochondrially-targeted expression of a cytoplasmic male sterility-associated orf220 gene causes male sterility in Brassica juncea[J]. BMC Plant Biol, 2010, 10: 231-240. DOI:10.1186/1471-2229-10-231 |
| [12] |
Jing B, Heng S, Tong D, et al. A male sterility-associated cytotoxic protein ORF288 in Brassica juncea causes aborted pollen development[J]. J Exp Bot, 2012, 63: 1285-1295. DOI:10.1093/jxb/err355 |
| [13] |
Stephens JC, Holland RF. Cytoplasmic male sterility for hybrid sorghum seed production[J]. Agron J, 1954, 46: 20-23. DOI:10.2134/agronj1954.00021962004600010006x |
| [14] |
卢庆善, 孙毅. 杂交高粱遗传改良[M]. 北京: 中国农业科学技术出版社, 2005.
|
| [15] |
Kante M, Rattunde HFW, Nébié B, et al. QTL mapping and validation of fertility restoration in West African sorghum A1 cytoplasm and identification of a potential causative mutation for Rf2[J]. Theor Appl Genet, 2018, 131(11): 2397-2412. DOI:10.1007/s00122-018-3161-z |
| [16] |
张福耀, 牛天堂, 韦耀明, 等. 高粱非买罗细胞质A2, A3, A4, A5, A6, 9E雄性不育系研究[J]. 山西农业科学, 1996, 24(3): 3-6. |
| [17] |
侯荷亭, 侯丽娟, 仪治本, 等. 高粱新胞质雄性不育遗传机理及三系配套的研究[J]. 华北农学报, 2001, 16(2): 16-19. DOI:10.3321/j.issn:1000-7091.2001.02.004 |
| [18] |
Tang HV, Pring DR, Shaw LC, et al. Transcript processing internal to a mitochondrial open reading frame is correlated with fertility restoration in male-sterile sorghum[J]. Plant J, 1996, 10: 123-133. DOI:10.1046/j.1365-313X.1996.10010123.x |
| [19] |
Jordan DR, Mace ES, Henzell RG, et al. Molecular mapping and candidate gene identification of the Rf2 gene for pollen fertility restoration in sorghum[Sorghum bicolor (L.) Moench][J]. Theor Appl Genet, 2010, 120: 1279-1287. DOI:10.1007/s00122-009-1255-3 |
| [20] |
Jordan DR, Klein RR, Sakrewski KG, et al. Mapping and characterization of Rf5:a new gene conditioning pollen fertility restoration in A1 and A2 cytoplasm in sorghum[Sorghum bicolor (L.) Moench][J]. Theor Appl Genet, 2011, 123: 383-396. DOI:10.1007/s00122-011-1591-y |
| [21] |
Fujii S, Kazama T, Yamada M, et al. Discovery of global genomic re-organization based on comparison of two newly sequenced rice mitochondrial genomes with cytoplasmic male sterility-related genes[J]. BMC Genomics, 2010, 11: 209-223. DOI:10.1186/1471-2164-11-209 |
| [22] |
Clifton SW, Minx P, Fauron CM, et al. Sequence and comparative analysis of the maize NB mitochondrial genome[J]. Plant Physiol, 2004, 136(3): 3486-3503. DOI:10.1104/pp.104.044602 |
| [23] |
Kim YJ, Zhang DB. Molecular control of male fertility for crop hybrid breeding[J]. Trends Plant Sci, 2018, 23(1): 53-65. DOI:10.1016/j.tplants.2017.10.001 |
| [24] |
Chen LT, Liu YG. Male sterility and fertility restoration in crops[J]. Annu Rev Plant Biol, 2014, 65: 579-606. DOI:10.1146/annurev-arplant-050213-040119 |