2. 南京农业大学,南京 210095
2. Department of Plant Pathology, Nanjing Agricultural University, Nanjing 210095
全球大规模商业化种植转基因作物(Genetically modified crops,GMC)自从1996年以来已有20多年的时间,种植面积现已经累计达到23亿hm2,涉及大豆、玉米、棉花和油菜等多种作物[1-2]。而在我国允许商业化种植的转基因作物只有转基因抗虫棉花和抗病毒番木瓜[3]。大豆是我国重要的粮食油料作物,同时大豆在榨油后的豆粕也是重要动物饲料来源[4]。我国是大豆消费大国,需求量从1990年的1 100万t增加到2015年的9 300万t[5]。但我国种植的大豆总产量远远不能满足国内需求,从1996起就开始大规模进口。据不完全统计,我国大豆的进口量从1996年的111万t持续增加到2017年的近亿吨,其中主要是从巴西、美国和阿根廷进口的转基因大豆[6-7]。尽管目前中国还没有开放转基因大豆的商业化种植,但每年进口的转基因大豆已经占据了我国大豆市场的90%[8]。按照国家规定进口转基因大豆只能作为榨油和饲料用途,而日常消费直接食用的大豆为非转基因大豆。因此,开发转基因大豆的快速检测方法对转基因大豆的实时监控具有重要意义。
所谓的转基因大豆就是将所需的外源目的基因通过一定的途径转入到大豆基因组中,与基因组整合并能稳定地遗传和表达,从而改造大豆的生物学性状[9]。目前转基因大豆品种主要有抗除草剂、改良大豆油脂的、抗虫的和复合性状转基因大豆[10]。耐除草剂大豆品种含有特异性的抗草甘膦、抗草胺磷或抗磺酰脲类等除草剂的目的基因,能够高效地去除杂草又确保作物安然无恙[11]。抗虫的转基因大豆将苏云金杆菌的杀虫蛋白Bt基因或其他杀虫基因转入到大豆基因组中,使得这些转基因大豆能够有效地预防害虫的发生。复合性状的转基因大豆就是把以上两个或几个基因同时整合到大豆中[10]。由于这些转基因大豆比非转基因大豆具有出油率高和价格便宜等诸多优势,因此自1996年转基因大豆商业化种植以来,全球转基因大豆的种植面积连年持续增加[12],而我国转基因大豆尚处于前期研究阶段,并没有实际的商业种植[13]。
根据检测原理的不同,目前常用的转基因植物检测技术主要有两大类:一类为蛋白水平的检测方法,如免疫试纸条法、蛋白质芯片和酶联免疫吸附法等[14]; 另一类为核酸水平上的鉴定方法,如PCR以及PCR相关的检测技术,还有核酸杂交和基因芯片等方法[15]。蛋白质水平的检测是基于所转的目的基因在转基因植株中得到了表达,通过检测这些蛋白的存在来确定转基因。由于这些鉴定方法只适用于新鲜的和未加工过的转基因材料,并且当转入的基因表达量低时也无法检测,因此这些方法并未得到广泛采用。DNA水平上的检测方法主要是基于PCR反应,但这些检测方法都需要热循环PCR仪,实验程序复杂并且检测时间较长,难以满足非实验室条件下的快速筛查和检测的需求[16]。最近几年出现了恒温条件下的等温扩增技术,其中研究较热的一个技术就是环介导等温扩增法(Loop-mediated isothermal amplification,LAMP)[17]。LAMP检测方法快速,能在恒温65℃下完成整个反应且实现了在反应管中肉眼对反应结果的判读,已经成功应用于转基因大豆的检测,但仍然需要恒温加热设备[18]。
重组酶聚合酶扩增技术(Recombinase polyme-rase amplification,RPA)是2006年Piepenburg等[19]研发出的一种等温扩增技术,它能在37-42℃恒温条件下快速完成数十亿DNA扩增[20]。RPA反应体系中主要包括重组酶(uvsX)、单链结合蛋白(Gp32)和链置换DNA聚合酶Bsu,其反应原理不同于体外DNA复制的PCR技术,重组酶uvsX在常温下就能打开DNA双链进行变性,因此不需要热循环PCR仪[21-22]。RPA反应技术灵敏度高,反应快速,并且通过试纸条可以直接进行观察,方法简便易行。因此,本文的目的是建立一个转基因大豆的RPA快速检测技术,利用试剂条显色反应实现肉眼鉴定样品中是否含有转基因成分,并对日常接触到的大豆食品和种植的大豆进行初步应用。
1 材料与方法 1.1 材料试验中所需的引物由金斯瑞生物科技有限公司合成并提供。待检测的豆芽、新鲜毛豆和大豆植株是随机从市场购买或田间采集。作为阳性对照的转基因大豆,含有花椰菜花叶病毒CaMV35S启动子和根癌农杆菌NOS终止子序列的转基因质粒载体pBinGFP2 DNA(50 ng/μL)由南京农业大学植保学院提供。
1.2 方法 1.2.1 大豆材料基因组DNA提取各种大豆材料的基因组DNA提取均采用“DNAsecure新型植物基因组DNA提取试剂盒”(天根,北京,货号DP320),并按使用说明书操作。
1.2.2 PCR扩增和反应产物的检测PCR试剂盒购自诺唯赞(Vazyme),货号为P505-d1/d2/d3。50 μL反应体系中包括25 μL 2×Phanta Max Buffer、1 μL dNTP Mix、1 μL Phanta Max Super-Fidelity DNA Polymerase、2 μL DNA模板,10 μmol/L上下游引物各2.0 μL。PCR反应程序为:95℃预变性3 min; 95℃变性15 s,63℃退火15 s,72℃延伸30 s,35次循环; 72℃延伸5 min。反应结束后取出PCR管,对PCR扩增产物进行2%琼脂糖电泳检测。
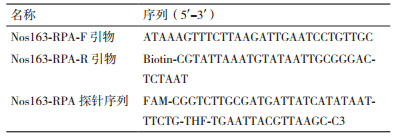
1.2.3 RPA引物的设计现有的转基因作物中大都有花椰菜花叶病毒CaMV35S启动子和根癌农杆菌NOS终止子[10],通过对这些保守DNA序列分析可以设计RPA引物。RPA引物长度一般在30-35 nt,扩增片段长度最好在100-200 bp,利用Primer3软件(http://primer3.ut.ee/),设计了5对RPA引物用作RPA试剂条引物的初步筛选(表 1)。
RPA反应体系包括一对特异性引物和一条nfo探针,其中下游引物修饰有生物素B,nfo探针修饰有fluorescein isothiocyanate荧光素FITC。根据Nos终止子以及Nos163引物的序列,我们设计了Nos163的RPA引物和探针,序列见表 2。如果反应体系中的模板DNA为转基因大豆基因组DNA,扩增产物会含有生物素B和FITC,扩增产物通过毛细作用从试纸条的加样点由下而上流动时,试纸条上会显现control band和test band两条条带。当为非转基因模板DNA或其他DNA时,试纸条上只显示control band一条带。
RPA试剂盒(TwistAmp nfo)和Milenia Hybridtech 1 strips(no nfo kits)试纸条均购自南京沃博生物科技有限公司,货号分别为TANFO02KIT和MILENIA01。RPA反应体系按试剂盒说明书进行混合,为了保证反应同时进行,2.5 μL 280 mmol/L醋酸镁溶液应该最后加在八连管的盖子上,小心翻转盖上,然后上下颠倒八连管混匀,离心。整个操作过程要在冰上进行。RPA反应在39℃恒温下反应4 min后,取出反应管进行涡旋震荡,点动离心,再在39℃恒温下反应26 min,反应结束后取5 μL扩增产物滴加至试纸条检测区,再将95 μL试纸条检测buffer加入到1.5 mL EP管中,最后将试纸条垂直放入试纸条检测buffer中,以质控线出现明显条带即可,3 min左右,待试纸条晾干后拍照。
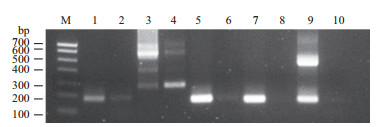
2 结果 2.1 PCR筛选特异性RPA引物为了筛选特异性且扩增效率高的RPA检测引物,我们首先利用常规PCR技术对5对RPA引物分别进行扩增,阳性对照模板DNA为含有CaMV35S启动子和NOS终止子的质粒载体pBinGFP2 DNA(50 ng/μL),阴性对照采用超纯水。如图 1所示,引物35s266和35s157对pBinGFP2 DNA扩增出两条或两条以上的条带(3和9),但从水中也扩增出条带(4),说明有污染。Nos183扩增的产物条带(1)较弱扩增效率低,且阴性对照条带有污染(2)。而Nos156和Nos163引物条带清晰(5和7)且没有污染(6和8),说明用Nos156和Nos163引物扩增DNA反应特异性强能够产生单一的条带,且扩增效率高,产物浓度高,因此选择这2对引物进行后续的RPA检测。
|
| M:DNA分子标准;1,3,5,7,9泳道分别为利用引物Nos183、35s266、Nos163、Nos156和35s157从pBinGFP2 DNA中PCR产物,为阳性对照;2,4,6,8,10泳道依此为这5对引物从超纯水中的PCR扩增片段,为阴性对照 图 1 五对RPA引物的常规PCR检测 |
为了进一步检测引物Nos156和Nos163反应的灵敏性,分别利用稀释后不同浓度的质粒pBinGFP2DNA作为模板进行PCR反应,如图 2所示引物Nos163的PCR反应比Nos156更为敏感,在相同template DNA浓度下,PCR条带更为明显,因此我们最终选择Nos163引物用来设计RPA试剂条。

|
| 图 2 引物Nos156和Nos163的PCR灵敏度检测 |
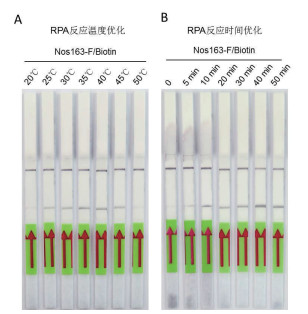
为了进一步优化RPA反应体系,以质粒载体pBinGFP2作为模板DNA进行了不同温度和反应时间的试剂条检测,如图 3-A所示,RPA体系反应温度为25-45℃,但最合适的反应温度为大约40℃。反应时间图 3-B所示从10 min-50 min都可以进行,最适合的反应时间大约为40 min。
|
| A:不同温度下的RPA反应;B:不同时间的RPA反应 图 3 RPA反应温度和反应时间的优化 |
以转基因大豆基因组DNA作为模板,反应温度为39℃,时间为40 min进行RPA反应,实验结果见图 4-A所示随着模板DNA浓度的降低,RPA条带浓度逐渐减弱,当模板DNA浓度从100 pg-100 ag时,RPA方法都能检测出转基因DNA。利用同样浓度的转基因大豆DNA进行PCR反应,结果如图 4-B所示,只有DNA为1 ng时PCR才能检测出转基因DNA的存在。PCR和RPA方法比较说明RPA反应灵敏度更高,在模板DNA浓度微量时(< 1 ng)RPA检测比PCR方法具有显著的检测优势。

|
| A:RPA试纸条反应结果;B:PCR产物电泳图 图 4 RPA反应体系与常规PCR灵敏度比较 |
为验证上述RPA技术的实用性,选取市场销售的豆芽菜,新鲜毛豆夹以及田间采摘的大豆样品共6种材料进行检测,实验过程中以2株已确定转基因的大豆作为阳性对照和1株已知非转基因大豆为阴性对照,结果(图 5-A)显示,两株转基因大豆(Transgenic1和2)均显示明显的两条带,为阳性,而非转基因大豆(WT)只有一条带,为阴性。所有的大豆样品均为阴性,显示为非转基因,说明南京周边检测到的市售大豆新鲜产品和种植的大豆没有转基因成分。现有的转基因大豆检测技术中最常用的为PCR,为了验证RPA检测体系的准确性,我们利用这些样品进行PCR检测,结果(图 5-B)显示,只有两株转基因大豆(Transgenic 1和Transgenic 2)显示为阳性,其他样品均为阴性,此结果与RPA检测相一致,进一步证明RPA技术的可靠性。

|
| A:不同样品的RPA检测结果;B:不同样品的PCR检测结果Soybean1-3分别为市场购买的豆芽菜和新鲜毛豆,Edamame1-6为田间采样的毛豆样品,Transgenic1和2为实验室保存的已知转基因大豆,WT:实验室保存的非转基因大豆 图 5 RPA和PCR检测豆芽菜和大豆样品 |
转基因大豆的大量进口和使用,对于缓解我国大豆短缺的问题具有重要意义,同时对进口转基因大豆的监控提出新的挑战。为了防止转基因大豆国内的非法种植和流通,因此迫切需要简便可行的转基因大豆快速检测方法。
现已建立的转基因大豆检测方法主要包括PCR和实时定量荧光PCR[25-26]。但这些方法都需要PCR仪或实时定量荧光PCR仪以及琼脂糖电泳,操作繁琐,检测过程长达几个小时,不适合实时快速监测。环介导等温扩增法是近年来应用较多的不依赖于复杂昂贵的仪器设备、简便快速DNA检测方法,并已应用于大豆的检测[27]。但环介导等温扩增法需60-65℃等温条件,并要设计4种引物。根据现有转基因大豆检测上的优缺点和RPA技术不需要专门的仪器,常温下能进行目的基因特异性扩增的优势,本研究设计建立转基因大豆的RPA试剂条快速检测技术,通过对不同浓度的转基因大豆基因组DNA扩增实验结果表明,该方法比常规PCR方法更灵敏,在微量的模板DNA浓度下(< 1 ng),PCR无法检测时RPA也能检测到转基因DNA的存在。同时利用此方法对市场随机购买的豆芽和毛豆样品以及田间种植的大豆样品进行检测,未发现转基因大豆的存在,此结果和常规PCR检测结果一致,进一步证明了本体系的可靠性和准确性。与环介导等温扩增法相比本方法具有无需恒温加热仪器的优点,RPA可以在25-45℃之间反应,简单的保温杯或人体温度甚至常温就可以实现。同时RPA的引物设计简单和PCR引物设计方法相似,无需复杂的分析,而环介导等温扩增法需要设计4种引物。总之,本文中的转基因大豆RPA快速检测方法具有常温下反应灵敏度高和结果可靠等优势,可以用于国内大豆的监控。
4 结论本研究建立的转基因大豆的RPA试剂条快速分子检测方法,简便易操作,无需PCR和电泳仪等昂贵的实验器材,检测周期短,试剂条结果肉眼可见。应用此方法初步检测南京市田间和市场大豆,在待检测的样品中没有发现转基因成分。
| [1] |
何超. 转基因作物20年[J]. 湖南农业, 2016, 9: 28. |
| [2] |
国际农业生物技术应用服务组织. 2017年全球生物技术/转基因作物商业化发展态势[J]. 中国生物工程杂志, 2018, 6: 1-8. |
| [3] |
沈平, 武玉花, 梁晋刚, 等. 转基因作物发展及应用概述[J]. 中国生物工程杂志, 2017, 37(1): 119-128. |
| [4] |
杨华, 彭城, 肖英平, 等. 转基因大豆SHZD32-1转化体普通PCR和qRT-PCR检测方法的研究[J]. 农业生物技术学报, 2017, 26(3): 492-501. |
| [5] |
查霆, 钟宣伯, 周启政, 等. 我国大豆产业发展现状及振兴策略[J]. 大豆科学, 2018, 37(3): 458-463. |
| [6] |
宋扬, 吴存祥, 侯文胜, 等. 对引进的美国大豆品种进行转基因成分的检测[J]. 大豆科学, 2005, 24(2): 116-120. DOI:10.3969/j.issn.1000-9841.2005.02.007 |
| [7] |
詹琳.全球转基因作物商业化进展情况及有关问题的分析[D].北京: 中国农业科学院, 2015. http://cdmd.cnki.com.cn/Article/CDMD-82101-1017139184.htm
|
| [8] |
刘慧. 国产大豆产量创十年来新高我国亟需打响"中国大豆"品牌[J]. 中国食品, 2018, 11: 94-95. |
| [9] |
Singh OV, Ghai S, Paul D, et al. Genetically modified crops:success, safety assessment, and public concern[J]. Appl Microbiol Biotechnol, 2006, 71(5): 598-607. DOI:10.1007/s00253-006-0449-8 |
| [10] |
崔宁波, 张正岩. 转基因大豆研究及应用进展[J]. 西北农业学报, 2016, 25(8): 1111-1124. |
| [11] |
王宜林, 李宏. 抗除草剂转基因植物的研究现状[J]. 生物学通报, 2005, 40(4): 15-17. |
| [12] |
郭兵福, 等. 不同大豆基因型对农杆菌敏感性研究及优异种质筛选[J]. 植物遗传资源学报, 2015, 16(3): 569-575. |
| [13] |
王志刚, 彭纯玉. 中国转基因作物的发展现状与展望[J]. 农业展望, 2010, 6(11): 51-55. DOI:10.3969/j.issn.1673-3908.2010.11.014 |
| [14] |
沈泓, 李超, 李珏. 分子生物学技术在转基因食品检测领域中的研究进展[J]. 中国农业信息, 2017, 15: 57-59. |
| [15] |
Kamle M, et al. Current perspectives on genetically modified crops and detection methods[J]. Biotech, 2017, 7(3): 219. |
| [16] |
Salisu IB, Shahid AA, Yaqoob A, et al. Molecular approaches for high throughput detection and quantification of genetically modified crops:A review[J]. Front Plant Sci, 2017, 8: 1670. DOI:10.3389/fpls.2017.01670 |
| [17] |
Randhawa GJ, et al. Loop-mediated isothermal amplification:rapid visual and real-time methods for detection of genetically modified crops[J]. J Agric Food Chem, 2013, 47: 11338-11346. |
| [18] |
周杰, 黄文胜, 邓婷婷, 等. 环介导等温扩增法检测6种转基因大豆[J]. 农业生物技术学报, 2017, 25(2): 335-344. |
| [19] |
吕蓓程, 严庆丰, 等. 用重组酶介导扩增技术快速扩增核酸[J]. 中国科学:生命科学, 2010, 40(10): 983-988. |
| [20] |
Piepenburg O, Williams CH, et al. DNA detection using recombin-ation proteins[J]. PLoS Biology, 2006, 4(7): 1115-1121. |
| [21] |
高威芳, 朱鹏, 黄海龙. 重组酶聚合酶扩增技术:一种新的核酸扩增策略[J]. 中国生物化学与分子生物学报, 2016, 32(6): 627-634. |
| [22] |
景志刚, 董浩, 狄栋栋, 等. 重组酶聚合酶扩增技术研究进展[J]. 生物技术通报, 2016, 32(6): 47-53. |
| [23] |
周延培, 孙海新, 赵美丽, 等. 一种转基因马铃薯PCR鉴别方法的建立[J]. 粮油食品科技, 2018(3): 67-70. DOI:10.3969/j.issn.1007-7561.2018.03.014 |
| [24] |
Xu C, Li L, Jin W, Wan Y. Recombinase polymerase amplification (RPA) of CaMV-35S promoter and nos terminator for rapid detection of genetically modified crops[J]. Int J Mol Sci, 2014, 15(10): 18797-18205. |
| [25] |
马建荣, 余永红. 市场销售大豆的转基因检测[J]. 广东化工, 2018, 45(2): 37-38. DOI:10.3969/j.issn.1007-1865.2018.02.016 |
| [26] |
钱云开, 高飞, 王海洋, 等. 16种转基因大豆品系的实时荧光PCR法筛查[J]. 中国油脂, 2017, 42(2): 141-145. DOI:10.3969/j.issn.1003-7969.2017.02.032 |
| [27] |
Liu M, Luo Y, Tao R, et al. Sensitive and Rapid Detection of Genetic Modified Soybean(Roundup Ready)by Loop-Mediated Isothermal Amplification[J]. Journal of the Agricultural Chemical Society of Japan, 2009, 73(11): 2365-2369. |