2. 中国农业大学农学院,北京 100193
2. College of Agronomy and Biotechnology, China Agricultural University, Beijing 100193
高粱[Sorghum bicolor(L.)Moench]为禾本科、高粱属、一年生草本植物,是人类最早栽培的重要谷类作物之一,其产量仅次于玉米、水稻、小麦和大麦,居世界第五位。高粱具有抗旱、耐涝、耐盐碱、耐瘠薄等特性,是部分干旱、半干旱、低洼易涝及瘠薄盐碱地区人们的主要粮食和饲料来源[1]。近年来,人口增长、耕地面积减少、水资源短缺及气候变化等问题日益严峻,严重影响世界粮食安全问题[2]。预计到2050年,全球粮食产量只有增长70%才能满足人们未来的粮食需求,因此培育高产的高粱品种将对缓解世界粮食安全问题起到重要作用。粒重是影响高粱产量的三要素之一,增加粒重是提高高粱产量的有效途径[3]。粒重是由数量性状基因控制的,遗传机理较复杂。20多年来,众多研究者利用数量性状基因座(Quantitative trait locus,QTL)定位的方法来挖掘控制高粱粒重的相关基因,以期利用分子标记辅助选择的方法进行育种,并揭示粒重基因的调控机理,取得了一定进展。本文对高粱粒重的遗传特性、高粱粒重的影响因素以及高粱粒重QTL定位研究进展进行了综述,对已定位的高粱粒重QTLs进行了比较分析,查找水稻和玉米中已克隆的粒重相关基因在高粱中的同源基因,并与高粱粒重QTLs定位区间进行比较,从而为高粱高产的分子标记辅助育种及高粱粒重主效QTLs的精细定位和克隆提供参考。
1 高粱粒重的遗传分析及影响因素高粱粒重与穗粒数和有效穗数共同构成高粱单位面积产量的三要素,提高粒重是增加高粱产量的重要育种方向[3]。
高粱粒重的遗传力高,亲本的粒重高低直接影响着F1代粒重的表现。卢庆善[4]认为高粱千粒重性状遗传力高,是由少数主效基因控制的。此外,杨伟光等[5]和程宝成等[6]研究发现高粱粒重的遗传符合加性-显性模型,高值亲本比低值亲本遗传传递力强,并且杂交一代多数接近或超过高值亲本,这对于高粱杂交育种亲本材料的选用具有重要的指导意义。
除遗传因素外,环境也会对高粱粒重造成影响,进而影响高粱产量。开花前发生干旱会影响穗的大小、穗粒数和籽粒产量,开花后发生干旱,则会使籽粒灌浆受阻,造成减产。研究表明,灌浆初、后期的温度和光照会对高粱粒重造成影响[7]。另外高粱病虫害也会对高粱产量造成影响。除上述生物和非生物胁迫以外,Kiniry[8]和Tao等[9]研究发现高粱千粒重和穗粒数呈负相关性。Murry等[10]对高粱籽粒和茎秆含糖量的遗传平衡进行研究发现茎糖产量和浓度的升高在一定程度上制约了籽粒产量的增加。
综上所述,高粱粒重主要是由遗传因素决定的,并间接受到环境影响。开展高粱粒重基因的定位和克隆研究对于进行高粱分子标记辅助育种具有重要意义。
2 高粱粒重QTL定位研究进展高粱为二倍体植物(2n=20),基因组小,其单倍体基因组DNA含量约为730 Mbp,这些特点使其成为研究甘蔗族及其他C4植物功能基因组学的模式植物[11]。随着高粱全基因组测序的完成以及大量新型分子标记的开发,完整覆盖高粱全基因组的高密度遗传连锁图谱已构建完成,一些控制高粱粒重的QTLs也通过分子标记定位于相应的遗传连锁群上。
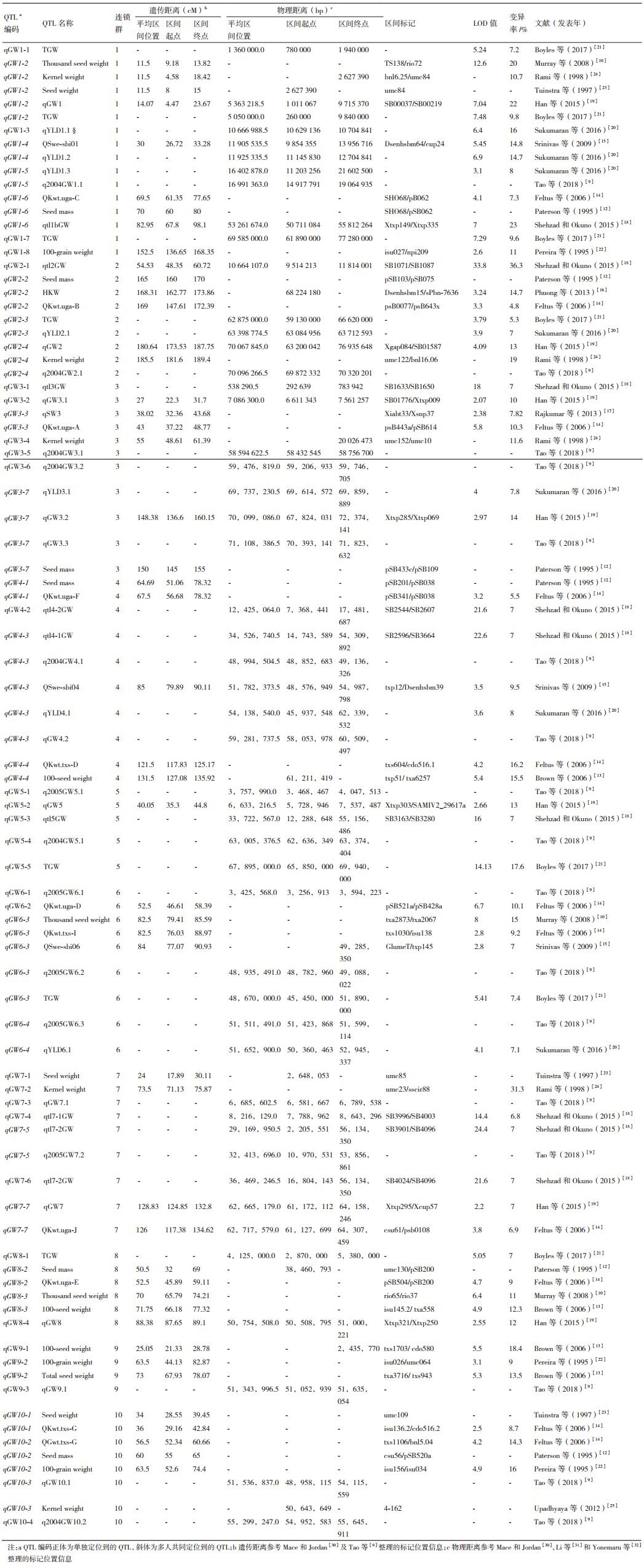
2.1 高粱粒重QTL初步定位研究进展近几十年来,越来越多的科学家利用QTL定位的方法来揭示作物复杂性状的遗传控制原理,以用于分子标记辅助选择育种或相关基因的克隆。目前已有来自16篇研究报道的108个控制高粱粒重的QTLs被定位到(表 1),做图群体有F2群体、F2:3群体、重组自交系群体和回交群体等,群体大小为90-370株,遗传连锁群数量为10-17个,遗传连锁图全长为878 cM-2225.3 cM,标记间的平均距离介于0.5 cM-14.8cM之间,具体定位结果汇总如下。
Paterson等[12]利用来自BTx623×S.propinquum的F2群体定位到6个控制高粱粒重的QTLs,分别位于1、2、3、6、8和10号染色体上。Brown等[13]用来自BTx623×IS3620C的119个重组自交系将3个高粱百粒重QTLs分别定位到4、8和9号染色体上,并在9号染色体上定位到一个高粱穗粒重QTL。Feltus等[14]用来自BTx623×IS3620C的重组自交系群体定位到3个控制高粱千粒重的QTLs(位于4、6和10号染色体),用来自BTx623×S. propinquum的F2群体定位到7个控制高粱千粒重的QTLs(位于1、2、3、4、6、7和8号染色体),解释的表型变异分别为35%和49%,其中在不同群体中定位到的两组QTLs(位于4号染色体上的QKwt.txs-D与Kwt.uga-F和位于6号染色体上的QKwt.txs-I与QKwt.uga-D)为相同的QTLs。Srinivas等[15]用来自296B×IS18551的168个重组自交系定位到3个高粱粒重QTLs,分别位于1、4和6号染色体上,解释的表型变异为7%-14.8%,来自大粒亲本296B的等位基因增加了粒重。Phuong等[16]利用来自IS2449×IS1488的100个重组自交系分别在干旱胁迫下和充足灌溉的环境下定位到1个(位于5号染色体)和2个(位于2和5号染色体)高粱百粒重QTLs,解释的表型变异为12.7%-18.9%,其中在不同环境下定位到的位于5号染色体的QTL为相同的QTL。Rajkumar等[17]利用来自E36-1×SPV570的184个重组自交系对高粱粒重QTL进行定位,将4个粒重QTLs分别定位到2(2个QTLs)、3和4号染色体上,解释的表型变异最高为7.82%。Shehzad和Okuno[18]利用Red Kafir×Takakibi组配的F2和F2:3家系对高粱产量相关性状进行QTL定位,结果在F2群体中定位到9个百粒重QTLs,分布于1、2、3、4、5和7号染色体上,其中位于2号染色体的qtl2GW解释的表型变异最高,为36.3%,LOD值为33.8,利用F2:3家系能够重复检测到5个相同的QTLs,qtl2GW的LOD值最高,为33.8,说明qtl2GW是一个控制高粱百粒重的主效QTL。Han等[19]利用来自SA2313×Hiro-1的F2群体对高粱粒重QTL进行定位,用种于北京的138株F2群体定位到3个QTLs,位于1、2和7号染色体上,1号染色体的qGW1解释了最高的表型变异,为22%,并用种于海南的F2群体中47株极端小粒个体和47株极端大粒个体对定位结果进行验证,定位到6个QTLs,其中位于1号和2号染色体的qGW1和qGW2能够被重复检测到,解释的表型变异率分别为40%和27%,是主效的QTLs。此外,在ATx623×SA2313和K-385×SA2313的两个F2群体中也检测到了qGW1,证明qGW1在不同的遗传背景中均能被检测到。Sukumaran等[20]利用测序基因分型(Genotyping-by-sequencing,GBS)的方法对定位群体进行基因型鉴定,结合高粱产量表型共定位到7个高粱产量QTLs,分布在1、2、3、4和6号染色体上,其中1号染色体的qYLD1.1在不同的种植环境下均能够被检测到,解释的表型变异最高,为16%。Boyles等[21]利用两个不同的定位群体定位到6个高粱千粒重QTLs,分布于1、2、5、6和8号染色体上,其中位于5号染色体上的QTL是最新发现的,解释了21%的遗传变异。Tao等[9]利用一个优质粒用高粱品种R931945-2-2和其野生祖先S. bicolor subsp. Verticilliflorum组配的BC1F5群体定位到17个控制高粱千粒重的QTLs,其中有5个QTLs与前人的定位结果相同。Pereira等[22],Tuinstra等[23],Rami等[24],Murray等[10]和Upadhyaya等[25]也对高粱粒重QTL的定位也进行了报道。近年来,Zhang等[26]和Boyles等[27]利用全基因组关联分析法(Genome-wide association study,GWAS)分别检测到10个和17个与高粱粒重相关联的基因区域。根据以上研究结果发现高粱粒重相关QTL在10条染色体上均有分布。
2.2 高粱粒重QTL精细定位研究进展虽然对高粱粒重QTL初步定位研究已有很多报道,但高粱粒重QTL的精细定位或基因克隆研究却寥寥无几,远远落后于水稻、玉米等其他作物粒重相关基因的研究。
Han等[19]首次对高粱粒重QTL精细定位进行了研究,他们选取在不同的环境和遗传背景下均能被检测到的主效QTL qGW1为研究对象进行精细定位,在F2群体中选取qGW1处为杂合,其它QTL位点处为隐性的大粒交换单株自交得到的F3群体为精细定位群体,将qGW1定位到1号染色体短臂标记HLJ-10和HLJ-18之间大约101 kb的范围内,在这个范围内包含13个候选基因,根据这13个基因在不同大粒和小粒品种间的测序结果,初步将Sobic.001G038300基因作为qGW1的候选基因,但没作进一步验证,韩立杰[28]还对另一个主效QTL qGW2进行了精细定位,将其定位到2号染色体标记Xtxp100和SB01474之间大约1 674 kb的范围内。
Boyles等[21]在5号染色体发现了一个新的高粱千粒重QTL,解释了较高的表型变异率为21%,该位点的优势等位基因来自小粒亲本BTxARG-1,经过进一步检测,最高峰处的标记位于Sobic.005G188400基因上,该基因编码一个功能未知的的remorin蛋白,主要在早期花序组织和发育的胚中表达;而水稻中的Grain setting defect1(GSD1)基因也编码一个remorin蛋白,通过调控胞间连丝的通透性,进而调控光合同化物的装载和分配,使光合同化物由光合位点向韧皮部转运过程受阻所致,最终影响水稻种子结实率[29]。
3 高粱粒重QTL的比较分析目前已有多个控制高粱粒重的QTLs被定位到,尽管研究者所用研究材料不尽相同,但有些QTLs可能是相同的,为了对这些定位结果进行有效的比较和利用,Mace和Jordan[30]与Tao等[9]分别将1995-2010年和1995-2018年报道的高粱粒重相关QTLs整合到同一遗传连锁图谱上。根据这些标记在高粱遗传连锁图上的距离及Li等[31]和Yonemaru等[32]发布的相关标记在高粱染色体上的物理距离,将已定位高粱粒重相关QTLs在染色体上的位置进行整理,见表 2。
从表 2中可以看出,来自16篇研究报道的108个控制高粱粒重的QTLs被整合为50个QTLs。1号、3号和7号染色体上富含控制高粱粒重的基因区域。1号染色体涵盖了数量最多的QTLs,占全部QTLs的16%,qGW1-2、qGW1-4、qGW1-5和qGW1-6 4个QTLs均被两篇及以上研究报道共同定位到,其中qGW1-2在5篇报道中均被定位到,解释的遗传变异最高为22%[19],该QTL与Mace和Jordan[30]报道的元QTL QKWT_meta1.1为相同的QTL。3号和7号染色体上各有7个QTLs,各占全部QTLs的14%,但是仅有qGW3-3、qGW3-7和qGW7-5、qGW7-7被两篇及以上研究报道共同定位到。2、4、6、8和10号染色体上各包含4个QTLs,其中2、4和10号染色体上各有3个QTLs,6和8号染色体上各有2个QTLs,在两篇及以上研究报道中被定位到。5号染色体上的5个QTLs均为单独定位到的QTL。9号染色体上的QTLs最少,仅有3个,其中qGW9-2被两篇研究报道共同定位到。在整合的50个QTLs中,有22个QTLs被两篇及以上研究报道共同定位到,这些QTLs均能解释较高的遗传变异,并且研究者所用的材料均不尽相同,说明高粱粒重相关QTL在不同遗传背景和环境条件下一致表达,具有重要的实际意义,不同报道中定位到的QTLs相互间也对定位结果的准确性进行了验证,这些QTLs和标记将帮助育种者对高粱粒重性状进行遗传改良,从而培育高产的高粱品种。
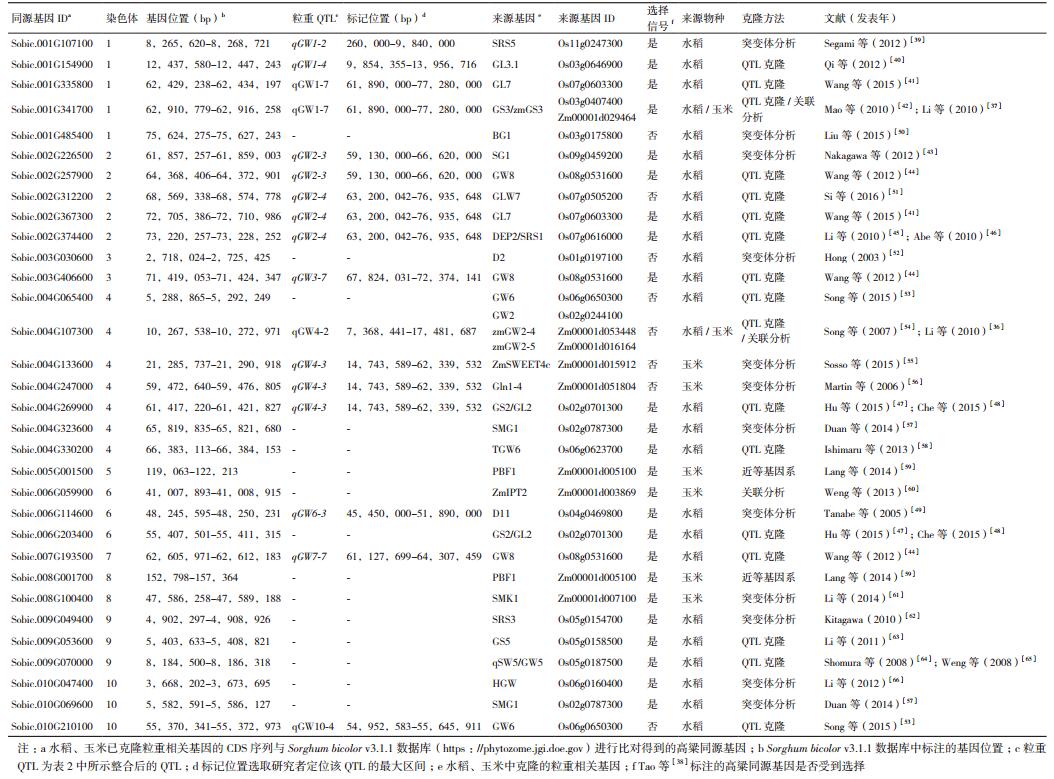
4 水稻和玉米已克隆粒重相关基因在高粱中的同源基因分析玉米、水稻、小麦、高粱和谷子等主要作物都是由其野生种驯化而来,在漫长的驯化过程中,一些共同的优良特性,如果实或种子的增大、种子落粒性的丧失、种子休眠的打破和生育期的同步等,被人们有意或无意的选择并保存下来[33]。这些相似表型变化的分子基础有些是不同作物间同源基因导致的。Lin等[34]在高粱中克隆的Sh1基因在水稻及玉米中的同源基因被证实可控制水稻及玉米的落粒,Liu等[35]在高粱中克隆的HD1基因被证实在高粱、水稻和谷子中受到平行选择,水稻中克隆的粒重相关基因GW2和GS3在玉米中的同源基因均被证实影响着玉米籽粒大小和粒重[36-37]。因此,在谷物驯化过程中,某些驯化性状基因的同源性和共线性被很好地保存下来,可能存在功能保守的基因组区域,是各种谷物种子大小变化的基础[12]。目前,水稻和玉米中已克隆了大量粒重相关基因,查找这些基因在高粱中的同源基因,并结合其在驯化过程中是否受到选择和QTL定位结果,对于克隆高粱粒重相关基因并阐明其分子机理具有重要意义。
Han等[19]和Tao等[38]均查找了水稻和玉米中已克隆粒重相关基因在高粱中的同源基因,Tao等对这些基因在驯化过程中是否受到人工选择也进行了研究,现将这些基因信息进行整理并与高粱粒重QTL整合信息进行比较,见表 3。将22个水稻粒重相关基因和8个玉米粒重相关基因的CDS序列与高粱基因组数据库Sorghum bicolor v3.1.1(https://phytozome.jgi.doe.gov)进行比对,得到32个同源基因,分布在高粱10条染色体上。将同源基因在基因组上的位置和表 2中整合的QTLs侧翼标记的物理距离进行比较,发现18个同源基因位于高粱粒重QTL区间内,其中有12个基因具有驯化选择信号,即水稻小圆粒基因SRS5[39]在高粱中的同源基因Sobic.001G107100位于qGW1-2所在区间内;水稻粒长基因GL3.1[40]的同源基因Sobic.001G154900位于qGW1-4所在区间内;水稻粒长基因GL7[41]在高粱基因组中存在2个同源性较高的基因,Sobic.001G335800位于qGW1-7所在区间内,Sobic.002G367300位于qGW2-4所在区间内;水稻粒长粒重基因GS3[42]和玉米粒重基因zmGS3[37]的同源基因为相同的基因Sobic.001G341700,位于qGW1-7所在区间内;水稻短粒基因SG1[43]的同源基因Sobic.002G226500位于qGW2-3所在区间内;水稻粒重基因GW8[44]在高粱基因组中存在3个同源性较高的基因,Sobic.002G257900位于qGW2-3所在区间内,Sobic.003G406600位于qGW3-7所在区间内,Sobic.007G193500位于qGW7-7所在区间内;水稻小圆粒基因DEP2[45]/SRS1[46]的同源基因Sobic.002G374400位于qGW2-4所在区间内;水稻籽粒大小基因GS2[47]/GL2[48]的同源基因Sobic.004G269900位于qGW4-3所在区间内;水稻小粒基因D11[49]的同源基因Sobic.006G114600位于qGW6-3所在区间内。
以上这些同源基因位于前人定位的高粱粒重QTL所在区间内,并在高粱驯化过程中表现出选择的特征,表明这些QTLs在高粱驯化过程中受到了影响,并且这些同源基因可能是调控高粱粒重的相关基因,在作物驯化过程中受到了平行驯化,但仍需要进一步验证。
5 高粱粒重基因的应用及展望进一步提高产量是高粱育种的重要目标,可通过传统杂交育种和分子标记辅助育种两种途径培育高粱高产新品种。由于高粱粒重超亲遗传现象较为普遍,亲本粒重的高低影响着杂交一代粒重的表现,因此在传统杂交育种中,亲本材料特别是母本(不育系)材料最好选用大粒材料。分子标记辅助育种是快速培育优质作物品种的有效途径,在水稻、玉米和小麦等作物中的应用已取得很大进展,通过QTL定位手段对高粱粒重关键基因进行定位和克隆,可为育种家选育高产的高粱新品种提供分子标记,加快育种速度,提高育种效率。
目前已定位到大量的高粱粒重相关QTLs,通过对不同研究者定位到的QTLs进行比较,可对QTLs进行相互验证,并能发现一些遗传变异率高,在不同遗传背景和环境下均能被检测到的主效QTLs,与之连锁的分子标记可用于高粱高产分子标记辅助育种,对这些主效QTLs可作进一步的精细定位和克隆研究。
在谷物驯化过程中,大粒的品种易受到人工选择,不同谷物间功能保守的大粒性状基因的同源性和共线性可以被很好的保存下来。查找水稻和玉米中已克隆粒重相关基因在高粱中的同源基因,研究其在驯化过程中是否受到选择,并与QTL定位结果进行比较,可发掘高粱粒重候选基因,对于克隆高粱粒重相关基因并阐明其分子机理具有重要意义。
| [1] |
梁小红, 仪治本, 赵威军. 高粱重要抗性性状的基因定位研究综述[J]. 作物杂志, 2005(3): 7-9. DOI:10.3969/j.issn.1001-7283.2005.03.003 |
| [2] |
Godfray HCJ, Beddington JR, Crute IR, et al. Food security:the challenge of feeding 9 billion people[J]. Science, 2010, 327(5967): 812-818. DOI:10.1126/science.1185383 |
| [3] |
高士杰. 高粱高产杂交种产量结构的分析[J]. 吉林农业科学, 1986(3): 16-19. |
| [4] |
卢庆善. 高粱学[M]. 北京: 中国农业出版社, 1999.
|
| [5] |
杨伟光, 韩立军, 牟金明, 等. 粒用高粱粒重的遗传研究[J]. 作物学报, 2001, 27(5): 627-632. DOI:10.3321/j.issn:0496-3490.2001.05.012 |
| [6] |
程宝成, 刘巧英, 江宏. 高粱粒重的双列分析[J]. 遗传, 1989, 11(3): 12-14. |
| [7] |
Kiniry JR, Musser RL, 王富德. 高粱粒重对灌浆初、后期环境的反应[J]. 国外农学-杂粮作物, 1989(6): 35-38. |
| [8] |
Kiniry JR, 王富德. 高粱随粒数减少而致的粒重增加[J]. 国外农学-杂粮作物, 1990(1): 5-9. |
| [9] |
Tao Y, Mace E, George-Jaeggli B, et al. Novel grain weight loci revealed in a cross between cultivated and wild sorghum[J]. The Plant Genome, 2018, 11(2): 1-10. |
| [10] |
Murray SC, Sharma A, Rooney WL, et al. Genetic improvement of sorghum as a biofuel feedstock:Ⅰ. QTL for stem sugar and grain nonstructural carbohydrates[J]. Crop Science, 2008, 48(6): 2165-2179. DOI:10.2135/cropsci2008.01.0016 |
| [11] |
Paterson AH, Bowers JE, Bruggmann R, et al. The sorghum bicolor genome and the diversification of grasses[J]. Nature, 2009, 457(7229): 551-556. DOI:10.1038/nature07723 |
| [12] |
Paterson AH, Lin YR, Li Z, et al. Convergent domestication of cereal crops by independent mutations at corresponding genetic loci[J]. Science, 1995, 269(5231): 1714-1718. DOI:10.1126/science.269.5231.1714 |
| [13] |
Brown PJ, Klein PE, Bortiri E, et al. Inheritance of inflorescence architecture in sorghum[J]. Theoretical and Applied Genetics, 2006, 113(5): 931-942. DOI:10.1007/s00122-006-0352-9 |
| [14] |
Feltus FA, Hart GE, Schertz KF, et al. Alignment of genetic maps and QTLs between inter- and intra-specific sorghum populations[J]. Theoretical and Applied Genetics, 2006, 112(7): 1295-1305. DOI:10.1007/s00122-006-0232-3 |
| [15] |
Srinivas G, Satish K, Madhusudhana R, et al. Identification of quantitative trait loci for agronomically important traits and their association with genic-microsatellite markers in sorghum[J]. Theoretical and Applied Genetics, 2009, 118: 1439-1454. DOI:10.1007/s00122-009-0993-6 |
| [16] |
Phuong N, Stützel H, Uptmoor R. Quantitative trait loci associated to agronomic traits and yield components in a Sorghum bicolor L. Moench RIL population cultivated under pre-flowering drought and well-watered conditions[J]. Agricultural Sciences, 2013, 4(12): 781-791. DOI:10.4236/as.2013.412107 |
| [17] |
Rajkumar FB, Kavil SP, Girma Y, et al. Molecular mapping of genomic regions harbouring QTLs for root and yield traits in sorghum (Sorghum bicolor L. Moench)[J]. Physiology and Molecular Biology of Plants, 2013, 19(3): 409-419. DOI:10.1007/s12298-013-0188-0 |
| [18] |
Shehzad T, Okuno K. QTL mapping for yield and yield-contributing traits in sorghum (Sorghum bicolor (L.) Moench) with genome-based SSR markers[J]. Euphytica, 2015, 203(1): 17-31. DOI:10.1007/s10681-014-1243-9 |
| [19] |
Han L, Chen J, Mace E, et al. Fine mapping of qGW1, a major QTL for grain weight in sorghum[J]. Theoretical and Applied Genetics, 2015, 128(9): 1813-1825. DOI:10.1007/s00122-015-2549-2 |
| [20] |
Sukumaran S, Li X, Li X, et al. QTL mapping for grain yield, flowering time, and stay-green traits in sorghum with genotyping-by-sequencing markers[J]. Crop Science, 2016, 56(4): 1429-1443. DOI:10.2135/cropsci2015.02.0097 |
| [21] |
Boyles RE, Pfieffer BK, Cooper EA, et al. Quantitative trait loci mapping of agronomic and yield traits in two grain sorghum biparental families[J]. Crop Science, 2017, 57(5): 2443-2457. DOI:10.2135/cropsci2016.12.0988 |
| [22] |
Pereira MG, Ahnert D, Lee M, et al. Genetic mapping of quantitative trait loci for panicle characteristics and kernel weight in sorghum[J]. Revista Brasileira De Genetica, 1995, 18(2): 249-257. |
| [23] |
Tuinstra MR, Grote EM, Goldsbrough PB, et al. Genetic analysis of post-flowering drought tolerance and components of grain development in Sorghum bicolor (L.) Moench[J]. Molecular Breeding, 1997, 3(6): 439-448. DOI:10.1023/A:1009673126345 |
| [24] |
Rami JF, Dufour P, Trouche G, et al. Quantitative trait loci for grain quality, productivity, morphological and agronomical traits in sorghum (Sorghum bicolor L. Moench)[J]. Theoretical and Applied Genetics, 1998, 97(4): 605-616. DOI:10.1007/s001220050936 |
| [25] |
Upadhyaya HD, Wang YH, Sharma S, et al. SSR markers linked to kernel weight and tiller number in sorghum identified by association mapping[J]. Euphytica, 2012, 187(3): 401-410. DOI:10.1007/s10681-012-0726-9 |
| [26] |
Zhang D, Li J, Compton RO, et al. Comparative genetics of seed size traits in divergent cereal lineages represented by sorghum (Panicoidae) and rice (Oryzoidae)[J]. G3:Genes, Genomes, Genetics, 2015, 5(6): 1117-1128. |
| [27] |
Boyles RE, Cooper EA, Myers MT, et al. Genome-wide association studies of grain yield components in diverse sorghum germplasm[J]. The Plant Genome, 2016, 9(2): 1-17. |
| [28] |
韩立杰.高粱粒重的QTL分析及qGW1的精细定位[D].北京: 中国农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10019-1016085111.htm
|
| [29] |
Gui J, Liu C, Shen J, et al. Grain setting defect1, encoding a remorin protein, affects the grain setting in rice through regulating plasmodesmatal conductance[J]. Plant Physiology, 2014, 166(3): 1463-1478. DOI:10.1104/pp.114.246769 |
| [30] |
Mace ES, Jordan DR. Integrating sorghum whole genome sequence information with a compendium of sorghum QTL studies reveals uneven distribution of QTL and of gene-rich regions with significant implications for crop improvement[J]. Theoretical and Applied Genetics, 2011, 123(1): 169-191. DOI:10.1007/s00122-011-1575-y |
| [31] |
Li ML, Yuyama N, Luo L, et al. In silico mapping of 1758 new SSR markers developed from public genomic sequences for sorghum[J]. Molecular Breeding, 2009, 24(1): 41-47. DOI:10.1007/s11032-009-9270-2 |
| [32] |
Yonemaru J, Ando T, Mizubayashi T, et al. Development of genome-wide simple sequence repeat markers using whole-genome shotgun sequences of sorghum[Sorghum bicolor (L.) Moench][J]. DNA Research, 2009, 16(3): 187-193. DOI:10.1093/dnares/dsp005 |
| [33] |
刘欢欢.高粱驯化相关性状遗传结构的解析[D].北京: 中国农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10019-1017000815.htm
|
| [34] |
Lin ZW, Li XR, Shannon LM, et al. Parallel domestication of the Shattering1 genes in cereals[J]. Nature Genetics, 2012, 44(6): 720-723. DOI:10.1038/ng.2281 |
| [35] |
Liu HH, Liu HQ, Zhou LN, et al. Parallel domestication of the Heading Date 1 gene in cereals[J]. Molecular Biology and Evolution, 2015, 32(10): 2726-2737. DOI:10.1093/molbev/msv148 |
| [36] |
Li Q, Li L, Yang XH, et al. Relationship, evolutionary fate and function of two maize co-orthologs of rice GW2 associated with kernel size and weight[J]. BMC Plant Biology, 2010, 10(1): 143-158. DOI:10.1186/1471-2229-10-143 |
| [37] |
Li Q, Yang XH, Bai GH, et al. Cloning and characterization of a putative GS3 ortholog involved in maize kernel development[J]. Theoretical and Applied Genetics, 2010, 120(4): 753-763. DOI:10.1007/s00122-009-1196-x |
| [38] |
Tao Y, Mace ES, Tai S, et al. Whole-genome analysis of candidate genes associated with seed size and weight in sorghum bicolor reveals signatures of artificial selection and insights into parallel domestication in cereal crops[J]. Frontiers in Plant Science, 2017, 8: 1237-1251. DOI:10.3389/fpls.2017.01237 |
| [39] |
Segami S, Kono I, Ando T, et al. Small and round seed 5 gene encodes alpha-tubulin regulating seed cell elongation in rice[J]. Rice, 2012, 5(1): 4-14. DOI:10.1186/1939-8433-5-4 |
| [40] |
Qi P, Lin YS, Song XJ, et al. The novel quantitative trait locus GL3. 1 controls rice grain size and yield by regulating Cyclin-T1;3[J]. Cell Research, 2012, 22(12): 1666-1680. DOI:10.1038/cr.2012.151 |
| [41] |
Wang Y, Xiong G, Hu J, et al. Copy number variation at the GL7 locus contributes to grain size diversity in rice[J]. Nature Genetics, 2015, 47(8): 944-948. DOI:10.1038/ng.3346 |
| [42] |
Mao H, Sun S, Yao J, et al. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Proceedings of the National Academy of Sciences, 2010, 107(45): 19579-19584. DOI:10.1073/pnas.1014419107 |
| [43] |
Nakagawa H, Tanaka A, Tanabata T, et al. SHORT GRAIN1 decreases organ elongation and brassinosteroid response in rice[J]. Plant Physiology, 2012, 158(3): 1208-1219. DOI:10.1104/pp.111.187567 |
| [44] |
Wang S, Wu K, Yuan Q, et al. Control of grain size, shape and quality by OsSPL16 in rice[J]. Nature Genetics, 2012, 44(8): 950-954. DOI:10.1038/ng.2327 |
| [45] |
Li F, Liu W, Tang J, et al. Rice DENSE AND ERECT PANICLE 2 is essential for determining panicle outgrowth and elongation[J]. Cell Research, 2010, 20(7): 838-849. DOI:10.1038/cr.2010.69 |
| [46] |
Abe Y, Mieda K, Ando T, et al. The SMALL AND ROUND SEED1 (SRS1/DEP2) gene is involved in the regulation of seed size in rice[J]. Genes Genetic Systems, 2010, 85(5): 327-339. DOI:10.1266/ggs.85.327 |
| [47] |
Hu J, Wang Y, Fang Y, et al. A rare allele of GS2 enhances grain size and grain yield in rice[J]. Molecular Plant, 2015, 8(10): 1455-1465. DOI:10.1016/j.molp.2015.07.002 |
| [48] |
Che R, Tong H, Shi B, et al. Control of grain size and rice yield by GL2-mediated brassinosteroid responses[J]. Nature Plants, 2015, 2(1): 15195-15226. |
| [49] |
Tanabe S, Ashikari M, Fujioka S, et al. A novel cytochrome P450 is implicated in brassinosteroid biosynthesis via the characterization of a rice dwarf mutant, dwarf11, with reduced seed length[J]. The Plant Cell, 2005, 17(3): 776-790. DOI:10.1105/tpc.104.024950 |
| [50] |
Liu L, Tong H, Xiao Y, et al. Activation of Big Grain1 significantly improves grain size by regulating auxin transport in rice[J]. Proceedings of the National Academy of Sciences, 2015, 112(35): 11102-11107. DOI:10.1073/pnas.1512748112 |
| [51] |
Si L, Chen J, Huang X, et al. OsSPL13 controls grain size in cultivated rice[J]. Nature Genetics, 2016, 48(4): 447-456. DOI:10.1038/ng.3518 |
| [52] |
Hong Z, Ueguchi-Tanaka M, Umemura K, et al. A rice brassinosteroid-deficient mutant, ebisu dwarf (d2), is caused by a loss of function of a new member of cytochrome P450[J]. The Plant Cell, 2003, 15(12): 2900-2910. DOI:10.1105/tpc.014712 |
| [53] |
Song XJ, Kuroha T, Ayano M, et al. Rare allele of a previously unidentified histone H4 acetyltransferase enhances grain weight, yield, and plant biomass in rice[J]. Proceedings of the National Academy of Sciences, 2015, 112(1): 76-81. DOI:10.1073/pnas.1421127112 |
| [54] |
Song XJ, Huang W, Shi M, et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics, 2007, 39(5): 623-630. DOI:10.1038/ng2014 |
| [55] |
Sosso D, Luo D, Li QB, et al. Seed filling in domesticated maize and rice depends on SWEET-mediated hexose transport[J]. Nature Genetics, 2015, 47(12): 1489-1493. DOI:10.1038/ng.3422 |
| [56] |
Martin A, Lee J, Kichey T, et al. Two cytosolic glutamine synthetase isoforms of maize are specifically involved in the control of grain production[J]. The Plant Cell, 2006, 18(11): 3252-3274. DOI:10.1105/tpc.106.042689 |
| [57] |
Duan P, Rao Y, Zeng D, et al. SMALL GRAIN 1, which encodes a mitogen-activated protein kinase kinase 4, influences grain size in rice[J]. The Plant Journal, 2014, 77(4): 547-557. DOI:10.1111/tpj.2014.77.issue-4 |
| [58] |
Ishimaru K, Hirotsu N, Madoka Y, et al. Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield[J]. Nature Genetics, 2013, 45(6): 707-711. DOI:10.1038/ng.2612 |
| [59] |
Lang Z, Wills DM, Lemmon ZH, et al. Defining the role of prolamin-box binding factor1 gene during maize domestication[J]. Journal of Heredity, 2014, 105(4): 576-582. DOI:10.1093/jhered/esu019 |
| [60] |
Weng J, Li B, Liu C, et al. A non-synonymous SNP within the isopentenyl transferase 2 locus is associated with kernel weight in Chinese maize inbreds (Zea mays L.)[J]. BMC Plant Biology, 2013, 13(1): 98-98. DOI:10.1186/1471-2229-13-98 |
| [61] |
Li XJ, Zhang YF, Hou MM, et al. Small kernel 1encodes a pentatricopeptide repeat protein required for mitochondrial nad7 transcript editing and seed development in maize (Zea mays) and rice (Oryza sativa)[J]. The Plant Journal, 2014, 79(5): 797-809. DOI:10.1111/tpj.2014.79.issue-5 |
| [62] |
Kitagawa K, Kurinami S, Oki K, et al. A novel kinesin 13 protein regulating rice seed length[J]. Plant Cell Physiology, 2010, 51(8): 1315-1329. DOI:10.1093/pcp/pcq092 |
| [63] |
Li Y, Fan C, Xing Y, et al. Natural variation in GS5 plays an important role in regulating grain size and yield in rice[J]. Nature Genetics, 2011, 43(12): 1266-1269. DOI:10.1038/ng.977 |
| [64] |
Shomura A, Izawa T, Ebana K, et al. Deletion in a gene associated with grain size increased yields during rice domestication[J]. Nature Genetics, 2008, 40(8): 1023-1028. DOI:10.1038/ng.169 |
| [65] |
Weng JF, Gu SH, Wan XY, et al. Isolation and initial characterization of GW5, a major QTL associated with rice grain width and weight[J]. Cell Research, 2008, 18(12): 1199-1209. DOI:10.1038/cr.2008.307 |
| [66] |
Li J, Chu H, Zhang Y, et al. The rice HGW gene encodes a ubiquitin-associated (UBA) domain protein that regulates heading date and grain weight[J]. PLoS One, 2012, 7(3): 34231-34243. DOI:10.1371/journal.pone.0034231 |