NAC名称来自矮牵牛(Petunia hybrida)的NAM及拟南芥的ATAF1/2和CUC2的第一个字母,最初由Souer等[1]和Aida等[2]发现。NAC是植物中最大的转录因子家族之一,全基因组研究表明拟南芥(Arabidopsis thaliana)中有117个NAC基因,水稻(Oryza sativa)中有151个,大豆(Glycine max)和烟草(Nicotiana tabacum)中各有101和152个[3-6]。
越来越多的研究已经证实植物的生长发育过程及胁迫条件下的生存与NAC转录因子有关,NAC转录因子家族成员的功能研究受到了公众的广泛关注。笔者总结了近年来NAC转录因子在调控细胞增殖与寿命、代谢物合成、细胞程序化死亡、胁迫响应、信号传导等方面的突破性进展(表 1)。可见,近年来关于NAC转录因子家族成员的功能逐渐成为研究热点。本文就植物NAC转录因子的最新研究进展展开综述,并预测未来的研究方向。
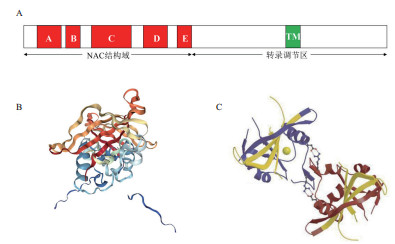
NAC结构域最初的特征在于来自NAM、ATAF1/2和CUC2的共有序列。NAC蛋白含有结合DNA的NAC结构域,其大约150个氨基酸位于蛋白质的N末端,通常分为5个子域(A-E),ACD亚结构域高度保守,B和E亚结构域可变,参与NAC蛋白的许多独特功能[7]。除了NAC结构域外,同一组中的大多数成员共享一个或多个基序。单子叶植物和双子叶植物中,SNAC组在保守结构域外的区域内具有一段28个氨基酸高度保守的基序。C末端转录调节区(Transcription regulatory region,TRR)可以激活或抑制转录。Nuruzzaman等[8]研究表明TRR基序对于一个特定的NAC亚家族是保守的,但在不同的亚家族中存在差异。因此,该区域赋予NAC蛋白质的个体功能变异。一些NAC转录因子还在C-末端区域中含有负责锚定到质膜或内质网的跨膜(Transmembrane,TM)基序。当植物处于特定发育阶段或者胁迫条件下,其被蛋白水解切割后移位至细胞核(图 1-A)[9]。但关于植物NAC蛋白三级结构的研究还很薄弱,目前,通过X光衍射仅获得ANAC019和OsNAC1 2个NAC转录因子保守域的晶体结构[10-11]。ANAC019是第一个进行晶体结构解析的NAC蛋白,其N-末端保守域由3个α-螺旋和6个反向平行的β-折叠组成,其同源二聚体模型也同时被报道(图 1-B,1-C)[10]。其实许多NAC蛋白存在与之类似的二聚体结合位点,表明大多数NAC蛋白均以同源二聚体的形式发挥调控作用[12]。
Ooka等[13]首次对水稻(单子叶植物)和拟南芥(双子叶植物)中的NAC家族基因进行了全面分析。通过序列相似性将已知的NAC家族蛋白分为两大组和18个亚组。随后,对NAC亚家族成员的研究均以Ooka的分类为依据进行展开。
2 NAC转录因子的功能 2.1 NAC转录因子在植物生长发育中的功能NAC转录因子首先在正向遗传筛选中被确定为发育过程的关键调节因子。NAC家族的基因已被证明可以调节广泛的发育过程,包括纤维发育[14]、次生壁合成[15]、细胞扩增[16]、叶片衰老[17]及果实成熟[18]等。最新研究表明NAC转录因子可以通过参与多种激素代谢途径调控植物生长发育。Huang等[14]确定了一个赤霉素(GA)信号级联,调节水稻的次生壁纤维素合成。NAC29/31和MYB61是水稻中的纤维素合成酶基因(CESA)调节剂,NAC29/31直接调节MYB61,后者反过来激活CESA表达;SLENDER RICE1(SLR1)是GA信号传导的关键阻遏物,与NAC直接相互作用,NAC-MYB-CESA信号被SLR1-NAC29/31所阻断。GA信号通过减弱SLR1和NAC之间的相互作用来促进纤维素合成。NAC转录因子还可以通过可选择的剪接实现对木材形成期间纤维细胞壁增厚等过程的调节。
毛果杨(Populus trichocarpa)的NAC转录因子PtrWND1B在次生木质部纤维细胞中发生可变剪接。两种PtrWND1B亚型在调控杨树纤维细胞分化期间细胞壁增厚中起拮抗作用。PtrWND1B-s过表达增强纤维细胞壁增厚,而PtrWND1B-l过表达抑制纤维细胞壁增厚[15]。NAC基因可以受乙烯调节参与细胞扩增。玫瑰(Rosa hybrida)中的RhNAC100转录物由乙烯通过miR164依赖的转录后调控进行调节,RhNAC100的过表达通过抑制花瓣细胞扩增显著减少了花瓣大小[16]。NAC家族的多个成员在拟南芥和其他植物物种中的叶子衰老调节中起作用。Ma等[17]发现番茄(Solanum lycopersicum)中ABA激活的NAC转录因子SlNAP2通过直接控制衰老相关基因SlSAG113和叶绿素降解相关基因SlSGR1和S1PAO的表达以及自我调节ABA含量,调控叶片衰老和果实产量。并且这种NAP调控机制在单子叶和双子叶中是高度保守的。水稻NAC转录因子OsNAC2直接激活叶绿素降解基因(OsSGR和OsNYC3)导致叶绿素水平降低、直接上调ABA生物合成基因(OsNCED3和OsZEP1)以及下调ABA分解代谢基因(OsABA8ox1)的表达而导致ABA水平的增加,最终加速叶片衰老[19]。
NAC转录因子还参与果实成熟的各个方面,研究发现AaNAC2、AaNAC3和AaNAC4结合猕猴桃萜合成酶1(AaTPS1)启动子,促使软枣猕猴桃(Actinidia arguta)果实中积累较多的单萜挥发物[18]。拟南芥JUB1可以抑制细胞内H2O2水平,降低衰老相关基因的水平,强烈延长植物寿命[20]。拟南芥转录因子ANAC087和ANAC046在柱状层和次生根中参与程序性细胞死亡的不同方面的调节[21]。
2.2 NAC转录因子在非生物胁迫中的功能NAC转录因子是植物应激反应过程中复杂信号传导的重要组成部分。考虑到来自不同植物的相对大量的NAC转录因子及其在复杂环境刺激下的未知和多样化作用,揭示其在非生物胁迫中的作用仍然是一个相当大的挑战。最近的一些新证据证实了NAC转录因子在调控与植物非生物反应相关的转录重编程中发挥的积极作用。
2.2.1 NAC转录因子对干旱胁迫的响应番茄JUN-GBRUNNEN1(JUB1)直接结合SlDREB1、SlDREB2和SlDELLA的启动子,增加了番茄的耐旱性。JUB1作为耐旱的转录调节因子,控制非生物胁迫相关基因的调控网络在拟南芥和番茄中相当保守[22]。拟南芥ANAC096与bZIP型转录因子ABRE结合因子和ABRE结合蛋白(ABF/AREB)合作以激活ABA诱导基因帮助植物在脱水和渗透下存活[23]。Wu等[24]发现枸橘(Poncirus trifoliata)中PtrNAC72是腐胺生物合成的阻遏物,并且可能通过调节腐胺相关的活性氧稳态而负面调节干旱胁迫响应。甘蓝轮枝菌(Verticillium longisporum)侵染拟南芥诱导束鞘细胞转分化成木质部,VND6和VND7在转分化中发挥关键作用并伴随植物耐旱性增加[25]。苜蓿(Medicago falcata)在干旱胁迫下,MfNACsa去棕榈酰化移位至细胞核,结合MtGlyl启动子,通过维持谷胱甘肽库处于还原状态而导致耐旱性[26]。
2.2.2 NAC转录因子对盐胁迫的响应Liu等[27]发现盐芥中获得的TsNAC1(拟南芥ANAC072的同系物)能够靶向一种重要的质子转运蛋白TsVP1,TsVP1利用来自PPi水解的能量与H+-ATPase合作将H+转移到液泡或细胞外环境中来提高植物的耐盐性,而拟南芥中AVP1对盐胁迫无反应,并且不受ANAC072的正调控,TsNAC1和ANAC072之间的这些差异有助于解释拟南芥和盐芥之间耐盐性的差异。Ana等[28]发现了一种新的“MdNAC047-MdERF3-乙烯-盐耐受性”调节途径,MdNAC047直接与MdERF3(乙烯反应因子)启动子结合并激活其转录,促进乙烯的释放并赋予植物耐盐性,为乙烯和盐胁迫之间的联系提出了新见解。
2.2.3 NAC转录因子对温度胁迫的响应在单子叶植物和双子叶植物中保守的C重复基序结合因子(CBF)在调节大量冷相关(COR)基因中起至关重要的作用,在冷信号网络中充当中心枢纽[29]。一些研究表明,NAC转录因子参与了CBF-COR途径。例如,GmNAC20通过直接结合DREB1A的启动子并抑制DREB1C的表达来调节COR并介导应激耐受[30];MaNAC1通过与MaCBF1相互作用参与冷应激[29]。这表明NAC-DREB/CBF-COR为重要的信号级联,调节植物对低温胁迫的反应。至今,热应激反应研究有限,仅有为数不多的NAC基因被证实响应植物的高温胁迫。例如,小麦(Triticum aestivum)TaNAC2L可通过调节应激反应基因的表达来改善耐热性[31]。Fang等[32]报道了一种应激反应的NAC基因SNAC3,其可以直接靶向ROS相关酶基因,通过调节ROS稳态来赋予热胁迫耐受性。
2.3 NAC转录因子对生物胁迫的响应大多数报道表明NAC转录因子是植物免疫系统,基础防御和系统获得性抗性方面的核心组成部分,这是一个活跃的研究领域[33]。ATAF2由于抑制发病相关(PR)基因表达导致对尖孢镰刀菌的易感性增加[34]。与ATAF2不同,ATAF1可通过假设的负调节因子间接调节PR基因的抑制作用,提高对强毒白粉菌的抗性和提高抗穿透性[35],表明ATAF亚家族调节不同的病原体显然具有保守但非冗余的功能。Huang等[33]发现番茄中6种NAC转录因子对番茄黄叶卷曲病毒感染有反应,这些转录因子还与其他蛋白质相互作用,包括蛋白磷酸酶和丝裂原活化蛋白激酶以及转录因子。NAC和其他转录因子之间存在的调节途径可能是植物应对病原体感染的机制的重要组成部分。JA反应性SNAC因子可能在生物和非生物胁迫反应中起作用是合理的。番茄中两种同源NAC转录因子JA2和JA2L通过不同的机制差异调节病原体诱导的气孔关闭和重新开放[36]。
2.4 NAC转录因子的其他功能除了在植物生长发育以及一些常见胁迫中发挥重要作用外,NAC转录因还具有诸多功能。ANAC78和ANAC53蛋白是蛋白酶体应激调节的中心调节剂,它通过提供足够的26S蛋白酶体和其他蛋白质稳态因子来减轻蛋白质毒性应激[37]。ANAC013通过与线粒体功能障碍基序顺式调节元件的直接相互作用介导线粒体逆行信号并增加氧化应激耐受性[38]。ANAC017在线粒体功能障碍期间激活AOX1a的转录,介导H2O2诱导的转录本丰度的变化,从而整合顺行和逆行调节途径[39]。ZmNAC84被钙/钙调蛋白依赖性蛋白激酶磷酸化,激活下游基因参与ABA诱导的抗氧化防御[40]。Hofmann等[41]发现SHYG(ANAC047)在洪涝胁迫下激活下游靶标ACO5来促进乙烯生物合成,通过涉及乙烯的正反馈回路改变了叶柄的角度,保持叶子与空气接触,在洪涝响应中起作用。小麦TaNAC2-5A可直接结合编码硝酸盐转运蛋白和谷氨酰胺合成酶的基因的启动子区域,提高根系生长和硝酸盐流入速率,从而提高了根系获得氮的能力并具有较高的籽粒产量[42]。拟南芥NAC1转录因子基因特异性表达以响应叶片外植体中的伤口,并在根尖再生中发挥作用[43]。
3 结论与展望为揭示NAC转录因子的作用而进行的科学研究已经证明,这类转录调节因子在植物生长发育、响应非生物胁迫和生物胁迫中都有明显的参与,具有广阔的应用前景。基因工程未来应该努力微调特定时间和空间模式中关键NAC转录因子的表达,以最大限度对植物生长和产量产生正面影响。
NAC转录因子的研究主要集中在拟南芥、番茄、水稻等模式植物,对非模式植物研究较少。而且大部分NAC转录因子尚处于基因克隆、鉴定和功能初步分析层面。除了挖掘出更多NAC功能基因外,未来的研究重点还应该放在确定NAC下游(如直接或间接调控的基因)或上游(如信号分子)的遗传组分,这将有助于提供它们参与不同信号转导通路更多的证据,全面认识其调控网络。此外,为了使NAC蛋白质发挥其作用优势,研究蛋白质-蛋白质相互作用将为阐明植物在生长发育和逆境胁迫下的生理、生化过程的分子机制提供新的线索和切入点。
| [1] |
Souer E, vanHouwelingen A, Kloos D, et al. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 1996, 85(2): 159-170. DOI:10.1016/S0092-8674(00)81093-4 |
| [2] |
Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation in Arabidopsis:an analysis of the cup-shaped cotyledon mutant[J]. Plant Cell, 1997, 9: 841-857. DOI:10.1105/tpc.9.6.841 |
| [3] |
Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290(5499): 2105-2110. DOI:10.1126/science.290.5499.2105 |
| [4] |
Xiong Y, Liu T, Tian C, et al. Transcription factors in rice:a genome-wide comparative analysis between monocots and eudicots[J]. Plant Molecular Biology, 2005, 59(1): 191-203. DOI:10.1007/s11103-005-6503-6 |
| [5] |
Rushton PJ, Bokowiec MT, et al. TOBFAC:the database of tobacco transcription factors[J]. BMC Bioinformatics, 2008, 9: 53. DOI:10.1186/1471-2105-9-53 |
| [6] |
Pinheiro GL, Marques CS, Costa MD, et al. Complete inventory of soybean NAC transcription factors:sequence conservation and expression analysis uncover their distinct roles in stress response[J]. Gene, 2009, 444: 10-23. DOI:10.1016/j.gene.2009.05.012 |
| [7] |
Olsen AN, Ernst HA, Leggio LL, et al. NAC transcription factors:Structurally distinct, functionally diverse[J]. Trends in Plant Science, 2005, 10(2): 79-87. DOI:10.1016/j.tplants.2004.12.010 |
| [8] |
Nuruzzaman M, Sharoni AM, Kikuchi S. Roles of NAC transcription factors in the regulation of biotic and abiotic stress responses in plants[J]. Front Microbiol, 2013, 4: 248. |
| [9] |
Li S, Wang N, Ji DD, et al. Evolutionary and functional analysis of membrane-bound NAC transcription factor genes in soybean[J]. Plant Physiol, 2016, 172(4): 1804-1820. |
| [10] |
Ernst HA, Olsen AN, Skriver K, et al. Structure of the conserved domain of ANAC, a member of the NAC family of transcription factors[J]. Embo Reports, 2004, 5(3): 297-303. DOI:10.1038/sj.embor.7400093 |
| [11] |
Chen Q, et al. A structural view of the conserved domain of rice stress-responsive NAC1[J]. Protein Cell, 2011, 2(1): 56-63. |
| [12] |
Welner DH, Lindemose S, Grossmann JG, et al. DNA binding by the plant-specific NAC transcription factors in crystal and solution:A firm link to WRKY and GCM transcription factors[J]. Biochemical Journal, 2012, 444(3): 395. DOI:10.1042/BJ20111742 |
| [13] |
Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Research, 2003, 10(6): 239-247. DOI:10.1093/dnares/10.6.239 |
| [14] |
Huang DB, Wang SG, Zhang BC, et al. A gibberellin-mediated Della-NAC signaling cascade regulates cellulose synthesis in rice[J]. Plant Cell, 2015, 27(6): 1681-1696. DOI:10.1105/tpc.15.00015 |
| [15] |
Zhao YJ, Sun JY, Xu P, et al. Intron-mediated alternative splicing of WOOD-ASSOCIATED NAC TRANSCRIPTION FACTOR1B regulates cell wall thickening during fiber development in Populus species[J]. Plant Physiol, 2014, 164(2): 765-776. DOI:10.1104/pp.113.231134 |
| [16] |
Pei HX, Ma N, Tian J, et al. An NAC transcription factor controls ethylene-regulated cell expansion in flower petals[J]. Plant Physiol, 2013, 163(2): 775-791. DOI:10.1104/pp.113.223388 |
| [17] |
Ma XM, Zhang YJ, Turečková V, et al. The NAC transcription factor SlNAP2 regulates leaf senescence and fruit yield in tomato[J]. Plant Physiol, 2018, 177(3): 1286-1302. DOI:10.1104/pp.18.00292 |
| [18] |
Nieuwenhuizen NJ, Chen XY, et al. Natural variation in monoter-pene synthesis in kiwifruit:transcriptional regulation of terpene synthases by NAC and ETHYLENE-INSENSITIVE3-like transcri-ption factors[J]. Plant Physiol, 2015, 167(4): 1243-1258. DOI:10.1104/pp.114.254367 |
| [19] |
Mao CJ, Lu SC, Lv B, et al. A Rice NAC transcription factor promotes leaf senescence via ABA biosynthesis[J]. Plant Physiol, 2017, 174(3): 1747-1763. DOI:10.1104/pp.17.00542 |
| [20] |
Wu AH, Allu AD, Garapati P, et al. JUNGBRUNNEN1, a reactive oxygen species-responsive NAC transcription factor, regulates longevity in Arabidopsis[J]. Plant Cell, 2012, 2: 482-506. |
| [21] |
Huysmans M, Buono RA, Skorzinski N, et al. NAC transcription factors ANAC087 and ANAC046 control distinct aspects of programmed cell death in the Arabidopsis columella and lateral root cap[J]. Plant Cell, 2018, 30(9): 2197-2213. DOI:10.1105/tpc.18.00293 |
| [22] |
Thirumalaikumar VP, Devkar V, Mehterov N, et al. NAC transcription factor JUNGBRUNNEN1 enhances drought tolerance in tomato[J]. Plant Biotechnol J, 2018, 16(2): 354-366. DOI:10.1111/pbi.2018.16.issue-2 |
| [23] |
Xu ZY, Kim SY, Hyeon DY, et al. The Arabidopsis NAC transcription factor ANAC096 cooperates with bZIP-type transcription factors in dehydration and osmotic stress responses[J]. Plant Cell, 2013, 25(11): 4708-4724. DOI:10.1105/tpc.113.119099 |
| [24] |
Wu H, Fu B, Sun PP, et al. A NAC transcription factor represses putrescine biosynthesis and affects drought tolerance[J]. Plant Physiol, 2016, 172(3): 1532-1547. DOI:10.1104/pp.16.01096 |
| [25] |
Reusche M, Thole K, Janz D, et al. Verticillium infection triggers VASCULAR-RELATED NAC DOMAIN7-dependent de novo xylem formation and enhances drought tolerance in Arabidopsis[J]. Plant Cell, 2012, 24(9): 3823-3837. DOI:10.1105/tpc.112.103374 |
| [26] |
Duan M, et al. A lipid-anchored NAC transcription factor is translocated into the nucleus and activates Glyoxalase I expression during drought stress[J]. Plant Cell, 2017, 7: 1748-1772. |
| [27] |
Liu C, et al. TsNAC1 is a key transcription factor in abiotic stress resistance and growth[J]. Plant Physiol, 2018, 1: 742-756. |
| [28] |
Ana JP, Yaoa JF, Xub RR, et al. An apple NAC transcription factor enhances salt stress tolerance by modulating the ethylene response[J]. Physiologia Plantarum, 2018, 164(3): 279-289. DOI:10.1111/ppl.2018.164.issue-3 |
| [29] |
Shan W, et al. Banana fruit NAC transcription factor MaNAC1 is a direct target of MaICE1 and involved in cold stress through interac-ting with MaCBF1[J]. Plant Cell Environ, 2014, 9: 2116-2127. |
| [30] |
Hao YJ, Wei W, Song QX, et al. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants[J]. Plant J, 2011, 68(2): 302-313. DOI:10.1111/j.1365-313X.2011.04687.x |
| [31] |
Guo WW, Zhang JX, Zhang N, et al. The wheat NAC transcription factor TaNAC2L is regulated at the transcriptional and post-translational levels and promotes heat stress tolerance in transgenic Arabidopsis[J]. PLoS One, 2015, 10(8): e0135667. DOI:10.1371/journal.pone.0135667 |
| [32] |
Fang Y, Liao K, Du H, et al. A stress-responsive NAC transcription factor SNAC3 confers heat and drought tolerance through modulation of reactive oxygen species in rice[J]. J Exp Bot, 2015, 66(21): 6803-6817. DOI:10.1093/jxb/erv386 |
| [33] |
Huang Y, Li T, Xu ZS, et al. Six NAC transcription factors involved in response to TYLCV infection in resistant and susceptible tomato cultivars[J]. Plant Physiol Biochem, 2017, 120: 61-74. DOI:10.1016/j.plaphy.2017.09.020 |
| [34] |
Delessert C, Kazan K, Wilson IW, et al. The transcription factor ATAF2 represses the expression of pathogenesis related genes in Arabidopsis[J]. Plant J, 2005, 43(5): 745-757. DOI:10.1111/tpj.2005.43.issue-5 |
| [35] |
Chen YJ, et al. The barley HvNAC6 transcription factor affects ABA accumulation and promotes basal resistance against powdery mildew[J]. Plant Mol Biol, 2013, 83(6): 577-590. DOI:10.1007/s11103-013-0109-1 |
| [36] |
Du MM, et al. Closely related NAC transcription factors of tomato differentially regulate stomatal closure and reopening during pathogen attack[J]. Plant Cell, 2014, 7: 3167-3184. |
| [37] |
Gladman NP, Marshall RS, Lee KH, et al. The proteasome stress regulon is controlled by a pair of NAC transcription factors in Arabidopsis[J]. Plant Cell, 2016, 28(6): 1279-1296. DOI:10.1105/tpc.15.01022 |
| [38] |
De Clercq I, Vermeirssen V, Van Aken O, et al. The membrane-bound NAC transcription factor ANAC013 functions in mitochondrial retrograde regulation of the oxidative stress response in Arabidopsis[J]. Plant Cell, 2013, 25(9): 3472-3490. DOI:10.1105/tpc.113.117168 |
| [39] |
Ng S, Ivanova A, Duncan O, et al. A membrane-bound NAC transcription factor, ANAC017, mediates mitochondrial retrograde signaling in Arabidopsis[J]. Plant Cell, 2013, 9:: 3450-3471. |
| [40] |
Zhu Y, Yan JV, Liu WJ, et al. Phosphorylation of a NAC transcription factor by a Calcium/Calmodulin-dependent protein kinase regulates abscisic acid-induced antioxidant defense in maize[J]. Plant Physiol, 2016, 171(3): 1651-1664. DOI:10.1104/pp.16.00168 |
| [41] |
Hofmann NR. A NAC transcription factor for flooding:SHYG helps plants keep their leaves in the air[J]. Plant Cell, 2013, 25(12): 4771. DOI:10.1105/tpc.113.251212 |
| [42] |
He X, Qu BY, Li WJ, et al. The nitrate-inducible NAC transcription factor TaNAC2-5A controls nitrate response and increases wheat yield[J]. Plant Physiol, 2015, 169(3): 1991-2005. |
| [43] |
Chen XD, Cheng JF, Chen LQ, et al. Auxin-independent NAC pathway acts in response to explant-specific wounding and promotes root tip emergence during de novo root organogenesis in Arabidopsis[J]. Plant Physiol, 2016, 170(4): 2136-2145. DOI:10.1104/pp.15.01733 |