日本结缕草抗逆性较强,在草坪方面应用广泛。在维持其草坪景观以及运动场需求方面,草坪修剪能够增强抗逆能力以及刺激基部分蘖,因此研究日本结缕草应对机械损伤的自我修复和促进芽分化的分子机制具有重要的借鉴意义。
随着科学技术的发展,植物部分基因在应答外界不同环境变化的信号转导或转录调控方面至关重要。相关研究指出,转录因子通常以家族形式出现,并直接或者间接地作用于目标基因的顺式作用元件上,正调控或负调控该基因的表达,从而应答外界逆境胁迫。在机械损伤过程中,AP2/EREBP、NAC以及WRKY转录因子起重要作用[1-3]。植物在遭受干旱、冷害以及高盐胁迫时,因细胞失水造成的机械损伤能够引起AP2/EREBP转录因子的应答,进而调控抗逆基因[3-4]。对于NAC转录家族来说,NAC不仅能够调节植株衰老以及营养物质的转运[2, 5],也能响应干旱、高盐、植物激素等诱导因子以及病害和组织损伤防御[6-7]。植物的WRKY是在植物中分布较为广泛地一类转录因子,不仅响应高温、高盐、冷害、机械损伤等非生物胁迫,也应答病菌、虫害等生物胁迫以及受到水杨酸(SA)和茉莉酸(JA)信号转导途径的调控[8-11]。但是对于日本结缕草应对机械损伤的自我调控机制来说,相关的AP2/EREBP、NAC以及WRKY转录因子还未挖掘。
通过对不同胁迫时间的机械损伤转录组的分析,发现众多转录组数据量较大,人工分析效率低且出错率较高。通过使用Mapman工具的使用,可大大提高对转录组数据分析的便捷,且能够对其验证。
本研究通过将转录组拼接得到的序列进行比对,将其数据转换为Mapman识别的探针ID,筛选AP2/EREBP、NAC和WRKY这三类转录因子的差异基因(DEG),并与转录组注释相结合,为研究日本结缕草应答机械损伤胁迫的调控机制奠定基础。
1 材料与方法 1.1 材料Li等[12]对日本结缕草机械损伤胁迫0(T7)、2(T8)和6 h(T9)的转录组数据。
1.2 方法将组装好的序列转换为Mapman识别的探针ID,将其载入Mapman中,Mapman对蛋白或代谢物注释编号,称之为BIN,在Mapman数据库收录的每一个蛋白序列或代谢物序列都有一个ID,筛选归类为AP2/EREBP、NAC以及WRKY的差异基因以及拼接的长度,将转录组数据中的Swiss-Prot与Nr数据库中的注释进行匹配,并通过Mapman将T8和T9较于T7的表达量差异可视化,T7(机械损伤0 h)为对照,T8(机械损伤2 h)与T9(机械损伤6 h)为处理组,T8-T7为T8与T7时差异基因表达量之差,T9-T7为T9与T7时差异基因表达量之差,T8-T7/T9-T7为两组差异基因表达热图。
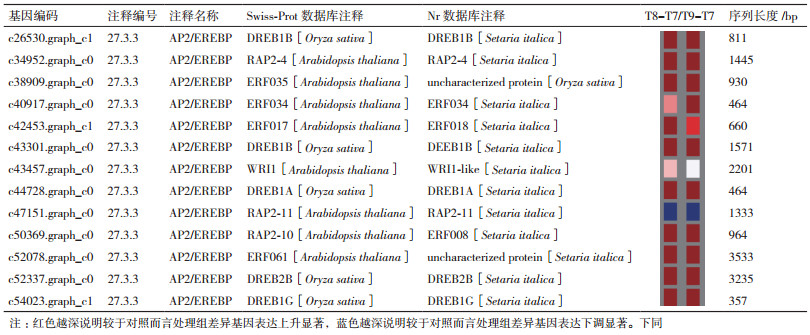
2 结果 2.1 AP2/EREBP转录因子相关的DEG信息分析通过mapman对转录组获得的序列进行匹配(表 1),AP2/EREBP转录因子获得DEGs差异基因13个,其组装的序列长度为464-3 533 bp。在转录组Swiss-Prot数据库注释到8个拟南芥物种基因,另外5个unigenes注释为水稻。除此之外,Nr数据库中的转录因子主要集中于谷子这一类农作物,其中有一个基因未能得到明确的注释结果;只有一个未得到注释蛋白匹配到水稻中。与T7相比,在T8和T9时,大部分AP2/EREBP转录因子呈上调趋势,只有注释为RAP-2-11的差异基因c47151.graph_c0表现为下调。机械损伤后日本结缕草叶片产生的相关AP2/EREBP转录因子,在适应机械损伤机制中起着重要的调节作用,能够增强抵抗高盐、干旱以及病害的能力。
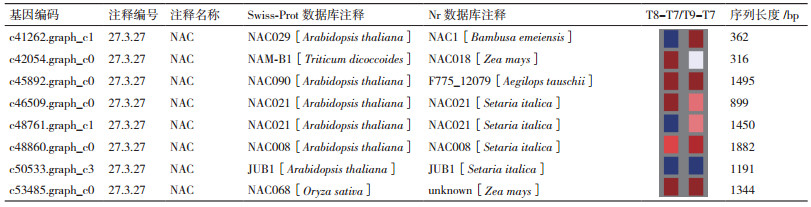
通过筛选共获得Bin编号27.3.27的NAC转录因子差异基因8个(表 2),其组装成的转录本长度最短为316 bp,最长为1 882 bp。在Swiss-Prot数据库中,差异基因主要注释为拟南芥,另2个差异基因分别注释为野生型小麦和水稻。对于Nr数据库,有4个差异基因注释为谷子,其次,有2个差异基因注释为玉米,最后的差异基因分别注释为竹子和节节麦中。与T7相比,在T8和T9时,有2个差异基因分别注释为NAC029(c41262.graph_c1)和NAC021(c48761.graph_c1),呈先下调再上调的趋势,注释为JUB1基因功能的c50533.graph_c3全程表达受到抑制,其余差异基因都是明显地上调。
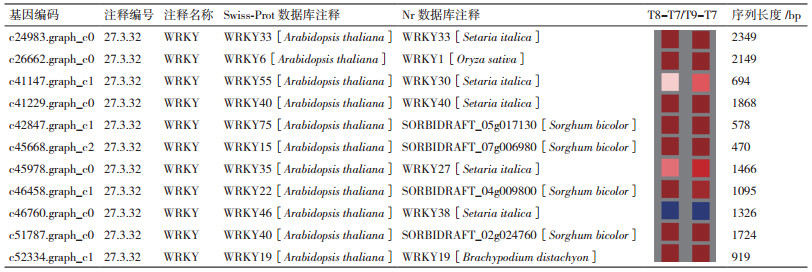
在拟南芥中,WRKY转录因子包括74个植物特异性响应植物胁迫的转录因子,主要在病害防御,机械损伤响应以及衰老过程中起着重要作用[1]。在筛选出的差异基因WRKY转录因子中,其共分为三大类WRKY即Ⅰ、Ⅱ和Ⅲ,即注释为WRKY33的c24983.graph_c0和抗病X-TIR-NBS-LRR-X即注释为WRKY19的c52334.graph_c1属于Ⅰ类,在Ⅱ类又分为子群,即注释为WRKY6的c26662.graph_c0为Ⅱb,注释为WRKY75的c42847.graph_c1为Ⅱc,注释为WRKY22的c46458.graph_c1和注释为WRKY35的c45978.graph_c0为Ⅱe,注释为WRKY15的c45668.graph_c2,注释为WRKY40的c41229.graph_c0和c51787.graph_c0为Ⅱd,以及注释为WRKY46的c46760.graph_c0和注释为WRKY55的c41147.graph_c1属于Ⅲ类。其序列长度分布为470-2 349 bp,在Nr数据库中,有5个基因注释到谷子中,其次注释到高粱的基因有4个,其余的2个基因分别注释到水稻和二穗短柄草中。这一类基因的大多数都与对照相比表达量上升,仅只有一个注释为WRKY46的c46760.graph_c0表达量明显下降。
3 讨论日本结缕草由于在非生物胁迫和生物胁迫中具有较强的抗逆性,管理粗放,广泛应用于园林景观以及运动场之中。然而目前对日本结缕草经常受到修剪后适应生长的特性机制不太了解,因此我们分析机械损伤日本结缕草转录组数据中受机械损伤诱导的相关的转录因子AP2/EREBP,NAC以及WRKY在这损伤过程中的作用。
AP2/EREBP类转录因子在应对非生物胁迫过程中都特异性地识别并绑定5'-[AG]CCGAC-3'的DAN序列,结合DRE元件介导高盐和脱水诱导的转录,并调节植物对ABA、病原、高盐、低温以及干旱胁迫的应答反应[4];在机械损伤日本结缕草转录组中,发现有5个DREB类转录因子注释到水稻中,而注释到拟南芥中的转录因子有4个ERF类、3个RAP类以及1个WRI类,除了RAP2-11转录因子与对照相比表现出明显地下调,大部分AR2/EREBP转录因子差异性表达上调,意味着机械损伤后提高对非生物和生物胁迫的反应能力,增强抗逆性。
有关研究表明,NAC转录因子不仅作用于植物的生长发育器官模式建成,也响应非生物胁迫和生物胁迫的应答[13-18]。NAC029一方面能够促进ABA的产生进而造成叶绿素的降解使植物表现出衰老的表型[5, 13],与JUB1延缓叶片衰老的功能相反[18],另一方面还能够响应高盐、干旱胁迫以及ABA处理,并且负调控MdDREB2A、MdCBF1和MdCBF4在干旱、高盐和低温胁迫的响应[16-17]。在本研究转录组数据中,NAC029和JUB1基因的表达在T8时都表现出较于T7明显地下调,仅有NAC029的表达在T9时较T7明显地上调,意味着叶片损伤部位的衰老与NAC029和JUB1相关,并且快速响应非生物胁迫,全程上调NAC068调控细胞程序性死亡以阻止病原体入侵[6]。对于得到注释为野生型小麦的NAM-B1的c42054.graph_c0来说,其功能主要负责促进叶片衰老以及将其营养物质转移到正在生长中的谷粒中[2],c46509.graph_c0和c48761.graph_c1共同注释为NAC021(NAC1),其主要被生长素诱导以及介导生长素信号转导机制促进侧根的生长,并且该转录因子激活下游生长素响应基因DBP和AIR3[19],加上NAC090和NAC008在应答外界胁迫时参与植物的体细胞胚胎发生[20-21],这些基因在损伤的日本结缕草叶片中显著地上调,说明这些NAC转录因子在叶片损伤部位传递信号至茎端分生组织并共同调控其生长。
大多数WRKY转录因子在病菌侵染时被快速诱导,进而通过SA途径介导的抗病相关基因PR1快速地进行防御,是一种植物系统获得抗性的表现,比如OsWRKY6直接绑定水杨酸合成关键酶OsICS1,促进SA的产生,使其激活OsPR10a提高植株的抗病性,而且在病害部位提高转录因子水平造成细胞程序性死亡组织阻止病菌进一步侵染[10, 22]。活性氧(ROS)的增多也能够诱导PeWRKY6-1的表达,随之与衰老相关的转录因子(SAG12-1)的表达量增多,造成ROS量多的叶片部位叶片衰老[23],是植物自我防御的反应机制。然而WRKY6和WRKY33在持续的虫害中通过提高JA水平来防御自身[24-25]。同样,WRKY22、WRKY46、WRKY47、WRKY53以及WRKY75通过SA信号转导机制正调控防御相关基因PR1对细菌和真菌产生基本的抗性[26-29];WRKY18、WRKY40和WRKY60相互作用也在病害防御方面起着重要作用,其中WRKY40与WRKY18相互拮抗,使得WRKY18在对P.syringae的抗性方面由于WRKY40的转录抑制使植物易感病[30]。除此之外,WRKY46还能快速响应干旱胁迫、盐胁迫以及氧化胁迫,能够调控参与细胞间的渗透保护剂的形成和氧化还原态稳态的相关基因的表达[11]。对于机械损伤日本结缕草叶片来说,只有注释为WRKY46功能的c46760.graph_c0出现明显的下调,由于机械损伤为非生物胁迫,该基因的下调导致ROS的大量积累,发生氧爆现象,进而诱导相关的WRKY转录因子进行自我防御,并且促进损伤部位的衰老来隔离外界环境对损伤部位的感染,起到对病菌感染的预防作用。
4 结论通过将转录组数据载入至mapman中得到的注释与转录组数据中的Swiss-Prot数据库注释相同,与部分Nr数据库注释相同。AP2/EREBP转录因子有13个差异基因,NAC转录因子有8个差异基因,WRKY转录因子有11个差异基因,这三类转录因子在机械损伤T8和T9时都出现与T7相比较大的表达量差异。日本结缕草在遭受机械损伤时AP2/EREBP和WRKY类转录因子能够增强对非生物胁迫和生物胁迫的应答能力,增强抗逆性,并且NAC类转录因子传递信号至茎端分生组织并调控其生长。
| [1] |
Eulgem T, Rushton PJ, Robatzek S, et al. The WRKY superfamily of plant transcription factors[J]. Trends in Plant Science, 2000, 5(5): 199-206. DOI:10.1016/S1360-1385(00)01600-9 |
| [2] |
Cristobal U, Assaf D, Tzion F, et al. A NAC Gene regulating senescence improves grain protein, zinc, and iron content in wheat[J]. Science, 2006, 314(5803): 1298-1301. DOI:10.1126/science.1133649 |
| [3] |
Gutterson N, Reuber TL. Regulation of disease resistance pathways by AP2/ERF transcription factors[J]. Curr Opin Plant Biol, 2004, 7(4): 465-471. DOI:10.1016/j.pbi.2004.04.007 |
| [4] |
Chen JQ, Meng XP, Yun Z, et al. Over-expression of OsDREB genes lead to enhanced drought tolerance in rice[J]. Biotechnol Lett, 2008, 30(12): 2191-2198. DOI:10.1007/s10529-008-9811-5 |
| [5] |
Guo Y, Gan S. AtNAP, a NAC family transcription factor, has an important role in leaf senescence[J]. Plant J, 2006, 46(4): 601-612. DOI:10.1111/tpj.2006.46.issue-4 |
| [6] |
Lee MH, Jeon HS, Kim HG, et al. An Arabidopsis NAC transcription factor NAC4 promotes pathogen-induced cell death under negative regulation by microRNA164[J]. New Phytol, 2017, 214(1): 343-360. DOI:10.1111/nph.14371 |
| [7] |
Xu Z, Gongbuzhaxi, Wang C, et al. Wheat NAC transcription factor TaNAC29 is involved in response to salt stress[J]. Plant Physiol Biochem, 2015, 96(2015): 356-363. |
| [8] |
Liu F, Li X, Wang M, et al. Interactions of WRKY15 and WRKY33 transcription factors and their roles in the resistance of oilseed rape to Sclerotinia infection[J]. Plant Biotechnol J, 2018, 16(4): 911-925. DOI:10.1111/pbi.2018.16.issue-4 |
| [9] |
Guo P, Li Z, Huang P, et al. A tripartite amplification loop involving the transcription factor WRKY75, salicylic acid, and reactive oxygen species accelerates leaf senescence[J]. Plant Cell, 2017, 29(11): 2854-2870. DOI:10.1105/tpc.17.00438 |
| [10] |
Choi C, Hwang SH, Fang IR, et al. Molecular characterization of Oryza sativa WRKY6, which binds to W-box-like element 1 of the Oryza sativa pathogenesis-related(PR)10a promoter and confers reduced susceptibility to pathogens[J]. New Phytol, 2015, 208(3): 846-859. DOI:10.1111/nph.13516 |
| [11] |
Ding ZJ, Yan JY, Xu XY, et al. Transcription factor WRKY46 regulates osmotic stress responses and stomatal movement independently in Arabidopsis[J]. Plant J, 2014, 79(1): 13-27. DOI:10.1111/tpj.2014.79.issue-1 |
| [12] |
Li L, He X, Zhao F, et al. WUS and PIN1-related genes undergo dynamic expressional change during organ regeneration in response to wounding in Zoysia japonica[J]. Mol Biol Rep, 2018, 45(6): 1733-1744. DOI:10.1007/s11033-018-4317-2 |
| [13] |
Jiading Y, Eric W, Michael U. A NAP-AAO3 regulatory module promotes chlorophyll degradation via ABA biosynthesis in Arabidopsis leaves[J]. Plant Cell, 2014, 26(12): 4862-4874. DOI:10.1105/tpc.114.133769 |
| [14] |
Kewei Z, Susheng G. An abscisic acid-AtNAP transcription factor-SAG113 protein phosphatase 2C regulatory chain for controlling dehydration in senescing Arabidopsis leaves[J]. Plant Physiol, 2012, 158(2): 961-969. DOI:10.1104/pp.111.190876 |
| [15] |
Araujo NCP, Afonso R, Bringela A, et al. Peroxides with antiplasmodial activity inhibit proliferation of Perkinsus olseni, the causative agent of Perkinsosis in bivalves[J]. Parasitol Int, 2013, 62(6): 575-582. DOI:10.1016/j.parint.2013.06.010 |
| [16] |
郭运娜. MdNAC29基因在苹果干旱和盐胁迫中的作用和机制[D].沈阳: 沈阳农业大学, 2018. http://cdmd.cnki.com.cn/Article/CDMD-10157-1018993300.htm
|
| [17] |
An JP, Li R, Qu FJ, et al. An apple NAC transcription factor negatively regulates cold tolerance via CBF-dependent pathway[J]. J Plant Physiol, 2018, 221(2018): 74-80. |
| [18] |
Wu A, Allu AD, Garapati P, et al. JUNGBRUNNEN1, a reactive oxygen species-responsive NAC transcription factor, regulates longevity in Arabidopsis[J]. Plant Cell, 2012, 24(2): 482-506. DOI:10.1105/tpc.111.090894 |
| [19] |
Xie Q, Frugis G, Colgan D, et al. Arabidopsis NAC1 transduces auxin signal downstream of TIR1 to promote lateral root development[J]. Genes Dev, 2000, 14(13): 3024-3036. |
| [20] |
Jin F, Hu L, Yuan D, et al. Comparative transcriptome analysis between somatic embryos(SEs)and zygotic embryos in cotton: evidence for stress response functions in SE development[J]. Plant Biotechnol J, 2014, 12(2): 161-173. DOI:10.1111/pbi.12123 |
| [21] |
Yi D, Alvim Kamei CL, Cools T, et al. The Arabidopsis SIAMESE-RELATED cyclin-dependent kinase inhibitors SMR5 and SMR7 regulate the DNA damage checkpoint in response to reactive oxygen species[J]. Plant Cell, 2014, 26(1): 296-309. DOI:10.1105/tpc.113.118943 |
| [22] |
Silke R, Somssich IE. Targets of AtWRKY6 regulation during plant senescence and pathogen defense[J]. Genes Dev, 2002, 16(9): 1139-1149. DOI:10.1101/gad.222702 |
| [23] |
Rosenvasser S, Mayak S, Friedman H. Increase in reactive oxygen species(ROS)and in senescence-associated gene transcript(SAG)levels during dark-induced senescence of Pelargonium cuttings, and the effect of gibberellic acid[J]. Plant Sci, 2006, 170(4): 873-879. DOI:10.1016/j.plantsci.2005.12.010 |
| [24] |
Skibbe M, Qu N, Galis I, et al. Induced plant defenses in the natural environment: Nicotiana attenuata WRKY3 and WRKY6 coordinate responses to herbivory[J]. Plant Cell, 2008, 20(7): 1984-2000. DOI:10.1105/tpc.108.058594 |
| [25] |
Birkenbihl RP, Diezel C, Somssich IE. Arabidopsis WRKY33 is a key transcriptional regulator of hormonal and metabolic responses toward Botrytis cinerea infection[J]. Plant Physiol, 2012, 159(1): 266-285. DOI:10.1104/pp.111.192641 |
| [26] |
Tsuneaki A, Guillaume T, Joulia P, et al. MAP kinase signalling cascade in Arabidopsis innate immunity[J]. Nat, 2002, 415(6875): 977-983. DOI:10.1038/415977a |
| [27] |
Hu Y, Dong Q, Yu D. Arabidopsis WRKY46 coordinates with WRKY70 and WRKY53 in basal resistance against pathogen Pseudomonas syringae[J]. Plant Science, 2012, 185-186(2012): 288-297. |
| [28] |
Moreau M, Degrave A, Vedel R, et al. EDS1 contributes to nonhost resistance of Arabidopsis thaliana against Erwinia amylovora[J]. Molecular Plant-Microbe Interactions, 2012, 25(3): 421-430. DOI:10.1094/MPMI-05-11-0111 |
| [29] |
Encinas-Villarejo S, Maldonado AM, Amil-Ruiz F, et al. Evidence for a positive regulatory role of strawberry(Fragariaxananassa)Fa WRKY1 and Arabidopsis AtWRKY75 proteins in resistance[J]. J Exp Bot, 2009, 60(11): 3043-3065. DOI:10.1093/jxb/erp152 |
| [30] |
Xu X, Chen C, Fan B, et al. Physical and functional interactions between pathogen-induced Arabidopsis WRKY18, WRKY40, and WRKY60 transcription factors[J]. Plant Cell, 2006, 18(5): 1310-1326. DOI:10.1105/tpc.105.037523 |