2. 上海海洋大学 水产科学国家级实验教学示范中心,上海 201306;
3. 上海海洋大学 上海水产养殖工程技术研究中心,上海 201306
2. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306;
3. Shanghai Engineering Research Center of Aquaculture, Shanghai Ocean University, Shanghai 201306
色素框同源蛋白(Chromobox homolog,CBX)是组成多梳蛋白复合体(Polycomb group complex,PcG)的主要核心蛋白[1-4],目前在哺乳动物中发现该蛋白家族包含CBX2、CBX4、CBX6、CBX7和CBX8 5个成员[5]。CBXs各蛋白虽然具有共同保守的结构域,但各个成员的分子大小、组织分布及生物学功能却存在较大的差异。由于近年来CBX2在哺乳动物人和小鼠性发育中作用的发现,逐渐成为研究的热点[5]。
cbx2基因最早于1992年在果蝇中被发现[6],在1997年被确认是PcG复合体的组成成分[7]。该复合体作为转录抑制物广泛参与细胞周期调控、X-染色体失活、干细胞分化、衰老及肿瘤发生等重要生命过程[8-10]。在人和鼠中,cbx2基因主要在性腺中表达,尤其在精巢中的表达量较高。Katoh-Fukui等[11]发现,在小鼠中对M33(cbx2)基因缺失或进行扰乱,会产生雄鼠向雌鼠转变的性逆转现象;在人类中,cbx2基因的缺失则会导致46,XY性别发育障碍(Disorders of sex development,DSD)[12],最新研究发现,cbx2基因有两个亚型cbx2.1和cbx2.2,cbx2.1即为上述的cbx2,cbx2.2如果发生突变也会丧失对性别发育相关基因表达的调节,从而导致严重的46,XY DSD缺陷[13]。这些研究表明cbx2可能通过调节性别发育相关基因的表达进而在哺乳动物(人和小鼠)的性发育中发挥着重要的调控作用。但迄今为止,关于cbx2在低等脊椎动物,尤其是鱼类性别分化和性腺发育中的作用未见报道。
牙鲆(Paralichthys olivaceus)隶属于鲽形目(Pleuronectiforms),是我国重要的海水养殖经济鱼类之一,雌性个体较雄性个体大且生长快[14],其性腺发育与分化的分子机制一直是近年来鱼类生殖发育研究的重点。鉴于cbx2基因在哺乳动物性发育中的重要作用,本研究利用PCR克隆和生物信息学方法鉴定了牙鲆cbx2基因,并采用RT-PCR技术分析了其在牙鲆不同组织的表达,旨为进一步阐明cbx2基因在牙鲆性腺发育和分化中的功能奠定重要基础。
1 材料与方法 1.1 材料牙鲆成鱼购自上海市铜川路水产市场,解剖取其精巢、卵巢、脑、肝脏、肌肉、胃及鳃组织。各组织样品经焦碳酸二乙酯处理水冲洗干净,立即置于Trizol(Invitrogen)中匀浆,再用于总RNA提取。
1.2 方法 1.2.1 牙鲆cbx2基因的分子克隆通过查询NCBI数据库(https://www.ncbi.nlm.nih.gov/),我们获得了来自牙鲆的cbx2 cDNA(XM_020094544)序列,该cDNA序列包括完整的开放阅读框(Open reading frame,ORF)。为保证克隆cDNA序列的准确性,设计3对基因特异性引物进行PCR扩增并测序,经拼接得到其全长cDNA并推导的氨基酸序列,见图 1。

|
| 图 1 牙鲆cbx2 cDNA及推导的氨基酸序列 |
首先通过ExPASy-ProtParam在线分析牙鲆CBX2蛋白质的理化性质与疏水性;利用SignalP 4.1预测其是否具有蛋白跨膜区和蛋白信号肽;利用SMART和swissmodel预测CBX2蛋白的二级和三级结构。
然后在NCBI中查阅各物种cbx2同源基因信息,并进行基因结构比较分析;采用BioEdit对牙鲆CBX2与其他物种进行氨基酸序列比对,并利用MEGA5.1构建其系统进化树。其他物种同源基因的序列登录号如下:杜氏鰤(Seriola dumerili),XP_022595013.1;尖吻鲈(Lates calcarifer),XP_018518344.1;大黄鱼(Larimichthys crocea),XP_019117076.1;尼罗罗非鱼(Oreochromis niloticus),XP_005469121.1;半滑舌鳎(Cynoglossus semilaevis),XP_008327382.1;大西洋鲑(Salmo salar),青鳉(Oryzias latipes),NP_001098386.1;人(Homo sapiens),NP_005180.1;斑马鱼(Danio rerio),NP_919354.1;小鼠(Mus musculus)NP_031649;热带爪蟾(Xenopus tropicalis),XP_017949680。
最后,通过Ensembl Genome Browser(http://www.ensembl.org/index.html)确定cbx2及其相邻基因在染色体上的定位。
1.2.3 RT-PCR检测cbx2基因的组织表达上述总RNA经DNase I(Promega)处理以去除基因组DNA,然后在M-MLV reverse transcriptase(Promega)作用下合成cDNA第一条链。使用Primer5.0设计牙鲆cbx2基因和内参基因18s的PCR引物,cbx2的上下游引物序列分别为:GTCACAGATGTCACCGCTAATC和TCAGAACCCAAATCCCCTC,18s的上下游引物序列分别为:AGTTGGTGGAGCGATTTG和CTCGGCGAAGGGTAGACA。
PCR反应在C1000 TouchTM Thermal Cycler(Bio-Rad)上进行,其反应体系为:cDNA1µL,上下游引物1 µL,Taq PCR Master Mix(上海生物工程公司)10 µL及7 µL灭菌水。PCR扩增程序为:95℃2 min;95℃ 30 s;60℃ 30 s;72℃ 30 s;进行35个循环反应;然后72℃延伸5 min。获得的PCR产物用于2%琼脂糖凝胶电泳,并在凝胶成像系统上观察、拍照,以检测cbx2在牙鲆各组织的表达情况。
2 结果 2.1 cbx2 cDNA的序列克隆及其同源基因结构比较经PCR克隆和测序获得的牙鲆cbx2 cDNA序列如图 1所示,其开放阅读框长为1 584 bp,编码527个氨基酸。
脊椎动物cbx2同源基因结构比较分析发现,cbx2基因在哺乳动物人和小鼠中包含6个外显子,其编码区与非编码区在整个基因的分布大致相同;而在珠鸡(Numida meleagris)、热带爪蟾、斑马鱼、青鳉、罗非鱼(Oreochromis spp.)和牙鲆中则只有5个外显子,其中牙鲆、青鳉、尼罗罗非鱼的cbx2基因的编码区与非编码区排列分布更为相似,均具有较小的外显子1、2、3、4和大的外显子5,外显子还包含了一个较大的3’-非翻译区(Untranslated region,UTR)。
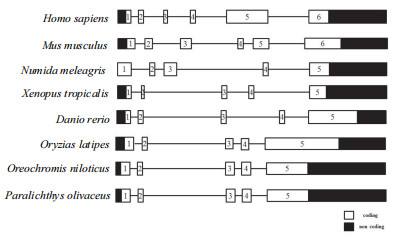
|
| 图 2 脊椎动物cbx2基因结构的比较分析 |
SMART和SWISS-MODEL预测牙鲆CBX2蛋白的二级与三级结构如图 3,其二级结构中包含1个染色质结构域和4个低复杂结构域(紫色区域),该染色质结构域是真核生物蛋白质模体之一,高度保守,含30-50个氨基酸,常结合甲基化氨基酸残基,存在于动植物细胞核内参与调节染色质结构的若干蛋白质中。三级结构分析发现,牙鲆CBX2蛋白包含13.28%的α-螺旋、11.20%的延伸链、3.24%的β转角和72.11%的不规则卷曲,其中以α螺旋和无规则卷曲为主,不均匀地分布于整个蛋白质多肽链上。

|
| 图 3 牙鲆CBX2蛋白的二级及三级结构 |
经SignalP 4.1分析,CBX2蛋白无信号肽、无跨膜区域,表明该蛋白既不是分泌蛋白质也不是跨膜蛋白质。在其编码的527个氨基酸序列中包含20种常见的氨基酸,其中Ser(S)含量最高(15.9%),其次为Lys(K)(9.7%),而Pyl(O)和Sec(U)的含量为0。通过ExPASy-ProtParam分析得到其分子式为C2418H3966N752O805S8,分子量为56 578.98 K,等电点为10.03,是一种不稳定的碱性亲水性蛋白。
2.3 牙鲆cbx2基因的同源性与系统进化分析氨基酸序列比对分析(图 4)发现,牙鲆cbx2基因cDNA推导出的氨基酸序列与杜氏鰤的同源性最高,为89%;其次是尖吻鲈和大黄鱼,其同源性分别为88%和85%;与尼罗罗非鱼、半滑舌鳎和大西洋鲑的同源性分别为77%、68%和60%,与青鳉、斑马鱼的同源性仅有57%和53%;与两栖类和哺乳动物的同源性较低,如与热带爪蟾的同源性为42.51%,与人类和小鼠分别为41%和40.52%。但是,CBX2蛋白的同源结构域在脊椎动物是保守的,尤其是在鱼类中高度一致。

|
| 图 4 牙鲆CBX2和其他物种的氨基酸序比对分析 黑色区域代表保守的氨基酸残基;其他颜色区域代表物种间的差异氨基酸 |
系统进化树分析发现,牙鲆CBX2与所有鱼类聚为一支(图 5)。基因同线性分析显示了cbx2基因与相邻基因在染色体上的相对位置,虽然在鱼类中与cbx2基因相邻的基因被定名的较少,但在哺乳类的人类、小鼠和鸟类的珠鸡中,与cbx2基因邻近的基因较为一致,均有cbx8和enpp7基因(图 6)。

|
| 图 5 脊椎动物CBX2蛋白的系统进化树 |
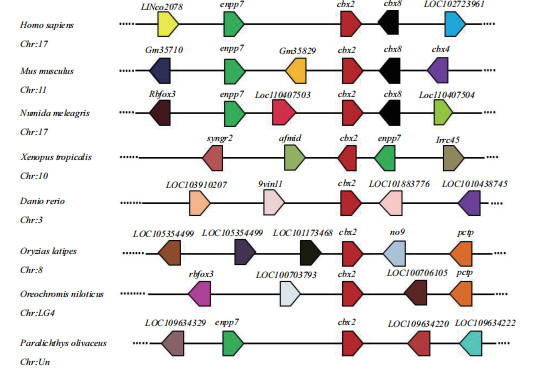
|
| 图 6 脊椎动物cbx2基因的同线分析 |
RT-PCR结果(图 7)显示,cbx2基因在成体牙鲆的各个组织中均有表达,但在不同组织的表达水平差异显著。其中牙鲆cbx2基因在精巢中表达量最高,卵巢次之,在脑中也有较高的表达,但在肌肉、胃、鳃和肝脏中的表达则较低。

|
| 图 7 牙鲆cbx2基因的组织表达 |
本研究通过分子克隆与生物信息学方法鉴定了牙鲆cbx2基因,基因结构、氨基酸同源性和系统进化分析表明cbx2基因在脊椎动物进化中较为保守,理化性质分析结果显示牙鲆CBX2蛋白是一种偏碱性的亲水性蛋白,编码该蛋白的氨基酸中以丝氨酸(Sr)的含量为最高,而丝氨酸富集区是磷酸化的主要位点[15]。研究表明该丝氨酸富集区的残基正是CBX2的磷酸化位点,且在细胞内是稳定磷酸化的[16]。从鱼类到人类的进化中,丝氨酸富集区的长度有所减少,其在鱼类中一般有24个丝氨酸残基,而在人类中有一个延伸的16个丝氨酸残基[17]。在成年小鼠的肝细胞中,CBX2的鼠同源物M33穿梭于细胞核与细胞质之间,在快速增殖的细胞中M33蛋白仅在细胞核中以高度磷酸化的形式存在,而在休眠细胞的细胞质中,M33则主要以去磷酸化的形式存在[8]。空间结构预测发现,牙鲆CBX2蛋白以α螺旋和无规则卷曲为主,其二级结构中含有1个染色质结构域和4个低复杂结构域。研究表明,该染色质结构域常结合甲基化氨基酸残基,主要调控异染色质与基因表达,此外还与H3K27me3结合,H3K27me3是另一种典型的表观遗传基因沉默标记,常位于发育相关基因的启动子区[18],这表明CBX2蛋白在表观遗传调控中发挥着重要的作用。
性别分化与性腺发育一直是生命科学领域的研究热点,目前关于cbx2基因在性发育中的研究主要集中在人类和小鼠中,而在低等脊椎动物包括鱼类中几乎未见报道。本研究发现,cbx2基因在牙鲆精巢的表达量最高,在卵巢和脑中也有较高的表达,而在其他组织的表达量较低。基于2014年Fagerberg等[19]的RNA测序,数据分析显示在人类正常组织中,cbx2在精巢的表达量最高,卵巢、胎盘次之,在肺中也有一定量的表达,而在其他组织中表达量较低;同样基于2014年Yue等[20]在小鼠中的测序,显示cbx2在脑、四肢表达量较高,在卵巢和性腺也有较高的表达,但在其他组织表达量则较低。研究表明,cbx2基因在哺乳动物的细胞周期变化[8, 21]、减数分裂、同源染色体的联会和生殖细胞增殖分化中发挥着较为重要的作用[22]。Katoh-Fukui等[23]早在1998年发现,在小鼠中通过对m33(cbx2)基因进行扰乱或基因缺失,会产生雄性小鼠向雌性转变的性反转现象,且由于m33的突变会导致雌性小鼠胚胎的生殖脊发育迟缓,性腺生长的缺陷在Y染色体特定的SRY(Sex region of Y chromosome)表达时会显现出来,暗示m33(cbx2)缺失可能是通过干扰SRY上游的某些因子从而导致了性反转,因而推测m33(cbx2)可能是小鼠性别决定的重要基因[11]。进一步研究发现,在m33(cbx2)基因敲除的小鼠中,m33(cbx2)是ad4bp/sf1表达的上游调控基因,能刺激靶基因同向表达;在人类中的研究表明cbx2通过激活相关的启动子区能刺激靶基因sf1/nr5a1的表达,如果cbx2突变将失去对靶基因sf1/nr5a1的调控,从而引起性别发育异常[12]。这与先前报道的CBX2作为PcGs的组成成分,是组蛋白表观遗传修饰的转录抑制因子不同,cbx2还可作为基因转录的激活因子发挥作用。事实上,cbx2一方面为精巢中雄性表达的特性基因;另一方面还通过调控Foxl2和Wnt4信号通路进而抑制雌性的某些信号通路[24]。因此,cbx2基因在牙鲆精巢和卵巢中的高表达,提示了该基因在牙鲆性腺发育中可能发挥着重要的作用,而具体的功能和作用机制非常值得进一步深入的研究。
4 结论本研究通过PCR克隆和测序获得了牙鲆cbx2基因的cDNA序列,采用多种生物信息学方法明确了其分子特性,并利用RT-PCR技术证明了其在牙鲆性腺(尤其是精巢)组织具有较高的表达,初步探讨了cbx2基因在牙鲆性腺发育中的作用。
| [1] |
Simon JA, Kingston RE. Mechanisms of polycomb gene silencing:knowns and unknowns[J]. Nat Rev Mol Cell Biol, 2009, 10(10): 697-708. |
| [2] |
Surface LE, Thornton SR, Boyer LA. Polycomb group proteins set the stage for early lineage commitment[J]. Cell Stem Cell, 2010, 7(3): 288-298. DOI:10.1016/j.stem.2010.08.004 |
| [3] |
Gao Z, Zhang J, Bonasio R, et al. PCGF homologs, CBX proteins, and RYBP define functionally distinct PRC1 family complexes[J]. Molecular Cell, 2012, 45(3): 344-356. DOI:10.1016/j.molcel.2012.01.002 |
| [4] |
Tavares L, Dimitrova E, Oxley D, et al. RYBP-PRC1 complexes mediate H2A ubiquitylation at polycomb target sites independently of PRC2 and H3K27me3[J]. Cell, 2012, 148(4): 664-678. DOI:10.1016/j.cell.2011.12.029 |
| [5] |
Ma RG, Zhang Y, Sun TT, et al. Epigenetic regulation by polycomb group complexes:focus on roles of CBX proteins[J]. Journal of Zhejiang University Science B, 2014, 15(5): 412-428. DOI:10.1631/jzus.B1400077 |
| [6] |
DeCamillis M, Cheng NS, Pierre D, Brock HW. The polyhomeotic gene of Drosophila encodes a chromatin protein that shares polytene chromosome-binding sites with Polycomb[J]. Genes Dev, 1992, 6(2): 223-232. |
| [7] |
Alkema MJ, Bronk M, Verhoeven E, et al. Identification of Bmi1-interacting proteins as constituents of a multimeric mammalian polycomb complex[J]. Genes Dev, 1997, 11(2): 226-240. |
| [8] |
Noguchi K, Shiurba R, Higashinakagawa T. Nuclear translocation of mouse polycomb m33 protein in regenerating liver[J]. Biochemical and Biophysical Research Communications, 2002, 291(3): 508-515. DOI:10.1006/bbrc.2002.6480 |
| [9] |
Morey L, Pascual G, Cozzuto L, et al. Nonoverlapping functions of the Polycomb group Cbx family of proteins in embryonic stem cells[J]. Cell Stem Cell, 2012, 10(1): 47-62. DOI:10.1016/j.stem.2011.12.006 |
| [10] |
Clermont PL, Sun L, Crea F, et al. Genotranscriptomic meta-analysis of the Polycomb gene CBX2 in human cancers:initial evidence of an oncogenic role[J]. British Journal of Cancer, 2014, 111(8): 1663-1672. DOI:10.1038/bjc.2014.474 |
| [11] |
Katoh-Fukui Y, Tsuchiya R, et al. Male-to-female sex reversal in M33 mutant mice[J]. Nat, 1998, 393(6686): 688-692. DOI:10.1038/31482 |
| [12] |
Biason-Lauber A, Konrad D, Meyer M, et al. Ovaries and female phenotype in a girl with 46, XY karyotype and mutations in the CBX2 gene[J]. Am J Hum Genet, 2009, 84(5): 658-663. |
| [13] |
Sproll P, Eid W, Gomes CR, et al. Assembling the jigsaw puzzle:CBX2 isoform 2 and its targets in disorders/differences of sex development[J]. Mol Genet Genomic Med, 2018, 6(5): 785-795. DOI:10.1002/mgg3.2018.6.issue-5 |
| [14] |
尤锋, 许建和, 倪静, 等. 牙鲆同质雌核发育人工诱导研究[J]. 高技术通讯, 2008, 18(8): 874-880. DOI:10.3772/j.issn.1002-0470.2008.08.019 |
| [15] |
O'Mahony DJ, Smith SD, Xie W, et al. Analysis of the phosphorylation, DNA-binding and dimerization properties of the RNA polymerase I transcription factors UBF1 and UBF2[J]. Nucleic Acids Research, 1992, 20(6): 1301-1308. DOI:10.1093/nar/20.6.1301 |
| [16] |
Kawaguchi T, Machida S, Kurumizaka H, et al. Phosphorylation of CBX2 controls its nucleosome-binding specificity[J]. Journal of Biochemistry, 2017, 162(5): 343-355. DOI:10.1093/jb/mvx040 |
| [17] |
Senthilkumar R, Mishra RK. Novel motifs distinguish multiple homologues of Polycomb in vertebrates:expansion and diversification of the epigenetic toolkit[J]. BMC Genomics, 2009, 10(1): 549-566. DOI:10.1186/1471-2164-10-549 |
| [18] |
Rajasekhar VK, Begemann M. Concise review:roles of polycomb group proteins in development and disease:a stem cell perspective[J]. Stem Cells, 2007, 25(10): 2498-2510. DOI:10.1634/stemcells.2006-0608 |
| [19] |
Fagerberg L, Hallstrom BM, Oksvold P, et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics[J]. Molecular & Cellular Proteomics:MCP, 2014, 13(2): 397-406. |
| [20] |
Yue F, Cheng Y, Breschi A, et al. A comparative encyclopedia of DNA elements in the mouse genome[J]. Nat, 2014, 515(7527): 355-364. DOI:10.1038/nature13992 |
| [21] |
Hirose S, Komoike Y, Higashinakagawa T. Identification of a nuclear localization signal in mouse polycomb protein, M33[J]. Zoological Science, 2006, 23(9): 785-791. DOI:10.2108/zsj.23.785 |
| [22] |
Baumann C, De La Fuente R. Role of polycomb group protein cbx2/m33 in meiosis onset and maintenance of chromosome stability in the Mammalian germline[J]. Genes, 2011, 2(1): 59-80. DOI:10.3390/genes2010059 |
| [23] |
Katoh-Fukui Y, Owaki A, Toyama Y, et al. Mouse Polycomb M33 is required for splenic vascular and adrenal gland formation through regulating Ad4BP/SF1 expression[J]. Blood, 2005, 106(5): 1612-1620. DOI:10.1182/blood-2004-08-3367 |
| [24] |
Eid W, Opitz L, Biason-Lauber A. Genome-wide identification of CBX2 targets:insights in the human sex development network[J]. Molecular Endocrinology, 2015, 29(2): 247-257. DOI:10.1210/me.2014-1339 |




