耐药菌感染被称为“当代最大的健康威胁之一”,特别是具有多重耐药能力的超级细菌,如产超广谱β-内酰胺酶的肠杆菌科细菌、耐甲氧西林金黄色葡萄球菌、耐万古霉素肠球菌、多重耐药铜绿假单胞菌等[1-4],2014年世界卫生组织发布的《抗菌素耐药:全球监测报告》显示:每年美国和欧盟范围内因感染超级耐药细菌而死亡的人数分别达到6.3和2.5万人。
海洋环境尤其是近岸海域容易受到耐药菌的污染,造成耐药菌在海洋环境的流行[5-7]。有学者从海水浴场中分离出8株具有多重耐药性的大肠杆菌[8],这些耐药菌对人体健康有较大威胁。国内外学者对海水浴引起的疾病风险进行系统评估的结果显示,浴者比非浴者有更高的胃肠道感染的风险,且涨潮和雨后会增加其风险[9-10],也有数据表明海水浴场中产超广谱β-内酰胺酶的细菌可能会引起海水浴者的尿路感染[11],因此,海水浴已成为人类感染耐药菌的潜在途径。国内大多数关于海水浴场耐药菌的研究都是基于粪便污染指示菌,研究对象比较单一,且对细菌耐药谱的研究不全面,本实验对从海水浴场筛选到的所有MDRO进行了耐药特征的分析,并对其进行种属鉴定,为海水浴场耐药细菌的污染状况提供了更加详细的数据。
1 材料与方法 1.1 材料 1.1.1 主要试剂R2A培养基与MH培养基(青岛高科园海博生物技术有限公司);抗生素:氨苄西林、氯霉素、链霉素、四环素、卡那霉素(北京索莱宝科技有限公司);药敏纸片(杭州微生物试剂有限公司);EasyPure Bacteria Genomic DNA Kit、2×TransTaq High Fidelity PCR SuperMix(北京全式金生物技术有限公司);引物合成及测序由美吉生物医药科技有限公司完成。
1.1.2 实验主要仪器TC型基因扩增仪-LifeECO型号(杭州博日科技有限公司);DYY-6C型电泳仪(北京六一生物科技有限公司);蓝盾501S可见光凝胶投射仪(厦门致善生物科技股份有限公司);德国SIGMA 1-16/16K小型台式离心机(德国Sigma离心机有限公司)。
1.2 方法 1.2.1 采样方法于2017年8月到青岛海水浴场采样,以浴者密度最大点、距浴者密度最大点两侧各50米处及距离海岸1公里处为海水采样点,以浴场近岸的潮上带、潮间带、潮下带的表面沙层5 cm以下为沙样采样点。各采样点设3个平行样,水样用孔径为0.22 µm的滤膜过滤,将滤膜与沙样收集盒冷藏于采样箱中。
1.2.2 细菌的分离与纯化在无菌条件下将剪碎的滤膜或沙样置于一定的生理盐水中,经振荡器充分震荡,用生理盐水将各个样本稀释为不同梯度浓度,分别涂布于R2A琼脂培养基,29℃培养48 h,根据形态、颜色、表面湿润程度、边缘形态等差异挑取单菌落纯化培养。
1.2.3 MDRO的筛选通过抗性筛选平板进行MDRO的筛选。将分离出的细菌在含有不同抗生素的R2A琼脂培养基上分别进行划线培养,筛选出能同时对三种及以上抗生素耐药的细菌。抗生素及其终浓度分别为氨苄西林(100 mg/L)、氯霉素(30 mg/L)、链霉素(30 mg/L)、四环素(30 mg/L)、卡那霉素(30 mg/L)。
1.2.4 药敏试验采用K-B药敏纸片扩散法对筛选到的MDRO做药敏试验。用生理盐水将培养18 h的菌液稀释至0.5麦氏比浊标准涂布于MH琼脂培养基,并将药敏纸片贴于培养基表面。质控菌株采用大肠埃希菌(ATCC 25922)。判读标准参照临床实验室标准化协会(Clinical and Laboratory Standards Institute,CLSI)[12]推荐的标准。
1.2.5 PCR扩增模板的制备使用EasyPure Bacteria Genomic DNA Kit试剂盒提取MDRO的基因组DNA,并用可见光凝胶透射仪观察结果,作为PCR反应的模板。
1.2.6 16S rDNA与耐药基因的PCR扩增PCR反应体系为:2×TransTaq High Fidelity PCR SuperMix1为10 µL,去离子水6µL,上下游引物各1 µL,DNA模板2 µL。16S rDNA的PCR扩增采用通用引物(27F,1492R),反应程序为:95℃预变性5 min;95℃变性1min、52℃退火1min、72℃延伸10 min,此3个程序进行35个循环;72℃终延伸10 min。耐药基因的PCR反应程序为:95℃预变性5 min;94℃变性30 s、退火30 s、72℃延伸5min,此3个程序进行30个循环;72℃终延伸5 min(退火温度及引物见表 3)。
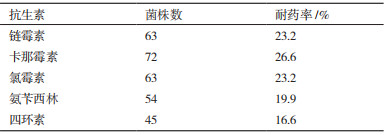
经过对样本细菌的分离培养,共得到271株可培养菌株,将其涂布于五种含不同抗生素的抗性筛选平板上,结果显示271株可培养菌株对五种抗生素的耐药率差异不大,其中对四环素耐药率最低(16.6%),见表 1。经筛选,对3种及以上抗生素耐药的MDRO有60株,MDRO的分离率为22.1%,其中有3株MDRO来自于距离岸边1公里处的水样中。
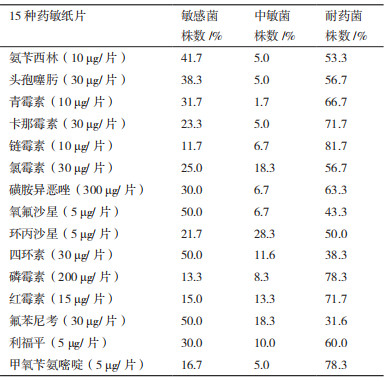
药敏试验结果(表 2)显示,60株MDRO对九大类15种抗生素的耐药率在31.6%-81.7%,除氟氧沙星、环丙沙星、四环素和氟苯尼考外,60株MDRO对其他11种抗生素的耐药率均在50%以上,其中对链霉素(81.7%)、磷霉素(78.3%)、甲氧苄氨嘧啶(78.3%)的耐药率较高,对氟苯尼考耐药率最低(31.6%)。从抗生素种类分析,60株MDRO对氨基糖苷类、磺胺类和磷霉素类抗生素耐药率较高,对喹诺酮类、四环素类和氟苯尼考类抗生素较敏感。
药敏试验结果(图 1)显示60株MDRO中至少对4种抗生素耐药,有5株MDRO对15种抗生素全部耐药,其中对10种以上抗生素耐药的有35(58.3%)株。值得注意的是编号为H2B3、H2B17、H2C27、H6C3、H6C11的MDRO对15种药敏纸片均表现为耐药,且均为条件致病菌。
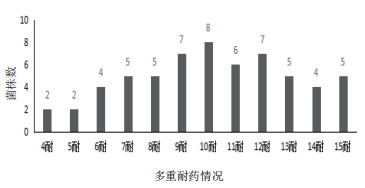
|
| 图 1 MDRO对15种抗生素的多重耐药性 |
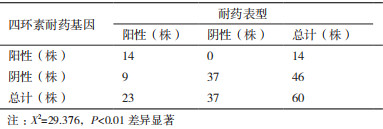
为探究MDRO的耐药表型与耐药基因间的相关性,选择链霉素和四环素耐药基因进行PCR检测,引物见表 3。结果显示,60株MDRO中有2株显示strA-strB阳性,31株显示aadA1阳性,33株链霉素耐药基因阳性菌株中有97%的菌株表现为耐药表型阳性;另外,有11株显示tetA阳性,3株显示tetB阳性,14株四环素耐药基因阳性的菌株全部表现为四环素耐药表型阳性。
2.4 耐药表型与耐药基因的相关性分析为进一步验证MDRO的耐药表型与耐药基因间的相关性,用SPSS 19.0软件对其进行卡方检验,结果显示,链霉素耐药基因阳性菌株中的耐药表型阳性菌株显著高于耐药基因阴性菌株,四环素耐药基因阳性菌株中的耐药表型阳性菌株显著高于耐药基因阴性菌株,表明细菌耐药性与耐药基因的检出率呈正相关(表 4,表 5)。
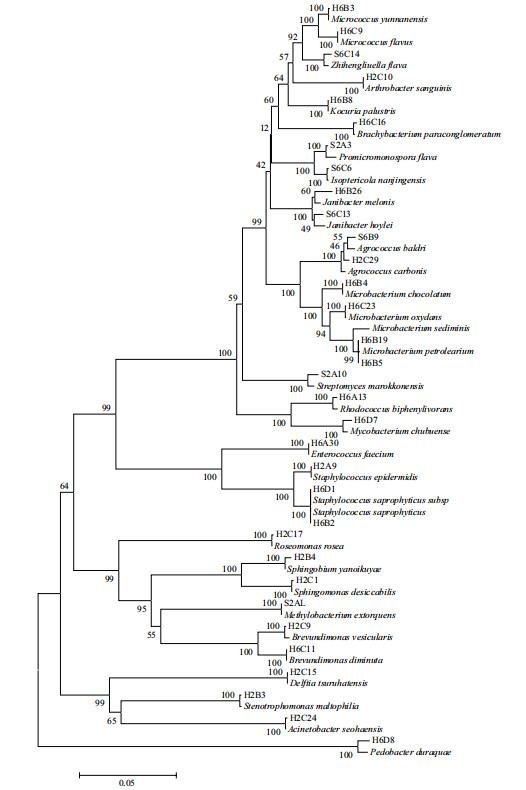
提取60株MDRO的基因组DNA作为模板,PCR法扩增其16s rDNA基因片段并通过琼脂糖凝胶电泳观察结果(图 2),将PCR产物送测序公司测序。通过Mothur软件将测序所得的基因序列进行操作分类单元聚类分析,将相似性≥97%的序列归为一个OTU,共分为33个OTU。选出每个OTU的代表序列,并通过NCBI网站进行比对,选出同源性最高的序列作为参考序列并下载。将33个代表序列及其参考序列用MEGA 5.0软件以邻接法构建系统发育树(图 3),初步确定耐药细菌所属菌属。
|
| 图 2 部分细菌样品16S rDNA基因片段电泳图 |
|
| 图 3 33个OTU代表序列及其参考序列构建的系统进化树 |
结合系统发育树和比对结果进行初步鉴定,从青岛海水浴场筛选出的60株MDRO属于6个纲,24个菌属,分别为α-变形菌纲的短波单胞菌属(Brevundimonas)、鞘脂菌属(Sphingobium)、鞘氨醇单胞菌属(Sphingomonas)、玫瑰单胞菌属(Rose-omonas)、甲基营养菌属(Methylobacterium);β-变形菌纲的戴尔福特菌属(Delftia);γ-变形菌纲的寡养单胞菌属(Stenotrophomonas)、不动杆菌属(Aci-netobacter);芽孢杆菌纲的葡萄球菌属(Staphylococcus)、肠球菌属(Enterococcus);放线菌纲的节杆菌属(Arthrobacter)、微杆菌属(Microbacterium)、土壤球菌属(Agrococcus)、红球菌属(Rhodococcus)、微球菌属(Micrococcus)、考克氏菌属(Kocuria)、两面神菌属(Janibacter)、短状杆菌属(Brachybacterium)、分枝杆菌属(Mycobacterium)、原小单孢菌属(Promicromonospora)、链霉菌属(Streptomyces)、白蚁菌属(Isoptericola)、刘志恒菌属(Zhihengliuella);拟杆菌纲的拟杆菌属(Pedobacter)。其中寡养单胞菌属(Stenotrophomonas)占比最大(16%),其次分别为微杆菌属(Microbacterium)、短波单胞菌属(Brevundimonas)、葡萄球菌属(Staphyloco-ccus)分别占比13%、8%、8%,其他菌属数量较少。值得注意的是,对15种药敏纸片均表现为耐药的H2B3、H2B17、H2C27菌株经初步鉴定为嗜麦芽窄食单胞菌,H6C3、H6C11菌株为缺陷短波单胞菌,均为条件致病菌。
3 讨论海水浴场受人类活动影响较大,通常含有较多耐药的致病菌,海水浴者通过皮肤接触或海水吞咽等方式易感染耐药致病菌,面临一定的健康风险[15],因此对海水浴场耐药菌的调查研究尤为重要。本实验对青岛海水浴场中MDRO分离率为22.1%,与De Oliveira等[16]的调查结果相比,要高于其在巴西东南部休闲海域MDRO分离率(6.9%-8.33%),揭示了青岛海水浴场的耐药菌污染较为严重。虽然耐药菌的存在通常与人类活动有关,但本研究中有3株MDRO来自于浴者罕至的距离岸边1公里处的水样中,甚至有关研究在人类活动罕至的南极地区也发现了具有多重耐药性的细菌[17],表明细菌的耐药性可以从海岸扩散到更远的海域。
抗生素的不合理使用是造成耐药菌流行的主要原因,有关研究对我国多地海水养殖场的耐药菌进行了调查,结果显示海水养殖区的耐药菌对氨基糖苷类和磺胺类抗生素表现出了高耐药率[18],对喹诺酮类、氟苯尼考类抗生素的耐药率较低[19],与本研究结果大致吻合,侧面反映出海水浴场细菌的耐药情况或与海水养殖中抗生素的使用有较大关系。本研究分离出的MDRO显示出了对链霉素的高耐药率,Andrade等[20]对圣维森特两个海滩水样及沙样中的大肠杆菌的耐药性进行了研究,也显示出了对链霉素的高耐药率,这可能与链霉素临床应用较早有关。另外,有数据显示海水浴场细菌的耐药性还与粪便污染程度和携带致病菌的浴者数量有关,人类粪便污染指数较高的海水浴场有更高的耐药率[20-22]。
有研究表明细菌的耐药表型与耐药基因的检出率相关[23],本研究对60株MDRO的链霉素耐药基因和四环素耐药基因进行了PCR检测,结果也显示了细菌耐药表型和耐药基因检出率的正相关性。60株MDRO分属于24个菌属,且多为条件致病菌,其中有3株对15种抗生素均耐药的MDRO经鉴定为嗜麦芽窄食单胞菌,该菌主要引起呼吸道感染,是重要的医院感染菌,其分离率在非发酵菌中,仅次于铜绿假单胞菌和鲍曼氏不动杆菌[24];另外,还有2株对15种抗生素均耐药的MDRO经鉴定为缺陷短波单胞菌,该菌虽然致病率低,但致病作用较强,已有报道表明其可引起颅内感染和腹膜炎[25-26],这些细菌的致病率取决于各种因素,例如水中的污染程度、暴露的时间及海滩游客的免疫状况,有研究显示,浴者暴露于海水中15分钟即可带入6×106 CFU的金黄色葡萄球菌[27],因此海水浴场中的耐药致病菌对浴者有较大的健康风险。建议相关部门把浴场的耐药菌污染情况作为浴场水质监测的项目之一,另外,由于海水中的耐药菌主要来自于人类和动物粪便、城镇污水的排放及水产养殖业,对于靠近海滨度假胜地的沿海地区,应加强对城镇污水处理系统及水产养殖业的监督,从源头控制耐药菌的扩散。
4 结论研究结果显示,青岛海水浴场的MDRO有较高的分离率(22.1%),对氨基糖苷类、磺胺类、磷霉素类抗生素耐药性较强,并分离到有5株对15种抗生素均有耐药性的MDRO,表明青岛海水浴场的耐药菌污染较严重。海水浴场的MDRO多样性丰富,60株MDRO属于6个纲,24个菌属,且多为条件致病菌,其中寡养单胞菌属占比最大(16%)。另外,研究表明MDRO对链霉素、四环素的耐药表型与其耐药基因的检出率显著相关。
| [1] |
Jevons MP. "Celbenin"-resistant staphylococci[J]. Br Med J, 1961, 14(4): 385-393. |
| [2] |
许建文, 史道华. 万古霉素耐药肠球菌耐药机制的研究进展[J]. 中国抗生素杂志, 2016, 41(5): 329-334. DOI:10.3969/j.issn.1001-8689.2016.05.002 |
| [3] |
Gerard D. The antibiotic resistome:the nexus of chemical and genetic diversity[J]. Nature Reviews Microbiology, 2007, 5(3): 175-186. DOI:10.1038/nrmicro1614 |
| [4] |
Davies SC, Fowler T, Watson J, et al. Annual Report of the Chief Medical Officer:infection and the rise of antimicrobial resistance[J]. Lancet, 2013, 381(9878): 1606-1609. DOI:10.1016/S0140-6736(13)60604-2 |
| [5] |
Ashbolt NJ, Amezquita A, Backhaus T, et al. Human health risk assessment(HHRA)for environmental development and transfer of antibiotic resistance[J]. Environ Health Perspect, 2013, 121(9): 993-1001. DOI:10.1289/ehp.1206316 |
| [6] |
Manaia CM. Assessing the risk of antibiotic resistance transmission from the environment to humans:non-direct proportionality between abundance and risk[J]. Trends Microbiol, 2017, 25(3): 173-181. DOI:10.1016/j.tim.2016.11.014 |
| [7] |
Chandrasekaran S, Venkatesh B, Lalithakumari D. Transfer and expression of a multiple antibiotic resistance plasmid in marine bacteria[J]. Curr Microbiol, 1998, 37(5): 347-351. DOI:10.1007/s002849900390 |
| [8] |
王玥, 苏洁, 明红霞, 关道明. 大连星海浴场中大肠杆菌耐药性分析[J]. 海洋环境科学, 2016, 35(1): 113-116. |
| [9] |
Leonard AFC, Gaze WH, Singer A, et al. Is it safe to go back in the water? A systematic review and meta-analysis of the risk of acquiring infections from recreational exposure to seawater[J]. International Journal of Epidemiology, 2018, 47(2): 572-586. DOI:10.1093/ije/dyx281 |
| [10] |
石岩, 明红霞, 马玉娟. 用肠球菌评价大连海水浴场人体健康风险[J]. 应用与环境生物学报, 2016, 22(6): 1161-1166. |
| [11] |
Soraas A, Sundsfjord A, Sandven I, et al. Risk factors for community-acquired urinary tract infections caused by ESBL-producing Enterobacteriaceae-a case-control study in a low prevalence country[J]. PLoS One, 2013, 8(7): 1-7. |
| [12] |
CLSI. M100-S24 Performance standards for antimicrobial susceptibility testing. Twentieth-four informational supplement[S]. Wayne: Clinical and Laboratory Standards Institute, 2014.
|
| [13] |
Sunde M, Norström M. The prevalence of, associations between and conjugal transfer of antibiotic resistance genes in Escherichia coli isolated from Norwegian meat and meat products[J]. The Journal of Antimicrobial Chemotherapy, 2006, 58(4): 741-747. DOI:10.1093/jac/dkl294 |
| [14] |
Ye C, Bai X, Zhang J, et al. Spread of Streptococcus suis Sequence Type 7, China[J]. Emerging Infectious Diseases, 2008, 14(5): 787-791. DOI:10.3201/eid1405.070437 |
| [15] |
W HO. Guidelines for safe recreational water environments:volume 1, coastal and fresh waters[M]. Geneva: World Health Organization, 2003.
|
| [16] |
Fernandes Cardoso de Oliveira AJ, Ranzani de França PT, Pinto AB. Antimicrobial resistance of heterotrophic marine bacteria isolated from seawater and sands of recreational beaches with different organic pollution levels in southeastern Brazil:evidences of resistance dissemination[J]. Environ Monit Assess, 2010, 169(1-4): 375-384. DOI:10.1007/s10661-009-1180-6 |
| [17] |
Tam HK, Wong CM, Yong ST, et al. Multiple-antibiotic-resistant bacteria from the maritime Antarctic[J]. Polar Biology, 2015, 38(8): 1129-1141. DOI:10.1007/s00300-015-1671-6 |
| [18] |
写腊月, 胡琳琳, 房文红. 海水养殖源弧菌耐药性调查与分析[J]. 海洋渔业, 2011, 33(4): 442-446. DOI:10.3969/j.issn.1004-2490.2011.04.012 |
| [19] |
李壹, 曲凌云, 朱鹏飞. 山东地区海水养殖区常见抗生素耐药菌及耐药基因分布特征[J]. 海洋环境科学, 2016, 35(1): 55-62. |
| [20] |
Andrade VD, Zampieri B, Ballesteros ER. Densities and antimicrobial resistance of Escherichia coli isolated from marine waters and beach sands[J]. Environ Monit Assess, 2015, 187:342.[J]. Environ Monit Assess, 2015, 187(6):342-342.
|
| [21] |
Abou-Elela GM, El-Sersy NA, Abd-Elnaby H. Distribution and bio-diversity of fecal indicators and potentially harmful pathogens in North Delta(Egypt)[J]. Australian Journal of Basic and Applied Sciences, 2009, 3(4): 3374-3385. |
| [22] |
Efuntoye MO. Study of antibiotic sensitivity pattern and enetrotoxigenicity of Staphylococci isolated from swimming pools in Ibadan, Nigeria[J]. World Applied Science Journal, 2010, 9(11): 1324-1327. |
| [23] |
支威, 马海燕, 仇永凤, 等. 猪源沙门氏菌耐药性及耐药基因的分析[J]. 生物技术通报, 2018, 34(3): 170-176. |
| [24] |
艾效曼, 胡云建, 胡志东. 2012年中国CHINET嗜麦芽窄食单胞菌耐药性监测[J]. 中国感染与化疗杂志, 2014, 14(6): 488-492. DOI:10.3969/j.issn.1009-7708.2014.06.006 |
| [25] |
耿会娟, 刘维生, 伊正君. 缺陷短波单胞菌致颅内感染1例[J]. 中国感染控制杂志, 2013, 12(2): 156-156. DOI:10.3969/j.issn.1671-9638.2013.02.026 |
| [26] |
冯福英, 郑宗富, 王淑萍, 李华. 缺陷短波单胞菌致腹膜炎1例[J]. 中华医院感染学杂志, 2009, 19(9): 1074-1074. DOI:10.3321/j.issn:1005-4529.2009.09.062 |
| [27] |
Elmir SM, Wright ME, Abdelzaher A, et al. Quantitative evaluation of bacteria released by bathers in a marine water[J]. Water Res, 2007, 41(1): 3-10. DOI:10.1016/j.watres.2006.10.005 |