甲基营养菌是一类广泛存在的可利用一碳化合物作为唯一碳源的微生物类群,其模式菌株M. extorquens AM1已成为研究微生物进化和合成代谢的良好材料[1-4],但受该菌遗传背景的影响,目前只有一套基于IncP复制子的遗传质粒操作系统[5-6],因此多元化的标记基因开发是开展遗传操作工作的基础,而高效稳定的遗传标记基因尤为重要。
荧光蛋白基因是一类广泛应用的报告基因,可通过对荧光强度的检测,表征细胞内一个或多个基因的表达水平。1962年Shimomura首次从水母(Aequorea victoria)体内发现并分离到绿色荧光蛋白(Green fluorescent protein,GFP),1999年Matz等[7]从珊瑚中分离到红色荧光蛋白(Red fluorescent protein,RFP),灵敏度和信噪比均比GFP高,但是具有成熟慢、易聚合和对细胞有一定的毒性等特性限制其应用,而后出现的一系列优化突变体尤以mCherry成熟快、单体性好,被广泛应用[8]。目前荧光蛋白按波长可分为深蓝色、蓝色、青色、绿色、黄色、橙色、红色、深红色和近红外系列等40多种,激发/发射波长范围从355/424 nm到684/708 nm[9-10]。绿色荧光蛋白eGFP因其灵敏度高,不需要其他辅助底物就能产生较强的荧光,且对细胞无毒害作用,不影响其他蛋白的表达,稳定性好,已在微生物、植物、动物中获得成功表达。在M. extorquens菌株中已报道表达的荧光蛋白有mCherry和GFP,其中mCherry表达显著,在荧光成像、荧光定位及基因表达强度鉴定方面表现出良好的特征[6, 11],而绿色荧光蛋白的种类多达6种以上[5, 12-15]。以上研究报道所涉及的荧光检测手段和取样测定点都不尽相同,荧光强度表征方式也不一而足,荧光蛋白之间没有详尽的荧光表达分析,因此各荧光基因在M. extorquens AM1中表达是否稳定,以及荧光强度在菌株生长过程中的变化趋势尚不明确。截至目前,其他荧光报告基因的使用在M. extorquens未见报道。
为进一步明确荧光报告基因在甲基营养菌生长过程中的表达变化,筛选最适的荧光报告基因,我们克隆了实验室现有的五种荧光基因egfp、yfp、wgfp、mcherry、rfp,利用甲基营养菌中常用的强启动子PmxaF控制表达[16],转化至M. extorquens AM1后筛选相应的转化子并进行测定和比较分析;借助先进的细胞微孔板成像仪器cytation5[17-18]分别对其荧光成像和荧光表达强度的测定,获得荧光成像展示及荧光表达水平分析;结合SDS-PAGE蛋白凝胶电泳确证荧光基因都能表达出相应的蛋白;通过相对荧光强度和稳定性比较,最终确定最优的荧光蛋白报告基因yfp,为后续实验中荧光标记基因的选择及荧光检测提供借鉴和数据支持。
1 材料与方法 1.1 材料甲基营养菌M. extorquens AM1和大肠杆菌E.coli DH5α均由本实验室保藏。质粒pCM80是可在大肠杆菌和甲基营养菌中复制的质粒,为本实验室保藏。pX458、pSWU-YFP、pSCV1-2、pSWU-mCherry、pSCV1-RFP质粒分别携带egfp、yfp、wgfp、mcherry、rfp荧光基因,来自购买或馈赠。
DH5α感受态细胞用于质粒的转化和扩增,培养与保藏用LB培养基:蛋白胨10 g/L、酵母粉5 g/L、Nacl 10 g/L,37℃培养。M. extorquens AM1菌株的培养采用Hypho培养基,具体配制方法参照文献[19],30℃条件下培养。所需抗生素四环素(tet,终浓度10 μg/mL)。分子克隆相关酶、DNA片段及质粒抽提纯化试剂盒购于Thermo公司。
1.2 方法 1.2.1 多种荧光报告基因差异比对及PCR扩增将本论文所用的五种荧光基因通过Bioedit中的clastalW软件进行DNA序列和编码蛋白比对分析,标记差异位点,设计通用引物对Fluo-F和Fluo-R,分别以质粒pSWU-mCherry、pX458、pSWU-YFP为模板进行PCR扩增mcherry、egfp、yfp的基因序列;设计RFP-F和RFP-R为引物对,以pSCV1-RFP质粒为模板,PCR扩增rfp序列;设计wGFP-F和wGFP-R引物对,以pSCV1-2质粒为模板PCR扩增wgfp序列。相关引物序列信息见表 1。
将PCR扩增得到的五种荧光基因片段用KpnⅠ- EcoRⅠ双酶切纯化回收后,分别与同样酶切的pCM80质粒进行连接,构建含有荧光报告基因的重组载体pCM80-egfp、pCM80-yfp、pCM80-wgfp、pCM80-mcherry和pCM80-rfp,并测序验证。将重组质粒与对照质粒pCM80分别电转化至M. extorquens AM1感受态细胞中,参数设定:1 800 V;1 mm电转杯;2 µL质粒混合50 µL感受态细胞;电击时间5 ms。在含有四环素的固体平板上筛选重组转化子,用于后续的分析鉴定。
1.2.3 重组菌株生长曲线测定将固体平板上的重组菌株单菌落转移到含有3 mL Hypho液体培养基,30℃、200 r/min,培养至指数期(OD600约0.6-0.8),转至含100 mL Hypho液体培养基,使培养液初始OD600=0.01,30℃、200 r/min培养,定时取菌用紫外分光光度计测定OD600值,直至OD600值开始出现下降为止,每个菌株3个生物学重复。
1.2.4 重组子荧光成像观察挑取重组菌株的单菌落接入带有Hypho液体培养基的试管中进行培养,分别取培养到0 h、24 h、48 h、72 h、96 h的菌液200 µL,离心去上清,洗涤2次,用同样体积的无菌水重悬,取20 µL加在烘干的载玻片上并加覆盖玻片,放入玻片适配器中,利用细胞成像微孔板检测系统-Cytation 5(BioTek)进行荧光成像检测,通过Gen5软件设定物镜观察范围,先自动聚焦再手动聚焦,并在[Brightfield(明场)]下找到菌体细胞,进而切换至相应的荧光场[Red:586,647]、[Green:469,525]下微调节焦距进行成像观察,拍照保存并分析。
1.2.5 荧光报告基因的蛋白SDS-PAGE电泳检测将50 mL的重组菌株的液体发酵液培养至OD600=0.8,收集菌体,离心洗涤,重悬于5 mL Tris-HCl(100 mmol/L,pH=8.0),在4℃下,用French press细胞破碎仪38 kPa破碎至澄清,制备粗酶液,离心,分离上清和细胞碎片沉淀(沉淀加适量无菌水重悬),分别取粗酶液、上清和细胞沉淀与上样缓冲液混合,采用SDS-PAGE(5%浓缩胶和10%分离胶)进行电泳检测,用考马斯亮蓝R-250染色观察荧光蛋白的表达。
1.2.6 相对荧光表达强度测定根据生长曲线取点,测定菌株在相应时间和OD下荧光表达强度。每次取200 µL菌液加至96微孔板中,加载至细胞成像微孔板检测系统-Cytation 5(BioTek)中,选择荧光检测,安装荧光滤光片(通道1:EX485/20,EM528/20;通道2:EX530/25,EM590/35),选取96孔板上相应的读取范围,测定荧光强度值,同时测定每孔的OD600值,则相对荧光强度=Δ荧光值/ΔOD600,即为荧光表达水平。测定前先测定900 nm和977 nm的吸收值(Abs900,Abs977),通过公式(Abs977-Abs900)/0.18将光程校正到1 cm对应的OD600的值。
2 结果 2.1 5种荧光蛋白基因的比对分析5种荧光蛋白eGFP、YFP、wGFP、mCherry和RFP,分别获得其基因序列和蛋白质序列,利用Bioedit软件中的ClustalW多序列比对模块分析发现,egfp与yfp的基因序列仅有9个碱基的差异,一致性达98.75%(碱基数比:711/720)(图 1-A);而mcherry的基因序列虽与egfp/yfp存在较大的碱基差异,但在5'端和3'端各有21 bp和25 bp的同源序列(图 1-A)。rfp与mcherry的基因序列一致性为97%(644/661)(图 1-B)。wgfp与egfp的一致性较低,核心序列一致性为77.1%(196/254)。进一步分析相应的荧光蛋白序列发现:YFP、wGFP分别与eGFP蛋白序列一致性为97.9%(234/239)和95.8%(229/239)(图 1-C);RFP作为单分子红色荧光蛋白与mCherry的蛋白序列一致性为90.8%(217/239)(图 1-D),而YFP、eGFP和wGFP分别与mCherry的一致性为28.8%-29.2%,与RFP为25.6%-26.5%,由此可见同色系列的荧光蛋白相似度很高。进一步对荧光蛋白的生色基团进行分析,已知eGFP是在野生型GFP基础上将原有的生色基团Ser65-Tyr66-Gly67(S-Y-G)中的Ser65突变为Thr65,即生色团为T-Y-G,使得荧光强度提高了5倍[20]。在本实验中黄色荧光蛋白YFP相应位点分别为G-Y-G,另一个酵母菌优化可用的wGFP蛋白在此处为S-Y-G,与野生型GFP位点相一致(图 1-C),推测其荧光强度会偏低。

|
| 图 1 荧光蛋白基因序列比对结果 A:egfp、yfp及mcherry基因序列比对;B:rfp与mcherry基因序列比对;C:eGFP、YFP、wGFP的蛋白序列比对;D:RFP与mCherry的蛋白序列比对。不同颜色背景分别表示碱基/氨基酸一致序列;无背景表示差异碱基/氨基酸;星号示共有序列;▲所示yfp与eGFP差异碱基;黑色方框所示为生色基团碱基。 |
将含有荧光基因的重组质粒pCM80-egfp、pCM80-yfp、pCM80-wgfp、pCM80-mcherry和pCM80-rfp,电转化至M. extorquens AM1感受态细胞中,挑选转化子进行荧光成像观察。菌株整体成像结果(图 2)显示,eGFP、YFP、wGFP和mCherry荧光活性较为显著,RFP则几乎看不到荧光。从4 d内的定点取样成像结果来看,0 h在Green和Red通道下相应的背景颜色比较高,不能检测到荧光;24 h的细胞培养物虽然菌落密度较低但已显示出很强的荧光;48-72 h细胞密度增加、荧光强度趋于稳定;96 h荧光强度开始下降,细胞密集,推测由于此时期内(衰退期)细胞裂解所致。含有pCM80质粒的菌株作为阴性对照,在培养后期出现了荧光点,推测与培养基残留及细胞代谢内溶物释放产生的背景干扰有关。

|
| 图 2 重组菌株的荧光成像观察 pCM80-egfp,pCM80-yfp和pCM80-wgfp转化子在绿色荧光场[Green:469,525]以及pCM80-rfp和pCM80-mcherry转化子在红色荧光场[Red:586,647]下分别从在0-96 h的荧光成像观察,pCM80作为阴性对照分别在两个场下进行观察。 |
为进一步探究Cytation5对不同荧光蛋白的辨别度,将含有pCM80-egfp、pCM80-mcherry及pCM80的菌株混合后制片,选择同一视野分别在[Bright Field]、[Red:586,647]、[Green:469,525]通道下成像,通过图像堆叠功能形成细胞成像差异图。结果显示当Red和Green叠加后,能有效检测出含有mCherry和eGFP蛋白的细胞(图 3-A);三场叠加后,对照细胞(黑色)也被显示出来(图 3-B)。三种细胞间没有交集,均能明显分离。

|
| 图 3 pCM80-egfp、pCM80-mcherry和pCM80重组转化菌株的叠加荧光成像 |
将pCM80-egfp、pCM80-mcherry和pCM80重组转化菌株进行液体培养至OD600=0.8,收集菌体高压破碎,取细胞破碎液全液(粗酶液)、破碎液离心后的上清和沉淀分别进行SDS-PAGE检测,结果重组菌株细胞破碎液全液和上清中在相对分子质量约27 kD处可见特异性的蛋白条带,与预期大小相符(图 4)。进一步对所有重组转化菌株细胞破碎液离心后的上清取样检测,均能检测到相应的目的条带(图 5,其中RFP略小为25.4 kD)。表明荧光蛋白在甲基营养菌内都有表达。

|
| 图 4 荧光蛋白电泳检测 a,b,c分别表示细胞破碎粗提物、离心后上清、离心后沉淀;1,2,3分别表示pCM80、pCM80-mcherry、pCM80-egfp转化子;M为蛋白分子量marker;箭头所示为目的蛋白条带 |

|
| 图 5 所有荧光蛋白电泳检测 1:wGFP;2:YFP;3:RFP;4:eGFP;5:mCherry;6:pCM80;M:蛋白分子量marker;箭头所示为目的蛋白条带 |
为鉴定最优的荧光报告基因,测定了重组菌株的生长曲线和荧光基因表达强度值。从生长曲线(图 6-A)来看,含有荧光基因的各转化子与对照pCM80转化子生长基本一致,荧光蛋白的表达未对菌株生长产生抑制。进一步测定各培养时期荧光量总值如图 6-B所示,当OD600=0.1(12 h),荧光总量显著增加,OD600=1.5左右(48 h)荧光总量达到最大,之后趋于稳定;菌株整体荧光量变化趋势与细胞OD600变化相一致,可见荧光蛋白的表达在细胞生长过程中相对比较稳定。荧光蛋白的整体荧光量程度比较为:YFP > eGFP > mCherry > wGFP > RFP。
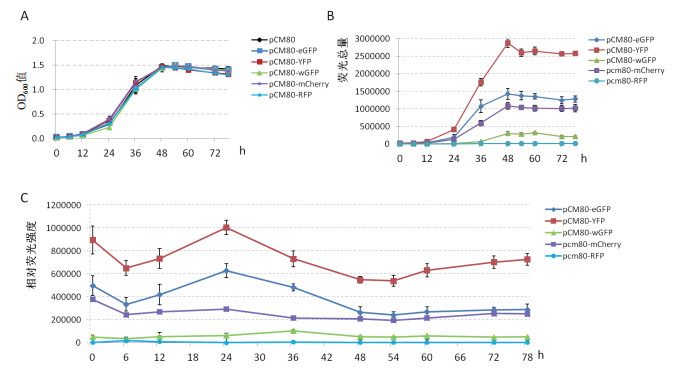
|
| 图 6 菌株生长曲线及荧光强度检测 A:转化子生长曲线;B:转化子荧光总量随时间变化;C:转化子的相对荧光强度随时间变化 |
分析每个荧光蛋白的相对荧光强度曲线(图 6-C)发现,在指数前期(24 h;OD600≈0.4)相对荧光强度最高,指数中期(36 h;OD600≈1.0)之后,相对荧光强度趋于稳定。YFP最大相对荧光强度是eGFP的1.6倍,而wGFP最大相对荧光强度仅为eGFP的0.1倍,mCherry是eGFP的0.47倍,RFP的荧光强度痕量。0-6 h,细胞处于延滞期,此时相对荧光均有下降,可计算出荧光蛋白半衰期分别是eGFP(9.1 h)、YFP(10.9 h)、wGFP(8.5 h)和mCherry(10.2 h),YFP的稳定性最高。
综上,在甲基营养菌中表达强度最明显的荧光蛋白是YFP,其次为eGFP和mCherry,wGFP荧光相对较弱,而RFP在甲基菌仅痕量。
3 讨论甲基营养菌由于受宿主背景和遗传操作工具的限制,需要不断开发和应用具有广宿主特性的遗传和分子模块,而高效稳定可靠的报告基因也是研究的重点,甲基营养菌中虽有利用绿色荧光蛋白和mCherry的研究[5, 6, 11-15],但仅限于用作筛选标记或报告基因指征一个或多个基因的表达水平,缺乏其内荧光蛋白之间相对荧光强度及表达稳定性的比较分析。因此,本论文将5种不同来源的荧光蛋白基因分别在M. extorquens AM1中表达,通过目的蛋白检测、荧光成像展示及荧光动态表达强度测定,指征荧光蛋白的表达水平,进一步分析比较各荧光蛋白性能。在M. extorquens AM1中各荧光蛋白的荧光强度顺序为:YFP > eGFP > mCherry > wGFP。RFP虽与mCherry蛋白序列相似度高,但在同样情况下没有检测到荧光,推测受宿主特性的影响,RFP蛋白在M. extorquens AM1中未能正确折叠或表达后无荧光活性,不适合应用于甲基营养菌。据文献报道,黄色荧光蛋白YFP是最强的荧光蛋白之一,其荧光强度是eGFP的1.5倍,红色荧光中mCherry荧光强度是eGFP的0.43倍[10]。本研究中YFP和mCherry的最大相对荧光强度分别是eGFP的1.6倍和0.47倍,与文献报道一致,可见三者具有较为广泛的宿主表达特性及稳定性。相比于常用的eGFP和mCherry,M. extorquens AM1中YFP表现出更好的荧光特性。
进一步通过荧光总量、相对荧光强度曲线和细胞生长曲线的分析发现:荧光总量与细胞的OD值变化相一致,最大荧光量出现在稳定期之前,之后保持稳定,表明荧光蛋白在M. extorquens AM1中稳定性强;相对荧光强度最大值出现在指数期前期,指数后期之后趋于稳定,表明相对荧光强度在细胞培养过程中并非一成不变而是处于动态变化之中,这是由细胞生长状态及荧光蛋白成熟后荧光衰减共同导致,根据此曲线可依不同的研究目的在细胞生长相应时期进行取点检测荧光。各荧光蛋白的半衰期在8.5-10.9 h之间,其中YFP半衰期最长,与eGFP相比不论是荧光强度还是稳定性都要强。mCherry比eGFP的半衰期略长,荧光强度低,但相比于RFP,是红色荧光蛋白的良选。
4 结论在甲基营养菌中成功表达了5种不同的荧光蛋白基因,并对其蛋白表达、荧光成像及荧光强度进行了检测、鉴定和细致分析,最终确定黄色荧光蛋白YFP的荧光稳定性及相对荧光强度都是最优的。
| [1] |
Chistoserdova L, Vorholt JA, Thauer RK, et al. C1 transfer enzymes and coenzymes linking methylotrophic bacteria and methanogenic Archaea[J]. Science, 1998, 281: 99-102. DOI:10.1126/science.281.5373.99 |
| [2] |
Lee MC, Chou HH, Marx CJ. Asymmetric, bimodal trade-offs during adaptation of Methylobacterium to distinct growth substrates[J]. Evolution, 2009, 63: 2816-2830. DOI:10.1111/evo.2009.63.issue-11 |
| [3] |
Chou HH, Chiu HC, Delaney NF, et al. Diminishing returns epistasis among beneficial mutations decelerates adaptation[J]. Science, 2011, 332: 1190-1192. DOI:10.1126/science.1203799 |
| [4] |
Vu HN, Subuyuj GA, Vijayakumar S, et al. Lanthanide-Dependent Regulation of Methanol Oxidation Systems in Methylobacterium extorquens AM1 and Their Contribution to Methanol Growth[J]. J Bacteriol, 2016, 198: 1250-1259. DOI:10.1128/JB.00937-15 |
| [5] |
Marx CJ, Lidstrom ME. Development of improved versatile broad-host-range vectors for use in methylotrophs and other Gram-negative bacteria[J]. Microbiology, 2001, 147: 2065-2075. DOI:10.1099/00221287-147-8-2065 |
| [6] |
Chubiz LM, Purswani J, Carroll SM, et al. A novel pair of inducible expression vectors for use in Methylobacterium extorquens[J]. BMC Res Notes, 2013, 6: 183. DOI:10.1186/1756-0500-6-183 |
| [7] |
Matz MV, et al. Fluorescent proteins from nonbiolumi-nescent Anthozoa species[J]. Nat Biotechnol, 1999, 17: 969-973. DOI:10.1038/13657 |
| [8] |
Shaner NC, Campbell RE, et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein[J]. Nat Biotechnol, 2004, 22: 1567-1572. DOI:10.1038/nbt1037 |
| [9] |
Shu X, Royant A, Lin MZ, et al. Mammalian expression of infrared fluorescent proteins engineered from a bacterial phytochrome[J]. Science, 2009, 324: 804-807. DOI:10.1126/science.1168683 |
| [10] |
Day RN, Davidson MW. The fluorescent protein palette: tools for cellular imaging[J]. Chem Soc Rev, 2009, 38: 2887-2921. DOI:10.1039/b901966a |
| [11] |
Michener JK, et al. Phylogeny poorly predicts the utility of a challenging horizontally transferred gene in Methylobacterium strains[J]. J Bacteriol, 2014, 196: 2101-2107. DOI:10.1128/JB.00034-14 |
| [12] |
Figueira MM, Laramee L, et al. Production of green fluorescent protein by the methylotrophic bacterium Methylobacterium extorquens[J]. FEMS Microbiol Lett, 2000, 193: 195-200. DOI:10.1111/fml.2000.193.issue-2 |
| [13] |
Marx CJ, Lidstrom ME. Development of an insertional expression vector system for Methylobacterium extorquens AM1 and generation of null mutants lacking mtdA and/or fch[J]. Microbiology, 2004, 150: 9-19. DOI:10.1099/mic.0.26587-0 |
| [14] |
Sy A, Timmers AC, Knief C, et al. Methylotrophic metabolism is advantageous for Methylobacterium extorquens during colonization of Medicago truncatula under competitive conditions[J]. Appl Environ Microbiol, 2005, 71: 7245-7252. DOI:10.1128/AEM.71.11.7245-7252.2005 |
| [15] |
Liang WF, Sun MY, Cui LY, et al. Cre/loxP-Mediated Multicopy Integration of the Mevalonate Operon into the Genome of Methylobacterium extorquens AM1[J]. Appl Biochem Biotechnol, 2018, 185: 565-577. DOI:10.1007/s12010-017-2673-3 |
| [16] |
Choi YJ, Morel L, Bourque D, et al. Bestowing inducibility on the cloned methanol dehydrogenase promoter(PmxaF)of Methylobacterium extorquens by applying regulatory elements of Pseudomonas putida F1[J]. Appl Environ Microbiol, 2006, 72: 7723-7729. DOI:10.1128/AEM.02002-06 |
| [17] |
Guiziou S, Sauveplane V, Chang HJ, et al. A part toolbox to tune genetic expression in Bacillus subtilis[J]. Nucleic Acids Res, 2016, 44: 7495-7508. |
| [18] |
Singh A, Azad M, Shymko MD, et al. The BH3 only Bcl-2 family member BNIP3 regulates cellular proliferation[J]. PLoS One, 2018, 13: e0204792. DOI:10.1371/journal.pone.0204792 |
| [19] |
Yang YM, Chen WJ, Yang J, et al. Production of 3-hydroxypropionic acid in engineered Methylobacterium extorquens AM1 and its reassimilation through a reductive route[J]. Microb Cell Fact, 2017, 16: 179. DOI:10.1186/s12934-017-0798-2 |
| [20] |
Heim R, Cubitt AB, Tsien RY. Improved green fluorescence[J]. Nat, 1995, 373: 663-664. |




