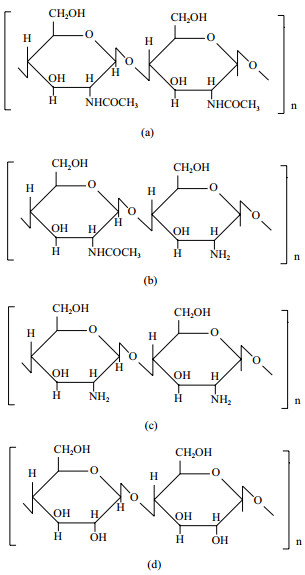
甲壳素(Chitin)是地球上数量仅次于纤维素的第二大天然高分子多糖,广泛存在于节肢动物、软体动物、环节动物、原生动物、腔肠动物及真菌和藻类的细胞壁中。地球上每年甲壳素的生物合成量约为100亿t[1]。1811年法国科学家Braconnot[2]首次发现甲壳素,纯态甲壳素为β-(1,4)-2-乙酰氨基-2-脱氧-D-吡喃葡聚糖,由二糖重复单元通过β-(1,4)-D-糖苷键连接成为线型高聚物(图 1-a),其化学结构和植物纤维素的结构非常相似;天然甲壳素因分子链中部分乙酰基被脱除,因此可视为2-乙酰氨葡萄糖和2-氨基葡萄糖的共聚物(图 1-b);进一步脱去结构中的乙酰基,就成为其最为重要的脱乙酰化衍生物壳聚糖(图 1-c)。
|
| 图 1 纯态甲壳素(a)、天然甲壳素(b)、壳聚糖(c)和纤维素分子结构(d) |
甲壳素/壳聚糖因具有良好的生物可降解性、生物相容性、无毒性和抑菌性,可用于纺织印染、食品医疗、化工环保等高附加值领域[3-5]。例如,由壳聚糖制成的人造皮肤,因其良好的透气性、渗出性、可抑菌止痛止痒等优点,并能促进皮肤细胞的生长从而加快伤口愈合[6],在临床医学上有着广泛的应用[7-8];利用壳聚糖设计生产纳米胶囊以实现微胶囊技术的应用,在药物输送、生物制剂以及疫苗生产等方面都有潜在的应用[9]。此外,由于甲壳素/壳聚糖分子中存在大量的活性官能团(氨基、羟基),有利于进一步对官能团进行修饰得到更加功能化的化合物,得到的衍生物在生物医学、药学、食品等领域也有着广泛的应用[10],壳聚糖衍生物作为疫苗佐剂可以增强机体对病原微生物的免疫应答[11]。
随着化石资源的日益减少,石油基合成高分子的降解性给生态环境造成的污染日益严重,开发有潜力的再生资源成为世界各国的研究热点,生产优良品质的甲壳素/壳聚糖成为开发应用这一材料重要的前提条件,为此本文对现有的甲壳素/壳聚糖的生物制备方法进行总结评述。
1 甲壳素/壳聚糖的来源甲壳素来源于节肢动物和低等植物的真菌和藻类,前者大量存在于海洋之中,以虾、蟹产量最大,且易于富集,甲壳素含量也高,干重含量在14%-27%之间,极具提取利用价值。目前,工业上生产甲壳素的主要原料便源于此[12]。
陆地上可以用于甲壳素原料的节肢动物有蚕蛹、家蝇的幼虫蛹壳和各类昆虫等。其中蚕蛹较为集中,其他则较为分散,收集困难,给工业化生产的实现带来一定的困难。但也有研究在无菌条件下繁殖家蝇,以蛆虫为原料提取甲壳素[13-14]。以此种资源提甲壳素,虽然比例小,但原料集中,便于工业化生产,有着良好的发展前景。在自然界中,除少数真菌外,其他真菌均含有甲壳素和壳聚糖。甲壳素与壳聚糖是真菌细胞壁的主要组成成分,例如,在子囊菌纲、担子菌纲等真菌细胞壁中,甲壳素的含量占菌体干重的2%-42%。
2 甲壳素制备在甲壳类动物中,甲壳素与蛋白质,以及附着于蛋白质上成壳的碳酸钙组成复杂的网状体系,甲壳素与蛋白之间的相互作用力较强,并且部分的蛋白质还参与了多糖-蛋白复合物的形成;此外,在原料中还存在些许脂肪和以虾青素为主的类胡萝卜素及其形成的酯。因此,生产甲壳素最主要的步骤是蛋白质和CaCO3的脱除,同时,脂肪和色素在以上脱除过程中一并去除[15]。有些情况下,需要添加脱色步骤用于脱除色素以达到甲壳素的生产要求。
传统生产甲壳素的方式是利用虾蟹壳作为原料的酸碱法,可归纳为“三脱”。一是脱除蛋白质,也是最为困难的一步,因为甲壳素和蛋白之间存在化学键的作用,脱蛋白试剂有NaOH、Na2CO3、NaHCO3、KOH、K2CO3、Ca(OH)2、Na2SO3、NaHSO3、Na3PO4和Na2S等,蛋白的脱除程度要具体对待,过度的脱蛋白会造成甲壳素的脱乙酰化和水解,相反,由于部分人群对甲壳类动物存在过敏的症状,所以蛋白存留会影响甲壳素在医药方面的应用;二是脱除无机盐,主要为CaCO3。脱盐的主要方法为酸处理,脱盐试剂有HCl、HNO3、H2SO4、CH3COOH、HCOOH等,最常用的是HCl。由于CaCO3易酸解,所以脱盐过程相对容易完成。一般来讲,提高温度有利于酸对甲壳素基质的浸透,提高脱盐效率[16],但同样会带来甲壳素的脱乙酰化和水解;三是脱除色素,目前,工业上甲壳素脱色主要通过日光照射和KMnO4氧化脱色的办法。日光照射脱色时间长,而且产物偏黄色。KMnO4脱色后需要加NaHSO3等还原剂进行还原处理。另外,未反应的KMnO4还会给环境造成压力。双氧水是工业上常用的氧化脱色剂,价格低廉且分解产物无污染,近年来双氧水脱色的研究引起了人们的关注[17-18]。
传统的酸碱法提取甲壳素存在很多缺陷:(1)使用了大量的酸碱,消耗能源的同时,环境污染严重;(2)甲壳素后续纯化过程复杂,增加了生产成本;(3)造成甲壳素分子量及乙酰化程度的降低,从而影响甲壳素产品品质。虽然,科研工作者在此基础上做了工艺上的改进。例如,用相对温和的EDTA、柠檬酸代替盐酸用于CaCO3等无机盐的脱除[19-20],但是始终无法从根本上解决这些问题。随着生物技术的发展,为了解决酸碱法带来的问题,不同的生物处理技术被研究用于甲壳素的提取,如蛋白酶酶解、微生物发酵等[21-22],另外,生物法与化学法相结合的处理手段也成为了研究的一个方向[23]。
2.1 酶法脱蛋白随着生物技术的发展,研究发现许多蛋白酶,如碱性蛋白酶、胃蛋白酶、木瓜蛋白酶、胰蛋白酶等,其中以碱性蛋白酶居多,有利于从甲壳动物外壳中将蛋白质分离出来,可以用于甲壳素的提取,并且该方法具有条件温和、环境友好、产品纯度高等优点[24-26],其作用原理是甲壳动物外壳中存在的蛋白质在蛋白酶的催化作用下与外壳分离,并且使分离过程中的去乙酰化作用和解聚作用最小化。Antunes-Valcareggi等[27]发现青蟹废料中添加3%(E/S)碱性蛋白酶ProteMax 750 L,在53℃,pH9条件下,作用120 min可以去除30%的蛋白质。
Younes等[28]对比了分别来源于Bacillus mojavensis A21、Bacillus subtilis A26、Bacillus. licheniformis NH1、Bacillus. licheniformis MP1、Vibrio metschnikovii J1及Aspergillus clavatus ES1所产的碱性蛋白酶对于虾壳蛋白的脱蛋白效率,研究发现相同条件下,其中来源于Bacillus mojavensis A21的蛋白酶脱蛋白效果最好,其蛋白质脱除率约为76%,而来源于Bacillus. licheniformis NH1的蛋白酶的蛋白脱除率为65%,Aspergillus clavatus ES1所产的蛋白酶的脱蛋白效率只有59%,可以看出虽然同属于碱性蛋白酶,但来源不同的酶对同一种生物资源底物蛋白的脱蛋白效果依旧有明显的差别。该研究进一步考察了脱盐后酶法脱蛋白的条件,并利用响应面法优化了酶、底物的添加比例、反应温度和反应时间等反应条件,源自Bacillus mojavensis A21的蛋白酶蛋白质脱除率可进一步提高到88%。另外,研究发现,同一种酶对于不同的生物资源的蛋白脱除率不尽相同,Hamdi等[29]从梭子蟹中提取了消化碱性蛋白粗酶,经分析,该粗酶中含有至少五种不同的蛋白酶。在50℃,5 U/mg添加量下,对虾壳作用3 h后,对蓝蟹壳的蛋白脱除率为84.69%±0.65%,但对虾壳中蛋白脱除率达到了91.06%±1.40%。如产自于Pseudomonas aeruginosa K-187的蛋白酶对天然虾壳的蛋白脱除率可到78%,对虾蟹壳粉末的蛋白脱除率为72%,而对于酸处理过的虾蟹壳粉末的蛋白脱除率只有45%[30]。上述结果说明相同的蛋白酶对于不同的底物的脱蛋白效果不尽相同,同一底物经不同的预处理过程后对蛋白酶的酶解作用也有明显的影响,脱蛋白酶作为一种酶催化剂的一种,对底物具有高度的选择性和特异性。
Carla等[31]对中试规模的化学法和酶法生产甲壳素的可持续性进行了分析和评估,通过在三个可持续维度的评估和整合以获得相对一致的比较基线,研究表明,由于酶法具有更加广阔的研究领域,上升空间巨大,未来可以获得显著的优势。酶法是可以替代化学法来完成甲壳素的提取,尤其是在考虑到水回收利用的时候,这一优势更加明显。
甲壳素生产过程中,酶催化作为新的蛋白质脱除方法,具有很大的潜力。相对于化学法而言,(1)酶法脱蛋白质的效率相对低,约5%-10%残留蛋白质与甲壳素结合在一起,还需要额外的添加NaOH,并在相对温和的条件下进行处理来提高甲壳素的纯度及结构;(2)在化学提取甲壳素的过程中,脱盐和脱蛋白质进行的先后顺序对甲壳素的最终产量和质量无明显的影响,但是,在酶法提取甲壳素时,矿物质的存在降低了蛋白酶的可及性,从而影响了虾壳的脱蛋白效率,所以脱盐的步骤应该在酶法脱蛋白之前。
2.2 微生物发酵制备甲壳素酶法脱蛋白提取甲壳素工艺虽然降低了碱所带来的环境污染,减少了能耗,但是存在耗时长、效率低、脱蛋白不充分等缺点,更重要的是所需酶制剂生产及使用问题亟待解决,这些都导致酶法提取甲壳素工艺成本较高,采用微生物发酵的提取工艺可以在一定程度上解决这一问题。微生物发酵工艺即以虾蟹壳的废料为原料,利用微生物在生长过程中所产的有机酸将虾、蟹等原料中的矿物质溶解起到脱盐的作用,所产“酵素”将蛋白质水解达到除蛋白的目的。微生物菌种可以来源于虾蟹壳等原料,也可以通过外加。虾蟹壳废料中的菌种为原始自带菌种,活力较差,调控较为复杂,发酵效果相对较差,因此,微生物发酵一般采用外加菌种的方式进行。Arbia等[32]对现有报道用于甲壳类废料发酵的微生物种类进行了总结,包括乳酸杆菌属(Lactobacillus)、芽孢杆菌属(Bacillus)、假单胞菌属(Pseudomonas)、片球菌属(Pediococcus)、沙雷菌属(Serratia)、黑曲霉(Aspergillus niger)等。根据微生物发酵过程中的作用方式不同,可以分为发酵脱除盐[33]、发酵脱除蛋白质[34]及发酵脱除盐和蛋白质[35]3种作用方式。此外,脱蛋白/盐的效率与发酵原料、初始pH、发酵类型和发酵时间等条件有关[36]。
Choorit等[37]利用响应面法优化了Pediococcus sp L1/2发酵虾头脱盐条件,结果表明,在37℃,糖浓度50 g/L,初始pH7.0,发酵36 h后脱盐率最高达到83.47%。Ghorbel-Bellaaj等[38]评价了6种蛋白水解芽孢杆菌对虾壳发酵脱蛋白的效果,6种芽孢杆菌分别为Bacillus pumilus A1、Bacillus mojavensis A21、Bacillus licheniformis RP1、Bacillus cereus SV1、Bacillus amyloliquefaciens An6及Bacillus subtilis A26。结果显示,6种芽孢杆菌均可以有效地脱除虾壳中的蛋白质,脱蛋白率在80%以上,但是只有不到67%的矿物被脱除;Zhang等[39]以虾壳粉为原料,利用连续三步微生物发酵技术制备甲壳素(所用菌种为Serratia marcescens B742,Lactobacillus plantarum ATCC 8014和Rhizopus japonicus M193),甲壳素产率为21.35%,回收率达74.67%。Neves等[33]发现Lactobacilus plantarum发酵对淡水虾壳脱蛋白质、脱盐的效果更佳,在其最佳发酵条件下发酵72 h后,蛋白质脱除率达到99%,脱盐率达到87%,有望替代传统的化学法提取甲壳素。
为了进一步提高发酵提取甲壳素的效率,充分发挥菌种的不同作用,实现最大限度的脱除蛋白质及钙盐,共培养和分步连续培养的发酵方式被引入发酵提取甲壳素提取工艺中[40-41]。Duan等[42]考察了Streptococcus thermophiles、Lactobacillus acidophilus和Lactobacillus bulgaricus共培养体系对虾壳中甲壳素的提取,8 h内发酵体系的pH降低到4.25,pH最低可达3.22,相对乳酸含量为0.25 mol/L,此酸性环境促进了钙和蛋白的脱除,发酵168 h,脱盐率和脱蛋白率分别为91.3%和97.7%,同时色素脱除率也达到了32.3%。Liu等[43]研究了产酶菌种Bacillus licheniformis 21886和产酸菌种Gluconobacter oxydans DSM-2003不同培养方式下用于虾壳废料中甲壳素的提取。发酵方式分别为:(1)21886菌种和2003菌种同时共培养发酵96 h,添加5%葡萄糖;(2)21886菌种先培养发酵60 h,之后接种2003菌种,同时添加5%葡萄糖,继续共培养发酵36 h;(3)2003菌种先培养发酵36 h,同时添加5%葡萄糖,之后接种21886菌种,继续共培养60 h。研究发现,第二种培养方式对甲壳素的提取效率最佳,在共发酵中,以乳酸、甲/乙酸为主的8种有机酸的含量达到了16 g/L,是Gluconobacter oxydans DSM-2003单独发酵培养产酸量的3倍多,发酵96 h后,脱盐率达到了87%,脱蛋白率达到了93.5%,发酵体系中甲壳素的含量达到了90.8%。同时,也有专利公开发酵生产甲壳素的方法,首先利用枯草芽孢杆菌和氧化葡萄糖杆菌协同作用加以葡萄糖来发酵虾皮和虾头进行脱盐和脱蛋白质,然后利用嗜热链球菌、嗜酸乳杆菌、保加利亚乳杆菌共生体系发酵一周进行脱钙和脱色,得到相应的甲壳素[44]。
微生物发酵法制备甲壳素具有条件温和、能耗低、环境友好等优点,但是现阶段的研究基本都停留在实验室阶段,距离最后的工业化生产阶段还有很长的距离。目前,菌种筛选、优化发酵条件、组合不同发酵方式等方法虽然已经用于甲壳素的提取,但其纯度依旧有限,符合工业级要求的较少。微生物发酵法依旧存在产酸和蛋白酶不足的问题,导致脱盐和脱蛋白质不够充分,并且产酸的速度相对较慢,常导致发酵过程中原料腐败。因此,筛选高产酸、产酶菌株,通过基因工程改造菌株,提高酸和蛋白酶的产量和产率是微生物发酵提取甲壳素的重点研发方向。
3 壳聚糖制备甲壳素溶解性能较差导致其应用受到限制,在脱乙酰作用下,当乙酰化程度(摩尔分数)低于50%时,即成为其最重要的脱乙酰衍生物壳聚糖,一般认为是甲壳素不同程度脱乙酰后的一类聚合物。与甲壳素相比,壳聚糖的溶解性能得到改善,分子间作用力相对较弱,糖基环中除含有活性羟基外,还存在游离氨基和羟基,化学修饰更加多样化[11, 45]。因此,壳聚糖的制备成为完善甲壳素衍生化学体系的重要前提。现阶段,甲壳素通过化学法或生物法均能够制备壳聚糖,在工业上,由于化学法的低投入以及稳定的规模化生产而被广泛采用。
从化学的角度看,酸或者碱都可以用于甲壳素的脱乙酰化,由于糖苷键对酸较为敏感,在酸条件下易断裂,通常脱乙酰化在碱性条件下进行。甲壳素在浓NaOH热溶液处理几个小时后,脱乙酰率在85%-99%之间[46],但这会造成N-乙酰-D-氨基葡萄糖和D-氨基葡萄糖不规则的分布于聚合物的主链中,导致壳聚糖在水溶液中更容易聚集而影响其溶解性,进一步影响壳聚糖的物理化学性质。例如,平均分子量、在水中的黏度等[47],同时也存在能耗大、浓碱废液和产物降解等一系列问题。生物法因其温和的作用条件,环境友好等优势而逐渐应用于壳聚糖的制备生产中。
3.1 酶法脱乙酰甲壳素脱乙酰酶(Chitin deacetylase,CDA,EC 3.5.1.41)能够催化甲壳素中N-乙酰胺基水解生成壳聚糖,该活性酶存在于多种真菌及昆虫物种,其中研究较多的脱乙酰酶分别提取于Mucor circinelloides[48]、Aspergillus nidulans[49]及Colletotrichum lindemuthianum[50]。上述CDA通常存在于胞质区域或分泌于菌种培养基中,并且脱乙酰酶具有良好的热稳定性(最优作用温度为50-60℃),并且对β-(1,4)-N-乙酰-D-葡萄糖胺聚合物具有很强的专一性。
研究表明,CDA属于糖脂酶家族4(Carbohydrate esterase family4,CE-4),其结构特性有很多相似性,来源于Colletotrachum landemuthaanum的CDA是由一个结构域组成的单核金属酶。其酶水解的作用机理也与CE-4成员类似,是一种多点进攻模式,即可以同时脱去多个位点的乙酰基[51]。另外,研究发现,相比较于晶态甲壳素,CDA更容易作用于无定型甲壳素,这是因为,CDA是通过接近甲壳素分子进而脱除乙酰基完成其作用的,当甲壳素结晶成为纤维状后,CDA便很难在进入内部,这就造成了乙酰基脱除的困难[52]。
甲壳素脱乙酰酶常被用于壳聚糖生产[53],代替现有的浓碱热解法以生产高品质的壳聚糖,与化学过程相比,酶法脱乙酰过程更为可控,从而更好地生产新型、具有明确定义的壳聚糖产物,同时该方法也可专门用于壳聚糖低聚物的生产,这些都是化学法无法实现的。值得注意的是,与其他种类酶的特性不同,由Colletotrichum lindemuthianum生产的甲壳素脱乙酰酶不存在产物(乙酸盐)抑制作用,因此,更具有潜在的生物技术应用前景[54-55]。
在壳聚糖制备过程中,无论是晶型的还是非晶型的甲壳素都很难溶解,非均相体系降低了脱乙酰酶和底物的可及性,从而导致脱乙酰酶作用效果较差,脱乙酰率较低(< 10%),这一点与纤维素的酶解类似。因此,在添加脱乙酰酶前对底物预处理可以有效地增加脱乙酰酶对底物的可及性,从而提高脱乙酰的效率。有研究验证了这一观点,窦勇等[56]以淡水小龙虾壳为原料,经EDTA法提取甲壳素,采用超声辅助CDA酶法制备壳聚糖,结果表明,经过超声功率476 W,时间60 min的预处理后,同样在酶添加量9.45%,酶解温度50℃,酶解时间3.5 h条件下,壳聚糖脱乙酰度高达91.09%,相对得率81.87%。此外,离子液体的使用可以使脱乙酰作用在均相体系中完成,研究发现,[Amim][Cl]可以使甲壳素脱乙酰环境趋于均相,与未添加离子液体的酶解体系相比较,脱乙酰酶的活性提高了39%[57],为壳聚糖的制备提供了新的思路。
现阶段,由于甲壳素脱乙酰酶的来源较窄,大部分来源于真菌,产酶菌株活力不高,分离提取不成熟,另外,酶解催化机理研究较少[58],这些都制约了酶法脱乙酰的发展,使其局限于实验室研究。
3.2 微生物发酵制备壳聚糖以上介绍的壳聚糖主要是从虾蟹壳中提取,然而由于虾蟹壳不易收集、且其质量无法保证稳定;虾蟹壳不易储备,时间长了容易发生腐败。近年来在发酵技术方面的研究进展表明,这些问题都可以通过培养生产壳聚糖的真菌来克服,尤其是接合真菌,这类菌含有1%-10%细胞干重的壳聚糖。相比较从虾、蟹壳中提取壳聚糖,微生物发酵法生产壳聚糖有以下优点:(1)作为原料的真菌大部分可以大规模培养,并且不受地域和季节的限制;(2)可以通过控制培养时间从而获得不同分子量的壳聚糖;(3)聚合度均匀,脱乙酰化程度高;(4)工艺简单,能耗低并且环境友好;(5)利用工业生产中产生的富含壳聚糖的废弃菌丝发酵生产壳聚糖,变废为宝的同时还可以较少环境污染。另外,由真菌发酵生产的壳聚糖具有较高的纯度和品质,适用于食品、医药、化妆品等行业。
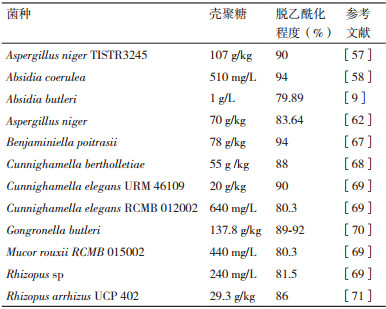
近些年来,利用菌种发酵生产壳聚糖的研究得到了越来越多的关注,如黑曲霉、酵母、甘格氏菌、毛霉菌和根霉菌等[59-60]。表 1中汇总了常见的接合真菌生产壳聚糖的产量和脱乙酰化程度。研究人员对几种不同真菌的生产潜力进行了评估。Pochanavanich等[61]考察了真菌Aspergillus niger、Rhizopus oryzae、Lentinus edodes、Pleurotus sajocaju、Zygosaccharomyces rouxii TISTR5058和Candida albicans TISTR5239在复合培养基中生产壳聚糖的能力。从上述菌株中分离出来的壳聚糖产量为10-140 mg/g细胞干重,脱乙酰度为84%-90%,分子量2.7×104-1.9×102 kD,黏度是3.1-6.2 cP。Rane等[62]研究了菌种Absidia. Coerulea、Absidia blakesleeana、G. butleri、Mucor rouxii和Phvcomyces blakesleeanus在简单的酵母提取物-蛋白胨-葡萄糖培养基中壳聚糖的生产,结果表明Absidia. coerulea壳聚糖最大产量为510 mg/L(培养48 h),脱乙酰度为94%,当介质中添加钴元素后,壳聚糖产率可增加20%。Muzzarelli等[63]进一步优化了培养条件,Absidia. coerulea培养72 h生物质产量为6-7 g干重/L,其中壳聚糖约占30%即1.8 g/L,脱乙酰率为95%,平均分子量为500 kD。Kim等[64]进一步报道Absidia. coerulea在pH 4.5,搅拌速度250 r/min和曝气率2 vvm等优化条件下可连续生产壳聚糖,产率可到52 mg/L/h。Tasara等[66]系统研究了采集于土耳其埃尔祖鲁姆地区的菌种Rhizopus oryzae PAS 17(基因库登录号KU318422.1)在非无菌条件下发酵生产壳聚糖,利用其可以在较低温度和pH条件下生长的特性以抑制其他微生物的污染。通过正交实验优化了对壳聚糖生产影响最明显的五个因素,分别是碳源(糖蜜)、金属离子(Mg2+)、接种量、转速和培养时间,得到最优的培养条件:糖蜜70 mL/L,MgSO4·7H2O 0.5 g/L,接种量6.7×106孢子/瓶,搅拌速度150 r/min,培养时间8 d。结果显示,壳聚糖和生物量产量分别是未优化实验的14.45和8.58倍。
利用发酵法得到的环保型壳聚糖可以作为抗菌剂应用于众多领域。Gharieb等[72]研究了3种真菌(Cunninghamella elegans RCMB 012002,Mucor rouxii RCMB 015002和Rhizopus.sp)生产的壳聚糖,以及壳聚糖对抗不同致病微生物的抗菌活性。通过实验发现Mucor rouxii,Rhizopus sp和Cunninghamella elegans生产的壳聚糖其脱乙酰度分别为80.3%、81.5%和80.3%。为进一步提高真菌的壳聚糖产量,优化了培养基、碳源、氮源、pH值及温度等培养条件,结果表明,以上3种真菌在YPG培养条件下壳聚糖产量分别提高到440、240和640 mg/L。通过对比表明以葡萄糖为碳源可以获得较高的壳聚糖产率,最适发酵温度为30℃,其中Mucor rouxii和Cunninghamella elegans最好的氮源为硝酸钠,最适pH为5;Rhizopus sp.最好的氮源为尿酸,最适pH为4.5。利用上述3种真菌生产的壳聚糖作为抗菌剂对抗致病微生物,如金黄色葡萄球菌(ATCC-47077)、大肠杆菌(ATCC-25922)、白色念球菌(ATCC-0231)和尖孢镰刀菌,结果表明以上3种真菌都有不同的抗菌活性,计算得到的最低抑菌浓度(Minimum inhibitory concentration,MIC)在100-1 500 ppm之间。
通过更换廉价的发酵碳源可以进一步降低发酵成本,Yang等[73]利用稀酸蒸爆预处理后富含木糖的玉米秸秆为碳源,研究了Rhizopus oryzae AS 3.819发酵生产壳聚糖的情况,玉米秸秆预处理料中含30 g/L木糖和10 g/L葡萄糖,发酵72 h后,壳聚糖产量为90 g/kg,壳聚糖的脱乙酰度(91.27%)高于利用天然贝类生产的商业化壳聚糖(87.25%),壳聚糖黏度(2.67 mPa·s)比商业化的壳聚糖黏度(22.25 mPa·s)有了较大程度的降低。
目前壳聚糖主要还是以虾蟹壳为原料生产,由于其生产成本高、聚合度不均匀、质量不稳定以及环境不友好等原因,导致其产品品质不高。微生物发酵法虽然也存在成本问题,但在一定程度上解决环境污染问题,若利用废弃菌丝变废为宝的同时还可以制得高品质的壳聚糖,从而满足在食品、医药及化妆等领域中的应用。
4 结语甲壳素/壳聚糖作为一种“动物性”纤维素,在纺织印染、造纸、农业、食品、化妆品及医疗卫生等领域有着越来越广泛的应用,逐渐成为了继木质纤维素后另一种可再生资源新体系。甲壳素/壳聚糖的提取制备工艺很多,酸碱法为现行的工业化甲壳素提取工艺,但是提取工程中使用大量的酸碱,消耗能源的同时还会造成环境污染,后续污染物处理耗费高,已经成为甲壳素衍生化学未来发展的瓶颈。酶法及微生物发酵法是节能、环境友好型的生产技术,可以生产优质的甲壳素和壳聚糖,但如何解决成本高且提取效率低的问题也成为了制约生物法工业化的关键所在,如酶催化发制备甲壳素存在蛋白残留率高,微生物发酵法制备甲壳素存在微生物产酸产酶速度慢,脱盐脱蛋白不够充分,酶法制备壳聚糖过程中产CDA菌株活力不高,提取技术不成熟等问题,这些都限制了生物法工业化的进程。鉴于目前存在的诸多问题,可以从以下几个方面进行改善:其一,通过诱变筛选产酶、产酸多的优势菌种,优化培养基和发酵参数,提高发酵效率;其二,开发新的提取工艺,如生物/化学法有机结合,提高产品纯度,扩大生产规模,以达到工业化的要求;其三,可以利用废弃的菌丝如青霉素、柠檬酸发酵的废菌丝体等,解决环境问题的同时获得高附加值的壳聚糖。另外,我国甲壳素/壳聚糖生产处于整个生产链的前端,存在产品种类相对单一,技术含量低,甲壳素/壳聚糖深加工及应用远远不足等问题,因此,今后应该在保证绿色环保生产甲壳素/壳聚糖的同时,进一步加大对甲壳素衍生物的研发力度,增加高附加值产品的产出,实现其在更多领域的应用。
| [1] |
程倩, 吴薇, 籍保平. 微生物发酵法提取甲壳素的国内外研究进展[J]. 食品科技, 2012, 37(3): 40-43. |
| [2] |
Muzzarelli RAA, Boudrant J, Meyer D, et al. Current views on fungal chitin/chitosan, human chitinases, food preservation, glucans, pectins and inulin:A tribute to Henri Braconnot, precursor of the carbohydrate polymers science, on the chitin bicentennial[J]. Carbohydrate Poymers, 2012, 87(2): 995-1012. DOI:10.1016/j.carbpol.2011.09.063 |
| [3] |
车小琼, 孙庆申, 赵凯. 甲壳素和壳聚糖作为天然生物高分子材料的研究进展[J]. 高分子通报, 2008(2): 45-49. |
| [4] |
施文涛, 陈鹏. 甲壳素酰化改性的研究进展[J]. 化工进展, 2016, 35(7): 2166-2172. |
| [5] |
张步宁, 崔英德, 陈循军, 等. 甲壳素/壳聚糖医用敷料研究进展[J]. 化工进展, 2008, 27(4): 520-526. DOI:10.3321/j.issn:1000-6613.2008.04.010 |
| [6] |
Cho YW, Cho YN, et al. Water-soluble chitin as a wound healing accelerator[J]. Biomaterials, 1999, 20(22): 2139-2145. DOI:10.1016/S0142-9612(99)00116-7 |
| [7] |
Patrulea V, Ostafe V, Borchard G, et al. Chitosan as a starting material for wound healing applications[J]. European Journal of Pharmaceutics and Biopharmaceutics, 2015, 97: 417-426. DOI:10.1016/j.ejpb.2015.08.004 |
| [8] |
Muñoz G, Valencia C, Valderruten N, et al. Extraction of chitosan from Aspergillus niger mycelium and synthesis of hydrogels for controlled release of betahistine[J]. Reactive and Functional Polymers, 2015, 91: 1-10. |
| [9] |
Elieh-Ali-Komi D, Hamblin MR. Chitin and chitosan:production and application of versatile biomedical nanomaterials[J]. International journal of advanced research, 2016, 4(3): 411-427. |
| [10] |
Aljawish A, Chevalot I, Jasniewski J, et al. Enzymatic synthesis of chitosan derivatives and their potential applications[J]. Journal of Molecular Catalysis B:Enzymatic, 2015, 112: 25-39. DOI:10.1016/j.molcatb.2014.10.014 |
| [11] |
Smith A, Perelman M, Hinchcliffe M. Chitosan:a promising safe and immune-enhancing adjuvant for intranasal vaccines[J]. Human vaccines & immunotherapeutics, 2014, 10(3): 797-807. |
| [12] |
Younes I, Rinaudo M. Chitin and chitosan preparation from marine sources. structure, properties and applications[J]. Marine drugs, 2015, 13(3): 1133-1174. DOI:10.3390/md13031133 |
| [13] |
张述智, 夏伟, 朱绍辉, 等.从蝇蛆中提取甲壳素的方法: CN103880979A[P]. 2014-06-25.
|
| [14] |
马翠翠, 任健. 蝇蛆壳制备壳聚糖的工艺条件研究[J]. 现代食品科技, 2013, 29(2): 331-334. |
| [15] |
Gadgey KK, Bahekar A. Study on extraction methods of chitin from crab shell and investigation of its mechanical properities[J]. International Journal of Mechanical Engineering and Technology, 2017, 8(2): 220-231. |
| [16] |
Truong T, Hausler R, Monette F, et al. Fishery industrial waste valorization for the transformation of chitosan by hydrothermo-chemial method[J]. Revve des Sciences de I'Eau, 2007, 20: 253-262. DOI:10.7202/016170ar |
| [17] |
高乐平, 杜予民, 等. 过氧化氢甲壳素脱色反应条件与分子量研究[J]. 武汉大学学报:理学版, 2002, 48(4): 453-456. |
| [18] |
陈雪姣, 姜启兴, 等. 南极磷虾甲壳素的脱色工艺研究[J]. 郑州轻工业学院学报(自然科学版), 2014, 29(3): 21-24. DOI:10.3969/j.issn.2095-476X.2014.03.005 |
| [19] |
张钟, 高佳欣. 柠檬酸-反相微胶团法提取虾壳中甲壳素的研究[J]. 食品工业科技, 2017, 38(5): 269-273. |
| [20] |
孟凡欣, 吴丽艳, 徐盼菊, 等. 虾壳甲壳素提取工艺优化及其辅助降血糖活性研究[J]. 食品研究与开发, 2016, 37(5): 95-100. DOI:10.3969/j.issn.1005-6521.2016.05.023 |
| [21] |
Gortari MC, Hours RA. Biotechnological processes for chitin recovery out of crustacean waste:a mini-review[J]. Electronic Journal of Biotechnology, 2013, 16(3): 14. |
| [22] |
Sedaghat F, Yousefzadi M, Toiserkani H, et al. Bioconversion of shrimp waste Penaeus merguiensis using lactic acid fermentation:an alternative procedure for chemical extraction of chitin and chitosan[J]. International Journal of Biological Macromolecules, 2017, 104: 883-888. DOI:10.1016/j.ijbiomac.2017.06.099 |
| [23] |
Valdez-Peña AU, Espinoza-Perez JD, Sandoval-Fabian GC, et al. Screening of industrial enzymes for deproteinization of shrimp head for chitin recovery[J]. Food Science and Biotechnology, 2010, 19(2): 553-557. DOI:10.1007/s10068-010-0077-z |
| [24] |
Vázquez JA, Noriega D, Ramos P, et al. Optimization of high purity chitin and chitosan production from Ⅲex argentinus pens by a combination of enzymatic and chemical processess[J]. Carbohydrate Polymers, 2017, 174: 262-272. DOI:10.1016/j.carbpol.2017.06.070 |
| [25] |
姜红鹰, 周玉玲, 张桂敏, 等. 酶法制备甲壳素系列衍生物的研究进展[J]. 氨基酸和生物资源, 2016, 38(4): 71-76. |
| [26] |
Kaur S, Dhillon GS. Recent trends in biological extraction of chitin from marine shell wastes:A review[J]. Critical Rev Biotechnol, 2015, 35(1): 44-61. DOI:10.3109/07388551.2013.798256 |
| [27] |
Antunes-Valcareggi SA, Ferreira SRS, Hense H. Enzymatic hydrolysis of blue crab(callinectes sapidus)waste processing to obtain chitin, protein, and astaxanthin-enriched extract[J]. International Journal of Environmental & Agriculture Research, 2017, 3(1): 81-92. |
| [28] |
Younes I, Ghorbel-Bellaaj O, Nasri R, et al. Chitin and chitosan preparation from shrimp shells using optimized enzymatic deproteinization[J]. Process Biochemistry, 2012, 47(12): 2032-2039. DOI:10.1016/j.procbio.2012.07.017 |
| [29] |
Hamdi M, Hammami A, Hajji S, et al. Chitin extraction from blue crab(Portunus segnis)and shrimp(Penaeus kerathurus)shells using digestive alkaline proteases from P. segnis viscera[J]. International Journal of Biological Macromolecules, 2017, 101: 455-463. DOI:10.1016/j.ijbiomac.2017.02.103 |
| [30] |
Oh YS, Shih L, Tzeng YM, et al. Protease produced by Pseudomonas aeruginosa K-187 and its application in the deproteinization of shrimp and crab shell wastes[J]. Enzyme and Microbial Technology, 2000, 27(1-2): 3-10. DOI:10.1016/S0141-0229(99)00172-6 |
| [31] |
Lopes C, Antelo LT, Franco-Uría A, et al. Chitin production from crustacean biomass:Sustainability assessment of chemical and enzymatic processes[J]. Journal of Cleaner Production, 2018, 172: 4140-4151. DOI:10.1016/j.jclepro.2017.01.082 |
| [32] |
Arbia W, Arbia L, Adour L, et al. Chitin extraction from crustacean shells using biological methods-A review[J]. Food Technology & Biotechnology, 2013, 51(1): 12-25. |
| [33] |
Nguyen TT, Barber AR, Smith P, et al. Appliaction and optimization of the highly efficient and environmentally-friendly microwave-intensified lactic acid demineralization of deproteinized Rock lobster shells(Jasus edwardsii)for chitin production[J]. Food and Bioproducts Processing, 2017, 102: 367-374. DOI:10.1016/j.fbp.2017.02.005 |
| [34] |
Ploydee E, Chaiyanan, S. Production of high viscosity chitosan from biologically purified chitin isoated by microbial fermentation and deproteinization[J]. International Journal of Polymer Science, 2014, 2014(5): 1-8. |
| [35] |
Neves AC, Zanette C, et al. Optimization of lactic fermentation for extraction of chitin from freshwater shrimp waste[J]. Acta Scientiarum Technology, 2017, 39(2): 125-133. DOI:10.4025/actascitechnol.v39i2.29370 |
| [36] |
Ghorbel-Bellaaj O, Hmidet N, Jellouli K, et al. Shrimp waste fermentation with Pseudomonas aeruginosa A2:optimization of chitin extraction conditions through Plackett-Burmanand response surface methodology approaches[J]. International Journal of biological Macromolecules, 2011, 48(4): 596-602. DOI:10.1016/j.ijbiomac.2011.01.024 |
| [37] |
Choorit W, Patthanamanee W, Manurakchinakorn S. Use of response surface method for the determination of demineralization efficiency in fermented shrimp shells[J]. Bioresour Technol, 2008, 99(14): 6168-6173. DOI:10.1016/j.biortech.2007.12.032 |
| [38] |
Ghorbel-Bellaaj O, Younes I, Maalej H, et al. Chitin extraction from shrimp shell waste using Bacillus bacteria[J]. International Journal of Biological Macromolecules, 2012, 51(5): 1196-1201. DOI:10.1016/j.ijbiomac.2012.08.034 |
| [39] |
Zhang H, Yu H, Qian Y, et al. Production of chitin and chitosan using successive three-step microbial fermentation[J]. Journal of Polymer Materials, 2017, 34(1): 123-127. |
| [40] |
Aytekin O, Elibol M. Cocultivation of Lactococcus lactis and Teredinobacter turnirae for biological chitin extraction from prawn waste[J]. Bioprocess and Biosystems Engineering, 2010, 33(3): 393-399. DOI:10.1007/s00449-009-0337-6 |
| [41] |
Zhang HC, Jin YF, Deng Y, et al. Production of chitin from shrimp shell powders using Serratia marcescens B742 and Lactobacillus plantarum ATCC 8014 successive two-step fermentation[J]. Carbohydrate Research, 2012, 362(2): 13-20. |
| [42] |
Duan S, Zhang YX, Lu TT, et al. Shrimp waste fermentation using symbiotic lactic acid bacteria[J]. Advanced Materials Research, 2011, 194: 2156-2163. |
| [43] |
Liu P, Liu SS, Guo N, et al. Cofermentation of Bacillus licheniformis and Gluconobacter oxydans for chitin extraction from shrimp waste[J]. Biochemical Engineering Journal, 2014, 91: 10-15. DOI:10.1016/j.bej.2014.07.004 |
| [44] |
段人钰.一种利用虾废料同时制取虾青素、壳聚糖的方法: CN106520984A[P]. 2017-03-22..
|
| [45] |
Hajji S, Younes I, Ghorbel-Bellaaj O, et al. Structural differences between chitin and chitosan extracted from three different marine sources[J]. International Journal of Biological Macromolecules, 2014, 65: 298-306. DOI:10.1016/j.ijbiomac.2014.01.045 |
| [46] |
Chang KLB, Tsai G, Lee J, et al. Heterogeneous N-deacetylation of chitin in alkaline soution[J]. Carbohyd Research, 1997, 303(3): 327-332. DOI:10.1016/S0008-6215(97)00179-1 |
| [47] |
Berger J, Reist M, Chenite A, et al. Pseudo-thermosetting chitosan hydrogels for biomedical application[J]. International Journal of Pharmaceutics, 2005, 288(2): 197-206. DOI:10.1016/j.ijpharm.2004.07.037 |
| [48] |
Kaczmarek MB, Struszczyk-Swita K, et al. Isolation, molecular coning and characterization of two genes coding chitin deacetylase from Mucor circinelloides IBT-83[J]. Progress of Chemistry & Application of Chitin & Its Derivatives, 2016, 21: 93-103. |
| [49] |
Liu Z, Gay LM, Tuveng TR, et al. Structure and function of a broad-specificity chitin deactylase from Aspergillus nidulans FGSC A4[J]. Scientific Reports, 2017, 7(1): 1746. DOI:10.1038/s41598-017-02043-1 |
| [50] |
Ramos-Puebla A, Santiago CD, Trombotto S, et al. Addition of abscisic acid increases the production of chitin deacetylase by Colletotrichum gloeosporioides in submerged culture[J]. Process Biochemistry, 2016, 51(8): 959-966. DOI:10.1016/j.procbio.2016.05.003 |
| [51] |
Blair DE, Hekmat O, Schüttelkopf AW, et al. Structure and mecha-nism of chitin deacetylase from the fungal pathogen Colletotrichum lindemuthianum[J]. Biochemistry, 2006, 45(31): 9416-9426. DOI:10.1021/bi0606694 |
| [52] |
Martinou A, Bouriotis V, et al. Mode of action of chitin deacetylase from Mucor rouxii on partially N-acetylated chitosans[J]. Carbohydrate Research, 1998, 311(1-2): 71-78. DOI:10.1016/S0008-6215(98)00183-9 |
| [53] |
王皓, 吴丽, 朱小花, 等. 甲壳素脱乙酰酶的研究概况及展望[J]. 中国生物工程杂志, 2015, 35(1): 96-103. |
| [54] |
Tsigos I, Bouriotis V. Purification and characterization of chitin deacetylase from Colletotrichum lindemuthianum[J]. J Biol Chem, 1995, 270(44): 26286-26291. DOI:10.1074/jbc.270.44.26286 |
| [55] |
Kang LX, Liang YX, Ma LX. Novel characteristics of chitin deacetylase from Colletotrichum lindemuthianum:Production of fully acetylated chitooligomers, and hydrolysis of deacetylated chitooligomers[J]. Process Biochemistry, 2014, 49(11): 1936-1940. DOI:10.1016/j.procbio.2014.07.011 |
| [56] |
窦勇, 胡佩红. 超声协同CDA酶法制备龙虾壳聚糖[J]. 食品与发酵工业, 2014, 40(11): 127-131. |
| [57] |
Aspras I, Kamińska M, Karzyński K, et al. The influence of selected ionic liquids on activity of chitin deacetylase[J]. Chemical and Process Engineering, 2016, 37(1): 77-82. |
| [58] |
Jaworska MM. Kinetics of enzymatic deacetylation of chitosan[J]. Cellulose, 2012, 19(2): 363-369. DOI:10.1007/s10570-012-9650-3 |
| [59] |
Ghormade V, Pathan EK, Deshpande MV. Can fungi compete with marine sources for chitosan production?[J]. International Journal of Biological Macromolecules, 2017, 104: 1415-1421. DOI:10.1016/j.ijbiomac.2017.01.112 |
| [60] |
Dhillon GS, Kaur S, Sarma SJ, et al. Integrated process for fungal citric acid fermentation using apple processing wastes and sequential extraction of chitosan from waste stream[J]. Industrial Crops and Products, 2013, 50: 346-351. DOI:10.1016/j.indcrop.2013.08.010 |
| [61] |
Pochanavanich P, Suntornsuk W. Fungal chitosan production and its characterization[J]. Letters in Applied Microbiology, 2002, 35(1): 17-21. DOI:10.1046/j.1472-765X.2002.01118.x |
| [62] |
Rane KD, Hoover DG. Production of chitosan by fungi[J]. Food biotechnology, 1993, 7(1): 11-33. DOI:10.1080/08905439309549843 |
| [63] |
Muzzarelli RAA, Ilari P, Tarsi R, et al. Chitosan from Absidia coerulea[J]. Carbohydrate Polymers, 1994, 25(1): 45-50. DOI:10.1016/0144-8617(94)90161-9 |
| [64] |
Kim WJ, Lee WG, Theodore K, et al. Optimization of culture conditions and continuous production of chitosan by the fungi, Absidia coerulea[J]. Biotechnology and Bioprocess Engineering, 2001, 6(1): 6-10. DOI:10.1007/BF02942243 |
| [65] |
Abdel-Gawad KM, Hifney AF, et al. Technology optimization of chitosan production from Aspergillus niger biomass and its functional activities[J]. Food Hydrocolloids, 2017, 63: 593-601. DOI:10.1016/j.foodhyd.2016.10.001 |
| [66] |
Tasar OC, Erdal S, Taskin M. Chitosan production by psychrotole-rant Rhizopus oryzae in non-sterile open fermentation conditions[J]. International Journal of Biological Macromolecules, 2016, 89: 428-433. DOI:10.1016/j.ijbiomac.2016.05.007 |
| [67] |
Doiphode N, Rajamohanan PR, et al. Chitosan production using a dimorphic zygomycetous fungus Benjaminiella poitrasii:role of chitin deacetylase[J]. Asian Chitin J, 2009, 5: 19-26. |
| [68] |
Amorim RVDS, Pedrosa RP, Fukushima K, et al. Alternative carbon sources from sugar cane process for submerged cultivation of Cunninghamella bertholletiae to produce chitosan[J]. Food Technology & Biotechnology, 2006, 44(4): 519-523. |
| [69] |
Amorim RVS, Souza W, Fukushima K, et al. Faster chitosan production by mucoralean strains in submerged culture[J]. Brazilian Journal of Microbiology, 2001, 32(1): 20-23. DOI:10.1590/S1517-83822001000100005 |
| [70] |
Vendruscolo F, Ninow JL. Apple pomace as a substrate for fungal chitosan production in an airlift bioreactor[J]. Biocatalysis and Agricultural Biotechnology, 2014, 3(4): 338-342. DOI:10.1016/j.bcab.2014.05.001 |
| [71] |
Cardoso A, Lins CIM, dos Santos E R, et al. Microbial enhance of chitosan production by Rhizopus arrhizus using agroindustrial substrates[J]. Molecules, 2012, 17(5): 4904-4914. DOI:10.3390/molecules17054904 |
| [72] |
Gharieb MM, El-Sabbagh SM, Shalaby MA, et al. Production of chitosan from different species of Zygomycetes and its antimicrobial activity[J]. Int. J. Sci. Eng. Res, 2015, 6: 123-130. |
| [73] |
Yang L, Li X, Lai C, et al. Fungal chitosan production using xylose rich of corn stover prehydrolysate by Rhizopus oryzae[J]. Biotechnology & Biotechnological Equipment, 2017, 31(6): 1160-1166. |