2. 农业部农业转基因生物安全评价(食用)重点实验室 中国农业大学,北京 100083;
3. 中国动物疫病预防控制中心 动物产品安全检测室,北京 102600
2. Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety)for the Ministry of Agriculture of P.R.China, Beijing 100083;
3. China Animal Disease Control Centre, Animal Products Safety Testing Room, Beijing 102600
食品安全是全球高度关注的、直接关系民生的公共卫生安全问题,因此食品安全检测变得尤为重要,食品安全的风险评估与检测技术成为研究热点。
食品被食源性致病菌污染后,会引起食品的变质腐败,且食源性致病菌的生长会产生特定的毒素,直接或间接地导致患病。常见的食源性致病菌包括致病性大肠杆菌(Escherichia coli O157:H7及肠出血性菌株Escherichia coli O103:H25,O26、O111、O115、O128及O145)、沙门氏菌(Salmonella spp.)、单核细胞增多性李斯特氏菌(Listeria monocytogenes)、金黄色葡萄球菌(Staphylococcus aureus)、致病性弧菌(Vibrio spp.)、弯曲杆菌(Campylobacter spp.)等。
传统微生物方法需要包含分离、培养、显微镜检测等步骤,虽然操作简单且结果稳定性好,但有几个局限。第一,此方法至少需要24 h培养。第二,后续鉴定检测繁琐,无法满足对食源性致病菌快速检测的需求。
此外,以PCR为代表的核酸分子检测技术可以实现对微生物的基因组DNA或RNA片段进行高效识别。该方法最为灵敏准确,广泛用于病原体的检测。但PCR检测需要完好无损伤的微生物DNA或RNA[1],且无法区分活菌和死菌。此外,常规PCR检测方法是需要合适的仪器、专业试剂及有经验的人员。
以抗原抗体为基础的免疫学方法也发展迅速,这些特异性抗体是以病原体特有的蛋白质或碳水化合物为靶标。此方法包括凝集试验、ELISA、Western blot分析等。免疫法灵敏度高,但保存和处理抗体需要特定条件[2],以防止其变性。因此,发展快速、成本效益好、可靠性高的微生物检测方法是十分必要的。如今,功能特点有望与抗体媲美的“核酸抗体”逐渐成为研究热点,我们称这种核酸抗体为“适配体”。
适配体(aptamer),由拉丁语“aptus”(合适)与德语“meros”(部分)演化而来,是运用SELEX技术从体外人工合成的随机寡核苷酸库中筛选得到的,能高亲和、特异性识别靶标物质的一段寡核苷酸序列(DNA/RNA)。适配体的特异性和亲和性与单克隆抗体类似。但与抗体相比,适配体具有其他大量优点:理论上几乎任何靶标(甚至是有毒的或无致免疫原性的)都有相应的适配体;适配体体积比较小(是单克隆抗体全尺寸1/20-1/25),可提高组织渗透;适配体是一个相对稳定的化合物,它可以通过标准寡核苷酸化学合成方法合成,必要时可以进行不同的化学修饰,而修饰可以进一步提高其稳定性,生物利用度和药物动力学,或使其在使用过程中易于被固定或被标记;与微生物表面的决定因素特异性结合的DNA或RNA适配体,可以作为新型检测系统的识别元件;热稳定、化学稳定、低成本、批次偏差小等这些特点,使得基于适配体的方法有望代替免疫学方法。此外,高亲和性和选择性使适配体可以区分高相关性的靶标,如蛋白质不同亚型[3]。
适配体生物传感器是利用适配体独特的立体结构可与靶标微生物特异性结合的原理,以适配体作为识别元件,与适当的信号转换器相结合,转变为电、光、声等可识别的信号,实现对食源性致病菌检测的生物传感器。
由于适配体筛选可以在实际检测模拟条件下进行,这对环境和食品样品中微生物的检测极其有利。在不影响其亲和力的情况下,可以对适配体改造以便固定化和标记报告分子。与抗体相比,适配体更容易被化学修饰和标记;并且作为核酸序列,可进行变性和复性的重复循环;这使得固定化生物组件功能可以再生利用[4-5]。
本综述主要介绍食源性致病菌SELEX技术、食源性致病菌适配体及其适配体生物传感器在食源性致病微生物检测中的研究进展,旨在为微生物检测方法开拓新领域,指引新方向。
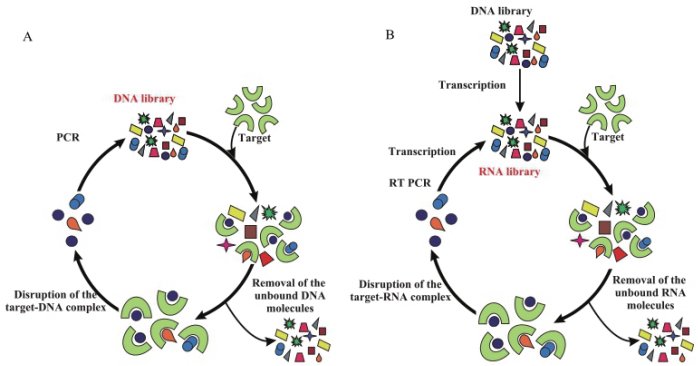
1 食源性致病菌适配体的SELEX筛选技术指数富集配体进化技术(Systematic evolution of ligands by exponential enrichment,SELEX),其基本方案包括3个主要阶段:文库和靶标进行孵育,适配体-靶标复合物与未结合寡核苷酸的分离,适配体的扩增(图 1-A)。基于RNA文库的SELEX另包括以下步骤:在体外转录获得一个RNA文库,对结合靶标的RNA进行反转录获得cDNA并进行扩增(图 1-B)。
在筛选过程中,文库中具有较高靶标亲和性的序列被富集。虽然能很快获得一些适配体,但大多需要5-15轮SELEX。在筛选后,对富集的序列进行克隆、测序,以确定每条适配体的序列。通过适配体的结构分析,找到共有区,以确定适配体与靶标结合所需的最小尺寸及适配体与靶标的亲和性。
到目前为止,SELEX技术应用于广泛的靶标,包括纯化蛋白质、小分子、活细胞、组织和微生物。
1.1 细胞-SELEX传统上,可溶性蛋白质纯化是SELEX中最常使用的靶标。基于纯化蛋白的SELEX方法被广泛应用的原因是其操作非常简单,可以在控制的条件下进行。但这种筛选是在非生理环境下完成的,重组蛋白往往不能折叠成正确的结构。因此,筛选的适配体可能不与天然构象的靶标识别。为了克服这个局限,以完整活细胞(cell-SELEX)作为一个复杂靶标,使得适配体的筛选过程可以在生理环境中进行。与基于蛋白质的SELEX技术相比,cell-SELEX过程中所有分子都处于其天然折叠结构和分布,这种方法可以提高在体内成功筛选适配体的可能性。
因此,cell-SELEX成为筛选细胞表面蛋白适配体的首选。其另一个优势是,对特定细胞类型的适配体进行筛选时,无需对其特定靶标有先验知识,这就导致可能得到多种识别特殊细胞表型的适配体,或发现新的细胞生物标记物。到目前为止,已筛选出一些不同细胞类型和其他复杂系统的适配体,如不同血清型的细菌。
1.2 磁珠-SELEX1997年,首次基于SELEX技术,利用磁珠固定靶标进行适配体筛选[7]。在此方法中,靶标大多为蛋白质,将蛋白质固定在磁珠上,与适配体结合后,通过磁分离,获得结合的适配体。2005年,有人提出了比磁珠-SELEX更加方便简单的FluMag-SELEX方法[8],此方法基于磁珠-SELEX,引入荧光标记的引物,经历PCR扩增后,使磁珠上靶标结合的适配体带有荧光基团。在FluMag-SELEX基础上发展了一种新方法,与前两种基于磁珠的SELEX方法不同,capture-SELEX是将DNA文库修饰在磁珠上,这种方法有利于小分子靶标适配体的筛选。磁珠-SELEX与微流体结合,开发一种自动化平台,用于快速获得高靶标亲和性的适配体。
1.3 毛细管电泳-SELEXSELEX技术最常用的变型是毛细管电泳-SELEX(CE-SELEX)[9],这种方法的原理是根据电泳迁移率来将适配体-靶标复合体与未结合序列分离。与传统方法相比,CE-SELEX可以大幅度降低筛选轮数。微自由流动电泳-SELEX技术(μFFE)[10]是CE-SELEX的改进技术,与传统SELEX和CE-SELEX相比,适配体的分离效率显著提高,这便于降低样本的使用量,降低实验成本。
1.4 One-step SELEX为了降低筛选轮数,一系列One-step SELEX技术陆续被开发,如MonolEX、NanoSelection等。MonolEX[11]是一种基于亲和层析方法的SELEX技术,其原理是将靶标固定在树脂上,亲和力强的适配体保留在树脂,而未结合序列被洗脱。NanoSelection的原理[12]是携带荧光标记寡核苷酸的粒子与固定在盖玻片的靶标孵育结合,通过荧光显微镜进行检测,在原子力显微镜下对结合的适配体进行提取。
2 食源性致病细菌适配体的研究进展适配体的寡核苷酸序列中间为20-60 nt随机序列,其中不同的序列具有不同的构象和结合能力。使用短的(< 50碱基)SELEX文库,其优势是,较短适配体可以高效的、便宜的、通过化学合成得到,同时也减少了文库的复杂性,降低了限制筛选的有效性。随机区域两侧通常是两个恒定序列,用于与引物结合,进行PCR扩增和转录(RNA文库)。在文库的设计上,引物区序列的长度是一个重要的部分。为开发片段较短的适配体,构建短的或无引物序列的文库越来越备受关注。此外,由于引物可能覆盖适配体序列总长度的一半以上,因此会很大程度上影响适配体的复杂性和适配体与靶标的结合能力。引物区采用较短序列(7-10个核苷酸)的方法已经开发出来。这些方法包括利用自身互补序列来隔绝恒定区域的序列,从而减少引物区对适配体与靶标结合的影响。有学者提出构建基于酶反应的无引物区的文库[13-14]。例如,Pan等[13]使用双链DNA核酸内切酶劈裂去除每个SELEX周期前的引物,在每轮筛选后再与引物连接重组为文库。
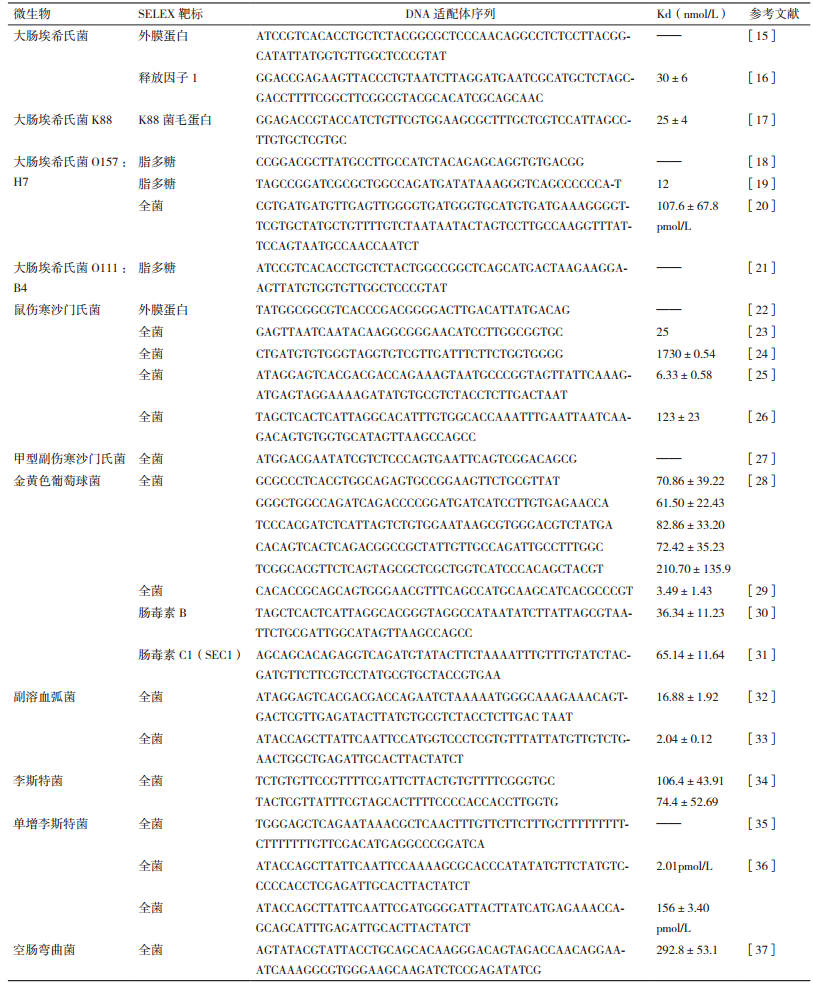
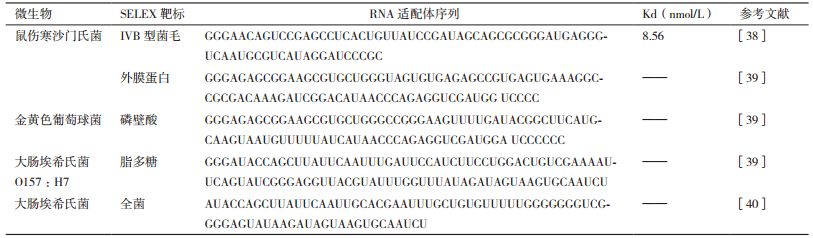
本文总结了目前国内外文献中报道的较为常见的食源性致病菌适配序列(表 1-表 3)。
在文库设计上,另一个需要考虑的因素是RNA或单链DNA的选择。一般来说,DNA文库扩增过程比RNA文库简单,并且DNA寡核苷酸更稳定和便宜。
2.2 RNA适配体RNA文库与DNA文库相比具有以下一些优势。首先,RNA分子更灵活,具有更高的折叠复杂性。此外,RNA适配体对细胞核酸酶更加敏感,引入化学修饰的保护基,以达到相对的稳定,甚至比ssDNA更好。适配体化学修饰最常见的方法是在核糖2' 端引入取代基和对磷酸核糖骨架进行修饰。在SELEX过程中,由于与酶促反应的兼容,2' -氟代和2' -氨代核苷可以被引入到初始文库。
2.3 劈裂适配体如今,适配体与靶标结合所需最小长度的研究越发被关注。Lee[41]对E.coli O157:H7特异结合的RNA适配体I-1,根据预测的二级结构进行劈裂,形成3个适配体衍生物:“长茎环结构”、“短茎环结构”及由长短茎环结构组成的“两茎环”结构。其中,“长、短茎环结构”均与靶标无法结合,但“两茎环”结构特异性识别E.coli O157:H7。
Chinnappan等[42]对基于Kolovskaya筛选的适配体进行劈裂,获得解离常数为3.2 nmol/L的劈裂适配体,并以此构建检测肠炎沙门氏菌的荧光共振转移传感器,其检测限为25 CFU/mL。
3 适配体结合力表征方法 3.1 流式细胞术流式细胞术是一种基于激光的方法,能够具有高重现性和准确性的表征分子的物理特性和化学特性[43]。因此,流式细胞术已经成为在表征适配体结合亲和性方面最常用的方法之一,一股单细胞悬浊液依次通过检测装置,来检测细胞群中被标记的细胞。结合相互作用是由标记细胞的荧光强度决定,其荧光强度高表明适配体与靶标细胞的结合能力强。直方图和点状图是流式细胞仪的两个主要输出方式,其中点状图可同时显示不同适配体与不同类型细胞结合的比较情况。目前,已经有研究利用流式细胞术,对适配体的结合力进行表征,这些适配体是利用不同靶标筛选得到的,如E.coli O157:H7[20]、鼠伤寒沙门氏菌[25]、副溶血弧菌[32]、空肠弯曲菌[37]、A族M型链球菌[44]、嗜酸乳杆菌[45]等。
3.2 基于光谱的方法紫外-可见吸收法(UV-Vis)是对适配体及其靶标结合特性进行表征的方法。通过同浓度适配体下,不同浓度的靶标的吸光值的改变来估算Kd值,但目前此方法应用较少。
荧光分光光度计可以通过对标记荧光基团的适配体与适配体结合荧光强度的检测,绘制解离曲线,以此来获得解离常数Kd值。由于荧光分光光度计使用较广泛,该方法得到广泛使用,在鼠伤寒沙门氏菌[26, 46]、金黄色葡萄球菌[29]等均有应用。
3.3 酶联分析类似于传统的、用于特定物质检测的酶联免疫吸附法(ELISA),可以将适配体偶联到酶联分析中,以确定其结合亲和力。1996年,Drolet[47]首次报道,这种方法分析适配体结合力的灵敏度高且可进行定量。因此,酶联分析也被常用来表征基于cell-SELEX方法获得的适配体结合亲和力,并应用于研究全细胞、细胞裂解液或细胞分泌物及分泌物裂解物。
3.4 放射性闪烁计数闪烁计数对放射性标记样品的检测具有高灵敏度,这使得闪烁计数成为检测适配体与其靶标细胞结合反应的有力工具。为准确测量适配体结合亲和性,分离结合靶标与未结合靶标的适配体是必不可少的,分离过程可以通过过滤或离心实现。基于闪烁计数的适配体结合分析被应用于32P标记的RNA适配体,此适配体与伤寒血清型沙门氏菌的pil操纵子编码的IVB型菌毛特异结合,通过硝化纤维膜对结合反应物过滤,对硝化纤维膜上的适配体-靶标进行闪烁计数[48]。基于闪烁计数法对32P标记的DNA适配体与金黄色葡萄球菌细胞的结合力进行考察,结果显示,与靶标结合的适配体通过闪烁计数方法被量化[28]。
3.5 荧光和共聚焦显微镜虽然不是半定量,但共聚焦显微镜和荧光显微镜是用来监测适配体与细胞结合的最常用的工具,它可以从视觉上直观显示适配体与其靶标细胞的特异性。同时荧光和共聚焦显微镜在荧光信号激发和发射的原理上是相似的,但共聚焦显微镜具有视野深度控制的能力,减少背景,并可以获得连续光学切片。目前,已经应用荧光和共聚焦显微镜来表征一些适配体的结合亲和性和特异性,如甲型副伤寒沙门氏菌[27]、空肠弯曲菌[37]、鼠伤寒沙门氏菌[49]等。活细胞和染色细胞均可用于荧光和共聚焦显微镜,而荧光信号的获得,可以直接在适配体上引入荧光标记,或间接地利用荧光标记抗体靶向一个在适配体上的特定标签。荧光图像通常显示一个细胞可结合多个适配体,如金黄色葡萄球菌[28]。
3.6 原子力显微镜(AFM)原子力显微镜(Atomic force microscope,AFM)是一种基于扫描探针的显微镜技术,用解析技术对大分子进行高分辨率成像,并在生理条件下测量单分子水平的双分子相互作用力。AFM无需对样品固定及脱水,可以在水溶液条件及其潜在形态下进行观察细胞。这种特性,使得AFM适用于对活细胞表面蛋白及拓扑结构。AFM的分析可以在力直方图和拓扑图像中进行。黏附(pN)测定的直方图峰值用于估算适配体与靶标之间的亲和力。pN值越高,结合力越大,这就意味着结合亲和力越好。拓扑成像显示靶标表面构象,从而以图片形式,显示适配体的结合如何改变靶标表面构象。在金表面修饰适配体,利用AFM动态力显微镜(DFS)测定适配体与沙门菌的相互作用力,并首次在沙门氏菌表面测定单个外膜蛋白的位置[50]。
3.7 表面等离子共振(SPR)表面等离子共振(Surface plasmon resonance,SPR)技术是一种高通量、无标记、实时技术,用于表征适配体与其靶标的结合亲和力。Ahn等[33]利用表面等离子共振技术对筛选获得的副溶血弧菌适配体的亲和性进行表征。生物素标记的适配体通过与链霉亲和素耦合的方式,修饰到SA芯片上,加入副溶血弧菌后,检测SPR信号强度来衡量亲和力。
3.8 高效液相色谱(HPLC)高效液相色谱(High performance liquid chromat-ography,HPLC)技术可以定性和定量地分析适配体与靶标的结合。其原理是先将适配体固定在吸附柱上,在加压条件下,使液体溶剂流通吸附柱,色谱图显示分析物浓度及保留时间,根据适配体、靶标、适配体-靶标复合物的平衡分布来估算Kd值[51]。
3.9 热力学表征等温滴定量热法(Isothermal titration calorimetry,ITC)和微尺度热泳(Microscale thermophoresis,MST)都是与热力学相关的技术,适用于表征适配体与其靶标之间的亲和力。等温滴定量热法中,指定浓度的适配体或靶标作为滴定标准液,被加入不同浓度的互补分子中,此互补分子为分析物。当三元复合物形成是唯一放热过程时,细胞温度与对照细胞温度保持一致,并避开所有放热源。利用等温滴定量热法,基于细胞相对能量变化,对适配体-靶标结合进行表征,能量变化与结合量成正比[52]。
3.10 实时定量荧光PCR(real-time qPCR)实时定量荧光PCR技术,是指在PCR反应体系中加入荧光染料或加入荧光探针,利用荧光信号的变化实时监测整个PCR进程中每一个循环扩增产物量的变化,最后通过Ct值和标准曲线对起始模板进行定量分析。Ahn等[33]在对每一轮SELEX产物的亲和性表征时,利用实时定量荧光PCR技术,通过对Ct值大小的比较,来间接衡量适配体与靶标菌的亲和力。
4 食源性致病细菌适配体传感器的研究进展目前,食源性致病细菌的适配体传感器主要包括:电化学传感器、光学传感器、压电晶体传感器、横向色谱法测试条等,其中电化学适配体传感器与光学适配体传感器应用最为广泛,因此围绕这两种传感器进行重点阐述。
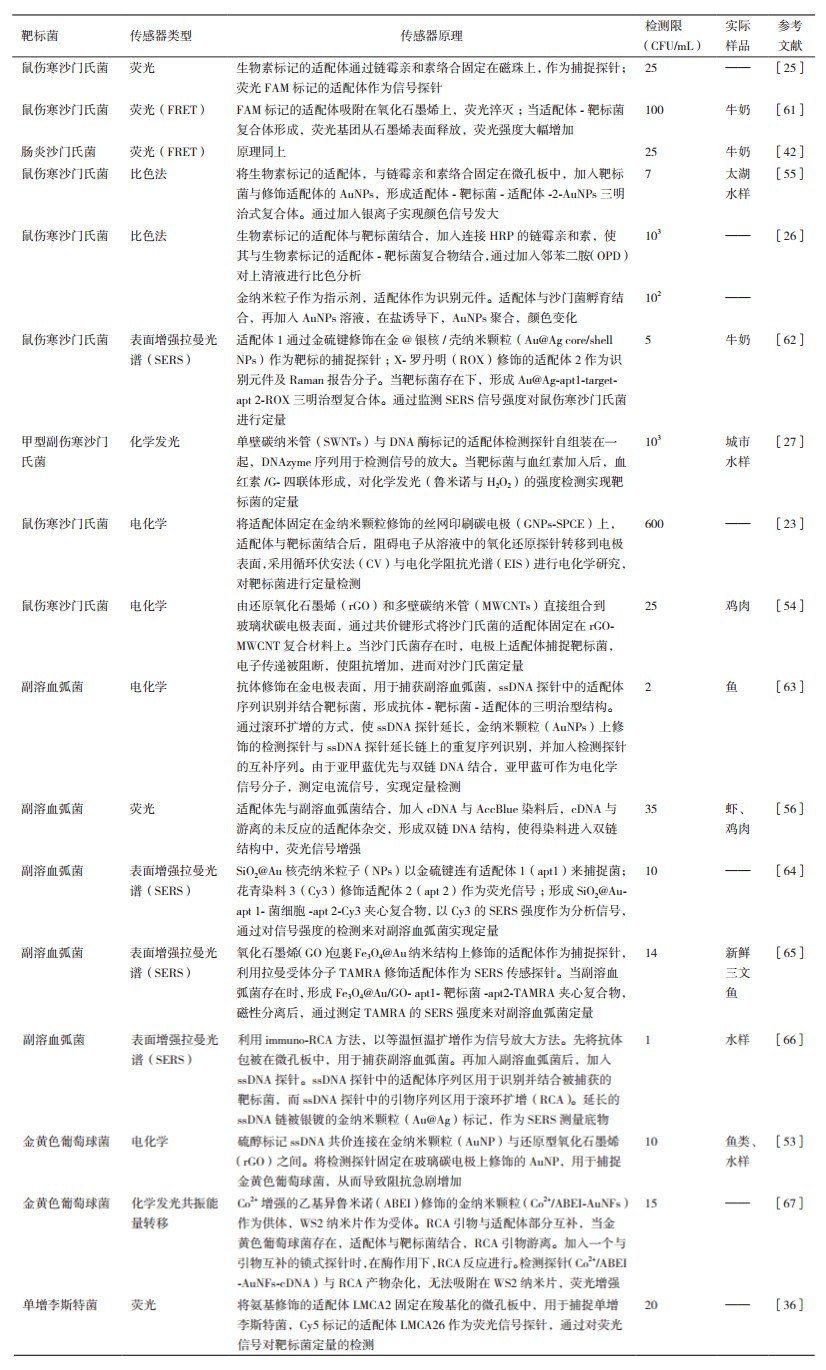
本文总结了目前国内外文献中报道的、较为常见的食源性致病细菌适配体传感器(表 4-表 5)。
在适当条件下,将适配体修饰到电极表面,靶标菌与适配体结合后,导致电极表面改变,从而导致电化学信号改变,如电流、电位、电导或阻抗。
电化学阻抗谱(Electrochemical impedance spec-troscopy,EIS)是一种检测电极表面界面性能变化的分析技术,分析物与电极表面的检测探针相互作用过后,使得电极表面界面性能改变。Peggy等[53]将Cao等[28]获得的其中一条适配体,进一步用于构建电化学阻抗适配体传感器。硫醇标记ssDNA共价连接在金纳米颗粒(Gold nanoparticle,AuNPs或GNPs)与还原型氧化石墨烯(Reduced graphene oxide,rGO)之间,将检测探针固定在玻璃碳电极上修饰的AuNPs,用于捕捉金黄色葡萄球菌,从而导致阻抗急剧增加,检测限为10 CFU/mL。
4.1.2 纳米管适配体传感器碳纳米管具有极好的电学特性、化学稳定性、比表面积大等优点,因此它被广泛应用于生物传感器,它的特性对于提高生物检测的灵敏度和稳定性具有巨大意义。段诺课题组[54]在其筛选获得的沙门氏菌适配体基础上,基于还原型氧化石墨烯(rGO)与多壁碳纳米管(Multiwalled carbon nanotube,MWCNT),构建一种电化学传感器,其检测限为25CFU/mL。
4.2 光学传感器 4.2.1 比色适配体传感器利用新型纳米材料的颜色可变性构建比色传感器,搭载适配体的新型纳米材料在溶液中呈分散状态,当靶标菌存在,适配体与之结合,使纳米颗粒聚集从而颜色改变。常用金作为比色传感器的指示剂,Yuan等[55]将适配体固定在微孔板中,加入靶标菌与修饰适配体的AuNPs,形成适配体-靶标菌-适配体-AuNPs三明治式复合体。通过加入银离子实现颜色信号发大。
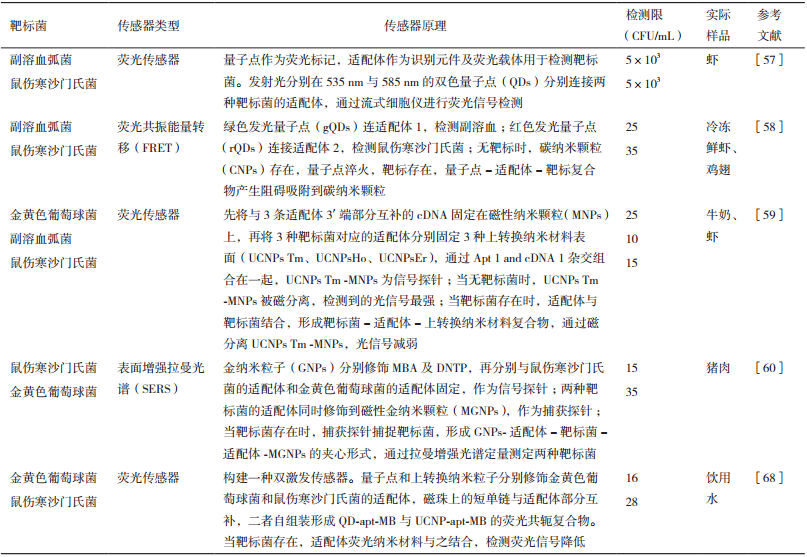
4.2.2 荧光适配体传感器荧光适配体传感器主要有两种,包括标记和无标记荧光传感器。如今,荧光传感器主要依赖于荧光基团标记的适配体或荧光纳米颗粒标记的适配体,通过靶标菌与适配体结合后,荧光偏振信号的产生或荧光强度的改变,来检测靶标菌。荧光强度的改变可以通过在适配体的两端标记荧光基团和淬灭基团,当存在靶标菌时,适配体构象改变,从而引起荧光信号的改变。标记荧光的方法困难、费时且昂贵,因此,开发便利且无标记的检测方法变得十分必要。PicoGreen、SYBRGreen dye与AccuBlue这些染料本身荧光十分微弱,然而与双链DNA结合时则具有高荧光性,但与单链DNA结合没有明显荧光变化。一种通用的荧光传感器具有信号“开”和“关”两种模式,这种传感器是基于AccuBlue染料与适配体,用于鼠伤寒沙门氏菌的检测[56]。量子点(Quantum dots,QDs)作为高效荧光纳米材料,可以作为高通量生物物理分析技术来定量检测细菌。量子点作为荧光标记,适配体作为识别元件及荧光载体用于检测靶标菌(副溶血弧菌、鼠伤寒沙门氏菌)。发射光分别在535 nm与585 nm的双色量子点分别连接两种靶标菌的适配体,通过流式细胞仪进行荧光信号检测,其检测限为5×103 CFU/mL[57]。基于双色量子点和碳纳米颗粒(Carbon nanoparticles,CNPs)利用荧光共振能量转移(FRET)原理,对鼠伤寒沙门氏菌与副溶血弧菌同时进行检测的传感器,其检测限分别为35 CFU/mL和25 CFU/mL[58]。氧化石墨烯(Graphene oxide,GO)是一种石墨烯制备的二维片层结构,具有优越的物理性能,如良好的导电性、机械强度高、比表面积大、生物相容性及可调控的光学特性。氧化石墨烯是荧光共振转移中良好的能量承载者,氧化石墨烯具有荧光淬灭的功能,是构建传感器的理想材料。
上转换纳米颗粒(Upconversion luminescent nanoparticles,UCNPs)作为荧光生物标记物,具有重要的光学和化学特性,如使用时间长、高耐光漂白和光化学降解。Wu[59]基于三种不同颜色的上转换纳米颗粒,对金黄色葡萄球菌、副溶血弧菌和鼠伤寒沙门氏菌进行检测,检测限分别为25 CFU/mL、10 CFU/mL和15 CFU/mL。
4.2.3 化学发光适配体传感器化学发光是某些化学反应伴随的一种光辐射现象。因为没有外激发光源存在,从而没有散射光背景干扰,因此具有灵敏度高、检测区间宽等优点。目前常见化学发光传感器,常在适配体上标记化学发光基团,或在适配体传感器体系中引入化学发光反应。杨明等[27]构建一种基于非共价的自组装检测方法,单壁碳纳米管(Single-walled carbon nanotube,SWNTs)与DNA酶标记的适配体检测探针自组装在一起,DNAzyme序列用于检测信号的放大。当靶标菌与血红素加入后,血红素/G-四联体形成,对化学发光(鲁米诺与H2O2)的强度检测实现靶标菌的定量,其检测限为103 CFU/mL。
4.2.4 表面增强拉曼光谱适配体传感器拉曼光谱是光子与分子相互碰撞发生方向和能量改变,从而产生散射光频率变化的联合散射光谱。为了增强光谱信号,对电极采用表面粗化的方法,即将分子吸附在粗糙金属表面或纳米粒子表面,从而获得表面增强拉曼光谱。GNPs分别修饰MBA及DNTP,再将鼠伤寒沙门氏菌的适配体和金黄色葡萄球菌的适配体分别固定在两种GNPs上,作为信号探针;将两种靶标菌的适配体分别修饰到磁性金纳米颗粒(MGNPs),作为捕获探针;当靶标菌存在时,捕获探针捕捉靶标菌,形成GNPs-适配体-靶标菌-适配体-MGNPs的夹心形式,通过拉曼增强光谱定量测定两种靶标菌检测限分别为15 CFU /mL和35 CFU /mL[60]。
5 展望目前,对食源性致病菌与适配体的结合位点及原理并未深入探索,因此食源性致病菌适配体的筛选技术仍以cell-SELEX为主。未来更加深入研究靶标与适配体的结合机制,对于构建快速筛选适配体的体系具有巨大推进作用。
食源性致病菌适配体生物传感器是一个学科交叉的新型检测方式,利用适配体传感器检测食源性致病菌目前仍局限于实验室,尚未应用于实际检测。其未来发展方向如下。
微生物单菌水平超灵敏检测技术的开发。目前的灵敏度仍待进一步提高,实际应用的检测限需达到1-10 CFU/mL水平。因此,利用各种信号放大手段来降低检测线尤为重要。目前,已有学者利用核酸扩增技术实现信号放大,检测限可低至1 CFU/mL[66]。
适配体与菌体表面识别机制有必要进一步解析。大多数适配体传感器是对某一靶标菌检测,适配体与菌的特异性取决于菌表面特定物质还是取决于特定构象,这些因素与菌的基因水平差异有何内在联系。有没有可能依据微生物基因组信息未来直接开发预测出靠谱的适配体序列。
微生物种类太多,变异性较大,目前适配体筛选的阳性菌株选择及阴性菌株选择存在者很大的随意性,后续有必要针对微生物的种属分类特点来提出并制定微生物适配体筛选评价指导手册或者规范,促进该行业的快速发展。
微生物适配体的亲和力评价手段很重要,不同方法结果不同,后续有必要开发适配体亲和性计算的模型及评价手段,适配体的结构裁剪规律同时也需要研究,以增强适配体的空间构象的稳定性,缩短适配体与靶物质的构象诱导的结合时间。
| [1] |
Kaittanis C, Santra S, Perez JM. Emerging nanotechnology-based strategies for the identification of microbial pathogenesis[J]. Advanced Drug Delivery Reviews, 2010, 62(5): 408-423. |
| [2] |
Shinde SB, Fernandes CB, Patravale VB. Recent trends in in-vitro nanodiagnostics for detection of pathogens[J]. Journal of Controlled Release, 2012, 159(2): 164-180. DOI:10.1016/j.jconrel.2011.11.033 |
| [3] |
Conrad R, Ellington AD. Detecting immobilized protein kinase C isozymes with RNA aptamers[J]. Analytical Biochemistry, 1996, 242(2): 261. |
| [4] |
Jayasena SD. Aptamers:an emerging class of molecules that rival antibodies in diagnostics[J]. Clinical Chemistry, 1999, 45(9): 1628. |
| [5] |
O'Sullivan CK. Aptasensors-the future of biosensing?[J]. Analytical & BioAnal Chem, 2002, 372(1): 44-48. |
| [6] |
Davydova A, Vorobjeva M, Pyshnyi D, et al. Aptamers against pathogenic microorganisms[J]. Critical Reviews in Microbiology, 2016, 42(6): 847-865. DOI:10.3109/1040841X.2015.1070115 |
| [7] |
Bruno JG. In vitro selection of DNA to chloroaromatics using magnetic microbead-based affinity separation and fluorescence detection[J]. Biochemical & Biophysical Research Communicat-ions, 1997, 234(1): 117-120. |
| [8] |
Stoltenburg R, Reinemann C, Strehlitz B. FluMag-SELEX as an advantageous method for DNA aptamer selection[J]. Analytical & BioAnal Chem, 2005, 383(1): 83-91. |
| [9] |
Mendonsa SD, Bowser MT. In vitro evolution of functional DNA using capillary electrophoresis[J]. J Am Chem Soc, 2004, 126(1): 20-21. DOI:10.1021/ja037832s |
| [10] |
Jing Meng, Bowser Michael T. Isolation of DNA aptamers using micro free flow electrophoresis[J]. Lab Chip, 2011, 11(21): 3703-3709. DOI:10.1039/c1lc20461k |
| [11] |
Nitsche A, Kurth A, Dunkhorst A, et al. One-step selection of Vaccinia virus-binding DNA aptamers by MonolEX[J]. BMC Biotechnol, 2007, 7: 48. DOI:10.1186/1472-6750-7-48 |
| [12] |
Peng L, Stephens BJ, Bonin K, et al. A combined atomic force/fluorescence microscopy technique to select aptamers in a single cycle from a small pool of random oligonucleotides[J]. Microscopy Research & Technique, 2007, 70(4): 372-381. |
| [13] |
Pan W, Xin P, Patrick S, et al. Primer-free aptamer selection using a random DNA library[J]. Journal of Visualized Experiments Jove, 2010, 629(41): 369-385. |
| [14] |
Lai YT, Destefano JJ. A primer-free method that selects high-affinity single-stranded DNA aptamers using thermostable RNA ligase[J]. Analytical Biochemistry, 2011, 414(2): 246-253. |
| [15] |
Bruno JG, Carrillo MP, Phillips T, et al. Anovel screening method for competitive FRET-aptamers applied to E.coli, assay development[J]. Journal of Fluorescence, 2010, 20(6): 1211-1223. DOI:10.1007/s10895-010-0670-9 |
| [16] |
Sando S, Ogawa A, Nishi T, et al. In vitro selection of RNA aptamer against Escherichia coli, release factor 1[J]. Bioorganic & Medicinal Chemistry Letters, 2007, 17(5): 1216-1220. |
| [17] |
Li H, Ding X, Peng Z, et al. Aptamer selection for the detection of Escherichia coli K88[J]. Canadian Journal of Microbiology, 2011, 57(6): 453-459. DOI:10.1139/w11-030 |
| [18] |
Wu W, Zhang J, Zheng M, et al. An aptamer -based biosensor for colorimetric detection of Escherichia coli O157:H7[J]. PLoS One, 2012, 7(11): e48999. DOI:10.1371/journal.pone.0048999 |
| [19] |
Kim SE, Su W, Cho MS, et al. Harnessing aptamers for electroche-mical detection of endotoxin[J]. Analytical Biochemistry, 2012, 424(1): 12-20. |
| [20] |
Amraee M, Oloomi M, Yavari A, et al. DNA aptamer identification and characterization for E.coli O157 detection using cell based SELEX method[J]. Analytical Biochemistry, 2017, 536: 36-44. DOI:10.1016/j.ab.2017.08.005 |
| [21] |
Bruno JG, Carrillo MP, Phillips T. In vitro, antibacterial effects of antilipopolysaccharide DNA aptamer-C1qrs complexes[J]. Folia Microbiologica, 2008, 53(4): 295-302. DOI:10.1007/s12223-008-0046-6 |
| [22] |
Joshi R, Janagama H, Dwivedi HP, et al. Selection, characterization, and application of DNA aptamers for the capture and detection of Salmonella enterica serovars[J]. Mol Cell Probes, 2009, 23(1): 20-28. DOI:10.1016/j.mcp.2008.10.006 |
| [23] |
Labib M, Zamay AS, Kolovskaya OS, et al. Aptamer-based viability impedimetric sensor for bacteria[J]. Anal Chem, 2012, 84(21): 8966-8969. DOI:10.1021/ac302902s |
| [24] |
Dwivedi HP, Smiley RD, Jaykus LA. Selection of DNA aptamers for capture and detection of Salmonella typhimurium using a whole-cell SELEX approach in conjunction with cell sorting[J]. Appl Microbiol Biotechnol, 2013, 97(8): 3677-3686. DOI:10.1007/s00253-013-4766-4 |
| [25] |
Duan N, Wu S, Chen X, et al. Selection and characterization of aptamers against Salmonella typhimurium using whole-bacterium Systemic Evolution of Ligands by Exponential Enrichment(SELEX)[J]. Journal of Agricultural & Food Chemistry, 2013, 61(13): 3229-3234. |
| [26] |
Lavu PSR, Mondal B, Ramlal S, et al. Selection and characterization of aptamers using a modified whole-Cell bacterium SELEX for the detection of Salmonella enterica serovar Typhimurium[J]. Acs Combinatorial Science, 2016, 18(6): 292-301. DOI:10.1021/acscombsci.5b00123 |
| [27] |
Yang M, Peng Z, Ning Y, et al. Highly specific and cost-efficient detection of Salmonella paratyphi a combining aptamers with single-walled carbon nanotubes[J]. Sensors(Basel), 2013, 13(5): 6865-6881. |
| [28] |
Cao X, Li S, Chen L, et al. Combining use of a panel of ssDNA aptamers in the detection of Staphylococcus aureus[J]. Nucleic Acids Research, 2009, 37(14): 4621-4628. DOI:10.1093/nar/gkp489 |
| [29] |
Moon J, Kim G, Park SB, et al. Comparison of whole-cell SELEX methods for the identification of Staphylococcus aureus-specific DNA aptamers[J]. Sensors(Basel), 2015, 15(4): 8884-8897. |
| [30] |
Mondal B, Ramlal S, Lavu PSR, et al. A combinatorial systematic evolution of Ligands by exponential enrichment method for selection of aptamer against protein targets[J]. Applied Microbiology & Biotechnology, 2015, 99(22): 9791-9803. |
| [31] |
Huang Y, Chen X, Duan N, et al. Selection and characterization of DNA aptamers against Staphylococcus aureus enterotoxin C1[J]. Food Chemistry, 2015, 166: 623-629. DOI:10.1016/j.foodchem.2014.06.039 |
| [32] |
Duan N, Wu S, Chen X, et al. Selection and identification of a DNA aptamer targeted to Vibrio parahemolyticus[J]. JAgric Food Chem, 2012, 60(16): 4034-4038. DOI:10.1021/jf300395z |
| [33] |
Ahn JY, Lee KA, Lee MJ, et al. Surface plasmon resonance aptamer biosensor for discriminating pathogenic bacteria[J]. Journal of Nanoscience & Nanotechnology, 2018, 18(3): 1599-1605. |
| [34] |
Suh SH, Dwivedi HP, Choi SJ, et al. Selection and characterization of DNA aptamers specific for Listeria species[J]. Analytical Biochemistry, 2014, 459(18): 39-45. |
| [35] |
Liu GQ, Lian YQ, Chao G, et al. In vitro Selection of DNA aptamers and fluorescence-based recognition for rapid detection Listeria monocytogenes[J]. Journal of Integrative Agriculture, 2014, 13(5): 1121-1129. DOI:10.1016/S2095-3119(14)60766-8 |
| [36] |
Lee SH, Ahn JY, Lee KA, et al. Analytical bioconjugates, aptamers, enable specific quantitative detection of Listeria monocytogenes[J]. Biosens Bioelectron, 2015, 68: 272-280. DOI:10.1016/j.bios.2015.01.009 |
| [37] |
Dwivedi HP, Smiley RD, Jaykus LA. Selection and characterization of DNA aptamers with binding selectivity to Campylobacter jejuni using whole-cell SELEX[J]. Appl Microbiol Biotechnol, 2010, 87(6): 2323-2334. DOI:10.1007/s00253-010-2728-7 |
| [38] |
Pan Q, Zhang XL, Wu HY, et al. Aptamers that preferentially bind type IVB pili and inhibit human monocytic-cell invasion by Salmonella enterica serovar typhi[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(10): 4052-4060. DOI:10.1128/AAC.49.10.4052-4060.2005 |
| [39] |
Maeng JS, Kim N, Kim CT, et al. Rapid detection of food pathogens using RNA aptamers-immobilized slide[J]. Journal of Nanoscience & Nanotechnology, 2012, 12(7): 5138. |
| [40] |
Dua P, Ren S, Sang WL, et al. Cell-SELEXBased identification of an RNA aptamer for Escherichia coli and its use in various detection formats[J]. Molecules & Cells, 2016, 39(11): 807-813. |
| [41] |
Lee YJ, Han SR, Maeng JS, et al. In vitro selection of Escherichia coli O157:H7-specific RNA aptamer[J]. Biochemical and Biophysical Research Communications, 2012, 417(1): 414-420. DOI:10.1016/j.bbrc.2011.11.130 |
| [42] |
Chinnappan R, Alamer S, Eissa S, et al. Fluorometric graphene oxide-based detection of Salmonella enteritis using a truncated DNA aptamer[J]. Mikrochimica Acta, 2017, 185(1): 61. |
| [43] |
Tan SY, Acquah C, Sidhu A, et al. SELEX modifications and bioanalytical techniques for aptamer-target binding characterisation[J]. Critical Reviews in Anal Chem, 2016, 46(6): 521-537. DOI:10.1080/10408347.2016.1157014 |
| [44] |
Alfavian H, Gargari SLM, Rasoulinejad S, et al. Development of specified DNA aptamer binding to group A Streptococcus serotype M3[J]. Canadian Journal of Microbiology, 2016, 63(2). |
| [45] |
Hamula Cl, Zhang H, Guan ll, et al. Selection of aptamers against live bacterial cells[J]. Anal Chem, 2008, 80(20): 7812-7819. DOI:10.1021/ac801272s |
| [46] |
Moon J, Kim G, Lee S, et al. Identification of Salmonella typhimurium-specific DNA aptamers developed using whole-cell SELEX and FACS analysis[J]. Journal of Microbiological Methods, 2013, 95(2): 162-166. DOI:10.1016/j.mimet.2013.08.005 |
| [47] |
Drolet DW, Moonmcdermott L, Romig TS. An enzyme-Linked oligonucleotide assay[J]. Nature Biotechnology, 1996, 14(8): 1021-1025. DOI:10.1038/nbt0896-1021 |
| [48] |
Pan Q, Zhang XL, Wu HY, et al. Aptamers that preferentially bind type IVB pili and inhibit human monocytic-cell invasion by Salmonella enterica serovar typhi[J]. Antimicrobial Agents and Chemotherapy, 2005, 49(10): 4052-4060. DOI:10.1128/AAC.49.10.4052-4060.2005 |
| [49] |
Wang QY, Kang YJ. Bioprobes based on aptamer and silica fluorescent nanoparticles for bacteria Salmonella typhimurium detection[J]. Nanoscale Research Letters, 2016, 11(1): 150. DOI:10.1186/s11671-016-1359-z |
| [50] |
Wang B, Park B, Xu B, et al. label-free biosensing of Salmonella enterica, serovars at single-cell level[J]. Journal of Nanobiotech-nology, 2017, 15(1): 40. DOI:10.1186/s12951-017-0273-6 |
| [51] |
Deng Q, German I, Buchanan D, et al. Retention and separation of adenosine and analogues by affinity chromatography with an aptamer stationary phase[J]. Anal Chem, 2001, 73(22): 5415-5421. DOI:10.1021/ac0105437 |
| [52] |
Stoltenburg R, Schubert T, Strehlitz B. In vitro selection and interaction studies of a DNA aptamer targeting protein A[J]. PLoS One, 2015, 10(7): e0134403. DOI:10.1371/journal.pone.0134403 |
| [53] |
Reich P, Stoltenburg R, Strehlitz B, et al. Development of an impedimetric aptasensor for the detection of Staphylococcus aureus[J]. Int J Mol Sci, 2017, 18(11): 2484. DOI:10.3390/ijms18112484 |
| [54] |
Jia F, Duan N, Wu S, et al. Impedimetric Salmonella, aptasensor using a glassy carbon electrode modified with an electrodeposited composite consisting of reduced graphene oxide and carbon nanotubes[J]. Microchim Acta, 2016, 183(1): 337-344. DOI:10.1007/s00604-015-1649-7 |
| [55] |
Yuan J, Tao Z, Yu Y, et al. A visual detection method for Salmonella Typhimurium based on aptamer recognition and nanogold Labeling[J]. Food Control, 2014, 37(1): 188-192. |
| [56] |
Duan N, Wu S, Ma X, et al. A universal fluorescent aptasensor based on AccuBlue dye for the detection of pathogenic bacteria[J]. Analytical Biochemistry, 2014, 454(1): 1-6. |
| [57] |
Duan N, Wu S, Yu Y, et al. A dual-color flow cytometry protocol for the simultaneous detection of Vibrio parahaemolyticus and Salmonella typhimurium using aptamer conjugated quantum dots as Labels[J]. Analytica Chimica Acta, 2013, 804(804C): 151-158. |
| [58] |
Duan N, Wu S, Dai S, et al. Simultaneous detection of pathogenic bacteria using an aptamer based biosensor and dual fluorescence resonance energy transfer from quantum dots to carbon nanoparticles[J]. Microchim Acta, 2015, 182(5-6): 917-923. DOI:10.1007/s00604-014-1406-3 |
| [59] |
Wu S, Duan N, Shi Z, et al. Simultaneous aptasensor for multiplex pathogenic bacteria detection based on multicolor upconversion nanoparticles Labels[J]. Anal Chem, 2014, 86(6): 3100. DOI:10.1021/ac404205c |
| [60] |
Zhang H, Ma X, Liu Y, et al. Gold nanoparticles enhanced SERS aptasensor for the simultaneous detection of Salmonella typhimurium and Staphylococcus aureus[J]. Biosens Bioelectron, 2015, 74: 872-877. DOI:10.1016/j.bios.2015.07.033 |
| [61] |
Duan YF, Ning Y, Song Y, et al. Fluorescent aptasensor for the determination of Salmonella typhimurium, based on a graphene oxide platform[J]. Microchim Acta, 2014, 181(5-6): 647-653. DOI:10.1007/s00604-014-1170-4 |
| [62] |
Duan N, Chang B, Zhang H, et al. Salmonella typhimurium detection using a surface-enhanced Raman scattering-based aptasensor[J]. Int J Food Microbiol, 2016, 218: 38-43. DOI:10.1016/j.ijfoodmicro.2015.11.006 |
| [63] |
Teng J, Ye Y, Yao L, et al. Rolling circle amplification based amperometric aptamer/immuno hybrid biosensor for ultrasensitive detection of Vibrio parahaemolyticus[J]. Microchim Acta, 2017, 184(9): 3477-3485. DOI:10.1007/s00604-017-2383-0 |
| [64] |
Duan N, Yan Y, Wu S, et al. Vibrio parahaemolyticus detection aptasensor using surface-enhanced Raman scattering[J]. Food Control, 2016, 63: 122-127. DOI:10.1016/j.foodcont.2015.11.031 |
| [65] |
Duan N, Shen M, Wu S, et al. Graphene oxide wrapped Fe3O4@Au nanostructures as substrates for aptamer-based detection of Vibrio parahaemolyticus, by surface-enhanced Raman spectroscopy[J]. Microchim Acta, 2017, 184(8): 1-8. |
| [66] |
Li Y, Ye Y, Teng J, et al. In vitro isothermal nucleic acid amplification assisted surface-enhanced raman spectroscopic for ultrasensitive detection of vibrio parahaemolyticus[J]. Anal Chem, 2017, 89(18). |
| [67] |
Hao L, Gu H, Duan N, et al. An enhanced chemiluminescence resonance energy transfer aptasensor based on rolling circle amplification and WS2 nanosheet for Staphylococcus aureus detection[J]. Analytica Chimica Acta, 2017, 959: 83-90. DOI:10.1016/j.aca.2016.12.045 |
| [68] |
Kurt H, Yüce M, Hussain B, et al. Dual-excitation upconverting nanoparticle and quantum dot aptasensor for multiplexed food pathogen detection[J]. Biosens Bioelectron, 2016, 81: 280-286. DOI:10.1016/j.bios.2016.03.005 |