2. 中国农业科学院生物技术研究所,北京 100081
2. Biotechnology Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081
角蛋白是非营养型的硬蛋白[1],起到保护机体的作用[2]。β角蛋白存在于鸟禽的羽毛中,因此又被称为羽毛角蛋白[3]。随着畜牧养殖业为我们提供大量肉制品的同时也产生大量毛发、羽毛和蹄等难降解物质。传统方法(填埋、焚烧)处理这些物质不仅占据大量空间污染环境还浪费蛋白质资源[4]。角蛋白酶(Keratinase,Ker)是专一水解角蛋白且对环境友好的一类酶[5],1899年,Ward首次报道了一株能降解角蛋白的细菌——真菌马甲团囊菌[6];1990年,Williams等[7]报道了一株能够降解羽毛的地衣芽孢杆菌PWD-1;1993年,Atalo和Gashe[8]得到了一株能够降解多种纤维性蛋白的嗜热杆菌,丁正民[9]分离纯化得到一株能够降解鸡毛的放线菌株SS-1;1996年,Frederich等[10]分离得到一株能够利用羽毛角蛋白的嗜热厌氧菌闪光杆菌,同年Santos等[11]发现了能够降解角蛋白的曲霉;1999年,Chitte等[12]得到一株能够高效降解角蛋的放线菌株嗜热链霉菌,Wang等[13]利用基因工程的手段表达了重组角蛋白酶。目前,角蛋白酶的研究主要集中在利用基因工程筛选并生产高酶活的角蛋白酶[14]。
自然界中很多菌属都含有角蛋白酶基因[15],如:耐辐射异常球菌属的小球菌(Deinococcus radiodurans R1)和戈壁异常球菌(D. gobiensis Ⅰ-0)。R1是Anderson等(1956年)在俄勒冈州经大剂量辐射灭菌仍然腐烂变质的肉类罐头中分离获得的一种微小球菌,备受生物界、环境工程界和医学界关注[16-17]。Ⅰ-0是本实验室从新疆戈壁沙漠环境中分离到的极具辐射抗性的微生物,比较基因组学分析发现,戈壁异常球菌与耐辐射异常球菌在系统进化上属于同一进化分支,两者均为异常球菌属。生物信息学分析显示,戈壁异常球菌Ⅰ-0基因组中Dgo_RS02895基因编码的蛋白与peptidases_s8_s53蛋白家族序列具有一定相似性。本研究克隆Dgker蛋白基因,并进行外源表达与纯化,探索它对羽毛的降解能力,旨在获得高比活的角蛋白酶。
1 材料与方法 1.1 材料 1.1.1 菌株及培养基野生型戈壁异常球菌(D. gobiensis Ⅰ-0)为本实验室保存;高频转化受体菌大肠杆菌E. coli Top10及表达菌株E. coli BL21(DE3)购自北京全式金生物技术有限公司;原核表达载体pET22b购自北京全式金生物技术有限公司。大肠杆菌于LB培养基(1% Typtone,0.5% Yeast extract,1% NaCl,pH 7.0,固体培养基含1.5%琼脂)中37℃培养。
1.1.2 主要试剂常规质粒提取试剂盒、凝胶回收试剂盒购自Magen公司;BSA、限制性内切酶购于New England Biolabs公司;PrimeSTAR HSDNA聚合酶、Taq DNA聚合酶、T4 DNA连接酶、2k plusⅡmarker购自北京全式金生物技术有限公司;Western blot化学发光试剂购自碧云天生物技术有限公司。引物合成和基因测序均由华大基因完成。
1.2 方法 1.2.1 Dgker蛋白生物信息学分析从NCBI中获取戈壁异常球菌Dgo_RSO2895基因的序列及所编蛋白的氨基酸序列信息。通过Compute pI/Mw tool(http://web.expasy.org/compute_pi/)对戈壁菌角蛋白酶蛋白的分子质量和等电点进行预测;使用http://bioinf.cs.ucl.ac.uk/psipred/网站在线预Dgker蛋白的二级结构,通过SignalP 4.1 Server(http://www.cbs.dtu.dk/Services/SignalP/)预测信号肽;利用TMHMMServer v. 2. 0(http://www.cbs.dtu.dk/services/TM-HMM/)预测跨膜结构域。
1.2.2 Dgker重组E.Coli菌株的构建根据目的基因Dgker的序列及表达载体pET-22b(+)的多克隆位点,利用Oligo 7软件设计基因扩增特异性引物Dgker-F(5′-ACCGGATCCGATGAACGGAGTCTTACCCT-3′,下划线处为BamH Ⅰ酶切位点)和Dgker-R(5′-ACCCTCGAGGAAGTTCAGGGTGTACAGCA-3′,下划线处为Xho Ⅰ酶切位点),引物序列由北京擎科生物股份有限公司合成。
采用试剂盒提取戈壁异常球菌Ⅰ-0基因组DNA,测定其浓度及纯度后,以此为模板,用已合成的引物Dgker-F和Dgker-R进行PCR扩增。将PCR产物用1%琼脂糖凝胶电泳分离检测。将目的条带胶块用凝胶回收试剂盒回收。PCR扩增体系(50 μL):5×PrimeSTAR Buffer(Mg2+ Plus)10 μL;2.5 mmol/L dNTP Mixture 4 μL;10 μmol/L上、下游引物各2 μL;200 ng/μL DNA模板2 μL;2.5 U/μL PrimeSTAR HSDNA聚合酶0.5 μL;加ddH2O至总体积50 μL。PCR反应条件:95℃变性5 min;95℃变性30 s,55℃退火35 s,72℃延伸1.5 min,共30个循环;72℃延伸5 min。
将上步回收获得的PCR产物与载体pET-22b质粒分别用限制性内切酶BamH Ⅰ和Xho Ⅰ 37℃酶切3 h,用1%琼脂糖凝胶电泳分离酶切产物并回收目的基因及载体的DNA片段,随后用T4 DNA Ligase连接,转化E. coli top10感受态细胞,获得阳性克隆菌落。运用质粒提取试剂盒提取质粒,随后进行双酶切鉴定,将验证正确的质粒交由北京六合华大基因科技有限公司测序。将测序正确的重组质粒,转化表达菌株BL21(DE3)获得重组菌株BL21(pET22b-Dgker)。
1.2.3 重组E.coli菌株诱导表达将实验的重组菌株以及对照菌株在LB平板上分别划线过夜培养,从活化的平板上分离单菌落,接种于20 mL含有氨苄抗生素的LB培养基,过夜培养作为母液,将母液以1%的接种量加入到100 mL含有氨苄抗生素的LB培养基配养2 h左右OD600达到0.6-0.8加IPTG使终浓度为0.3 mmol/L诱导,37℃诱导4 h。
1.2.4 重组E.coli菌株功能试验将实验的重组菌株以及对照菌株在LB平板上分别划线过夜培养,从活化的平板上挑取单菌落,接种于20 mL含有氨苄抗生素的LB培养基过夜培养作为母液。将母液以1%的接种量加入到10 mL含有氨苄抗生素的羽毛培养基中37℃ 220 r/min培养,同时把菌液点在含脱脂奶粉的平板上37℃培养,观察有无降解圈以及降解圈的变化。
1.2.5 羽毛粉为底物酶活力的测定参照蔡成岗[18]测定角蛋白酶酶活方法,略有改动:实验组10 mg羽毛粉与2 mL Tris-HCl(50 mmol/L pH8.5)混匀,加入1 mL粗酶液30℃反应1 h后加入2 mL10%三氯乙酸(TCA)终止反应;冰浴10 min,10 000 r/min离心10 min,取上清紫外分光光度计测定280 nm处吸光值。空白组10 mg羽毛粉与2 mLTris-HCl(50 mmol/L pH8.5)混匀,加入1 mL粗酶液和2 mL10%三氯乙酸(TCA)30℃反应1 h后终止反应;冰浴10 min,10 000 r/min离心10 min,取上清紫外分光光度计测定280 nm处吸光值。
1.2.6 角蛋白酶最适温度和最适pH的测定用pH8.5的50 mmol/L Tris-HCl缓冲液混合底物,在不同温度(20-80℃)下摇床振荡反应1 h测Dgker的酶活力,由结果绘制最适温度曲线。以pH4.0-11.0的缓冲液混合羽毛粉底物,在60℃水浴反应1 h测Dgker的酶活力,根据测定结果绘制Dgker的最适pH曲线。
2 结果 2.1 Dgker蛋白生物信息学分析通过分析戈壁异常球菌基因组发现,Dgo_RS02895位于D. gobiensis Ⅰ-0染色体上,Dgker由412个氨基酸组成,分子量为41.17 kD,等电点(pI)为6.07。在线预测Dgker蛋白的二级结构,多以α螺旋和卷曲结构存在,对该蛋白和来自B.licheniformis PWD-1的Ker A蛋白保守性分析,结果显示前50左右的氨基酸(图 1-A红色框内)与跨膜结构有关,成熟蛋白区(图 1-A红色括号内)有3个主要氨基酸分别是171位的天冬氨酸、203位的组氨酸、358位的丝氨酸。氨基酸序列预测图表明该蛋白前端有一段序列形成前导肽(图 1-B),跨膜结构分析结果还显示该蛋白存在跨膜结构(图 1-C),另外对该蛋白的三级结构进行模拟(图 1-D),结果显示前50左右的氨基酸并没有参与主结构的形成,主结构是从第51个氨基酸开始和模型相似。

|
| 图 1 Dgker序列分析和结构预测 A:Dgker和来自B.licheniformis PWD-1的Ker A蛋白保守性分析;B:Dgker蛋白的序列分析;C:Dgker蛋白的跨膜结构分析:红色部分为跨膜结构区域;D:Dgker蛋白的三级结构序预测 |
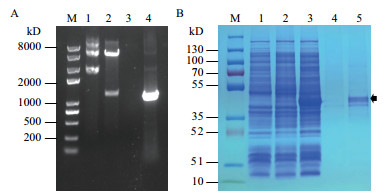
以提取的耐辐射异常球菌基因组DNA为模板PCR扩增目的基因,获得的条带大小与预期一致约为1 239 bp(图 2-A)。将回收产物连接pET-22b后转化到E. coli TOP10获得pET-22b-Dgker重组菌株,对重组菌株提取质粒进行双酶切验证(图 2-A),重组质粒8 000 bp左右,双酶切得到一条1 300 bp左右的目的条带和一条大于5 000 bp的载体片段。同时送公司进行测序,结果证明目的基因已经成功连入表达载体。
|
| 图 2 Dgker验证及纯化图 A:M:DNA marker(2k plus Ⅱ),1:重组质粒,2:双酶切产物,3:PCR对照,4:pcr产物.B:M:蛋白Marker,1:空载全蛋白,2:重组菌株未诱导全蛋白,3:重组菌株诱导全蛋白,4:洗脱液,5:含目的蛋白的洗脱液,黑色箭头所指为目的蛋白 |
对Dgker蛋白进行诱导表达,经亲和层析过柱纯化,获得纯化后的蛋白。SDS-PAGE分析结果(图 2-B)显示,以仅含空载体pET22b的大肠杆菌和未诱导的重组菌为对照,0.3 mmol/L IPTG诱导下出现1条与Dgker蛋白大小(41.1 kD)吻合的条带(图 2-B中黑色箭头所指)。
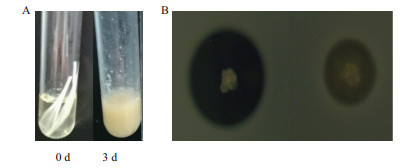
2.3 Dgker降解羽毛实验以OD 0.1转接种子液于加入羽毛的LB培养基中培养,观察记录到重组菌株在3 d左右的时间里把羽毛分解(图 3-A),同时在奶粉培养基上诱导表达获得的重组菌株粗酶液、空载菌株粗酶液出现降解圈的时间同步(图 3-B),对比可知含角蛋白基因的重组菌株粗酶液降解能力比空载菌株强,说明角蛋白基因有作用。
|
| 图 3 Dgker降解羽毛图 A:左:加入羽毛0 d,右:加入羽毛3 d;B:左:重组菌株粗酶液、降解圈半径比5.3,右:空载菌株粗酶液、降解圈半径比2.8 |
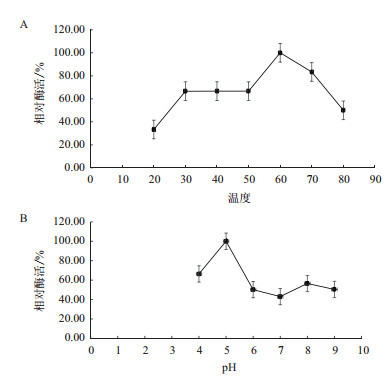
用pH8.5的缓冲液,分别在20、30、40、50、60、70和80℃下,测定Dgker酶活力。如图 (4-A)所示,Dgker的最适温度是60℃,当温度从20℃上升到60℃时,Dgker的酶活随之有上升的趋势,60℃到80℃酶活随温度升高而降低;温度在30-50℃之间酶活比较稳定,有利于在工业上应用推广。在60℃条件下,分别在pH为4.0、5.0、6.0、7.0、8.0和9.0的缓冲液测定Dgker酶活力。由图 4-B可知,Dgker的最适pH是5.0,pH从4.0上升到5.0时,酶活有个上升的过程,pH5.0-pH9.0酶活呈变化状态总趋势是下降。
|
| 图 4 Dgker在不同温度、pH下的酶活图 A:不同温度下Dgker酶活百分比;B:不同pH下Dgker酶活百分比 |
Dgker蛋白是相对分子量在30-50 kD的酶类,对羽毛有一定的分解作用、对头发和蹄分解作用不明显。本研究通过生物信息学分析发现Dgker蛋白分子量为41.1 kD与耐辐射异常球菌的蛋白亲缘关系最近且与同属蛋白具有较高的序列相似性,是一个异常球属特异性蛋白。异常球属微生物可以在极端环境中生存,其拥有非凡的适应机制以抵抗极端环境造成的损伤。Dgker来源于戈壁异常球菌,该菌分离于戈壁沙漠,推测其可能更耐酸碱及高温,对羽毛处理有重要作用。
目前为止,有关角蛋白酶的研究多集中在筛选新菌株[19-20]、发酵和处理条件优化[21-22]、基因工程等方面,研究表明角蛋白酶能破坏角蛋白中的二硫键[23]和肽键,可以在动物饲料加工和皮革制品鞣制过程中发挥重要作用[24]。本研究从戈壁异常球菌中克隆了一个新的角蛋白酶编码基因,氨基酸序列前段有前导肽与Wu等[15]在文章中分析的一致,并且在相似的位置形成跨膜结构域,此结构的存在对于蛋白的纯化有一定的影响。模拟Dgker的三维结构时还发现这个区域的氨基酸没有参与主结构的模拟,在后续实验中可以考虑把它们突变掉观察酶活性变化,用来提高生产效率。对Dgker和kerA蛋白保守性分析结果显示有很相似的区域如前导肽、成熟蛋白区都没有尾巴结构(C-terminal pro-peptide[15]),成熟蛋白区都有3个主要的氨基酸(Asp、His、Ser)在水解角蛋白时发挥作用。通过大肠杆菌原核表达系统进行表达并纯化后的蛋白经检测具有角蛋白水解活性,该菌株以后可以用于处理废弃羽毛等,为进一步发掘工业用角蛋白酶提供了基因资源。实验得出Dgker粗酶液酶活最适温度是60℃,此温度比B.licheniformis PWD-1生长温度45-50℃[7]更耐高温,该蛋白又是在大肠杆菌中表达方便培养利于生产。Dgker的最适pH是5.0与已知的大多数蛋白酶最适的值在7.5-10之间相比该蛋白更耐酸有利于生产成本的降低。该基因编码的前50个氨基酸对Dgker的影响将在接下来的工作中进行探索[25],同时将进一步研究Dgker在工业上的应用前景。
4 结论本研究确定了戈壁异常球菌(D. gobiensis Ⅰ-0)中Dgo_RS02895基因编码的蛋白与peptidases_s8_s53家族蛋白相似,是角蛋白酶的一种,命名为Dgker。成功构建了表达Dgker的重组菌株E. coli pET22b-Dgker/BL21,降解羽毛实验表明Dgker有降解羽毛的功能。粗蛋白的酶活测定结果表明Dgker酶活最适温度在60℃,最适pH是5.0。
| [1] |
陈莹, 王宇新. 角蛋白及其提取[J]. 材料导报, 2002, 16(12): 65-67. DOI:10.3321/j.issn:1005-023X.2002.12.020 |
| [2] |
庄媛, 周美华, 赵晓祥. 羽毛角蛋白的提取及其应用进展[J]. 环境科学与技术, 2013(3): 65-69. |
| [3] |
贾如琰, 何玉凤, 王荣民, 等. 角蛋白的分子构成、提取及应用[J]. 化学通报, 2008, 7(4): 265-271. |
| [4] |
赵玲, 周华龙, 华坚. 羽毛角蛋白的资源化利用新进展[J]. 中国皮革, 2011(5): 36-40. |
| [5] |
Dettmer A, Anjos PSD, Gutterres M, et al.杜卫宁编译.酶在皮革工业中的应用综述[J].西部皮革, 2013(22): 40-47. http://www.cnki.com.cn/Article/CJFDTotal-XBPG201322016.htm
|
| [6] |
Jain PC, Agrawal SC. A note on the keratin decomposing capability of some fungi[J]. Transactions of the Mycological Society of Jzpan, 1980, 21(4): 513-517. |
| [7] |
Williams CM, Richter CS, Mackenzie JM, et al. Isolation, identification and characterization of a feather-degrading bacterium[J]. Appl Environ Microbiol, 1990, 56(6): 1509-1515. |
| [8] |
Atalo K, Gashe BA. Protease production by a thermophilic Bacillus species(P-001A)which degrades various kinds of fibrous proteins[J]. Biotechnology Letters, 1993, 15(11): 1151-1156. DOI:10.1007/BF00131207 |
| [9] |
丁正民, 冯缵平, 刘达先, 等. -株分解鸡毛角蛋白的放线菌[J]. 微生物学报, 1993, 3: 227-232. |
| [10] |
Friedrich AB, Antranikian G. Keratin degradation by Fervidobacterium pennavorans, a novel thermophilic anaerobic species of the order thermotogales[J]. Appl environ Microbiol, 1996, 62(8): 2875-2882. |
| [11] |
Santos R, Finnino AaP, Desa CM, et al. Keratinolytic activity of Aspergillus fumigates fresenius[J]. Current Microbiology, 1996, 33(6): 364-370. DOI:10.1007/s002849900129 |
| [12] |
Chitte RR, Nalawade VK, et al. Keratinolytic activity from the broth of a feather-degrading thermophilic Streptomyces thermoviolaceus strain SD8[J]. Lett Appl Microbiol, 2010, 28(2): 131-136. |
| [13] |
Wang JJ, Shih JCH. Fermentation production of keratinase from Bacillus licheniformis PWD-1 and a recombinant B. subtilis, FDB-29[J]. J Ind Microbiol Biotechnol, 1999, 22(6): 608-616. DOI:10.1038/sj.jim.2900667 |
| [14] |
王秋影, 廖美德. 角蛋白酶的研究进展[J]. 中国饲料, 2012(22): 12-16. DOI:10.3969/j.issn.1004-3314.2012.22.005 |
| [15] |
Wu WL, Chen MY, Tu I, et al. The discovery of novel heat-stable keratinases from Meiothermus taiwanensis WR-220 and other extremophiles[J]. Scientific Reports, 2017, 7(1): 1-12. DOI:10.1038/s41598-016-0028-x |
| [16] |
Brim HS, McFarland SC, Fredrickson JK, et al. Deihococcu radiodurah for metal remediation in radioactive Engineering mixed waste environments[J]. Nature Biotechnology, 2000, 18: 85-90. DOI:10.1038/71986 |
| [17] |
Wood RD, Mitchell M, Sgouros J, et al. Human DNA repair genes[J]. Science, 2001, 291: 1284-289. DOI:10.1126/science.1056154 |
| [18] |
蔡成岗.角蛋白酶生产菌株选育, 发酵与分离纯化研究[D].杭州: 浙江大学, 2007.
|
| [19] |
Pereira JQ, Lopes FC, Petry MV, et al. Isolation of three novel Antarctic psychrotolerant feather-degrading bacteria and partial purification of keratinolytic enzyme from Lysobacter sp. A03[J]. International Biodeterioration & Biodegradation, 2014, 88(1): 1-7. |
| [20] |
Oliveira CTD, Pellenz L, Pereira JQ, et al. Screening of bacteria for protease production and feather degradation[J]. Waste & Biomass Valorization, 2016, 7(3): 447-453. |
| [21] |
Osman Y, Elsayed A, Mowafy AM, et al. Bioprocess enhancement of feather degradation using alkaliphilic microbial mixture[J]. British Poultry Science, 2017, 58(3): 319-328. DOI:10.1080/00071668.2017.1278627 |
| [22] |
Maciel JL, Werlang PO, et al. Characterization of protein-rich hydrolysates produced through microbial conversion of waste feathers[J]. Waste & Biomass Valorization, 2017, 8(4): 1-10. |
| [23] |
齐志国, 张铁鹰, 董杰丽, 等. 微生物角蛋白酶的研究进展及其在饲料中的应用前景[J]. 中国饲料, 2012(15): 31-35. DOI:10.3969/j.issn.1004-3314.2012.15.011 |
| [24] |
孙国龙, 陈丽, 黄金, 等. 酸性蛋白酶在皮革鞣后湿处理中的应用[J]. 西部皮革, 2013, 35(14): 19-22. DOI:10.3969/j.issn.1671-1602.2013.14.006 |
| [25] |
Fang Z, Zhang J, Liu B, et al. Enhancement of the catalytic efficiency and thermostability of Stenotrophomonas sp. keratinase KerSMD by domain exchange with KerSMF[J]. Microbial Biotechnology, 2016, 9(1): 35-46. |




