2. 乌鲁木齐市动物疾病控制与诊断中心,乌鲁木齐 830063;
3. 新疆畜牧科学院兽医研究所,乌鲁木齐 830000;
4. 中国农业科学院兰州兽医研究所,兰州 730046
2. Animal Disease Control and Diagnosis Center in Urumqi, Urumqi 830063;
3. Institute of Veterinary Medicine, Xinjiang Academy of Animal Science, Urumqi 830000;
4. Lanzhou Veterinary Research Institute, Chinese Academy of Agricultural Sciences, Lanzhou 730046
单核细胞增生李斯特菌(Listeria monocytogenes,LM)是一种革兰氏阳性人兽共患胞内寄生菌[1],能引起人和动物的李斯特菌病。作为重要的食源性致病菌之一[2],LM广泛存在于自然环境中,可在高盐、低温和低pH值条件下生存,严重威胁食品安全和公共卫生[3-5]。同时,LM也是一种最常见的腐生菌,易污染动物饲草料,给畜牧业的发展造成严重的损失[6]。研究发现,LM的环境耐受能力和致病性与其基因组上编码的多种应激因子(VirR、Sigma B)和调控因子(PrfA、CodY)有着密切的关系[7],而非编码RNAs(non-coding RNAs,ncRNAs)与这些蛋白分子组成了复杂的调控网络,对LM毒力和环境应激相关基因在转录、转录后和翻译水平进行精细调控[8]。
RibonucleaseⅢ(RnaseⅢ)是一种保守性较高的双链RNA特异性核酸内切酶,其中细菌RNase Ⅲ主要在rRNA成熟,mRNA降解和sRNA加工、转换和sRNA依赖性mRNA降解中起作用[9-10]。前期研究证实[11],LM RnaseⅢ是一种依赖于二价金属阳离子的磷酸二酯酶,当镁离子存在时,该酶可发挥降解双链结构的RNA的活性,且随镁离子浓度不同其降解活性不同。然而,LM RnaseⅢ发挥酶活中的关键氨基酸位点至今尚未报道。鉴于此,本研究利用SOE-PCR技术构建LM- RnaseⅢ-D50A、LM- RnaseⅢ-E122A突变体,通过体外酶活试验检测其对RNA降解活性的变化,为深入揭示LM RnaseⅢ的作用特点和调控ncRNAs的分子机制奠定前期基础。
1 材料与方法 1.1 材料LM-SB5野毒株由石河子大学动物疾病防控兵团重点实验室保存;大肠杆菌DH5α、BL21(DE3)和pET-32a(+)原核表达载体由本实验室保存;重组RnaseⅢ RncS由本实验室前期构建保存;Taq DNA聚合酶、dNTPs、pMD19-T载体、限制性核酸内切酶EcoRⅠ、XhoⅠ、T4 DNA Ligase、DL-2 000 DNA Marker、DL-5 000 DNA Marker、蛋白Marker、Trizol购自TaKaRa公司;MgCl2和MnCl2均购自北京北化精细化学品有限责任公司;兔抗LM阳性血清由本室制备;细菌基因组DNA提取试剂盒、质粒小量提取试剂盒、琼脂糖凝胶回收试剂盒、辣根过氧化物酶标记的山羊抗兔IgG、低背景化学发光检测试剂盒购自TIANGEN公司。
1.2 方法 1.2.1 引物设计根据前期扩增的rncS基因序列及生物信息学分析结果,通过Primer 5.0软件设计扩增rncS基因突变型D50A、E122A的特异性引物:D50A、E122A fragment1的扩增引物分别为H1、H2和R1、R2;D50A、E122A fragment1的扩增引物分别为H3、H4和R3、R4;重叠延伸PCR的引物分别为H1、H4和R1、R4;分析表达载体pET-32a(+)多克隆位点的情况,在H1、H4和R1、R4引物的5’端分别加酶切位点EcoRⅠ和XhoⅠ(下划线)及保护性碱基(斜体)(表 1)。引物由北京六合华大基因生物公司合成。
应用在线生物信息学分析软件SMART(http://smart.embl-heidelberg.de/smart/)预测RnaseⅢ RncS蛋白结构域;在NCBI中检索大肠杆菌、金黄色葡萄球菌、嗜热杆菌、布鲁氏菌等的RnaseⅢ氨基酸序列,利用DNAMAN 5.2.2软件进行多序列比对,选择高度保守的关键氨基酸进行突变。
1.2.3 LM rncS突变基因D50A、E122A的构建与测序鉴定提取LM-SB5菌株基因组DNA,以基因组DNA为模板,用H1/H2和H3/H4两对引物分别扩增D50A fragment1和fragment2,用R1/R2和R3/R4两对引物分别扩增E122A fragment1和fragment2。SOE-PCR采用25 μL反应体系:水14.8 μL,fragment1和fragment2各3 μL,10 × PCR buffer 2.5 μL,2.5 mmol/Lol/L dNTPs 1.5 μL,TaqDNA聚合酶0.2 μL。PCR反应条件:94℃预变性5 min;94℃变性45 s,55℃退火40 s,72℃延伸45 s,13个循环;再加入H1/H4和R1/R4引物各0.5 μL扩增片段的全长,反应条件:95℃预变性4min;95℃变性50 s,65℃退火40 s,72℃延伸1 min 40 s,30个循环;72℃延伸l0 min。经1%琼脂糖凝胶电泳鉴定后回收目的片段,并将目的片段克隆至pMD19-T Simple载体中,挑取阳性单克隆进行测序,构建突变质粒pMD19-T-D50A和pMD19-T-E122A。
1.2.4 突变型重组表达载体的构建与鉴定将pET-32a(+)原核表达载体与测序正确的pMD19-T-D50A和pMD19-T-E122A质粒用限制性核酸内切酶EcoRⅠ和XhoⅠ同时进行双酶切。回收目的片段和载体片段后用T4 DNA连接酶连接,构建突变型重组表达载体。经菌液PCR和双酶切鉴定,将鉴定正确的突变型重组表达质粒命名为pET32a(+)-D50A和pET32a(+)-E122A。
1.2.5 突变型重组蛋白的诱导表达、纯化及Western blot鉴定将鉴定正确的突变型重组表达质粒转化至大肠杆菌BL21(DE3)中,在Amp抗性的液体LB培养基中37℃培养至OD600 nm为0.6-0.7,用IPTG进行诱导8 h,同时设置空载体对照,然后进行SDS-PAGE分析,利用镍离子亲和层析法纯化突变型重组蛋白,同时,以兔抗LM阳性血清为一抗,辣根过氧化物酶标记的山羊抗兔IgG为二抗,进行Western blot分析。
1.2.6 突变型重组RnaseⅢ D50A和RnaseⅢ E122A对RNA降解活性研究提取LM总RNA,分析LM重组RnaseⅢ RncS突变对RNA降解活性的影响。设立RNA的空白对照和未突变的重组RnaseⅢ RncS对照,同时,设立不同浓度的MgCl2和MnCl2,采用20 μL反应体系:RNA 7 μL、RnaseⅢ RncS、RnaseⅢ D50A和RnaseⅢ E122A均3 μL、MgCl2(MnCl2)X,加Urtrapure water 10-X μL。将纯化好的重组RnaseⅢ RncS及突变型重组LM RnaseⅢ D50A和LM RnaseⅢ E122A适当稀释,在不同浓度MgCl2(MnCl2),37℃条件下水浴15 min,反应产物经琼脂糖凝胶电泳后用凝胶成像系统观察,定性地分析LM重组RnaseⅢ RncS突变对RNA降解活性的变化。同时,选取最佳浓度的MgCl2和MnCl2,按上述操作步骤,37℃条件下水浴15 min后分别测定各组的RNA浓度,并利用统计学方法定量分析LM重组RnaseⅢ RncS突变对RNA降解活性的变化。
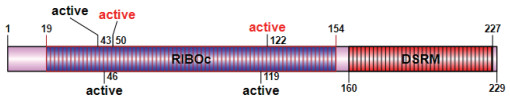
2 结果 2.1 LM rncS结构域分析及突变位点的选择SMART软件分析结果显示,LM-RnaseⅢ蛋白氨基酸序列含有1个核酸酶结构域(RIBOc,19-154)和1个双链RNA结合结构域(DSRM,160-227),其中核酸酶结构域RIBOc中有含有5个活性位点(active,43、46、50、119、122)(图 1)。通过DNAMAN多序列比对发现,这5个活性位点高度保守。
|
| 图 1 LM-RnaseⅢ RncS蛋白结构域模式图 |
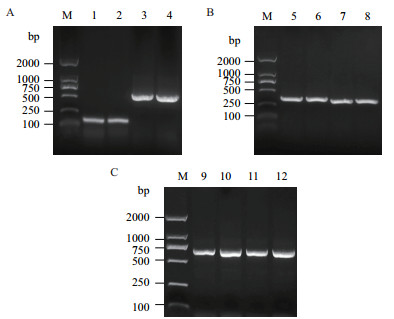
以LM基因组DNA为模板,用H1/H2、H3/H4及R1/R2、R3/R4特异性引物分别扩增突变型D50A、E122A的fragment1和fragment2,分别扩增得到161 bp、529 bp、374 bp、316 bp的目的条带,与预期结果一致。采用重叠延伸PCR技术融合fragment1和fragment2,得到与预期值690 bp相符的目的条带(图 2);阳性克隆pMD19-T-D50A和pMD19-T-E122A测序后的对比分析表明D50A、E122A已成功突变。
|
| 图 2 rncS基因突变株的克隆结果 M:DNA分子质量标准;1-2、5-6:突变型D50A、E122A的fragment1 PCR扩增产物;3-4、7-8:突变型D50A、E122A的fragment1 PCR扩增产物;9-10:突变型D50A的SOE-PCR扩增产物;11-12:突变型E122A的SOE-PCR扩增产物 |
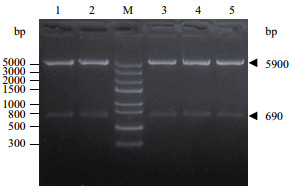
突变型重组质粒pET-32a(+)-D50A和pET-32a(+)-E122A用EcoR I/Xho I双酶切得到均为690 bp的目的条带和5 900 bp的pET-32a(+)片段(图 3),表明成功构建了pET-32a(+)-D50A和pET-32a(+)-E122A突变型重组原核表达载体。
|
| 图 3 rncS基因突变型重组质粒双酶切鉴定 |
将构建好的突变型重组表达载体pET-32a(+)-D50A和pET-32a(+)-E122A转化至大肠杆菌BL21(DE3)感受态细胞,筛选阳性菌,经IPTG诱导表达8 h后,通过SDS-PAGE分析,突变型重组菌pET-32a(+)-D50A和pET-32a(+)-E122A均出现了分子质量为42.5 kD的特异性蛋白条带。纯化蛋白经SDS-PAGE分析显示,在相对分子质量约42.5 kD处可见单一条带,大小与预期值一致,结果表明纯化的蛋白效果较好。经Western blot分析鉴定,该突变型重组蛋白能与兔抗LM阳性血清发生特异性反应,表明克隆的LM rncS基因突变型D50A、E122A在大肠杆菌中获得表达(图 4)。

|
| 图 4 突变型重组蛋白SDS-PAGE和Western blot分析 M1、M2:蛋白分子质量标准;1、7:1:pET-32a(+)质粒经IPTG诱导8 h的表达产物;2-3:pET-32a(+)-D50A和pET-32a(+)-E122A质粒经IPTG诱导8h的表达产物;4-5:纯化的重组蛋白RnaseⅢ-D50A和RnaseⅢ-E122A;6、8:重组蛋白RnaseⅢ-D50A和RnaseⅢ-E122A与LM阳性血清反应 |
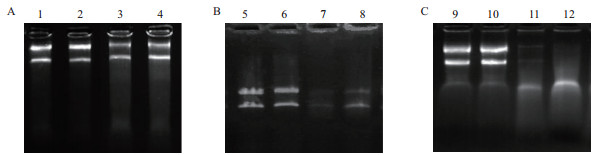
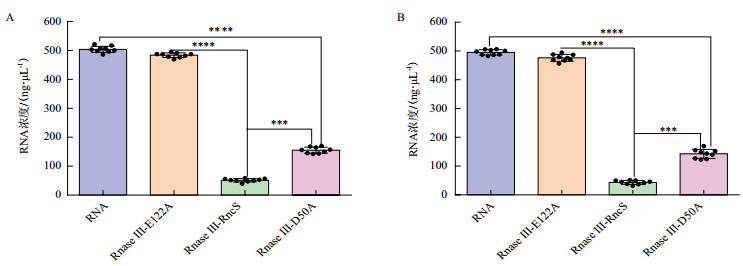
结果(图 5、图 6)显示,当缺乏Mn2+时,RnaseⅢ不能降解RNA,在低浓度的Mn2+和Mg2+条件下,RnaseⅢ的降解活性都较低,随着离子浓度的增大,其降解活性也升高,其中Mn2+浓度为50 mmol/L,Mg2+浓度为10 mmol/L时,RnaseⅢ具有较强的降解活性,但当Mn2+浓度大于100 mmol/L时,其降解活性减弱;在Mn2+浓度为50 mmol/L,Mg2+浓度为10 mmol/L的条件下,与对照组相比,将RnaseⅢ RncS第50位的天冬氨酸突变后,其降解活性减弱(P < 0.001),第122位的谷氨酸突变后降解活性显著下降(P < 0.000 1)(图 7),表明RnaseⅢ发挥降解活性依赖于Mn2+或Mg2+,其中第122位的谷氨酸是维持RnaseⅢ RncS活性的关键位点。

|
| 图 5 Mn2+对RnaseⅢ及其突变型降解活性的检测 A:不同Mn2+对RnaseⅢ的降解活性的影响;1、3-7:Mn2+浓度依次为0 mmol/L、1.25 mmol/L、7.5 mmol/L、5 mmol/L、50 mmol/L、100 mmol/L;B、C:分别为1.25 mmol/L、50 mmol/L的Mn2+对不同RnaseⅢ的降解活性;2、8、13:RNA对照;9、12:Rnase Ⅲ E122A;10、15:RnaseⅢ-D50A;11、14:RnaseⅢ-RncS |
|
| 图 6 Mg2+对RnaseⅢ及其突变型降解活性的检测 A、B、C:分别为1.25 mmol/L、7.5 mmol/L、10 mmol/L的Mg2+对不同RnaseⅢ的降解活性;2、6、10:RNA对照;3、7、12:RnaseⅢ-RncS;1、5、9:RnaseⅢ-E122A;4、8、11:RnaseⅢ-D50A |
|
| 图 7 RnaseⅢ及其突变型降解活性的检测 A:50 mmol/L的Mn2+;B:10 mmol/L的Mg2+;***表示P < 0.001,****表示P < 0.0001 |
RnaseⅢ是一种广泛存在于细菌、真核生物及噬菌体中的能够切割具有双链结构RNA的核酸酶,Wanger[12]和Darfeuille[13]等在大肠杆菌和沙门氏菌中研究证实,RnaseⅢ均参与了ncRNA的调控,影响基因的表达,从而调控细菌在宿主体内的生存。研究发现,ncRNA在LM毒力和环境应激适应过程具有重要的作用[14],然而,目前有关RnaseⅢ介导的ncRNA对其调控作用尚未研究。
序列比对发现,不同来源的细菌RNase Ⅲ是一种保守性较高的酶家族,均含有9个保守氨基酸(ERLEFLGDA)组成的RNase Ⅲ识别基序,是维持其酶降解活性的重要位点。RNase Ⅲ起同源二聚体的作用,通过核糖核酸酶结构域发生二聚化,两个核糖核酸酶结构域组合形成单个加工中心,每个结构域有助于双链底物的一条RNA链的水解[15]。Blaszczyk等[16]在嗜热杆菌(Aquifex aeolicus)Aa-RNase Ⅲ与dsRNA复合体晶体结构中研究发现,两个RNase Ⅲ单体通过NucD区的疏水作用和氢键结合形成二聚体结构,在两个单体作用界面形成一个催化谷(valley)结构用于结合RNA。因为单体蛋白靠近,来自同一亚基的五个酸性残基E38、E41、D45、D114和E117和一个来自伴侣亚基的E65簇位于催化谷的两端组成一个活性中心。在大肠杆菌中研究发现,E38参与蛋白质二聚化;E65参与底物识别和裂变键选择[15];D45、D114和D117螯合Mn2+离子[17];E41、D45、D114和E117进行易断裂键的水解[18]。本研究通过生物信息学分析发现,LM-RNaseⅢ含有1个核酸内切酶结构域(RIBOc)和1个dsRNA结合结构域(DSRM),且核酸酶结构域RIBOc中的5个活性位点在其它细菌RNaseⅢ中高度保守。
目前,细菌RNaseⅢ蛋白是研究最广泛的一类磷酸二酯酶[19]。Sun等[17]研究发现大肠杆菌RNaseⅢ发挥降解活性依赖于Mn2+或Mg2+。另外,Blaszczyk等[20]在嗜热杆菌中研究发现,Mn2+或Mg2+离子为RNase Ⅲ发挥活性所必须;当其第110位的E突变为K后,Aa-RNase Ⅲ-E110K能与dsRNA结合,但失去切割活性。Lioliou等[21]将金黄色葡萄球菌RNase Ⅲ中的残基E135和D63均突变成丙氨酸后,证实Sa-RNase Ⅲ-D63A和Sa-RNase Ⅲ-E135A降解活性显著降低。许先进[22]将布鲁氏菌RNase Ⅲ中的E54、D61、E81、E133均突变成丙氨酸后,证实这四种突变蛋白均能与布鲁氏菌自身编码的dsRNA BM-pre-0015结合,但RNaseⅢ-E133A的结合能力和切割活性显著降低,表明E133是其发挥酶活所必须的。本研究通过体外酶活试验检测发现,不同浓度的Mn2+或Mg2+对LM-RNaseⅢ的降解活性不同,其中Mn2+为50 mmol/L、Mg2+为10 mmol/L时具体较好的降解活性。另外,构建的LM-RNase Ⅲ-E122A突变体降解活性显著降低,提示LM-RNase Ⅲ中的第122位谷氨酸是其维持酶活性的关键位点,为进一步揭示RnaseⅢ对LM ncRNA的作用方式和调控机制奠定前期基础。
4 结论本研究利用生物信息学软件分析了LM-RNaseⅢ结构域,结果显示,LM-RnaseⅢ氨基酸序列含有1个核酸酶结构域(RIBOc)和1个双链RNA结合结构域(DSRM),其中结构域RIBOc中有含有5个活性位点;应用SOE-PCR技术构建了突变型LM-RnaseⅢ-D50A和LM-RnaseⅢ-E122A,并通过体外酶活实验检测显示,RnaseⅢ发挥降解活性依赖于Mn2+或Mg2+,将其第50位天冬氨酸突变后,RnaseⅢ RncS的降解活性有所降低;第122位谷氨酸突变后,RnaseⅢ RncS降解活性显著下降,提示第122位谷氨酸是维持LM RnaseⅢ RncS酶活性的关键位点。
| [1] |
Radoshevich L, Cossart P. Listeria monocytogenes: towards a complete picture of its physiology and pathogenesis[J]. Nature Reviews Microbiology, 2018, 16(1): 32. |
| [2] |
Gandhi M, Chikindas ML. Listeria: A foodborne pathogen that knows how to survive[J]. International Journal of Food Microbiology, 2017, 113(1): 1-15. |
| [3] |
孙静娟, 邱景璇, 曾海娟, 等. 单增李斯特菌CdaA的抗原表位分析及抗体的制备[J]. 生物技术通报, 2018, 34(5): 163-171. |
| [4] |
Neuhaus K, Satorhelyi P, Schauer K, et al. Acid shock of Listeria monocytogenes at low environmental temperatures induces prfA, epithelial cell invasion, and lethality towards Caenorhabditis elegans[J]. BMC Genomics, 2013, 27(14): 285. |
| [5] |
Galié S, Garcíagutiérrez C, Miguélez EM, et al. Biofilms in the food industry: Health aspects and control methods[J]. Frontiers in Microbiology, 2018, 9: 898. DOI:10.3389/fmicb.2018.00898 |
| [6] |
Van d VS, Van SS, Molenaar D, et al. The SOS response of Listeria monocytogenes is involved in stress resistance and mutagenesis[J]. Microbiology, 2010, 156(2): 374-384. |
| [7] |
Grubaugh D, Regeimbal JR, Ghosh P, et al. The VirAB ABC transporter is required for VirR regulation of Listeria monocytogenes virulence and resistance to nisin[J]. Infection & Immol/Lunity, 2018, 86(3): IAI. 00901-17. |
| [8] |
张再超, 彭叶龙, 孟庆玲, 等. 单核细胞增生李斯特菌ncRNA研究进展[J]. 生命的化学, 2013, 53(4): 87-91. |
| [9] |
Arraiano CM, Andrade JM, Domingues S, et al. The critical role of RNA processing and degradation in the control of gene expression[J]. Fems Microbiology Reviews, 2010, 34(5): 883-923. DOI:10.1111/j.1574-6976.2010.00242.x |
| [10] |
Afonyushkin T, Večerek B, Moll I, et al. Both RNase E and RNase Ⅲ control the stability of sodB mRNA upon translational inhibition by the small regulatory RNA RyhB[J]. Nucleic Acids Research, 2005, 33(5): 1678-1689. DOI:10.1093/nar/gki313 |
| [11] |
王立霞, 孟庆玲, 蔡扩军, 等. 单核细胞增生李斯特菌核糖核酸酶RNaseⅢ RncS的表达及其生物学活性研究[J]. 中国预防兽医学报, 2018(7): 581-585. |
| [12] |
Wagner EG, Altuvia S, Romby P. Antisense RNAs in bacteria and their genetic elements[J]. Advances in Genetics, 2002, 46(3): 361-398. |
| [13] |
Darfeuille F, Unoson C, Vogel J, et al. An antisense RNA inhibits translation by competing with standby ribosomes[J]. Molecular Cell, 2007, 26(3): 381-392. |
| [14] |
Lebreton A, Cossart P. RNA-and protein-mediated control of Listeria monocytogenes virulence gene expression[J]. RNA Biol, 2017, 14(5): 460-470. DOI:10.1080/15476286.2016.1189069 |
| [15] |
Xiao J, Feehery CE, Tzertzinis G, et al. E. coli RNase Ⅲ(E38A)generates discrete-sized products from long dsRNA[J]. RNA, 2009, 15(5): 984-991. DOI:10.1261/rna.1196509 |
| [16] |
Blaszczyk J, Tropea JE, Bubunenko M, et al. Crystallographic and modeling studies of RNase Ⅲ suggest a mechanism for double-stranded RNA cleavage[J]. Structure, 2001, 9(12): 1225-1236. DOI:10.1016/S0969-2126(01)00685-2 |
| [17] |
Sun W, Nicholson AW. Mechanism of action of Escherichia coli ribonuclease Ⅲ. Stringent chemical requirement for the glutamic acid 117 side chain and Mn2+ rescue of the Glu117Asp mutant[J]. Biochemistry, 2001, 40(16): 5102-5110. DOI:10.1021/bi010022d |
| [18] |
Gan J, Tropea JE, Austin BP, et al. Structural insight into the mechanism of double-stranded RNA processing by ribonuclease Ⅲ[J]. Cell, 2006, 124(2): 355-366. DOI:10.1016/j.cell.2005.11.034 |
| [19] |
Robertson HD, Webster RE, Zinder ND. A nuclease specific for double-stranded RNA[J]. Virology, 1967, 32(4): 718-719. |
| [20] |
Blaszczyk J, Gan J, Tropea JE, et al. Noncatalytic assembly of ribonuclease Ⅲ with double-stranded RNA[J]. Structure, 2004, 12(3): 457-466. |
| [21] |
Lioliou E, Sharma CM, Caldelari I, et al. Global regulatory functions of the, Staphylococcus aureus, endoribonuclease Ⅲ in gene expression[J]. PLoS Genetics, 2012, 8(6): 3202-3212. |
| [22] |
许先进.布鲁氏菌核糖核酸酶RibonucleaseⅢ的酶活性研究[D].武汉: 华中农业大学, 2014.
|





