2. 福建农林大学福建省农业生态过程与安全监控重点实验室,福州 350002
2. Fujian Provincial Key Laboratory of Agroecological Processing and Safety Monitoring, Fujian Agriculture and Forestry University, Fuzhou 350002
大肠杆菌(Escherichia coli)是广泛存在于人体和动物肠道中的一种革兰氏阴性菌,也是临床感染中最为常见的人畜共患病原菌,可引起严重腹泻和败血症[1]。使用抗生素是应对致病性大肠杆菌感染的有效策略,但是由于抗生素使用广泛且使用不当,导致当今细菌的耐药性问题日益严峻,耐药菌株不断增多,并且出现多重耐药菌株[2]。根据国家卫生部细菌耐药监测网显示,大肠杆菌对一些药物的耐药率已超过50%,对全球公共健康构成严重威胁[3]。
在防治大肠杆菌感染的药物中,最常见的是喹诺酮类抗生素。巴洛沙星(Balofloxacin)是一种新型的第四代喹诺酮类药物,可以用于治疗呼吸道感染和泌尿道感染,具有广谱抗菌活性,特别是对革兰氏阴性菌的抗菌效果较好[4]。但是当前已有越来越多的研究报道发现对巴洛沙星耐药的临床菌株出现,使得细菌耐药的形势更加严峻[5]。为了进一步探究大肠杆菌应对巴洛沙星的分子机制,本研究以模式菌株大肠杆菌为研究对象,利用iTRAQ标记结合高分辨质谱鉴定的定量蛋白质组学方法,比较大肠杆菌K12在巴洛沙星胁迫下,蛋白质水平的表达变化,以期发现细菌应对抗生素的分子机制,为细菌耐药机制的研究提供理论依据。
1 材料与方法 1.1 材料本研究中使用的菌株为大肠杆菌(Escherichia coli)K12 BW25113,该菌为常见工程菌,由中山大学彭宣宪教授惠赠。
1.2 方法 1.2.1 MIC值及生长曲线的测定采用二倍稀释法,用LB培养基将巴洛沙星进行梯度稀释,使其浓度为5、2.5、1.25、0.625、0.313、0.156、0.078 g/mL,将活化过夜的菌液进行稀释,并分别加入含有巴洛沙星LB培养基中,不含抗生素的LB培养基作为阴性对照[6]。然后于37℃静置培养16 h,记录MIC值,并使用酶标仪测定各浓度的OD600nm。根据测得的MIC值,使用全自动生长曲线分析仪测定大肠杆菌在不同浓度巴洛沙星下的生长情况,浓度梯度为1/8MIC、1/4MIC、1/2MIC、1×MIC和2×MIC,以及空白对照,每组重复3次,于37℃每隔1 h测定并记录OD600nm,连续测定12 h,使用Prism 5软件绘制图。
1.2.2 细菌培养将大肠杆菌转接入LB培养基中,置于37℃摇床,以200 r/min条件下过夜培养,然后将菌株按1:100稀释到含有1/4MIC巴洛沙星的新鲜LB培养基中培养至OD600nm=1.0,该处理组为实验组,同时将未经抗生素处理的稀释液作为对照组。
1.2.3 全菌蛋白的提取在5 000 ×g转速下将培养好的菌株离心10 min,用生理盐水洗涤两次,接着用5 mL含有PMSF的PBS重悬细菌,之后进行超声破碎。在15 000 ×g转速下离心30 min清除碎片,并将上清液用20 % TCA在冰上沉淀1 h,用预冷丙酮在15 000 ×g、4℃,离心10 min洗涤2次。用200 μL裂解液(8 mol/L尿素、0.1%SDS、2%Triton X-100溶解在pH8.5的0.5 mol/L三乙基碳酸氢铵缓冲液(TEAB))重悬沉淀得到蛋白样品。最后利用BCA方法测定蛋白质浓度并将蛋白质分装保存于-80℃。
1.2.4 iTRAQ标记与LC-MALDITOF/TOF约100 μg的蛋白质样品在5 mmol/L TCEP中37℃还原1 h,然后用终浓度为10 mmol/L的S-甲基硫代甲磺酸盐(MMTS)在室温下进行烷基化处理10 min,再用0.5 mmol/L TEAB将上述样品稀释8倍,并按1:20(W/V)的比例加入胰蛋白酶,37℃酶解过夜。根据iTRAQ标记试剂盒说明书将酶解的多肽样品进行标记合并,然后利用Sep-Paks Vac C18柱除盐、干燥,最后将重悬于0.5% TFA中的多肽样品经LC- 4800 Plus MALDI TOF/TOF(AB SCIEX)分析鉴定。
1.2.5 生物信息学分析将两次重复的质谱数据合并,然后利用Proteinpilot 4.2软件对原始质谱数据在大肠杆菌K12数据库中进行搜索,检索结果通过严格标准进行筛选(1% FDR),然后再筛选肽段匹配数≥2以及P-Value < 0.05的蛋白进行生物信息学分析,iTRAQ比值> 2.0或 < 0.5的蛋白为差异表达蛋白。然后借助OmicsBean(http://www.omicsbean.com:88)在线软件对差异蛋白进行Gene Ontology(GO)分析,并利用STRING 10.0软件分析KEGG代谢通路以及蛋白相互作用网络[7]。
1.2.6 qPCR验证相关基因在mRNA水平的表达变化用1/4MIC的巴洛沙星处理大肠杆菌,以未处理为对照,分别培养至OD600nm=1.0,收集菌体,用TRIZOL法提取细菌总RNA,并反转录为cDNA,作为qPCR的模板检测基因在mRNA水平上的表达情况,以16S rRNA作为内参基因[8]。
2 结果 2.1 大肠杆菌对巴洛沙星的MIC值及生长曲线根据大肠杆菌BW25113在不同浓度的巴洛沙星下的生长情况发现,其对巴洛沙星的MIC值为0.625 μg/mL。然后,依照测定的MIC值,分别用1/8MIC、1/4MIC、1/2MIC、1×MIC和2×MIC的巴洛沙星处理大肠杆菌,测定其生长曲线。与对照相比,抗生素处理之后大肠杆菌的生长受到了不同程度的抑制(图 1),而在1/4MIC处理下,抑制作用较为明显,但同时该菌还具有较高的活性,故选择1/4MIC作为巴洛沙星的处理浓度。

|
| 图 1 不同浓度巴洛沙星胁迫下大肠杆菌的生长曲线 |
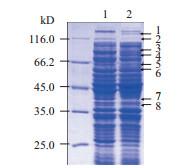
用1/4MIC浓度的巴洛沙星处理大肠杆菌E.coli BW25113,培养至OD600nm=1.0,提取全菌蛋白进行SDS-PAGE分离。研究显示,与对照相比,抗生素处理之后蛋白条带发生了明显的差异表达(图 2)。
|
| 图 2 巴洛沙星胁迫下大肠杆菌全蛋白SDS-PAGE 1:对照组;2:处理组。图中箭头和数字表示差异条带 |
将100 μg处理后的蛋白样品进行还原和烷基化,加入胰蛋白酶降解成多肽,然后进行iTRAQ标记。最后,将除盐后的混合多肽样品,在LC-MALDI TOF/TOF上进行分离鉴定,共鉴定出764个蛋白。与未添加抗生素处理组为对照,定量分析发现118个差异蛋白(差异倍数±2倍),其中包括52个表达上调蛋白和66个表达下调蛋白。
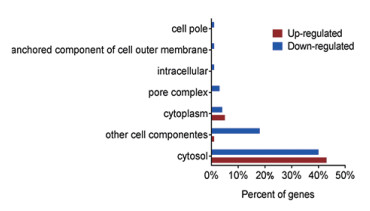
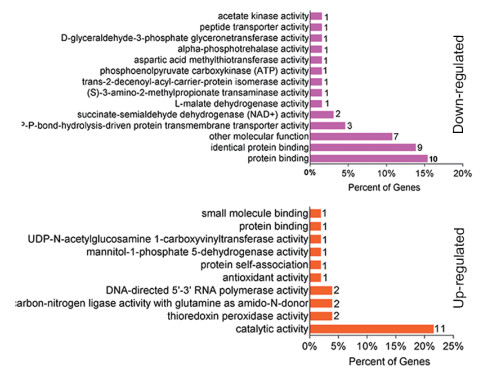
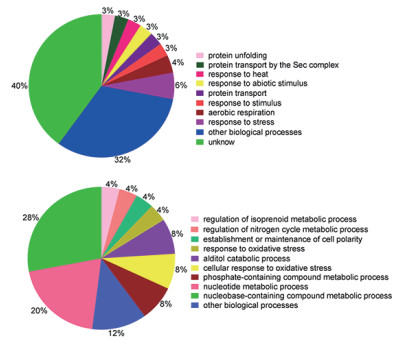
2.4 差异蛋白的功能分类与注释为进一步了解大肠杆菌在巴洛沙星胁迫中差异表达蛋白的生物学功能,利用OmicsBean在线软件对表达差异的蛋白进行GO注释,从细胞组分(Cellular component,CC)分子功能(Molecular function,MF)和生物学途径(Biological Process,BP)3个分类进行富集分析。图 3为表达差异蛋白的细胞组分分析结果,从图中可以看出,除了在“其他细胞组分”分类中下调蛋白数目较多以外,大部分上调或者下调蛋白的细胞组分并没有发生多大变化,均属于胞浆蛋白;在分子功能中,下调蛋白主要参与了蛋白结合功能(Potein binding),还有不少蛋白涉及到能量代谢相关酶的活性。此外,有一些蛋白具有蛋白跨膜转运(Protein transmembrane transporter activity)以及肽段转运活性(Peptide tra-nsporter activity),如OmpF、SecA、SecG和OppD等蛋白。而上调蛋白主要参与了催化作用(Catalytic activity)中。另外,各有2个蛋白参与了硫氧还蛋白过氧化物酶、连接酶、RNA多聚酶等酶活性,还有一些蛋白与小分子或蛋白结合作用相关(图 4);在生物学过程中,分别有6%和3%的下调蛋白参与了胁迫响应以及抗生素刺激的响应。此外,还有一些蛋白参与了蛋白转运过程(Protein transport),其中包括OppD、SecA和SecG等蛋白。在上调蛋白中发现,大部分蛋白参与了核酸代谢过程。还有一些蛋白涉及到磷酸盐的化合物代谢过程以及氮循环代谢过程的调节(图 5)。
|
| 图 3 巴洛沙星胁迫下大肠杆菌差异蛋白的细胞组分分析 柱子右侧数值表示相关的蛋白数目,同下 |
|
| 图 4 巴洛沙星胁迫下大肠杆菌中差异蛋白的分子功能(MF)分析 |
|
| 图 5 巴洛沙星胁迫下大肠杆菌中差异蛋白的生物学过程(BP)分析 |
借助STRING 10.0软件,对大肠杆菌在巴洛沙星胁迫中的差异表达蛋白进行了KEGG代谢通路分析。由图 6可以看出,下调蛋白参与了很多代谢过程,比如碳代谢(10个)、甲烷代谢(4个)、细菌不同环境下代谢(14个)、牛磺酸与牛磺酸代谢(2个)等,而且涉及能量代谢途径的蛋白也不在少数,如糖酵解途径(6个)、丙酮酸途径(9个)、TCA循环(4个)。而在上调蛋白相关代谢途径中,也有17个蛋白参与了细胞代谢过程,此外上调蛋白更多集中在DNA或者RNA的合成代谢相关途径,如有7个蛋白参与嘌呤代谢,6个参与嘧啶代谢,以及2个蛋白参与RNA多聚酶(Rif,RpoZ),提示在巴洛沙星处理下,细菌的核酸生物合成过程受到了压力。

|
| 图 6 巴洛沙星胁迫下大肠杆菌中差异蛋白的KEGG代谢通路分析 图中红色箭头代表下调蛋白,绿色箭头代表上调蛋白 |
蛋白质在行使生物学功能的过程中不是孤立的,往往形成复杂的蛋白-蛋白相互作用网络,应对外界条件的变化。利用STRING在线软件并结合GO与KEGG分析结果,本研究构建了大肠杆菌在巴洛沙星胁迫中的差异表达蛋白的蛋白互作网络(图 7)。与上述GO与KEGG分析结论类似,大部分表达下调的蛋白参与了能量生成相关的代谢途径并形成了复杂的相互作用网络。例如,巴洛沙星胁迫导致碳代谢(图 7中虚线圈2)途径中10个蛋白表达差异,其中除了TalB,AccB和Ppc蛋白上调以外,均为下调蛋白。而嘌呤与嘧啶代谢途径差异蛋白则与上调蛋白为主。例如,在图 7虚线圈1中6个嘧啶代谢相关差异蛋白中,只有一个蛋白表达下降(Pnp蛋白)。
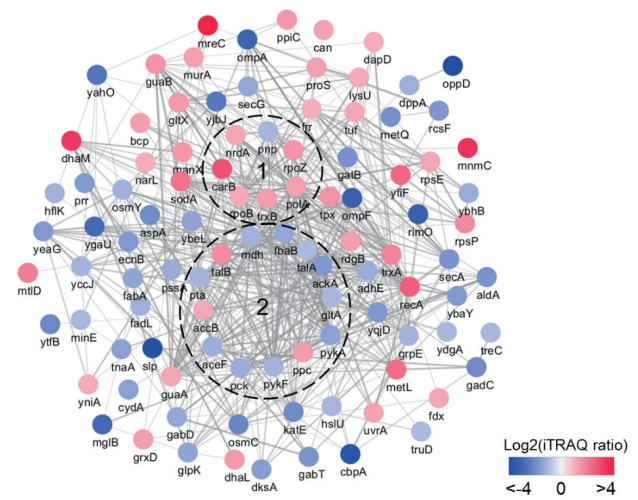
|
| 图 7 巴洛沙星胁迫下大肠杆菌中差异蛋白的蛋白相互作用网络分析 图中虚线圈1中为嘧啶代谢(Pyrimidine metabolism);2为碳代谢(Carbon metabolism)相关差异蛋白。红色代表上升蛋白;蓝色代表下调蛋白,以log2(iTRAQ比值)为参考值 |
根据蛋白相互作用网络的分析结果,本文选取了与碳代谢以及嘧啶代谢途径相关的基因,利用q-PCR技术分析了这些基因在mRNA水平上表达量的变化。在大肠杆菌响应巴洛沙星的胁迫过程中,以16S rRNA为内参基因,这些基因在mRNA水平上的表达量如图 8所示。在11个与碳代谢相关的基因中,除了accB在mRNA水平表达与蛋白水平相反,其他基因均与蛋白水平变化趋势一致,均为下调表达。在嘧啶代谢途径,有4个基因mRNA水平的表达变化与蛋白水平相同,分别是ndrA、pnp、ropB和trxB;而其他3个基因则呈相反趋势。可见,选取的大部分基因在mRNA水平上的表达变化与定量蛋白质组学的结果一致,说明本研究中蛋白质组学分析的结果具有一定可信度。
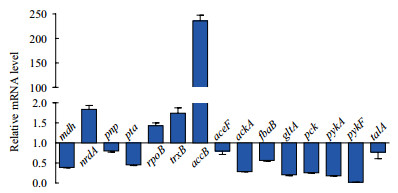
|
| 图 8 巴洛沙星胁迫下大肠杆菌部分基因在mRNA水平上的表达情况 |
喹诺酮类药物的抑菌作用机制主要是作用于细菌的脱氧核糖核酸,阻碍细菌拓扑异构酶的功能,从而终止DNA的复制,使细菌细胞不再分裂。如今,关于细菌对喹诺酮类抗生素耐药分子机制的研究有很多,大多与DNA旋转酶中GyrA和GyrB以及拓扑异构酶中ParC和ParE的突变对细菌耐药的影响有关[9]。此外,还有研究报道一些重要耐药蛋白如通道蛋白、外膜蛋白和外排泵蛋白等,在细菌耐药中起重要作用[10-11]。但是,还有很多蛋白的耐药功能及其分子机制有待进一步研究。
本研究中用第四代喹诺酮类药物巴洛沙星处理大肠杆菌,利用定量蛋白质组学的方法,共得到118种差异蛋白,其中52种上调,66种下调。在这些差异蛋白中,有一些蛋白的耐药功能已被广泛报道。例如,文献报道在大肠杆菌中缺失涉及DNA修复的uvrA基因对环丙沙星和甲硝唑的敏感性显著增强,而缺失另一个DNA重组修复相关基因recA,则对钠啶酮酸敏感[12]。本研究中UvrA和RecA蛋白在巴洛沙星胁迫下表达均升高,提示这些蛋白可能通过提高核酸修复能力来减轻喹诺酮类抗生素对核酸的破坏。变化的差异蛋白还包括4个表达下调的外膜蛋白(OmpC、OmpA、FadL和Slp)。前期研究发现OmpC与OmpF受细菌双调节系统EnvZ/OmpR的调控,通过控制外膜孔蛋白孔径的大小,调节外膜通透性,缺失ompC后可导致大肠杆菌对萘啶酮酸、碳青霉烯类及头孢类抗生素敏感[13-15]。此外研究发现OmpA与FadL等外膜蛋白均在萘啶酮酸、卡那霉素等抗生素胁迫下也发生了变化,提示这些外膜蛋白可能在细菌耐药过程中起重要的作用[16-17]。
研究表明,细菌耐药的形成除了几个关键耐药蛋白以外,胞内代谢通路的改变也可以改变细菌耐药特性[18]。例如,Su等[19]通过外源添加代谢物逆转细菌体内代谢流,与抗生素联用可显著杀死耐药细菌。本研究通过生物信息学分析发现,巴洛沙星的胁迫也导致细菌胞内代谢通路发生显著的波动,在能量生成相关的代谢途径,如TCA循环、丙酮酸代谢以及糖酵解/糖异生等途径中,则显著下降。该表现与嗜水气单胞菌应对土霉素胁迫中的行为类似,提示能量代谢相关蛋白下调可能是细菌应对抗生素压力的一个普遍机制[20-22]。此外,在上调蛋白通路中,嘌呤与嘧啶代谢途径相关蛋白表达普遍上升,提示细菌可能通过上调核酸代谢过程中相关蛋白的数量,如DNA多聚酶I(PolA)和RNA多聚酶亚基(RpoZ),以此减轻抗生素攻击靶点的压力,“稀释”抗生素的相对作用浓度,从而使细菌存活。综上所述,本研究通过对巴洛沙星胁迫下大肠杆菌定量蛋白质组学的研究,为研究细菌的耐药机制以及今后新型抗生素的研发提供了理论依据。
4 结论大肠杆菌K12 BW25113菌株在1/4 MIC巴洛沙星浓度胁迫下,共鉴定到了118个差异蛋白,包括52个上调表达蛋白和66个下调表达蛋白,这些差异蛋白主要参与了能量代谢以及核酸代谢来调整细菌耐药状态。
| [1] |
李丰田, 杨继勇, 叶丽艳, 等. 2007-2012年肠杆菌科细菌分布及耐药性分析[J]. 中华医院感染学杂志, 2014, 24(24): 6004-6005. |
| [2] |
Levy SB. Antibacterial resistance worldwide:causes, challenges and responses[J]. Nature Medicine, 2004, 10(12): 122-129. |
| [3] |
肖永红, 沈萍, 魏泽庆, 等. Mohnarin 2010年度全国细菌耐药监测[J]. 中华医院感染学杂志, 2011, 21(23): 4896-4902. |
| [4] |
吕瑞勤, 郗砚彬, 吕金玲, 等. 第四代喹诺酮类抗菌药物研究进展[J]. 中国新药杂志, 2012, 21(13): 1485-1493. |
| [5] |
Chen XH, Zhang BW, Li H, et al. Myo-inositol improves the host's ability to eliminate balofloxacin-resistant Escherichia coli[J]. Scientific Reports, 2015, 5(1): 10720. DOI:10.1038/srep10720 |
| [6] |
Lin X, Lin L, Yao Z, et al. An integrated quantitative and targeted proteomics reveals fitness mechanisms of Aeromonas hydrophila under oxytetracycline stress[J]. Journal of Proteome Research, 2015, 14(3): 1515-1525. DOI:10.1021/pr501188g |
| [7] |
Szklarczyk D, Franceschini A, Wyder S, et al. STRING v10:protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Res, 2015, 43(D1): D447-D452. DOI:10.1093/nar/gku1003 |
| [8] |
Clifford RJ, Milillo M, Prestwood J, et al. Detection of bacterial 16S rRNA and identification of four clinically important bacteria by real-time PCR[J]. PLoS One, 2012, 7(11): e48558. DOI:10.1371/journal.pone.0048558 |
| [9] |
Betanzoscabrera G, Juárezverdayes MA, Gonzálezgonzález G, et al. Gatifloxacin, moxifloxacin, and balofloxacin resistance due to mutations in the gyrA and parC genes of Staphylococcus epidermidiss trains isolated from patients with endophthalmitis, corneal ulcers and conjunctivitis[J]. Ophthalmic Research, 2009, 42(1): 43-48. |
| [10] |
Masi M, Réfregiers M, Pos MK, et al. Mechanisms of envelope permeability and antibiotic influx and efflux in Gram-negative bacteria[J]. Nature Microbiology, 2017, 2(3): 17001. DOI:10.1038/nmicrobiol.2017.1 |
| [11] |
Peng B, Wang C, Li H, et al. Outer membrane proteins form specific patterns in antibiotic-resistant Edwardsiella tarda[J]. Frontiers in Microbiology, 2017, 8(69): 1-12. |
| [12] |
Tamae C, Liu A, et al. Determination of antibiotic hypersensitivity among 4, 000 single-gene-knockout mutants of Escherichia coli[J]. J Bacteriol, 2008, 190(17): 5981-5988. DOI:10.1128/JB.01982-07 |
| [13] |
Nikaido H. Molecular basis of bacterial outer membrane permeabilityr evisited[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 593-656. DOI:10.1128/MMBR.67.4.593-656.2003 |
| [14] |
Lin XM, Li H, Wang C, et al. Proteomic analysis of nalidixic acid resistance in Escherichia coli:identification and functional characterization of OM proteins[J]. Journal of Proteome Research, 2008, 7(1): 2399-2405. |
| [15] |
Liu YF, Yan JJ, Lei HY, et al. Loss of outer membrane protein C in Escherichia coli contributes to both antibiotic resistance and escaping antibody-dependent bactericidal activity[J]. Infection and Immunity, 2012, 80(5): 1815-1822. DOI:10.1128/IAI.06395-11 |
| [16] |
Lin XM, Wang C, et al. Differential regulation of OmpC and OmpF by AtpB in Escherichia coli exposed to nalidixic acid and chlortetracycline[J]. J Proteomics, 2012, 75(18): 5898-5910. DOI:10.1016/j.jprot.2012.08.016 |
| [17] |
Peng XX, Xu CX, Ren HX, et al. Proteomic analysis of the sarcosine-insoluble outer membrane fraction of Pseudomonas aeruginosa responding to ampicilin, kanamycin, and tetracycline resistance[J]. Journal of Proteome Research, 2005, 4(6): 2257-2265. DOI:10.1021/pr050159g |
| [18] |
Peng B, Su YB, Li H, et al. Exogenous alanine and/or glucose plus kanamycin kills antibiotic-resistant bacteria[J]. Cell Metabolism, 2015, 21(2): 249-262. DOI:10.1016/j.cmet.2015.01.008 |
| [19] |
Su YB, Peng B, Li H, et al. Pyruvate cycle increases aminoglycoside efficacy and provides respiratory energy in bacteria[J]. Proc Natl Acad Sci USA, 2018, 115(7): 1578-1587. DOI:10.1073/pnas.1714645115 |
| [20] |
Li WX, Yao ZJ, Sun LN, et al. Proteomics analysis reveals a potential antibiotic cocktail therapy strategy for Aeromonas hydrophila infection in biofilm[J]. Journal of Proteome Research, 2016, 15(6): 1810-1820. DOI:10.1021/acs.jproteome.5b01127 |
| [21] |
Cheng ZX, Yang MJ, Peng B, et al. The depressed central carbon and energy metabolisms is associated to the acquisition of levofloxacin resistance in Vibrio alginolyticus[J]. J Proteomics, 2018, 181: 83-91. DOI:10.1016/j.jprot.2018.04.002 |
| [22] |
George AJ. Mechanisms of resistance to quinolones[J]. Clinical Infectious Diseases, 2005, 41: S120-S126. DOI:10.1086/428052 |




