随着全球人口的增长和气候环境的变化,粮食安全问题是许多国家面临的紧迫问题。通过转基因技术创制转基因作物并应用于商业化种植,有助于解决粮食安全问题。自1996年转基因作物商业化种植以来,全球转基因作物的种植面积逐年增加,2017年转基因作物种植面积达到1.898亿hm2,带来巨大的社会效益、经济效益和生态效益[1]。商业化种植的转基因事件一般都是从大量转化事件中筛选得到。由于转基因作物的安全性问题受到广泛关注,商业化种植的转基因作物都经过严格的安全性评价,许多国家已经出台了一系列法规和管理方法。
在转基因安全评价中,外源基因在基因组中的整合位点是必须提供的信息。鉴定外源基因在基因组中的整合位点对转基因作物的应用具有重要意义。目前,主要通过PCR技术鉴定外源基因的整合位点,包括TAIL-PCR(Thermal asymmetric interlaced PCR,TAIL-PCR)、反向PCR(Inverse PCR,I-PCR)、T接头PCR(T-linker PCR)等方法[2-6]。TAIL-PCR技术简单易行,反应高效灵敏,产物特异性好,重复性好,应用较为广泛。Xu等[7]通过TAIL-PCR方法分离了水稻转基因事件Bt-ZJ22中外源基因整合位点3'端旁侧序列,并根据旁侧序列建立了TaqMan实时定量PCR鉴定方法。Yang等[8]通过TAIL-PCR方法鉴定了转基因番茄华番l号外源基因整合位点的旁侧序列。Liu等[9]对抗除草剂转基因玉米事件Aro203进行分析,鉴定了外源基因整合位点两侧旁侧序列,并建立了事件特异性的PCR鉴定方法。
基于PCR技术获得的旁侧序列相对较短,如果旁侧序列比对到多条染色体,则不能通过旁侧序列确定外源基因整合的染色体。对外源基因进行遗传定位,可以确定外源基因在基因组中的物理位置,有助于全面了解外源基因在基因组中的整合情况。
本课题组前期将Cry1Ie和bar转化玉米,获得大量抗虫转基因玉米新材料[10]。本研究以其中一个抗虫转基因玉米事件IE34为研究对象,通过TAIL-PCR获得外源基因整合位点上游旁侧序列,并通过遗传定位的方法确定外源基因整合的染色体位置,以期为转基因玉米的安全性评价提供重要依据。
1 材料与方法 1.1 材料植物材料为转Cry1Ie抗虫玉米IE34。转基因玉米与玉米自交系B73杂交构建F2代分离群体用于遗传定位。转基因玉米材料申报在北京市和海南省的中间试验并进行种植。
1.2 方法 1.2.1 DNA的提取取200 mg苗期生长旺盛的玉米叶片,采用改良的CTAB法[11]提取DNA。
1.2.2 TAIL-PCR依据Liu等[5]方法进行TAIL-PCR,在外源基因的左边界处设计特异性引物与随机引物组合进行扩增(TAIL-PCR的随机引物为LAD1-1和LAD1-2,巢式引物为AC1;根据CaMV35S polyA终止子序列设计特异性引物35S-0、35S-1和35S-2(表 1))。扩增产物经琼脂糖凝胶电泳检测,回收,与T载体连接,随机挑选5个阳性克隆进行测序。
利用抗虫转基因玉米IE34的旁侧序列以及外源片段中的CaMV35S polyA序列,设计引物(上游引物IE34F:5'-AGGGGCTCCTCTGTTGTTGTA-3';下游引物IE34R:5'-TCGCTCATGTGTTGAGCATATAA-3'),建立IE34事件及其衍生产品的定性PCR鉴定方法。反应条件为95℃ 5 min;94℃ 30 s,58℃ 30 s,72℃ 1 min,共35个循环;72℃ 7 min。
1.2.4 BSR-Seq分析转基因玉米IE34与自交系B73构建的F2分离群体植株长至5叶期时,用草铵膦涂抹叶片,5 d后,根据叶片受害情况鉴定转基因植株与非转基因植株。分别取50株转基因玉米和非转基因玉米的叶片样品进行混池,提取RNA,送北京贝瑞和康生物技术股份有限公司建库测序。测序数据进行BSA分析,初步确定外源基因在玉米基因组上的整合位点。
1.2.5 外源基因整合位点的初定位验证在MaizeGDB(http://www.maizegdb.org/)网站上下载初定位处的SSR标记,通过PAGE凝胶电泳筛选出在综31和B73双亲之间存在多态性的引物。然后以分离群体中非转基因植株DNA为模板进行PCR扩增,聚丙烯酰胺凝胶电泳检测,硝酸银染色,筛选交换单株,验证外源基因整合位点。
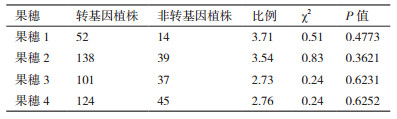
2 结果 2.1 TAIL-PCR扩增与序列分析前期通过Southern杂交确定转基因玉米IE34中外源基因单拷贝整合[10]。以转基因玉米IE34叶片基因组DNA为模板,非转基因玉米综31基因组DNA为对照,用5'端特异性引物与随机引物组合进行TAIL-PCR扩增。在第2轮及第3轮PCR后,转基因玉米IE34扩增到一条特异性条带,而非转基因玉米综31没有扩增到条带(图 1)。
|
| 图 1 TAIL-PCR扩增旁侧序列 M:DNA分子量Marker;1-3:第一轮TAIL-PCR;4-6:第二轮TAIL-PCR;7-9:第三轮TAIL-PCR;1、4、7:转基因玉米IE34;2、5、8:非转基因玉米综31;3、6、9:水 |
将第3轮PCR扩增到的特异PCR产物回收并克隆到T载体,选取阳性单克隆进行测序、比对,发现该序列1-776 bp为玉米基因组序列,777-982 bp为外源基因的序列(图 2)。BLAST比对结果表明,1-776 bp的玉米基因组序列为重复序列,存在于玉米基因组的多条染色体上。

|
| 图 2 转基因玉米IE34外源DNA整合位点5'端旁侧序列 1-776 bp:玉米基因组序列;777-982:外源基因序列 |
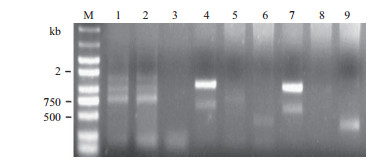
依据5'端旁侧序列的信息,分别在旁侧序列和5'端载体序列上设计上游、下游引物进行PCR扩增,只有转基因玉米IE34亲本、杂种F1和后代及其叶片、种子的DNA能扩增出312 bp特异性目的条带,水、非转基因植株和质粒均无扩增条带(图 3),从而建立了转基因玉米IE34的特异性PCR鉴定方法。
|
| 图 3 转基因玉米IE34事件特异性PCR检测 M:DL2000 plus DNA marker;1:水;2:质粒p3301UbiIe;3:非转基因玉米综31;4-5:转基因玉米IE34 T1代叶片;6:转基因玉米IE34 T2代叶片;7:转基因玉米IE34 T3代叶片;8:转基因玉米IE34 T2代种子;9:转基因玉米IE34 T3代种子;10:转基因玉米IE34杂交F1代叶片 |
外源基因整合位点旁侧序列比对到玉米多条染色体。为了进一步确定外源基因整合位点所在的染色体,构建遗传分离群体并进行定位分析。标记基因Bar与目的基因紧密连锁,可以通过在叶片上涂抹草铵膦的方法区分转基因玉米与非转基因玉米植株。将转基因玉米IE34与自交系B73杂交构建的F2代群体种植于田间,在苗期涂抹草铵膦后进行抗性鉴定并统计分离比,卡方测验表明F2代群体抗性分离比符合3:1(表 2),表明转基因玉米中只有1个外源基因的整合位点。
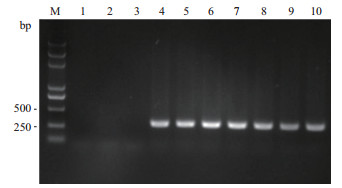
从F2代分离群体中随机取50株转基因植株和50株非转基因植株的叶片构建2个混池,提取叶片RNA。RNA样品进行转录组测序后,运用测序所得的SNP标记进行BSA分析。发现在第1和第5染色体上均有信号较强的峰(图 4),分别位于玉米第1染色体短臂36.2-51.7 Mb和第5染色体短臂2.74-7.77 Mb。
|
| 图 4 BSR-Seq分析对外源基因整合位点的初定位 |
在MaizeGDB网站(http://www.maizegdb.org/)下载BSR-Seq分析的2条染色体初定位区间的SSR引物,通过PAGE凝胶电泳筛选在双亲之间存在多态性的引物。其中第5染色体处的多态性引物有umc2539、umc2591和umc1679,第1染色体处的多态性引物有umc1403和phi109275。以分离群体中192个非转基因植株的DNA为模板,验证BSR-Seq分析得到的初定位区间,结果(表 3)显示,第1染色体处的标记引物扩增出2个亲本以及杂合型3种条带,第5染色体处的标记引物只扩增出亲本B73以及杂合型2种条带,说明外源基因的整合位点位于第5染色体。
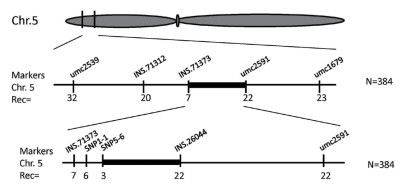
在第5染色体定位区间内筛选到3个InDel标记引物,分别为INS.71312、INS.71373和INS.26044。通过对定位群体筛选将bar定位在INS.71373和INS.26044 2个标记之间。进而依据BSR-Seq结果分析这个区间内SNP位点,通过测序筛选出具有多态性的SNP位点,得到SNP1-1和SNP5-6 2个SNP多态性位点。用这8对多态性引物对F2代群体中384个非转基因植株进行检测,发现左侧标记检测的交换单株从32株递减到3株,右侧标记检测到的交换单株呈现从23株到22株的递减趋势,说明外源基因位于SNP5-6与INS.26044 2个标记之间,这两个标记分别位于第5染色体2.35和2.61 Mb的位置(图 5)。综上所述,外源基因整合到了玉米第5染色体2.35-2.61 Mb约260 kb的区间内。
|
| 图 5 外源基因整合位点的精细定位 |
随着转基因作物研究的快速发展,需要对转基因作物进行有效监管。在创制有育种价值转基因新材料时,希望外源基因的整合不影响受体作物的有利性状,而且没有质粒骨架整合到受体基因组中。因此,确定外源基因在受体基因组中的整合位点对转基因作物安全性评价具有重要意义。检测外源基因整合位点旁侧序列的方法有多种,如TAIL-PCR、反向PCR、染色体步移技术等[12-14]。本研究采用TAIL-PCR方法获得了转基因玉米事件IE34中外源基因整合位点5'端的旁侧序列,但是没有获得整合位点3'端旁侧序列,表明TAIL-PCR方法也有局限性,扩增不成功的原因可能是引物不匹配,或者旁侧序列结构复杂导致无法扩增到目的条带。
如果知道外源基因整合位点两端旁侧序列,可以建立三引物PCR方法鉴定纯合转基因植株及杂合转基因植株[9, 15-16]。虽然本研究没有得到转基因玉米IE34中外源基因整合位点下游旁侧序列,但利用外源基因5'端的旁侧序列建立了转基因玉米IE34的特异性PCR检测方法,便于对其转基因后代进行检测。
Blast比对发现外源基因整合到转基因玉米IE34基因组中的重复序列区。此重复序列存在于玉米基因组多条染色体上,因此无法确定外源基因所整合的染色体及具体位置。在构建的F2代分离群体中转基因玉米与非转基因玉米呈现3:1的分离比,表明外源基因在玉米基因组中只有一个整合位点,因此可以利用遗传定位的方法对外源基因整合位点进行定位,进一步确定外源基因在玉米染色体上的位置。采用BSR-Seq对外源基因整合位点进行初定位,确定外源基因整合在第5染色体。通过进一步的精细定位确定外源基因的整合位点在玉米第5染色体2.35-2.61 Mb约260 kb的区间内,但还需要通过其他方法来确定整合位点下游的旁侧序列。Guo等[14]利用基因组重测序方法鉴定到转基因大豆中外源基因整合位点的旁侧序列。直接利用高通量测序的方法有可能鉴定到转基因玉米IE34中外源基因整合位点的下游旁侧序列。
4 结论转基因玉米IE34中的外源基因整合在5号染色体2.35-2.61 Mb约260 kb的区间内,通过PCR可以特异性检测转基因玉米IE34。
| [1] |
国际农业生物技术应用服务组织. 2017年全球生物技术/转基因作物商业化发展态势[J]. 中国生物工程杂志, 2018, 38: 1-8. |
| [2] |
Liu YG, Mitsukawa N, Oosumi T, et al. Efficient isolation and mapping of Arabidopsis thaliana T-DNA insert junctions by thermal asymmetric interlaced PCR[J]. Plant J, 1995, 8: 457-463. DOI:10.1046/j.1365-313X.1995.08030457.x |
| [3] |
Fujimoto S, Matsunaga S, Murata M. Mapping of T-DNA and Ac/Ds by TAIL-PCR to analyze chromosomal rearrangements[J]. Methods Mol Biol, 2016, 1469: 207-216. DOI:10.1007/978-1-4939-4931-1 |
| [4] |
Ochman H, Gerber AS, Hartl DL. Genetic applications of an inverse polymerase chain reaction[J]. Genetics, 1988, 120: 621-623. |
| [5] |
Liu YG, Chen Y. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J]. Biotechniques, 2007, 43: 649-656. DOI:10.2144/000112601 |
| [6] |
Yuanxin Y, Chengcai A, Li L, et al. T-linker-specific ligation PCR(T-linker PCR): an advanced PCR technique for chromosome walking or for isolation of tagged DNA ends[J]. Nucleic Acids Res, 2003, 31: e68. DOI:10.1093/nar/gng068 |
| [7] |
Xu J, Wang X, Chen X, et al. Qualitative detection and quantification of a Cry1A(b)transgene present in rice cv. Zhejing 22[J]. European Food Research & Technology, 2011, 233: 259-266. |
| [8] |
Yang C, Zhang D, Yang L. Development of event-specific PCR detection methods for genetically modified tomato Huafan No. 1[J]. Journal of the Science of Food & Agriculture, 2013, 93: 652-660. |
| [9] |
Liu YJ, Zhang YW, Liu Y, et al. Metabolic effects of glyphosate on transgenic maize expressing a G2-EPSPS gene from Pseudomonas fluorescens[J]. Journal of Plant Biochemistry and Biotechnology, 2015, 24: 233-241. DOI:10.1007/s13562-014-0263-9 |
| [10] |
Zhang YW, Liu YJ, Ren Y, et al. Overexpression of a novel Cry1Ie gene confers resistance to Cry1Ac-resistant cotton bollworm in transgenic lines of maize[J]. Plant Cell Tissue Organ Culture, 2013, 115: 151-158. DOI:10.1007/s11240-013-0348-5 |
| [11] |
Murray MG, Thompson WF. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Res, 1980, 8: 4321-4325. DOI:10.1093/nar/8.19.4321 |
| [12] |
Trinh Q, Zhu P, Shi H, et al. A-T linker adapter polymerase chain reaction for determining flanking sequences by rescuing inverse PCR or thermal asymmetric interlaced PCR products[J]. Analytical Biochemistry, 2014, 466: 24-26. DOI:10.1016/j.ab.2014.07.022 |
| [13] |
Zhang L, Huang X, Zhu S. An event-specific real-time PCR detection system for the transgenic rice line 114-7-2 of producing functional human serum albumin[J]. European Food Research & Technology, 2014, 239: 403-408. |
| [14] |
Guo B, Guo Y, Hong H, et al. Identification of genomic insertion and flanking sequence of G2-EPSPS and GAT transgenes in soybean using whole genome sequencing method[J]. Frontiers in Plant Science, 2016, 7: 1009. |
| [15] |
郭超, 何行健, 邓力华, 等. 转基因水稻BarKasalath-01事件特异性检测[J]. 分子植物育种, 2017, 15: 4466-4475. |
| [16] |
杨阳, 王叶, 范金杰, 等. 转基因棉花MON757转化体特异性PCR检测方法及应用[J]. 农业生物技术学报, 2016, 24: 908-918. |