多菌灵(Methyl-1H-benzimidazol-2-yl carbamate, MBC)是一种农林业中常用的广谱农药产品, 对由真菌引起的病害有防治效果, 在我国的使用范围广泛[1]。多菌灵也是多种常用杀菌剂(苯菌灵、甲基噻吩酯等)的有效成分或水解产物[2]。多菌灵为可疑致癌物、致突变物和内分泌干扰物, 在环境中较稳定, 对哺乳动物有毒害, 其残留可引起肝病和染色体畸变, 土壤中的多菌灵残留也可进入地下水系统危害水生生物[2-4]。它的使用在很多发达国家被严格控制, 但在我国的使用量较大[5]。土壤和水中的多菌灵残留通常经微生物自然降解, 如结核分枝杆菌属、诺卡氏菌和红球菌等[6-10]。然而, 天然微生物降解效率和底物特异较低。同时, 化学杀真菌剂的长期使用会改变土壤中微生物菌群的平衡, 影响降解效能[11]。其他的物理或化学降解方法起效较慢, 价格昂贵, 设施复杂, 不利于大规模推广[12]。因此, 开发合理的人工微生物降解方法对降低或去除多菌灵残留有重要的意义。
大肠埃希氏菌(Escherichia coli)作为常用的基因工程载体, 具有快速繁殖和易于进行靶基因表达调控的优势。选择适合的表达系统对靶基因的调控具有重要意义, 如诱导型表达系统可限时表达靶基因, 减少过表达引发的多效性效应, 并通过规避代谢负担获得产品的最大量产[13-14]。常用的诱导系统通过添加化学诱导剂如异丙基β-D-1硫代氨基吡喃糖苷(IPTG)、脱水四环素(ATC)、阿拉伯糖或调整物理化学因素如pH、温度或紫外光(UV)达到[15-17]。化学诱导剂价格昂贵, 有毒性, 需人为添加和分离, 并且不易去除, 难以精准调控[15]。物理化学诱导方法或引起蛋白质热聚集(温度诱导), 导致可溶性蛋白产量低; 或诱发细胞分子结构变化和生化反应(Ph诱导); 或产生细胞光毒性(紫外光诱导)[17-20]。光诱导具有廉价、环保、无细胞毒性, 可随时添加/去除, 无需分离等优势, 避免了上述诱导剂的劣势, 在合成生物学和生物工程中具有广泛的应用价值[21]。
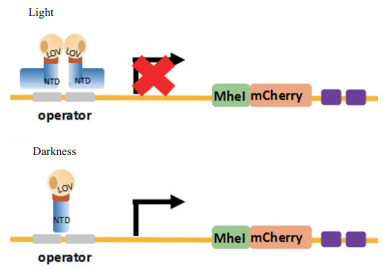
近年来, 利用重组大肠埃希氏菌降解土壤农残的报道逐年增加, 此类应用在大规模解毒过程中具有优势[6, 22-24]。大多数的异源基因表达采用的是传统的化学诱导方法。本研究根据一种由华东理工大学建立的新型的大肠埃希氏菌光控基因表达系统[25-26], 构建一株可利用光源作为诱导剂合成多菌灵水解酶(MBC hydrolyzing esterase)的重组大肠埃希氏菌。该光控基因表达系统的原理为:将从粗糙链孢霉(Neurospora crassa)中分离的光氧电压(LOV)结构域蛋白(蓝光传感器VIVID), 克隆到大肠埃希氏菌LexA repressor的C端, 构成一个名为LEV1(LexA-LOV)的光敏融合蛋白, 在光照条件下可形成同源二聚体, 抑制下游基因的表达; 而黑暗条件下, 同源二聚体自然分解, 下游基因得以表达(图 1)。试图证明该光控系统可表达具有生物活性的多菌灵水解酶, 具备廉价, 高效, 可操控等技术优势。可望为多菌灵类农药残留的生物降解提供新的技术手段和数据支持。
|
| 图 1 大肠埃希氏菌光控诱导表达系统的原理 光照条件下,光敏融合蛋白LEV1(LexA-LOV)形成同源二聚体并结合在操作子序列上,通过阻断RNA聚合酶的结合从而抑制启动子的功能;光源被剥夺后,LEV1二聚体逐渐分离,下游基因的表达被启动 |
大肠埃希氏菌DH5a菌株(TaKaRa Code No.9057), 质粒DNA纯化试剂盒plasmid purification kit (TaKaRa Code No.9760), 多菌灵MBC农药标准品(上海楷岳生物科技), 多菌灵水解酶酶联免疫反应试剂盒(上海通蔚实业有限公司), 大肠埃希氏菌细菌总蛋白提取试剂盒(贝博生物), SDS-PAGE蛋白凝胶配置试剂盒(诩圣生物), 限制性内切酶EcoRI (TaKaRa Code No.1040S), KpnⅠ (TaKaRa Code No.1068S), BglⅡ (TaKaRa Code No.1021S), Taq DNA聚合酶(TaKaRa Code No.2011A), 青链霉素(Solaria)。
1.1.2 仪器医用型洁净工作台(苏净安泰SW-CJ-1F), 压力蒸汽灭菌器(雅玛拓立式SN310C), 电热恒温培养箱(一恒DHP系列-立式), 凝胶成像仪(BioRad), 高速冷冻离心机(Sigma), 多功能酶标仪(Biotek), NanoDrop ND-1000分光光度计(IMPLEN), 紫外分光光度计(岛津), 多功能PCR机(LongGene)。
1.2 方法 1.2.1 构建生产多菌灵水解酶的光控基因表达载体光控基因表达载体使用的载体骨架为pCDFDuet1, 其中的启动子和MCS序列被去除。组成型启动子(bba_J23116, http://parts.igem.org/Promoters/Catalog/Constitutive)、光敏融合蛋白LEV1和两个大肠埃希氏菌终止子rrnB (从pBAD/His载体获得)插入其中的EcoRI和BglⅡ酶切位点, 获得pLEV1质粒。在此基础上, PColE启动子(含有一个可供光敏蛋白结合的操纵子序列)、多菌灵水解酶基因mheⅠ (从分枝杆菌中获得, Gene bank:KX698097.2)、和报告基因mCherry均根据公布的基因序列商业合成获得(广州华大基因研究院), 并插入到pLEV1质粒中的KpnⅠ和BgⅠ Ⅱ的酶切位点中, 获得pLEV1-mheⅠ-mCherry质粒(图 2), 并通过菌落PCR验证。菌落PCR的引物序列为:mheⅠ-F (5'-GCGGCGTAGCTTTTATGCTG-3'), mheⅠ-R (5'-CCATGTTATCCTCCTCGCCC-3')。PCR产物通过琼脂糖凝胶电泳检测。另外我们还设计了不含mheⅠ基因, 只有mCherry报告基因的质粒, 命名为pLEV1-mCherry, 用于阴性对照。

|
| 图 2 大肠埃希氏菌pLEV1-mheⅠ-mCherry的质粒构建展示(局部) 黑色箭头表示mheⅠ引物的位置 |
将pLEV1-mheⅠ-mCherry转化到DH5a菌株, 37℃过夜黑暗培养。从平板上挑取单克隆到5 mL的LB液体培养基(含1 µg/mL青链霉素)中37℃黑暗培养至OD600为0.6-0.8。将培养基中的菌液稀释100倍到两个20 mL的LB液体培养基(含青链霉素)中, 分别光照(自然光, 光照强度为普通日照)和黑暗(双层锡箔纸包裹)过夜培养, 温度为37℃。重组DH5a生产的粗蛋白酶液通过超声破碎的方式提取(贝宝大肠埃希氏菌总蛋白提取试剂盒)。BCA蛋白定量试剂盒用于测定可溶性蛋白溶液, 聚丙烯酰胺凝胶电泳(SDS-PAGE)用以检测全蛋白液, 酶联免疫法测定粗酶液中多菌灵水解酶的含量。
1.2.3 多菌灵水解酶的活性测定多菌灵水解酶的活性测定方法参照Lei等[6]的方法。具体操作为:在5 mL浓度为20 mmol/L的Tris-HCl (pH 7.4)的缓冲液中加入40 μmol的MBC作为底物和多菌灵水解酶作为反应物, 在30℃孵化1 h产生反应。反应以加入0.1 µg/mL的含多菌灵水解酶的粗蛋白酶液开始, 到加入同等体积的乙酸乙酯终止。反应完成后, 提取上层有机相, 用紫外分光光度计检测MBC在287 nm的吸光度。一个单位的酶的活性被定义为在30℃时, 1 min内1 μmol的底物MBC被水解成二级产物氨基苯并咪锉(2-AB)的所需酶的量。含LEV1-mheⅠ-mCherry质粒的菌落在光照条件下提取出的粗酶液作为测试品。在光抑制条件下提取出的粗酶液经过高温沸水浴, 以及不含mheⅠ基因的pLEV1-mCherry空载体作为阴性对照。
1.2.4 mheⅠ水解酶用于降解土壤中的MBC土壤样品从深圳某私家花园的场地中获取。将从地表到地下约10 cm处的土壤用收集器收集, 高压高温(121℃)灭菌30 min以去除所含野生微生物[19]。底物MBC以10 mg/kg的浓度被加入到约50 g的灭菌土壤样品和50 g的未灭菌样品, 底物在无菌环境下被充分混合后, 再加入4 mL的含多菌灵水解酶的粗酶液, 充分混合。另取含底物MBC的未灭菌土壤作为阴性对照。每个土壤样品的湿度调节到约30%, 在30℃中培育76 h后, 每12-16 h提取土壤样品检测其MBC的含量。方法参考Wang等[10]的操作并稍作改进, 具体如下:5 g土壤样品与5 mL的甲醇:水溶液(4:1, V/V)混合, 在200 r/min的摇床混合15分钟, 离心收集上清液。此步骤重复3次共获取约15 mL的上清液。上清液中加入10 mL的石油醚, 在200 r/min的摇床混合20 min, 去除石油醚层。剩余的水溶液在真空旋转蒸发仪和65℃烘箱中蒸干。残渣溶解于2 mL的甲醇, 用紫外分光光度计在287 nm检测吸光值(参考1.2.3)。
1.2.5 统计分析采用SPSS软件(SPSS Statistics 21)进行统计分析与检验。两组多菌灵水解酶的酶联反应结果使用独立样本t检验进行分析; 不同实验组多菌灵水解酶的酶活力差异, 以及土壤样本中MBC降解随时间的变化采用单因素方差分析(one-way ANOVA)和最小显著差异法(LSD)比较。以P < 0.05为差异有统计学意义。
2 结果 2.1 大肠埃希氏菌光控基因表达系统对mheⅠ基因的表达首先通过菌落PCR的方式证实重组质粒pLEV1-mheⅠ-mCherry的成功构建(图 3)。含此质粒的重组大肠埃希氏菌在黑暗诱导下合成多菌灵水解酶和mCherry的融合蛋白, 菌液经过夜摇瓶培养, 可产生肉眼可见的粉紫色颜色变化, 该颜色变化表明报告基因mCherry被成功表达; 而在可见光诱导条件下, 菌液为原色(图 4)。

|
| 图 3 琼脂糖凝胶电泳检测显示重组质粒pLEV1-mheⅠ-mCherry的成功构建 Marker:DL2000 DNA标准品;mheⅠ:PCR的扩增产物(包含一部分启动子、mheⅠ编码和一部分mCherry报告基因序列) |

|
| 图 4 重组大肠埃希氏菌经光控诱导成功表达显色蛋白 黑暗诱导条件下菌液呈粉紫色(小图:菌体沉淀),显示重组蛋白的生成(左);在光照条件下菌液呈原色(右) |
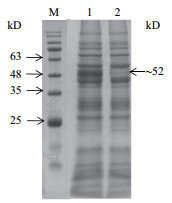
由于多菌灵水解酶与mCherry为融合蛋白, 颜色反应间接表明多菌灵水解酶被成功合成。SDS-PAGE的结果证实, 在光抑制条件下, 胞内上清含有疑似多菌灵水解酶(MHE)和mCherry的融合蛋白(~52 kD), 而在光照条件下, 该融合蛋白的表达量低(图 5)。多菌灵水解酶酶联反应的定性实验结果(图 6)表明, 光照条件下产生的多菌灵水解酶的量很低, 而在黑暗条件下, mheⅠ基因被成功超量表达, 多菌灵水解酶的产量有显著的提高(P < 0.05)。该结果同时也表明我们采用的这种光控表达系统的泄露率较低, 在非诱导(光照)条件下产物生产量极少。
|
| 图 5 SDS-PAGE凝胶电泳检测在光抑制(lane 1)或光照(lane 2)条件下产生的粗酶液的蛋白组成 误差线为标准误(n=3),统计分析用t-test检验。*P < 0.05光抑制状态对比光照状态 |

|
| 图 6 多菌灵水解酶酶联免疫法检测在光抑制(mheⅠ dark)或光照(mheⅠ light)条件下粗酶液与底MBC的反应情况 |
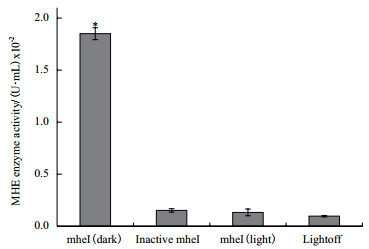
检测光控基因表达系统下生成的融合蛋白的酶活能力。从含pLEV1-mheⅠ-mCherry质粒的菌液在黑暗的诱导条件下提取的粗酶液(含多菌灵水解酶), 与从含pLEV1-mheⅠ-mCherry质粒的菌液在光照条件下提取的粗酶液(含多菌灵水解酶), 和含pLEV1-mheⅠ-mCherry质粒的菌液在光抑制条件下提取的粗酶液(含多菌灵水解酶)经过高温沸水浴使其中的酶失活, 以及含pLEV1-mCherry对照载体的菌液在黑暗条件下提取的粗酶液(不含多菌灵水解酶), 作为对比。结果(图 7)表明, 在黑暗诱导条件下提取的含多菌灵水解酶的粗酶液具有显著的酶活性(F=477.928, P < 0.05), 可分解溶液中的MBC底物, 其他对照组的粗酶液均不具有多菌灵水解酶的活性。
|
| 图 7 不同粗酶液中多菌灵水解酶的酶活性测定 mheⅠ(dark)为光抑制条件下提取的含多菌灵水解酶的粗酶液,Inactive mheⅠ为光抑制条件下提取的粗酶液经过高温失活,mheⅠ(light)为光照条件下提取的粗酶液,LightOFF为不含mheⅠ基因的空载体。误差线为标准误(n=3),统计分析用单因素方差分析,*表示P < 0.05 |
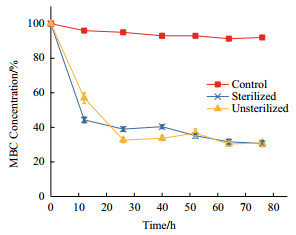
土壤样品在混合MBC底物并孵化76 h后, 绝大部分(>95%)的MBC底物可从该土壤样品中检测出, 说明MBC较稳定, 不易分解。当加入含多菌灵水解酶的粗蛋白液后, MBC底物被快速降解。此降解在前12 h较显著, 在随后的60 h内逐渐变缓, 可推测为多菌灵水解酶的含量逐渐减少所致(图 8)。对于加入了含多菌灵水解酶的粗蛋白液的土壤样品, 无论事先灭菌与否, MBC底物的浓度在76 h后均下降到不足30%, 表明多菌灵水解酶能够在土壤环境中有效降解MBC, 不受其他野生微生物存在的影响。
|
| 图 8 不同土壤样品中MBC的降解情况 “对照组”为不含多菌灵水解酶的未灭菌土壤样品,“灭菌”为加入含多菌灵水解酶的灭菌土壤样品,“未灭菌”为加入含多菌灵水解酶的未灭菌土壤样品。数据取自三个重复实验的平均值。误差线为标准误(n=3),统计分析用单因素方差分析(F=42.543,P < 0.05) |
常见的细菌异源基因表达系统中, 传统的化学或物理方法虽然能够诱导靶基因的表达, 但自身的局限性和外源添加的操作特点限制了其在生产领域的广泛应用。光源, 尤其是可见光, 基于其廉价, 低毒性、易操作、可调控等特点, 成为一种新的诱导方法[21]。我们根据一种已公布的新型的大肠埃希氏菌光控诱导系统, 构建一株通过可见光诱导, 可异源表达从分枝杆菌中分离的多菌灵水解酶基因(mheⅠ)的工程菌, 从而有效生产多菌灵水解酶。实验结果表明此方法生产出的多菌灵水解酶具有生物活性, 可降解溶液以及土壤中的多菌灵农药残留, 对于土壤的修复具有一定的意义。该光控基因表达系统通过光抑制进行诱导表达, 不仅操作简便, 还可进行精确的调控, 可望成为一个具有较好应用前景的新型的酶产品的生产模式。我们采用的这个光控系统只需一个单组件(LEV1融合光敏蛋白), 便可在可见光条件下进行诱导, 与其他之前报道的同类光控系统相比(PhyB-PIF3系统由两个融合蛋白组成且只能在远红外线下诱导[27], EnvZ/OmpR/Cph1系统有两个组件且诱导表达不可逆[28]), 可实现更简便、快捷、精确和可逆的诱导表达。
目前常用的土壤修复方法分为微生物修复(Microbial bioremediation)和酶生物修复(Enzymatic bioremediation)[6, 29-31]。相对于微生物修复, 酶生物修复具有更强的底物亲和性和更高的特异性, 同时, 酶在天然土壤中可自然降解[32]。土壤中有机磷等农残已广泛采用酶生物修复的方法去除[22-23, 33], 而多菌灵残留还大多局限于天然微生物修复。因此, 开发适合的生物酶用于多菌灵的去除以及土壤修复具有广泛的现实意义。基因工程中, 利用重组大肠埃希氏菌生产酶产品已是通用的技术手段[22-24, 30, 33-35], 但利用光源作为诱导剂的报道还鲜有见闻。我们第一次提出了此类操作在去除多菌灵农残上的应用, 其优势不仅在于工业生产成本的降低, 操作程序的简化, 还在于其可逆和可调控性。我们的工程菌平时在光照条件下不启动, 在需要时通过光抑制方法诱导生成水解酶, 并适时引入光照停止合成, 可有效延长工程菌的传代寿命。
众多可降解多菌灵农药的微生物, 如分歧杆菌属和诺卡氏菌等, 均具备先将多菌灵分解为二级代谢产物, 再将其作为碳源使用的特性[6-10], 本次重组的大肠埃希氏菌仍需开发相应的功能, 以提高该系统的实用性。此外, 重组大肠埃希氏菌虽可诱导生产水解酶, 却无法直接分泌到胞外。近年来, 已有通过添加信号肽(如Tat pathway)获得胞外分泌水解酶的工程菌的报道, 或以革兰氏阳性菌, 如枯草芽孢杆菌(Bacillus subtilis)为宿主, 利用其自身含有的胞外表达系统分泌水解酶[22-23, 36]。这些方法已成为构建"活菌降解器"(Biocatalysts)的主要途径[22, 36]。活菌降解器同时具备微生物修复的便捷, 和酶生物修复的高活性和底物专一性, 成为未来土壤农残降解的新趋势。下一步我们将尝试利用上述方法获得既可光控诱导又可胞外分泌水解酶的大肠埃希氏"活菌降解器", 可望成为新型的土壤降解多菌灵的生物工具。
4 结论本研究首次提出将大肠埃希氏菌光控基因表达系统用于生物降解土壤中的多菌灵农残。研究结果表明改建的光控系统可表达出有生物活性的多菌灵水解酶, 在水溶液和土壤样品中可有效水解多菌灵底物。此光控系统仅由可见光调控, 具有稳定、快速、可逆、无毒害、无后续分离等优势, 可成为生物降解农残的新应用趋势。
| [1] |
McCarroll NE, Protzel A, Ioannou Y, et al. A survey of EPA/OPP and open literature on selected pesticide chemicals:Ⅱ.Mutagenicity and carcinogenicity of carbendazim[J]. Mutation Research, 2002, 512: 1-35. DOI:10.1016/S1383-5742(02)00026-1 |
| [2] |
Sandahl M, Mathiasson L, Jonsson JA. Determination of thiophanate-methyl and its metabolites at thrace level in spiked natural water using the supported liquid membrane extraction and the microporous membrane liquid-liquid extraction techniques combined on-line with high performance liquid chromatography[J]. Journal of Chromatography Application, 2000, 893: 123-131. DOI:10.1016/S0021-9673(00)00697-X |
| [3] |
Boudina A, Emmelin C, Baaliouamer A. Photochemical behaviour of carbendazim in aqueous solution[J]. Chemosphere, 2003, 50: 649-655. DOI:10.1016/S0045-6535(02)00620-3 |
| [4] |
Kiigemagi U, Inman RD, Mellenthin WM, et al. Residues of benomyl (determined as carbendazim) and captan in postharvest-treated pears in cold storage[J]. Journal of Agricultural Food Chemistry, 1991, 39: 400-403. DOI:10.1021/jf00002a035 |
| [5] |
华小梅, 单正军. 我国农药的生产、使用状况及其污染环境因子分析[J]. 环境科学进展, 1996, 4(2): 33-44. |
| [6] |
Lei J, Ren L, Hu SB. The Characterization and bioremediation potential of degradation-enzyme from a newly isolated carbendazim-degrading Pseudomonas putida strain djl-1B[J]. International Journal of Simulation System, Science & Technology, 2015, 16(4B): 178. |
| [7] |
张桂山, 贾小明, 马晓航, 等. 一株多菌灵降解细菌的分离、鉴定及系统发育分析[J]. 微生物学报, 2004, 44(4): 417-421. DOI:10.3321/j.issn:0001-6209.2004.04.002 |
| [8] |
田连生, 陈菲. T2-2菌株对多菌灵的降解特性及生物修复试验[J]. 微生物学报, 2009, 49(7): 925-930. DOI:10.3321/j.issn:0001-6209.2009.07.013 |
| [9] |
Zhang XJ, Huang YJ, Harvey PR, et al. Isolation and characterization of carbendazim-degrading Rhodococcus erythropolis djl-11[J]. PLoS One, 2013, 8(10): e74810. DOI:10.1371/journal.pone.0074810 |
| [10] |
Wang Z, Wang Y, Gong F, et al. Biodegradation of carbendazim by a novel actino bacterium Rhodococcus jialingiae djl-6-2[J]. Chemsphere, 2010, 81(5): 639-644. DOI:10.1016/j.chemosphere.2010.08.040 |
| [11] |
Wang YS, Huang YJ, Chen WC, et al. Effect of carbendazim and pencycuron on soil bacterial community[J]. Journal of Hazard Materials, 2009, 172: 84-91. DOI:10.1016/j.jhazmat.2009.06.142 |
| [12] |
Mazellier P, Leroy E, Laat JT, Legube B. Degradation of carbendazim by UV/H2O2 investigated by kinetic modelling[J]. Environmental Chemistry Letter, 2003, 1: 68-72. DOI:10.1007/s10311-002-0010-7 |
| [13] |
Lee JM, Lee J, Kim T, et al. Switchable gene expression in Escherichia coli using a miniaturized photobioreactor[J]. PLoS One, 2013, 8(1): e52382. DOI:10.1371/journal.pone.0052382 |
| [14] |
Cho HS, Seo SW, Kim YM, et al. Engineering glyceraldehyde-3-phosphate dehydrogenase for switching control of glycolysis in Escherichia coli[J]. Biotechnology Bioengineering, 2012, 109: 2612-2619. DOI:10.1002/bit.v109.10 |
| [15] |
Schein CH. Production of soluble recombinant proteins in bacteria[J]. Nat Biotech, 1989, 7: 1141-1149. DOI:10.1038/nbt1189-1141 |
| [16] |
Menart V, Jevsevar S, Vilar M, et al. Constitutive versus thermoin-ducible expression of heterologous proteins in Escherichia coli based on strong PR, PL promoters from phage lambda[J]. Biotechnology Bioengineering, 2003, 83: 181-190. DOI:10.1002/(ISSN)1097-0290 |
| [17] |
Rinas U. Synthesis rates of cellular proteins involved in translation and protein folding are strongly altered in response to overproduc-tion of basic fibroblast growth factor by recombinant Escherichia coli[J]. Biotechnology Progress, 1996, 12: 196-200. DOI:10.1021/bp9600039 |
| [18] |
Lee SK, Keasling JD. Propionate-regulated high-yield protein production in Escherichia coli[J]. Biotechnology Bioengineering, 2006, 93: 912-918. DOI:10.1002/(ISSN)1097-0290 |
| [19] |
Jana S, Deb JK. Strategies for efficient production of heterologous proteins in Escherichia coli[J]. Applied Microbiological Biotechnology, 2005, 67: 289-298. DOI:10.1007/s00253-004-1814-0 |
| [20] |
Young DD, Deiters A. Photochemical activation of protein expression in bacterial cells[J]. Angewandte Chemistru International Edition, 2007, 46: 4290-4292. DOI:10.1002/(ISSN)1521-3773 |
| [21] |
Drepper T, Krauss U, Berstenhorst SMZ, et al. Lights on and action!Controlling microbial gene expression by light[J]. Applied Microbiology Biotechnology, 2001, 90(1): 23-40. |
| [22] |
Kang DG, Lim GB, C ha, H J. Functional periplasmic secretion of organophosphorous hydrolase using the twin-arginine translocation pathway in Escherichia coli[J]. Journal of Biotechnology, 2005, 118: 379-385. DOI:10.1016/j.jbiotec.2005.05.002 |
| [23] |
Yang C, Fredual F, Qiao C. Export of methyl parathion hydrolase to the periplasm by the Twin-arginine translocation pathway in Escherichia coli[J]. Journal of Agricultural Food Chemistry, 2009, 57: 8901-8905. DOI:10.1021/jf901739g |
| [24] |
Pandey G, Dorrian SJ, Russell RJ, et al. Cloning and biochemical characterization of a novel carbendazim (methyl-1H-benzimidazol-2-ylcarbamate)-hydrolyzing esterase from the newly isolated Nocardioides sp.strain SG-4G and its potential for use in enzymatic bioremediation[J]. Applied Environmental Microbiology, 2010, 76(9): 2940-2945. DOI:10.1128/AEM.02990-09 |
| [25] |
马正才, 刘韧玫, 杨弋.光激活基因表达系统在大肠杆菌中的应用[J/OL].中国科技论文在线, 2014.
|
| [26] |
Chen X, Liu R, Ma Z, et al. An extraordinary stringent and sensitive light-switchable gene expression system for bacterial cells[J]. Cell Research, 2016, 26(7): 854-847. DOI:10.1038/cr.2016.74 |
| [27] |
Shimizu-Sato S, Huq E, Tepperman JM, et al. A light-switchable gene promoter system[J]. Nat Biotech, 2002, 20: 1041-1044. DOI:10.1038/nbt734 |
| [28] |
Levskaya A, Chevalier AA, Tabor JJ, et al. Synthetic biology:Engineering Escherichia coli to see light[J]. Nature, 2005, 438: 441-442. DOI:10.1038/nature04405 |
| [29] |
Liang B, Lu P, Li HH, et al. Biodegradation of fomesafen by strain Lysinibacillus sp.ZB-1 isolated from soil[J]. Chemosphere, 2009, 77: 1614-1619. DOI:10.1016/j.chemosphere.2009.09.033 |
| [30] |
Mulbry WW, Karns JS. Purification and characterization of three parathion bacterial strains[J]. Applied Environmental Microbiology, 1989, 55(2): 289-293. |
| [31] |
Munnecke DM. Enzymatic detoxification of waste organophosphate pesticides[J]. Agricultural Food Chemistry, 1980, 28: 105-111. DOI:10.1021/jf60227a025 |
| [32] |
Munnecke DM, Hsieh PD. Microbial decontamination of parathion and p-nitrophenol in aqueous media[J]. Applied Microbiology, 1974, 28(2): 212-217. |
| [33] |
Fang H, Wang YQ, Gao CM, et al. Isolation and characterization of Pseudomonas sp.CBW capable of degrading carbendazim[J]. Biodegradation, 2010, 21(6): 939-946. DOI:10.1007/s10532-010-9353-0 |
| [34] |
Richins RD, Kaneva I, Mulchandani A, et al. Biodegradation of organophosphorus pesticides by surface-expressed organophosp-horus hydrolase[J]. Nat Biotech, 1997, 15: 984-987. DOI:10.1038/nbt1097-984 |
| [35] |
王德正, 吴辉, 李志敏, 叶勤. 重组大肠杆菌发酵生产谷胱甘肽的氨基酸添加策略优化[J]. 生物技术通报, 2015, 31(9): 197-203. |
| [36] |
Jongbloed JDH, Grieger U, Antelmann H, et al. Two minimal Tat translocasesin in Bacillus[J]. Molecular Microbiology, 2004, 54: 1319-1325. DOI:10.1111/j.1365-2958.2004.04341.x |



