近年来, 发热伴血小板减少综合征(俗称"蜱咬病")、施马伦贝格病等的新发引起人类对布尼亚病毒的广泛关注。布尼亚病毒是一类传染性强、致死率高的负链RNA病毒, 许多布尼亚病毒可引起严重的人类疾病。如汉坦病毒科的汉坦病毒可引起严重的人畜共患病, 世界卫生组织已将其列为可通过气溶胶传播的生物武器[1]。该病原体于1978年在韩国汉坦河附近被分离到。1993年, 美国的亚利桑那州、科罗拉多州、新墨西哥州和犹他州暴发的"死亡的峡谷", 病原即为汉坦病毒, 死亡率高达50%[2]。在南美洲和中美洲流行的人畜共患病——奥罗普切热的病原为周布尼亚病毒科的奥罗普切病毒(Oropouche virus, OROV)。在过去的60年中, 巴西、秘鲁、巴拿马、特立尼达和多巴哥报道了30多起流行病和超过50万例OROV感染的临床病例。在巴西, 奥罗普切热被认为是除登革热以外第二种最常见的虫媒病毒性发热疾病[3]。1965年以来我国新疆巴楚地区爆发的出血热疫情的病原为克里米亚-刚果出血热病毒(Crimean-Congo hemorrhagic fever virus, CCHFV), 此后该病在南疆地区一直流行, 死亡率达30%[4]。2010年夏季, 我国河南等地爆发的由蜱虫叮咬引发的"蜱咬病"的病原为发热伴血小板减少综合症病毒(Severe fever with thrombocytopenia syndrome virus, SFTSV), 死亡率达12%-30%[5]。此外, 裂谷热病毒(Rift valley fever virus, RVFV)、布尼韦尔病毒(Bunyamwera virus, BUNV)、拉克罗斯病毒(La Crosse encephalitis virus、LACV)、巴泰病毒(Batai, BATV)及阿里病毒(Ngari, NRIV)则能够在家畜、鸟类和人类中引发疾病。
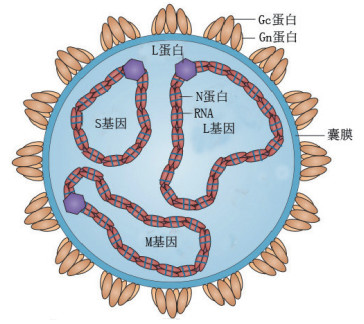
1 布尼亚病毒的基因组结构与功能布尼亚病毒是目前已知最大的RNA病毒家族, 已发现350多种病毒[6]。由于布尼亚病毒数目庞大, 2016年召开的第10次国际病毒分类委员会(ICTV)会议上将布尼亚病毒科升级为布尼亚病毒目, 由周布尼亚病毒科(Peribunyaviridae)、白细病毒科(Phenuiviridae)、汉坦病毒科(Hantaviridae)和内罗病毒科(Nairoviridae)等9个科组成。绝大多数布尼亚病毒基因组均由3条单股负链RNA组成, 分别为大(L)、中(M)、小(S)三段。它们都含有一个编码区以及编码区两侧的3'UTRs和5'UTRs。其中同一种属内的基因组RNA片段大小一致, L基因约为6.9 kb, M基因约为4.5 kb和S基因约为1.0 kb (图 1), 其中S基因编码N蛋白, M基因编码糖蛋白前体, L基因编码RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase, RdRp)。糖蛋白前体为多蛋白体, 其中含有非结构蛋白NSm。M基因的mRNA在宿主蛋白酶作用下翻译产生3种蛋白质, 编码顺序是Gn-NSm-Gc。布尼亚病毒颗粒呈球形, 直径为80-140 nm, 包括4种结构蛋白:两种表面糖蛋白, 分别称为Gn和Gc, 主要介导病毒的入侵; 两种内部蛋白质, 分别称为N蛋白和L蛋白, 其中N蛋白为核衣壳蛋白, 它将布尼亚病毒的L、M和S三个RNA节段基因组包裹, 并与RdRp形成核糖核蛋白复合物(Ribonucleoprotein complex, RNP), 并被包裹在脂质囊膜中, 负责病毒转录和复制。L蛋白质为RdRp, 在病毒转录和复制中起到重要的作用。
2 自然界中的布尼亚病毒重排布尼亚病毒之所以种类繁多, 是因为其基因组易发生变异的关系。布尼亚病毒基因组的分段性质, 增加了病毒成员之间基因重排的机率, 这种现象在几年前已经被反复验证。最新的ICTV分类建议列出了48种汉坦病毒, 除澳大利亚和南极洲以外的所有大洲均有分布[7]。在美国发现汉坦病毒肺综合征及其病原体辛诺柏病毒(Sin nombre virus, SNV)后, 很快就检测到它的重组体。尤其是SNV中的NMR11和CC107这两种亲缘关系密切的毒株感染同一个啮齿动物时, 基因重排的现象非常普遍[2]。Luis等和Saeed等[8-9]用SNV的毒株NMR11和CC107混合感染Vero E6细胞时产生了294个子代噬斑, 其中25个噬斑为重组体, 重组率为8.5%。贾托巴病毒(Jatobal, JATV)是在1985年从巴西帕拉州的南美长鼻浣熊中分离出来的, JATV和OROV序列在核苷酸水平上的相似性为65.6%, 其中S片段与OROV的秘鲁基因型相似性高达95.9%-97.3%。这些结果表明, JATV很可能是含有OROV的S片段的重配体[9]。伊基托斯病毒(Iquitos, IQTV)于1999年首次从秘鲁伊基托斯的一名发烧患者身上分离到。它也是OROV的重组体[10]。这种病毒拥有OROV的S和L片段。核酸序列分析表明, 欧洲2011新发的SBV的M片段与萨苏伯里病毒(Sathuperi virus, SATV)的日本株的序列相似性在81.8%-82.2%之间, 与沙门达病毒(Shamonda virus, SHAV)的S片段相似性为97%, L片段的相似性是94%, SBV可能是SATV与SHAV的基因重排产物[11]。AKAV的分离株之间的基因重排也非常频繁, Kobayashi等[12]对日本、台湾、澳大利亚和肯尼亚的赤羽病病毒(Akabane virus, AKAV)以及澳大利亚和日本发现的蒂纳罗病毒(Tinaroo virus, TINV)进行了测序和系统遗传学分析发现, TINV的S和L片段来自AKAV。此外, 研究表明在自然界中, NRIV是BUNV和BATV产生的一个重组株, 艾诺病毒(Aino virus, AINOV)和皮通病毒PEAV (Peaton virus, PEAV)之间也产生了一种重组株[13-14]。
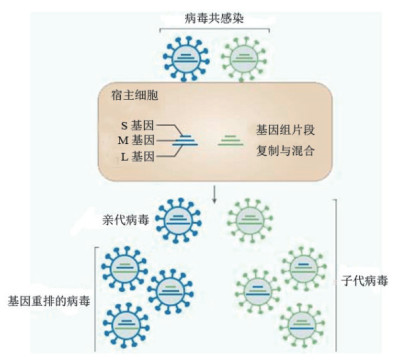
3 布尼亚病毒的基因重排机理 3.1 布尼亚病毒的基因重排的分子机制所有分节段的RNA病毒在共感染过程中都具有交换基因组片段的能力, 具体而言, 当两种或更多种病毒感染单个宿主细胞时, 它们可将彼此的基因组片段包装到新生病毒粒子中, 从而产生杂交后代, 流感病毒大流行中变异毒株的出现证实了上述观点[15]。与所有负链RNA病毒一样, 布尼亚病毒依赖RNA聚合酶(RdRp)缺乏核酸校对功能, 病毒复制而产生的基因组数量庞大, 这有助于基因变异的产生。由于布尼亚病毒结构的"分节段"性, 使它们能够更容易、更快速重组它们的基因组片段, 当两种不同的病毒同时感染单个细胞, 除了可以产生与亲本病毒相同的后代外, 基因重排还可产生6种潜在的重组病毒(图 2)[16]。然而, 这种重排的发生也是有限制的, 在Iroegbu和Pringle等[17]进行的体外实验证明, 基因重排只发生在关系密切的布尼亚病毒之间, 且这些病毒的基因组片段相互兼容, 才可能产生有效的重配株。对某些不相容的病毒基因组片段组合还有更多的限制。相关研究还显示, 现有的布尼亚病毒重组体倾向于L和S片段来自亲本病毒, M片段来自供体病毒[17]。因为L和S片段编码病毒复制必不可少的N蛋白以及病毒RNA聚合酶, 而M片段编码Gn和Gc蛋白, 其可以和宿主细胞上的附着受体和进入受体结合, 在病毒传播和媒介范围选择方面起到重要作用。因此, 来自供体的M片段使重组子代病毒会含有新的遗传信息, 潜在地改变该病毒对组织和宿主的嗜性[18]。
3.2 蚊子体内布尼亚病毒基因重排的发生机制Chastel认为, 被虫媒叮咬后无感染症状的宿主可能会成为"特洛伊木马", 将流行的虫媒病毒传入非疫区[16]。脊椎动物与作为病毒载体的节肢动物虫媒发生短时间密切接触后可以充当病毒的"生物容器"。当蚊子同时感染两种不同的布尼亚病毒时, 就可能导致基因重排。因为虫媒病毒能够在嗜血节肢动物和脊椎动物中交替地复制, 对脊椎动物的细胞有溶解性的破坏, 但对节肢动物的细胞很少或没有致病力。而且, 节肢动物宿主是不产生抗体的, 更有利于病毒的感染和复制[19]。
以吸食血液为生的节肢动物在其生命周期内可能会吸食多次血液, 这就增加了双重病毒感染的机率, 因此也会增加基因重排的机会。绝大多数重组病毒被预测是由蚊子而不是由蜱传播而来的, 这可能是由于双翅类动物的觅食方式决定, 因为蚊子可以在不同的脊椎动物身上进食, 而这些脊椎动物可能会感染不同的病毒, 因而基因组片段的重排机率很高; 而蜱的寿命虽然较长, 但却很少进食, 从而减少了双重感染的机率[19]。布尼亚病毒在蚊子体内进行基因重排的前提是蚊子必须感染两种不同的病毒, 在自然界中存在以下几种感染机制。一是口服感染, 同一只蚊子吸入两种感染不同病毒的宿主血液, 在这个过程中, 它可能会多次吸食血液, 或者是吸食一个宿主的血液时被打断, 接着在另一个寄主身上吸血结束[20]。自然界中共感染的发生又受双重感染抗性的限制, 即在感染第一种病毒后一段时间内, 再次感染第二种病毒受到抑制。这种双重感染阻力的现象在布尼亚病毒中同时存在, 多发生在感染第一种病毒后的几天内并随着时间的推移而加重, 限制了基因重排的发生。第二种机制是经卵感染, 如果雌蚊本身携带病毒, 它的子代也会携带与母体相同的病毒[21]。第三种机制是交配传播病毒, 如LACV或TAHV可与另一种加利福尼亚血清群的正布尼亚病毒发生双重感染。原因可能是经卵感染一种病毒的雄蚊对感染异种病毒的雌蚊进行授精, 导致雌蚊的双重感染。由于经卵感染的蚊子可能要比口服感染相同病毒的蚊子在中肠中的病毒滴度要低得多, 所以使双重感染阻力减小, 经卵感染一种布尼亚病毒的蚊子还能感染另一种布尼亚病毒[22-23]。
4 基因重排的潜在危害基因重排可使现有的变异株基因库进行随机重新分配, 使病毒能够适应不断变化的生态环境, 并且还支持不同病毒之间的重排, 这样更容易产生新的遗传性状, 从而改变子代病毒的毒性。尤其是基因型漂移和转变会改变病毒的致病性, 而且多表现为毒性增加。根据亲代病毒和子代的特征, 基因重排可产生病原性、媒介和宿主范围扩大的新型病毒。这给布尼亚病毒诊断、预防及控制带来了更大的困难, 增加了对公共卫生的危害。据报道, 在东非地区NRIV可导致人类严重的发热性疾病。研究证据表明, NRIV是在BUNV和BATV同时感染同一宿主时, 经天然基因重组而产生的一种新病毒。因为NRIV拥有BATV的M段, 同时与BUNV的S和L段相结合[24]。BUNV引起人体轻微症状, 如发热、头痛、关节痛和皮疹。儿童感染症状偏多, 免疫功能低下的患者可能会发展为严重的脑炎。在家畜中, 特别是反刍动物中, 感染后会出现严重的症状如自然流产和致畸[25]。BATV引起人类轻度的流感样疾病, 但可使反刍动物产生严重的疾病, 其表现为流产、早产和致命性出血热。NRIV致病力与母本病毒相比显著增强, 它能引起人的致命性出血热, 临床表现与RVFV类似。在1998-1999年的肯尼亚和索马里及2010年毛里塔尼亚都有爆发[26]。
2011年, 在德国和荷兰首次发现SBV, 家畜感染这种病毒后会产下畸形甚至是死胎。从发现后的一年时间席卷整个欧洲; 2012-2013年间, 在欧洲的27个国家都有发现, 涉及8 000多个农场, 严重威胁欧洲的畜牧业生产[27]。根据遗传发育证据显示, SBV与萨苏伯里病毒、沙门达病毒在基因组序列上十分接近。SBV是这两种病毒或突变的重组体。SATV和SHAV先后于1999年与2002年在日本发现, 但两种病毒感染动物后无明显症状。SBV的致病能力明显高于SATV和SHAV, 并能造成严重的经济损失。
5 展望对于分节段基因组的病毒而言, 基因重排和基因重组既是一种非常强大的进化压力, 促使其获得许多关键的适应性突变, 也是一种快速产生新病毒的方式, 使其致病性增加、宿主范围扩大。因此, 基因重排本质上增大了布尼亚病毒对宿主的危害, 从而对人类公共卫生造成了更大的威胁。尽管现有的布尼亚病毒重排研究工作为病毒间的相互作用研究提供了大量有效信息, 但是在变异方向、变异频率等方面仍存在许多未解之谜。所以, 监测、研究病毒间、尤其是已知的可以感染哺乳动物的布尼亚病毒间的基因重排和重组的发生仍是研究的重点。
由于很多布尼亚病毒可借助媒介在宿主动物间传播, 要完全消灭这些病毒难度极大。基于传统致弱减毒的疫苗也可能由于与自然界的野生型毒株发生遗传交换而恢复或增加其致病性, 目前也尚无可供使用的布尼亚病毒疫苗。因此, 要对重要的布尼亚病毒进行深入研究, 进一步探究病毒感染的遗传决定因素、节肢动物在布尼亚病毒传播中的作用、传播介质阻断病毒感染或传播的机制以及驱动布尼亚病毒基因重排的内在机理, 从而助推基因工程疫苗的开发及本病的免疫防控。
| [1] |
Muyangwa M, Martynova EV, Khaiboullina SF, et al. Hantaviral proteins:Structure, functions, and role in hantavirus infection[J]. Front Microbiol, 2015, 6: 1326. |
| [2] |
Llah ST, Mir S, Sharif S, et al. Hantavirus induced cardiopulmonary syndrome:A public health concern[J]. J Med Virol, 2018, 90(6): 1003-1009. DOI:10.1002/jmv.v90.6 |
| [3] |
Sakkas H, Bozidis P, Franks A, et al. Oropouche Fever:A Review[J]. Viruses, 2018, 10(4): pii E175. |
| [4] |
Bente DA, Forrester NL, et al. Crimean-Congo hemorrhagic fever:history, epidemiology, pathogenesis, clinical syndrome and genetic diversity[J]. Antiviral Res, 2013, 100(1): 159-189. DOI:10.1016/j.antiviral.2013.07.006 |
| [5] |
Zhan J, Wang Q, et al. Current status of severe fever with thromboc-ytopenia syndrome in China[J]. Virol Sin, 2017, 32(1): 51-62. |
| [6] |
Elliott RM. Emerging viruses:the Bunyaviridae[J]. Mol Med, 1997, 3(9): 572-577. DOI:10.1007/BF03401814 |
| [7] |
Forbes KM, Sironen T, Plyusnin A. Hantavirus maintenance and transmission in reservoir host populations[J]. Curr Opin Virol, 2018, 28: 1-6. DOI:10.1016/j.coviro.2017.09.003 |
| [8] |
Luis L, Jessica H, Clarence J, et al. Genetic reassortment among viruses causing hantavirus pulmonary syndrome[J]. Virology, 1998, 242: 99-106. DOI:10.1006/viro.1997.8990 |
| [9] |
Saeed MF, Wang H, Suderman M, et al. Jatobal virus is a reassortant containing the small RNA of Oropouche virus[J]. Virus Research, 2001, 77(1): 25-30. |
| [10] |
Aguilar PV, Barrett AD, Saeed MF, et al. Iquitos virus:a novel reassortant Orthobunyavirus associated with human illness in Peru[J]. PLoS Negl Trop Dis, 2011, 5(9): e1315. DOI:10.1371/journal.pntd.0001315 |
| [11] |
Garigliany MM, Bayrou C, Kleijnen D, et al. Schmallenberg virus:a new Shamonda/Sathuperi-like virus on the rise in Europe[J]. Antiviral Res, 2012, 95(2): 82-87. DOI:10.1016/j.antiviral.2012.05.014 |
| [12] |
Kobayashi T, Yanase T, Yamakawa M, et al. Genetic diversity and reassortments among Akabane virus field isolates[J]. Virus Res, 2007, 130(1-2): 162-171. |
| [13] |
Dutuze MF, Nzayirambaho M, Mores CN, et al. A review of Bunyamwera, Batai, and Ngari viruses:Understudied Orthobunyaviruses with potential one health implications[J]. Front Vet Sci, 2018, 5: 69. |
| [14] |
Yanase T, Aizawa M, Kato T, et al. Genetic characterization of Aino and Peaton virus field isolates reveals a genetic reassortment between these viruses in nature[J]. Virus Res, 2010, 153(1): 1-7. |
| [15] |
Beaty BJ, Bishop DH. Bunyavirus-vector interactions[J]. Virus Res, 1988, 10(4): 289-301. |
| [16] |
Elliott RM. Orthobunyaviruses:recent genetic and structural insights[J]. Nat Rev Microbiol, 2014, 12(10): 673-685. |
| [17] |
Iroegbu CU, Pringle CR. Genetic interactions among viruses of the Bunyamwera complex[J]. J Virol, 1981, 37(1): 383-394. |
| [18] |
Beaty BJ, Holterman M, Tabachnick W, et al. Molecular basis of bunyavirus transmission by mosquitoes:role of the middle-sized RNA segment[J]. Science, 1981, 211(4489): 1433-1435. DOI:10.1126/science.6781068 |
| [19] |
Briese T, Calisher CH, Higgs S. Viruses of the family Bunyaviridae:are all available isolates reassortants?[J]. Virology, 2013, 446(1-2): 207-216. |
| [20] |
Borucki MK, Chandler LJ, Parker BM, et al. Bunyavirus superinfection and segment reassortment in transovarially infected mosquitoes[J]. J Gen Virol, 1999, 80(Pt 12): 3173-3179. |
| [21] |
Chandler LJ, Hogge G, Endres M, et al. Reassortment of La Crosse and Tahyna bunyaviruses in Aedes triseriatus mosquitoes[J]. Virus Res, 1991, 20(2): 181-191. |
| [22] |
Thompson WH, Beaty BJ. Venereal transmission of La Crosse (California encephalitis) arbovirus in Aedes triseriatus mosquitoes[J]. Science, 1977, 196(4289): 530-531. DOI:10.1126/science.850794 |
| [23] |
Borucki MK, Kempf BJ, Blitvich BJ, et al. La Crosse virus:replication in vertebrate and invertebrate hosts[J]. Microbes Infect, 2002, 4(3): 341-350. DOI:10.1016/S1286-4579(02)01547-2 |
| [24] |
Yanase T, Kato T, et al. Genetic characterization of Batai virus indicates a genomic reassortment between orthobunyaviruses in nature[J]. Arch Virol, 2006, 151(11): 2253-2260. DOI:10.1007/s00705-006-0808-x |
| [25] |
Medlock JM, Snow KR, Leach S. Possible ecology and epidemiology of medically important mosquito-borne arboviruses in Great Britain[J]. Epidemiol Infect, 2007, 135(3): 466-482. DOI:10.1017/S0950268806007047 |
| [26] |
Bowen MD, Trappier SG, Sanchez AJ, et al. A reassortant bunyavirus isolated from acute hemorrhagic fever cases in Kenya and Somalia[J]. Virology, 2001, 291(2): 185-190. |
| [27] |
Beer M, Conraths FJ, van der Poel WH. 'Schmallenberg virus'-a novel orthobunyavirus emerging in Europe[J]. Epidemiol Infect, 2013, 141(1): 1-8. DOI:10.1017/S0950268812002245 |