氮素(N)是植物必需的大量元素之一,是植物体内蛋白质、核酸及叶绿素等的重要组分,在植物生长发育过程中发挥着十分重要的作用,其供给的充足与否,直接影响着植物体内众多物质和能量代谢活动,因此,被称为生命元素[1-3]。氮素以多种形态存在于自然生态系统中,大气中的氮主要以氮气的形式存在,土壤中能被植物吸收利用的氮素则主要为硝态氮(NO3-)和铵态氮(NH4+)等无机氮及尿素、氨基酸等有机氮,也有少数一些豆科植物可与固氮菌形成共生关系进而利用大气中的氮气作为其氮素营养的主要来源[4]。在自然条件下,旱田土壤中NO3-的含量要远高于NH4+。因此,在植物氮素吸收转运机制方面的研究中,NO3-得到了学术界更多的关注[4-7]。NO3-不仅是植物的重要氮源,还可作为信号分子在打破种子休眠、调控侧根发育、诱导叶片生长及调节相关基因表达等方面发挥关键作用[5, 7]。植物对NO3-的吸收和运输主要通过两种NO3-转运系统完成,即低亲和转运系统(Low-affinity transporter system,LATS)和高亲和转运系统(High-affinity transporter system,HATS)。当外界的NO3-浓度低于0.5 mmol/L时,NO3-的转运主要由高亲和转运系统负责执行;当外界NO3-的浓度高于0.5 mmol/L时,NO3-的转运主要依赖于低亲和转运系统[5, 8]。
植物根系对NO3-的吸收及NO3-在植株体内的转运主要由NO3-转运蛋白(Nitrate transporters,NRTs)介导[5, 8]。近年来,已有众多学者发现NRT家族成员蛋白不仅作为NO3-转运体在植物NO3-的吸收和转运过程中发挥重要作用,而且参与调控植物对多种逆境胁迫的抵御和适应过程。有关NRT类蛋白的结构特征及它们在植物正常生长发育过程中NO3-的吸收转运中的功能已有一些综述报道[9-13],但有关NRT类蛋白在植物抵御和适应逆境胁迫过程中的作用尚未见系统性的总结。鉴于此,本文重点综述了近年来关于NRT类蛋白在植物适应低NO3-、低K+、盐、干旱及重金属镉等胁迫中重要作用的研究进展,以期为今后进一步深入探究植物抗逆机理提供依据和参考。
1 NO3-转运蛋白在植物生长发育过程中NO3-转运中的功能概述高等植物中已发现的NO3-转运蛋白主要包括NRT1(根据系统发生关系更名为NPF家族蛋白)和NRT2两类家族蛋白[5, 8, 14]。在模式植物拟南芥中,已发现50多个NRT1家族成员,其中10个成员功能已被研究清楚。
AtNRT1.1(AtNPF6.3)是植物中首个被克隆和鉴定的NO3-转运蛋白[15],其编码基因主要在根的皮层及内皮层细胞中表达[16]。研究发现,AtNRT1.1是一种双亲和性NO3-转运蛋白,可参与高亲和与低亲和性NO3-的吸收[5]。AtNRT1.2(AtNPF4.6)在拟南芥根表皮细胞中表达,介导根系低亲和性NO3-的吸收[17]。AtNRT1.4(AtNPF6.2)主要在叶脉及叶柄细胞的液泡膜中表达,主要参与调控叶柄和叶片中NO3-的稳态平衡[18]。AtNRT1.5(AtNPF7.3)为低亲和性NO3-转运蛋白,主要在根中木质部周围中柱鞘细胞的细胞膜上表达,介导NO3-向木质部的装载及其向地上部的长距离运输[19];而定位在木质部薄壁细胞的AtNRT1.8(AtNPF7.2)参与介导木质部NO3-的卸载[20],AtNRT1.9(AtNPF2.9)主要表达于根韧皮部伴胞,参与根系韧皮部NO3-的装载[21],这三者共同参与调节NO3-在根系和地上部的分配[20-21]。AtNRT1.6(AtNPF2.12)仅表达于角果的维管组织和胚珠的珠柄,且在植株授粉后其表达量显著增加,该基因的主要功能是通过参与NO3-向胚胎的运输而直接影响胚胎的早期发育[22]。AtNRT1.7(AtNPF2.13)在老叶的韧皮部细胞中表达,定位于质膜上,参与调控NO3-从老叶向新叶的转运以实现NO3-的再利用[23]。而主要定位于叶片主脉韧皮部伴胞质膜上的AtNRT1.11和AtNRT1.12亦参与调节NO3-向新生幼嫩叶片的分配[24]。拟南芥NRT2家族有7个成员,它们主要介导高亲和性NO3-的吸收,在可利用的硝酸盐有限时被激活并发挥主导作用[5]。
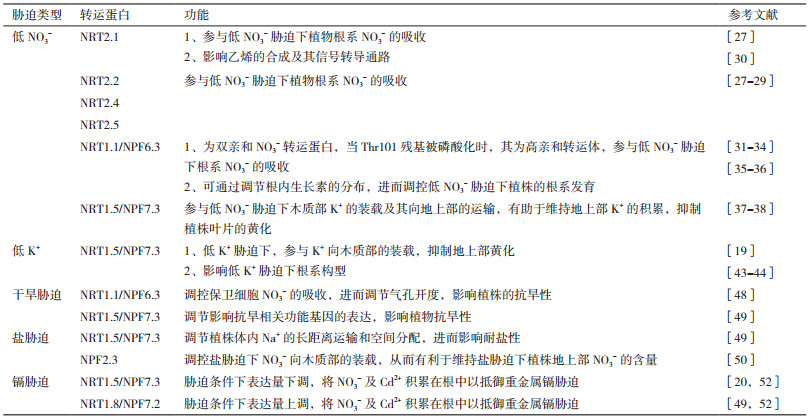
2 NO3-转运蛋白在植物适应逆境胁迫中的功能 2.1 NO3-转运蛋白在植物适应低NO3-胁迫中的作用植物对NO3-十分敏感,其供应不足会使叶片迅速黄化衰老[25]。大量研究表明,NRT2家族蛋白主要参与低NO3-胁迫下植物根系NO3-的吸收[5, 26-29]。在拟南芥中,AtNRT2.1主要表达于拟南芥根的成熟区表皮、皮层和内皮层细胞,该基因突变后,外界低浓度NO3-条件下植株根系NO3-吸收能力下降50%-70%,因此,AtNRT2.1是低NO3-胁迫下介导植物根系NO3-吸收的主要功能蛋白[27]。进一步研究发现,低NO3-胁迫可显著诱导AtNRT2.1的表达,一方面促进植株根系对NO3-的吸收;另一方面影响乙烯的合成及其信号转导通路,进而调控植株对低NO3-胁迫的适应过程[27, 30]。研究表明,AtNRT2.2突变后,低NO3-胁迫下植物根系NO3-的吸收能力仅下降19%,但当AtNRT2.1突变后,AtNRT2.2的表达丰度上调了3倍,表明AtNRT2.2是AtNRT2.1功能的一个补充者。AtNRT2.4属于极高亲和NO3-转运蛋白,其编码基因主要表达于侧根的表皮细胞[28]。高浓度NO3-条件下,AtNRT2.4的表达量极低,但当外界NO3-的浓度降低时,其表达量随着处理时间的延长持续增加[28]。在极低浓度NO3-(0.025 mmol/L)条件下,atnrt2.4突变体NO3-吸收速率明显低于野生型,表明AtNRT2.4参与极低浓度NO3-胁迫下的NO3-吸收过程[28]。AtNRT2.5主要表达于根表皮和根毛区皮层细胞,并受氮饥饿的显著诱导;在低NO3-胁迫下,拟南芥atnrt2.5突变体NO3-的吸收能力显著降低[29],且同时敲除AtNRT2.1、AtNRT2.2、AtNRT2.4和AtNRT2.5中任意3个基因的突变体植株在低NO3-条件下的生长均受到显著抑制[28-29]。可见,上述蛋白在低NO3-胁迫下植物根系NO3-的吸收过程中发挥着重要作用。
除上述高亲和性NO3-转运蛋白外,植物中几个低亲和性NO3-转运蛋白亦可参与介导低NO3-胁迫下植物根系NO3-的吸收。研究发现,AtNRT1.1是一种双亲和性NO3-转运蛋白,其对NO3-的亲和性随土壤中NO3-浓度的不同而发生转换,这一过程依赖于其氨基酸序列第101位苏氨酸残基(Thr101)的磷酸化和去磷酸化:当Thr101发生去磷酸化后,AtNRT1.1表现出低亲和性NO3-转运蛋白活性;而当Thr101残基被磷酸化后,AtNRT1.1则表现出高亲和性NO3-转运蛋白活性,参与低NO3-胁迫下根系NO3-的吸收过程[31]。Ho等[32]发现,与CBL(B类磷酸酶蛋白)互作的蛋白激酶CIPK23可使AtNRT1.1的T101位点发生磷酸化,进而使AtNRT1.1表现出高亲和性NO3-转运活性。在水稻中发现有2个NRT1.1同源基因(OsNRT1.1a和OsNRT1.1b),外界高NO3-条件下,它们的超表达均会显著增加植株体内氮素的累积,进而使得植株地上部干物质重量显著增加。然而,只有OsNRT1.1b的超表达能显著增加植株在低NO3-(0.125 mmol/L)环境下的氮素的累积,表明水稻OsNRT1.1b蛋白可参与低NO3-条件下水稻根系NO3-的吸收[33]。最新研究表明,在低氮的大田条件下,水稻OsNRT1.1a的超表达株系比野生型早熟9-13 d,且单株种子数、千粒重和分蘖数都多于野生型,从而使单株籽粒产量较野生型增加了32%-50%,因此,超表达OsNRT1.1a可有效提高氮素利用效率,增加低氮条件下水稻籽粒产量[34]。NRT1.1不仅参与NO3-的吸收,还可通过调控生长素在植物根中的分配进而调节植物的根系发育。
在低NO3-胁迫下,AtNRT1.1通过促进生长素的向基运输,而使侧根中生长素浓度降低,抑制侧根生长;当NO3-浓度增加时,AtNRT1.1抑制生长素的向基运输,侧根中生长素浓度增加,促进侧根生长[35]。另有研究表明,AtNRT1.1也可以感知外界较低浓度(0.05-0.50 mmol/L)的NO3-信号,缓解谷氨酸对植株主根生长的抑制,从而促进主根的生长[36]。
除NRT1.1外,低NO3-胁迫下NRT1.5也可通过调控植物叶片的衰老而影响植物的生长发育[37]。利用atnrt1.5单突变体和atnia1、atnia2、atnrt1.5三突变体对比研究表明,NO3-饥饿过程中atnrt1.5叶中NO3-的浓度高于三突变体,而三突变体老叶的衰老率反而低于atnrt1.5,因此,AtNRT1.5抑制叶片衰老的过程并不是通过影响NO3-转运来实现的[37]。进一步分析发现,在NO3-饥饿时,atnrt1.5叶片中的K+水平会显著降低,补充K+后叶片的衰老表型能基本得到恢复[37]。不仅如此,NO3-饥饿胁迫下atnrt1.5植株中与K+吸收相关的HAK5、RAP2.11及ANN1的表达量显著下调,这表明NRT1.5可以通过感知NO3-饥饿的信号,进而促进叶片中K+的积累来抑制叶片的衰老[37]。另有研究发现,AtNRT1.5可参与介导木质部K+的装载,与K+外整流通道SKOR共同调节不同的K+/NO3-条件下K+从根向地上部的长距离运输,其中,SKOR主要在高NO3低K+时活性较高,而AtNRT1.5则在低NO3条件下发挥作用[38]。
2.2 NO3-转运蛋白在植物适应低K+胁迫中的作用K+是植物体内含量最丰富的阳离子,与植物体的正常发育及农作物的产量等密切相关,K+从根向地上部的运输及在地上部的稳态平衡是植物保持营养平衡、维持正常生长和适应逆境环境的决定性因素之一[39-40]。大量研究发现植物体内NO3-和K+在吸收、转运、再分配过程中都存在密切的联系[41-42]。研究表明,AtNRT1.5的功能缺失会导致植株在低K+胁迫下表现出冠部提前发黄、根部持续生长的低K+敏感表型。同时,atnrt1.5突变体根部K+含量较野生型显著升高,地上部K+含量较野生型显著降低;而低K+胁迫下atskor突变体并未表现出明显缺K+表型,其根冠内的K+含量也与野生型相比无显著差异,且atnrt1.5/atskor双突变体与atnrt1.5的表型一致[43]。进一步利用非洲爪蟾卵母细胞异源表达系统的分析表明,AtNRT1.5是K+/H+反向转运蛋白,直接参与细胞K+外排[43]。由此可见,在低K+胁迫下AtNRT1.5可参与介导根部木质部薄壁细胞中K+向木质部的装载,从而促进根部K+向地上部的运输,缓解地上部因缺K+造成的胁迫损伤,并且此过程并不依赖于K+外整流通道SKOR[43]。然而有趣的是,Lin等[19]研究发现,低K+胁迫可显著抑制拟南芥AtNRT1.5的表达,但该基因转录水平上的变化模式与AtNRT1.5蛋白参与调控低K+条件下K+从根部向地上部运输的功能之间的关系有待进一步的研究。此外,除对地上部的影响之外,K+与植物根系的发育及形态结构也关系密切[41-42]。Zheng等[44]研究发现,在低K+且NO3-充足的条件下,atnrt1.5突变体植株的侧根密度显著减小,而侧根的发育受生长素的影响。因此,推测NRT1.5参与调控K+饥饿条件下植物根系生长素的稳态平衡。
2.3 NO3-转运蛋白在植物抵御盐和干旱胁迫中的作用氮素的吸收转运在植物适应盐和干旱逆境中发挥着重要作用[45-47]。Guo等[48]发现,AtNRT1.1参与调控植物的耐旱能力:在干旱胁迫下,拟南芥野生型植株叶片严重萎蔫黄化,而atnrt1.1突变体植株则叶片饱满且保持绿色。这主要是因为AtNRT1.1也在拟南芥保卫细胞中大量表达,参与介导保卫细胞NO3-的吸收;当AtNRT1.1突变后,保卫细胞中NO3-的积累量较野生型显著降低,进而降低气孔开度,这有利于减少由蒸腾作用导致的水分散失,从而提高了植株的耐旱能力。Chen等[49]发现,在盐和渗透胁迫(模拟干旱胁迫)下,拟南芥根中AtNRT1.5的表达丰度显著下降;与野生型植株相比,盐处理下atnrt1.5突变体木质部汁液中Na+含量显著下降,植株地上部Na+含量降低而根中Na+显著增加,使得atnrt1.5突变体耐盐能力明显增强;同时,与野生型植株相比,渗透胁迫下atnrt1.5突变体根和地上部抗旱相关功能基因P5CS1和RD29A的表达丰度显著增加,进而使得atnrt1.5的抗旱能力也明显强于野生型植株。可见,AtNRT1.5参与调节植株体内Na+的长距离运输和空间分配,并影响逆境响应相关基因的表达,进而在拟南芥耐盐抗旱性中起到“负调控”作用[49]。此外,主要定位于植物根系中柱鞘细胞的NO3-转运蛋白NPF2.3可通过调控盐胁迫下NO3-向木质部的装载,从而有利于维持盐胁迫下植株地上部NO3-的含量,进而在植物适应盐胁迫的过程中发挥重要作用[50]。
2.4 NO3-转运蛋白在植物抵御重金属隔胁迫中的作用镉(Cd)是生物毒性极强的一种重金属,且比其他重金属更易被植物吸收积累[51]。研究发现,调控NO3-在植物体内再分配的NRT1.8编码基因在Cd2+胁迫下的表达丰度显著上调,且Cd2+处理显著减少了NO3-由拟南芥根部向地上部的转运;在Cd2+胁迫下,atnrt1.8突变体表现出较野生型敏感的表型,并且atnrt1.8突变体对Cd2+敏感的表型依赖于介质中NO3-浓度[20]。进一步分析发现,atnrt1.8地上部Cd2+含量显著高于野生型,且atnrt1.8中Cd2+由根部向地上部转运也增加,因此,NRT1.8可通过将Cd2+滞留在植物根部,进而减少地上部Cd2+的积累,从而在植物抵御Cd2+毒害的过程中发挥重要作用[20]。有趣的是,AtNRT1.5对Cd2+胁迫的响应模式与AtNRT1.8相反[49],两者可共同参与调节植物对Cd2+胁迫的适应。在Cd2+胁迫时,负责将NO3-从木质部卸载至根部的NRT1.8表达量上调,而负责NO3-木质部装载的NRT1.5表达量下调,从而使更多的NO3-积累在植物根部以应对Cd2+胁迫[52]。此外,在许多的生物和非生物胁迫中AtNRT1.5和AtNRT1.8往往表现出相反的表达模式[20, 53],因此推测植物体内NO3-的分配可能参与调控植物对多种胁迫的响应[20, 26]。近年来,已有学者对这一响应模式在生物化学及分子水平上做了解释[52]。Cd2+等胁迫会激活植物乙烯(ET)信号途径和茉莉酸(JA)信号途径,使转录因子ERFs通过与AtNRT1.8启动子结合而诱导其表达,同时使EIN3/EIL1直接与AtNRT1.5启动子区域结合,抑制AtNRT1.5的表达,这两者的反向表达有利于NO3-在根中的积累,从而调节植株的抗逆性与植株生长之间的平衡[52]。
3 总结与展望NO3-转运蛋白不仅介导植物正常生长发育过程中的NO3-的吸收和转运,而且还参与调控植物对多种逆境胁迫的适应过程。虽然NO3-转运蛋白在植物适应低NO3-、低K+、盐、干旱及重金属镉胁迫中的重要作用已引起了学术界的关注,但对NO3-转运蛋白在植物逆境适应过程中的作用机制仍不清楚。目前相关研究大多以模式植物拟南芥为材料,该植物抵御盐和干旱等胁迫的能力十分有限,因此,以拟南芥为材料研究NO3-转运蛋白在植物抗逆性中的作用机制存在一定的局限性。
此外,目前,学术界仅关注到NO3-转运蛋白在植物适应上述几种胁迫中的重要作用,但对其在植物适应其他胁迫中的功能仍不明确。鉴于此,今后的研究可从以下几方面进行:(1)以具有极强抗逆能力的植物如盐渍和荒漠生境中广为分布的盐生和旱生植物为材料,克隆NO3-转运蛋白基因,利用CRISPR-Cas9、基因敲除、超表达等技术系统研究其调控活性及其在这些植物适应盐和干旱等逆境中的生理功能和相关作用机制;(2)进一步深入研究NO3-转运蛋白在植物适应低NO3-和低K+胁迫过程中的作用机制的同时,关注NO3-转运蛋白在植物适应磷、镁等其他重要营养元素亏缺条件中的功能。上述研究的完成可为进一步深入探究植物抗逆机理、筛选抗逆功能基因提供重要的理论依据。
| [1] |
Amtmann A, Armengaud P. Effects of N, P, K and S on metabolism:new knowledge gained from multi-level analysis[J]. Current Opinion in Plant Biology, 2009, 12(3): 275-283. DOI:10.1016/j.pbi.2009.04.014 |
| [2] |
王宇通, 邵新庆, 黄欣颖, 等. 植物根系氮吸收过程的研究进展[J]. 草业科学, 2010, 27(7): 105-111. |
| [3] |
李建勇, 龚继明. 植物硝酸根信号感受与传导途径[J]. 植物生理学报, 2011, 47: 111-118. |
| [4] |
Crawford NM, Glass ADM. Molecular and physiological aspects of nitrate uptake in plants[J]. Trends in Plant Science, 1998, 3(10): 389-395. DOI:10.1016/S1360-1385(98)01311-9 |
| [5] |
Wang YY, Hsu PK, Tsay YF. Uptake, allocation and signaling of nitrate[J]. Trends in Plant Science, 2012, 17(8): 458-467. DOI:10.1016/j.tplants.2012.04.006 |
| [6] |
Xu G, Fan X, Miller AJ. Plant nitrogen assimilation and use efficiency[J]. Annual Review of Plant Biology, 2012, 63(1): 153-182. DOI:10.1146/annurev-arplant-042811-105532 |
| [7] |
Wang YY, Cheng YH, Chen KE, et al. Nitrate transport, signaling, and use efficiency[J]. Annual Review of Plant Biology, 2018, 69(1): 85-122. DOI:10.1146/annurev-arplant-042817-040056 |
| [8] |
Dechorgnat J, Nguyen CT, Armengaud P, et al. From the soil to the seeds:the long journey of nitrate in plants[J]. Journal of Experimental Botany, 2011, 62(4): 1349-1359. DOI:10.1093/jxb/erq409 |
| [9] |
童依平, 蔡超, 刘全友, 等. 植物吸收硝态氮的分子生物学进展[J]. 植物营养与肥料学报, 2004, 10(4): 433-440. DOI:10.3321/j.issn:1008-505X.2004.04.018 |
| [10] |
李静, 张冰玉, 苏晓华, 等. 植物中的铵根及硝酸根转运蛋白研究进展[J]. 南京林业大学学报:自然科学版, 2012, 36(4): 133-139. |
| [11] |
马清, 管超, 夏曾润, 等. 高等植物氮素转运蛋白研究进展[J]. 兰州大学学报:自然科学版, 2015, 51(2): 217-227. |
| [12] |
张合琼, 张汉马, 梁永书, 等. 植物硝酸盐转运蛋白研究进展[J]. 植物生理学报, 2016(2): 141-149. |
| [13] |
宋田丽, 周建建, 徐晨曦, 等. 植物硝酸盐转运蛋白功能及表达调控研究进展[J]. 上海师范大学学报:自然科学版, 2017, 46(5): 740-750. |
| [14] |
Léran S, Varala K, Boyer JC, et al. A unified nomenclature of NITRATE TRANSPORTER 1/PEPTIDE TRANSPORTER family members in plants[J]. Trends in Plant Science, 2014, 19(1): 5-9. |
| [15] |
Tsay Y, Schroeder JI, Feldmann KA, et al. The herbcide sensitivity gene CHL1 of Arabidopsis encodes a nitrate inducible nitrate transporter[J]. Cell, 1993, 72(5): 705-713. DOI:10.1016/0092-8674(93)90399-B |
| [16] |
Huang NC, Chiang CS, Crawford NM, et al. CHL1 encodes a component of the low-affinity nitrate uptake system in Arabidopsis and shows cell type-specific expression in roots[J]. The Plant Cell, 1996, 8(12): 2183-2191. DOI:10.1105/tpc.8.12.2183 |
| [17] |
Huang NC, Liu KH, Lo HJ, et al. Cloning and functional characterization of an Arabidopsis nitrate transporter gene that encodes a constitutive component of low-aflinity uptake[J]. The Plant Cell, 1999, 11(8): 1381-1392. DOI:10.1105/tpc.11.8.1381 |
| [18] |
Chiu CC, Lin CS, Hsia AP, et al. Mutation of a nitrate transporter, AtNRT1:4, results in a reduced petiole nitrate content and altered leaf development[J]. Plant & Cell Physiology, 2004, 45(9): 1139-1148. |
| [19] |
Lin SH, Kuo HF, Canivenc G, et al. Mutation of the Arabidopsis NRT1.5 nitrate transporter causes defective root-to-shoot nitrate transport[J]. The Plant Cell, 2008, 20(9): 2514-2528. DOI:10.1105/tpc.108.060244 |
| [20] |
Li JY, Fu YL, Pike SM, et al. The Arabidopsis nitrate transporter NRT1.8 functions in nitrate removal from the xylem sap and mediates cadmium tolerance[J]. The Plant Cell, 2010, 22(5): 1633-1646. |
| [21] |
Wang YY, Tsay YF. Arabidopsis nitrate transporter NRT1.9 is important in phloem nitrate transport[J]. The Plant Cell, 2011, 23(5): 1945-1957. |
| [22] |
Almagro A, Lin SH, Tsay YF. Characterization of the Arabidopsis nitrate transporter NRT1.6 reveals a role of nitrate in early embryo development[J]. The Plant Cell, 2008, 20(12): 3289-3299. DOI:10.1105/tpc.107.056788 |
| [23] |
Liu W, Sun Q, Wang K, et al. Nitrogen Limitation Adaptation (NLA) is involved in source-to-sink remobilization of nitrate by mediating the degradation of NRT1.7 in Arabidopsis[J]. New Phytologist, 2016, 214(2): 734-744. |
| [24] |
Hsu PK, Tsay YF. Two phloem nitrate transporters, NRT1.11 and NRT1.12, are important for redistributing xylem-borne nitrate to enhance plant growth[J]. Plant Physiology, 2013, 163(2): 844-856. |
| [25] |
Guiboileau A, Sormani R, Meyer C, et al. Senescence and death of plant organs:nutrient recycling and developmental regulation[J]. Comptes Rendus Biologies, 2010, 333(4): 382-391. DOI:10.1016/j.crvi.2010.01.016 |
| [26] |
Gojon A, Gaymard F. Keeping nitrate in the roots:an unexpected requirement for cadmium tolerance in plants[J]. Journal of Molecular Cell Biology, 2010, 2(6): 299-301. DOI:10.1093/jmcb/mjq019 |
| [27] |
Li W, Wang Y, Okamoto M, et al. Dissection of the AtNRT2.1:AtNRT2.2 inducible high affinity nitrate transporter gene cluster[J]. Plant Physiology, 2007, 143(1): 425-433. |
| [28] |
Kiba T, Feria-Bourrellier A, Lafouge F, et al. The Arabidopsis nitrate transporter NRT2.4 plays a double role in roots and shoots of nitrogen-starved plants[J]. The Plant Cell, 2012, 24(1): 245-258. |
| [29] |
Lezhneva L, Kiba T, Feria BA, et al. The Arabidopsis nitrate transporter NRT2.5 plays a role in nitrate acquisition and remobilization in nitrogen-starved plants[J]. The Plant Journal, 2014, 60(2): 230-241. |
| [30] |
Zheng D, Han X, An YI, et al. The nitrate transporter NRT2.1 functions in the ethylene response to nitrate deficiency in Arabidopsis[J]. Plant Cell & Environment, 2013, 36(7): 1328-1337. |
| [31] |
Liu KH, Tsay YF. Switching between the two action modes of the dual-affinity nitrate transporter CHL1 by phosphorylation[J]. The EMBO Journal, 2003, 22(5): 1005-1013. DOI:10.1093/emboj/cdg118 |
| [32] |
Ho CH, Lin SH, Hu HC, et al. CHL1 functions as a nitrate sensor in plants[J]. Cell, 2009, 138(3): 1184-1194. |
| [33] |
Fan X, Feng H, Tan Y, et al. A putative 6-transmembrane nitrate transporter OsNRT1.1b plays a key role in rice under low nitrogen[J]. Journal of Integrative Plant Biology, 2016, 58(6): 590-599. DOI:10.1111/jipb.v58.6 |
| [34] |
Wang W, Hu B, Yuan D, et al. Expression of the nitrate transporter gene OsNRT1.1A/OsNPF6.3 confers high yield and early maturation in rice[J]. The Plant Cell, 2018, 30(3): 638-651. |
| [35] |
Krouk G, Crawford NM, Coruzzi GM, et al. Nitrate signaling:adaptation to fluctuating environments[J]. Current Opinion in Plant Biology, 2010, 13(3): 266-273. |
| [36] |
Walch LP, Forde BG. Nitrate signaling mediated by the NRT1.1 nitrate transporter antagonises L-glutamate-induced changes in root architecture[J]. The Plant Journal, 2008, 54(5): 820-828. DOI:10.1111/j.1365-313X.2008.03443.x |
| [37] |
Meng S, Peng JS, He YN, et al. Arabidopsis, NRT1.5 mediates the suppression of nitrate starvation-induced leaf senescence by modulating foliar potassium level[J]. Molecular Plant, 2016, 9(3): 461-470. |
| [38] |
Drechsler N, Zheng Y, Bohner A, et al. Nitrate-dependent control of shoot K homeostasis by NPF7.3/NRT1.5 and SKOR in Arabidopsis[J]. Plant Physiology, 2015, 169(4): 2832-2847. |
| [39] |
Maathuis FJM. Physiological functions of mineral macronutrients[J]. Current Opinion in Plant Biology, 2009, 12(3): 250-258. DOI:10.1016/j.pbi.2009.04.003 |
| [40] |
Sharma T, Dreyer I, Riedelsberger J. The role of K+ channels in uptake and redistribution of potassium in the model plant Arabidopsis thaliana[J]. Frontiers in Plant Science, 2013, 4(2): 224. |
| [41] |
Forde B, Lorenzo H. The nutritional control of root development[J]. Plant & Soil, 2001, 232(1/2): 51-68. |
| [42] |
LoPez-Bucio J, Cruz-RamãRez A, Herrera-Estrella L. The role of nutrient availability in regulating root architecture[J]. Current Opinion in Plant Biology, 2003, 6(3): 280-287. DOI:10.1016/S1369-5266(03)00035-9 |
| [43] |
Li H, Yu M, Du XQ, et al. NRT1.5/NPF7.3 functions as a proton-coupled H+/K+ antiporter for K+ loading into the xylem in Arabidopsis[J]. The Plant Cell, 2017, 29(8): 2016-2026. DOI:10.1105/tpc.16.00972 |
| [44] |
Zheng Y, Drechsler N, Rausch C, et al. The Arabidopsis nitrate transporter NPF7.3/NRT1.5 is involved in lateral root development under potassium deprivation[J]. Plant Signaling & Behavior, 2016, 11(5): 2832-2847. |
| [45] |
Wilkinson S, Davies WJ. ABA-based chemical signaling:the co-ordination of responses to stress in plants[J]. Plant Cell and Environment, 2002, 25(2): 195-210. |
| [46] |
Dodd IC, Tan LP, He J. Do increases in xylem sap pH and/or ABA concentration mediate stomatal closure following nitrate deprivation?[J]. Journal of Experimental Botany, 2003, 54: 1281-1288. DOI:10.1093/jxb/erg122 |
| [47] |
Schahram B, Sharyar B, Peter W, et al. Improvement of water use and N fertilizer efficiency by subsoil irrigation of winter wheat[J]. European Journal of Agronomy, 2008, 28(1): 1-7. DOI:10.1016/j.eja.2007.03.004 |
| [48] |
Guo FQ, Young J, Crawford NM. The nitrate transporter AtNRT1.1(CHL1) functions in stomatal opening and contributes to drought susceptibility in Arabidopsis[J]. The Plant Cell, 2003, 15(1): 107-117. |
| [49] |
Chen CZ, Lv XF, Li JY, et al. Arabidopsis NRT1.5 is another essential component in the regulation of nitrate reallocation and stress tolerance[J]. Plant Physiology, 2012, 159: 1582-1590. DOI:10.1104/pp.112.199257 |
| [50] |
Taochy C, Gaillard I, Ipotesi E, et al. The Arabidopsis root stele transporter NPF2.3 contributes to nitrate translocation to shoots under salt stress[J]. Plant Journal for Cell & Molecular Biology, 2015, 83(3): 466-479. |
| [51] |
荆红梅, 郑海雷, 赵中秋, 等. 植物对镉胁迫响应的研究进展[J]. 生态学报, 2001, 21(12): 2125-2130. DOI:10.3321/j.issn:1000-0933.2001.12.022 |
| [52] |
Zhang GB, Yi HY, Gong JM. The Arabidopsis ethylene/jasmonic acid-NRT signaling module coordinates nitrate reallocation and the trade-off between growth and environmental adaptation[J]. The Plant Cell, 2014, 26(10): 3984-3998. DOI:10.1105/tpc.114.129296 |
| [53] |
Zimmermann P, Hirsch-Hoffmann M, Hennig L, et al. GENEVESTIGATOR:Arabidopsis microarray database and analysis toolbox[J]. Plant Physiology, 2004, 136(1): 2621-2632. |