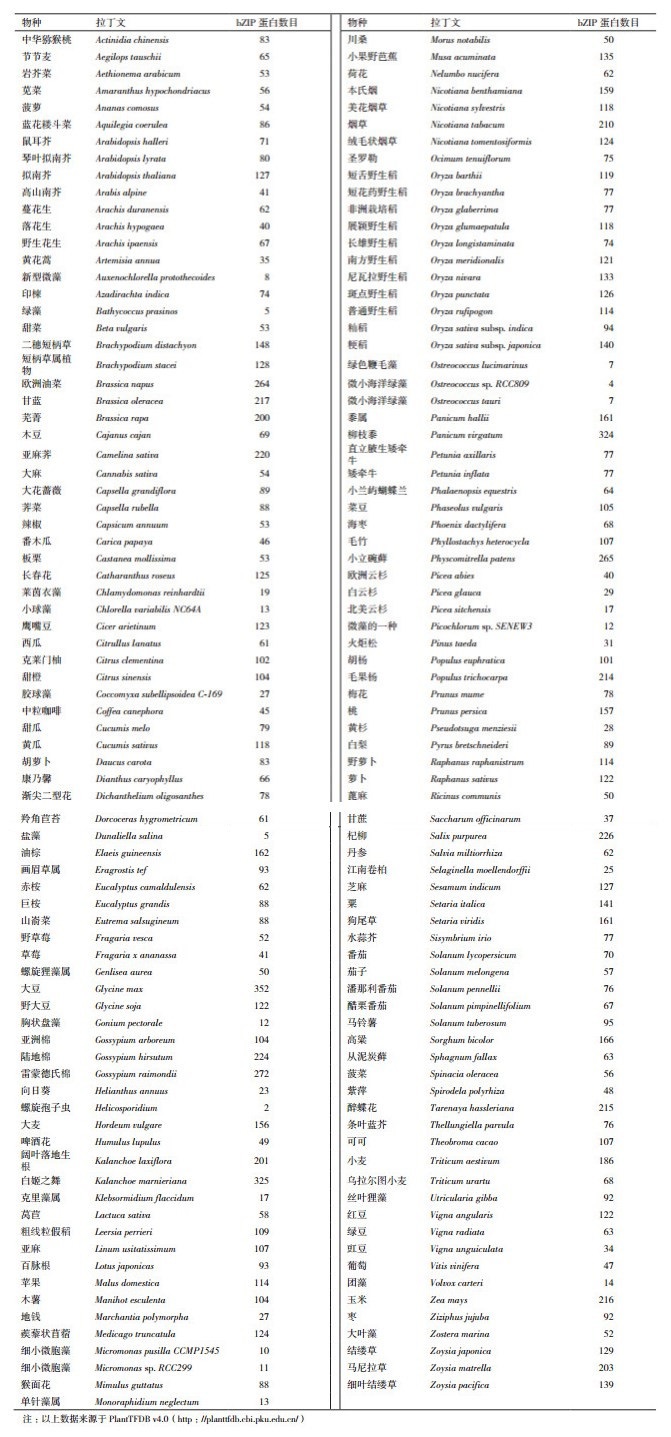
bZIP转录因子为最大的转录因子调控家族之一,在植物生物、非生物胁迫应答和发育等生理过程中发挥重要作用[1]。bZIP类转录因子可受干旱、盐害、冷害和脱落酸等因素的诱导,通过与胁迫相关基因启动子区域的顺式作用元件相互作用,调控靶基因的转录水平,进而调控植物的耐逆性。本文对bZIP类转录因子的分布、分类、结构特点、作用机理及其耐逆相关功能等方面的最新研究进展进行了较全面地综述。
1 bZIP转录因子的分布与分类 1.1 bZIP转录因子的分布目前,在几乎所有真核生物中均已鉴定了大量bZIP转录因子。但较之于微生物和动物,植物中具有更多的bZIP类转录因子[2]。迄今为止,在大豆(Glycine max)、亚洲棉花(Gossypium hirsutum)、玉米(Zea mays)、毛果杨(Populus trichocarpa)、烟草(Nicotiana tabacum)、小麦(Triticum aestivum)、拟南芥(Arabidopsis thaliana)、黄瓜(Cucumis sativus)、水稻(Oryza sativa)及番茄(Solanum lycopersicum)中分别发现了352、224、216、214、210、186、127、118、89和70个bZIP转录因子[3];而在酵母、果蝇和线虫中仅分别发现25、21和21个bZIP转录因子[4](表 1)。
bZIP转录因子的分类方法有多种,目前主要依据其氨基酸丰富度、DNA结合位点、保守序列以及系统进化关系等进行分类。根据特定氨基酸丰富度进行分类是较为常用的一种方法。利用该方法将芸苔(Brassica rapa)中的bZIP家族分为9组,它们的结构域中分别富含谷氨酰胺(Q)、天冬氨酸(D)、脯氨酸(P)、天冬酰胺(N)、丝氨酸(S)、甘氨酸(G)以及仅有简单结构域、无简单结构域或仅有一个跨膜结构域(Transmembrane domain,TMD)[5]。根据bZIP基本结构域和铰链区的氨基酸序列及DNA结合位点,可将水稻中的bZIP基因分为Ⅰ-Ⅺ共11组,每组分别包含7、3、6、22、3、14、16、3、11、3和1个OsbZIP基因[6],同样的方法可将蓖麻(Ricinus communis)中的bZIP基因分为Ⅰ-Ⅺ共11组,它们分别包含3、2、1、13、1、9、7、2、7、1、1个RcbZIP基因[7];根据系统发育进化关系,可将黄瓜中的64个bZIP基因分为Ⅰ-Ⅵ共6组,每组分别包含20、10、14、5、14和1个CsbZIP基因[1]。苹果(Malus domestica)中的114个bZIP基因分为A-I(A、B、C、D、E、F、G、H、I)和S共10个亚组,其中最大的一个亚组为S组,包含21个MdbZIP基因[8]。番茄bZIP基因可分成A-I共9组,分别包含16、12、6、2、12、2、3、4和12个SlbZIP基因[9]。木豆(Cajanus cajan)bZIP基因可分为A-I、S、U共11组,每组分别包含12、1、4、11、3、0、5、2、8、14和1个CcbZIP[10]。草莓(Fragaria vesca)bZIP基因可分为A-I、S、U共11组,每组分别包含8、2、7、4、2、2、5、2、6、9和3个FvbZIP[11]。葡萄(Vitis vinifera)bZIP基因可分为A-I、J、U共11组,每组分别包含13、3、10、5、4、6、2、1、2、7和2个VvbZIP[12]。
2 bZIP转录因子的结构特点与作用机制 2.1 bZIP转录因子的结构特点bZIP转录因子根据其共有的bZIP保守结构域来命名,bZIP结构域包含60-80个氨基酸,其结构特征为:(1)N末端为18个碱性氨基酸组成的碱性区域,含有一个核定位信号(Nuclear localization signal,NLS)和一个与特异DNA序列相结合的N-x7-R/K基序;(2)C末端为亮氨酸拉链区域,由于其独特的氨基酸组成,该区域往往经疏水面的相互作用形成α-螺旋形式的同源或异源二聚体[2-3]。例如,拟南芥花粉中的bZIP18、bZIP34转录因子可相互作用形成异源二聚体,共同调节脂类代谢途径,进而影响花粉壁的合成[14]。bZIP的转录激活结构域则富含脯氨酸、谷氨酰胺或酸性氨基酸[15]。
2.2 bZIP转录因子的作用机制bZIPs通过二聚化、磷酸化修饰或与其它蛋白之间的相互作用,改变其与DNA结合的特异性、亲和力,并影响其它基因的激活,同时影响bZIPs自身的稳定性和亚细胞定位[16]。bZIP转录因子形成同源或异源二聚体,并通过其碱性区域结合特定基因启动子,进而调控下游相关基因的表达[3]。植物bZIP通常优先结合ACGT核心的回文或假回文(Pseudo-palindromic)顺式作用元件,如G-box(CACGTG)、C-box(GACGTC)、A-box(TACGTA)及ABRE(CC-ACGTGG)[2-3]。
另外,某些植物转录因子可以结合H-box(CCTACC)、PB(TGAAAA)和GLM(GCN4-like mo-tif,GTGAGTCAT)等非回文结构的启动子序列,如烟草RSG、水稻RF2a和番茄VSF-1可分别结合非回文结构的启动子序列rbeS(CCCAAAGTCCAGC-TTGAAATG)、AC-I(CCCACCTACCA)和vs-1(TC-ACCAACCGTTGGATGTGG)[2-3, 17-18]。拟南芥bZIP家族bZIP的C组和S组成员通过同源或异源二聚化,在细胞核中产生多种具有不同调控特点的转录复合物,如C组bZIP成员AtbZIP10与S组(N端短、C端长)bZIP成员AtbZIP53形成异源二聚体,该二聚体与脯氨酸脱氢酶的启动子特异性结合,进而在低渗胁迫中激活脯氨酸脱氢酶的表达(图 1-A)。
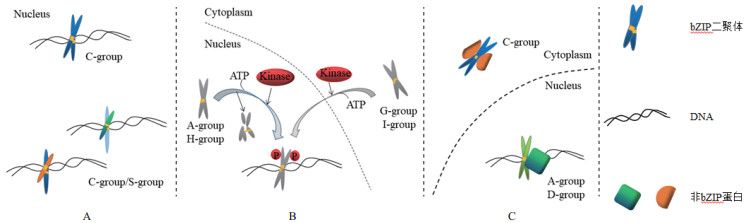
|
| 图 1 拟南芥bZIP蛋白调控机制及bZIP家族(A、C、D、G、H、I和S组)成员之间的相关性 A:同源或异源二聚化:bZIP家族C组(N端具有疏水性或酸性转录激活结构域)、S组(N端短、C端长)成员通过同源或异源二聚化,在细胞核中产生具有不同调控特点的多种转录复合物(如拟南芥);B:磷酸化修饰:磷酸化修饰可调控bZIP家族的A组(bZIP的保守序列中具有磷酸化位点)、H组(可与COP1蛋白相互作用)成员的活性和稳定性,bZIP家族的G组(N端有富含脯氨酸的转录激活结构域)和I组(基本结构域的-10位点上有保守的赖氨酸)成员在磷酸化修饰后进行核转位;C:bZIP家族的A、C和D组(bZIP可与DNA中TGACGT核心序列相结合)成员与非bZIP蛋白相互作用,在细胞核或细胞质中形成特异性调控复合物[16]。 |
磷酸化作用在植物的ABA途径及耐逆胁迫应答中具有重要作用,其产生的负电荷可改编蛋白质的构象及其排斥与吸引力,图 1-B所示为一些蛋白激酶和磷酸酶参与ABA信号途径。
图 1-C显示bZIP类转录因子的A、C、D组成员与非bZIP蛋白相互作用,在细胞核或细胞质中形成特异性调控复合物,其中A组bZIP的保守序列中具有磷酸化位点,C组bZIP的N端具有疏水性或酸性转录激活结构域,D组bZIP可与DNA中TGACGT核心序列相结合(表 2)。
拟南芥中bZIP转录因子ABF1、ABF2、ABF3和ABF4受逆境胁迫的诱导激活,并与顺式作用元件ABRE结合,即通过ABRE与bZIP蛋白之间的相互作用,调控下游诸多耐盐、耐旱相关基因的表达[19]。水稻bZIP转录因子RF2a、RF2b与水稻东格鲁杆状病毒(Rice tungro bacilliform virus,RTBV)的顺式作用元件BoxⅡ结合,即通过BoxⅡ与bZIP蛋白之间相互作用,激活水稻东格鲁杆状病毒增殖基因的表达,使得RTBV在水稻维管组织中大量复制,引发水稻东格鲁病症(Rice tungro disease,RTD),但过量表达bZIP转录因子RF2a和RF2b的水稻对东格鲁病的抗性增强[2]。在ABA、盐和干旱等胁迫诱导下,刚毛柽柳(Tamarix hispida)bZIP1转录因子与胁迫反应基因的启动子顺式元件结合,调控下游基因的表达。值得一提的是,ThbZIP1与启动子顺式元件C-box、G-box和A-box的结合亲和力依次由强到弱[20]。总之,bZIP转录因子通过与应答基因启动子区域特异性顺式调控序列相互作用,调控胁迫应答基因的表达,并对植物的耐逆性进行调控[18]。
3 bZIP参与植物非生物胁迫的应答高盐、干旱、低温以及损伤等非物胁迫对植物的生长发育具有重大影响,甚至会导致植株死亡。植物对多种非生物胁迫的响应通常需要多组分信号通路的交叉应答调控。转录因子bZIP、NAC、AP2/ERF及MYB等主要的植物转录因子家族形成了对非生物胁迫进行应答的调节网络,其中bZIP在植物对盐害、干旱、冷害、机械损伤以及渗透胁迫的应答中担任重要作用[21]。
3.1 bZIP参与盐胁迫应答盐胁迫可引起植物大范围的生理和基因表达的响应,而植物bZIP过量表达可有效提高转基因植株的耐盐性[22]。
拟南芥AtbZIP17直接或间接地激活盐胁迫应答基因如ATHB-7的表达,对盐胁迫进行响应,进而提高拟南芥的耐盐性[22]。异源表达拟南芥AtbZIP60的烟草、水稻和湿地松(Pinus elliottii)对盐胁迫的抗性增强,其超氧化物歧化酶的活性提高,而脂质过氧化反应则减慢[23]。水稻OsbZIP71、Oshox22、OsHBP1b等bZIP转录因子也参与盐胁迫的应答。OsbZIP71与渗透调控基因OsNHX1的启动子结合,将细胞质中多余的Na+、K+运至液泡,通过减少细胞质中Na+浓度,进而提高水稻耐盐性[24];Oshox22经脱落酸介导的信号途径参与盐胁迫响应,水稻Oshox22过表达使植株ABA含量增加,进而降低水稻的耐盐性[25]。较之于野生型烟草,盐处理下OsHBP1b的转基因烟草过氧化氢含量降低,超氧化物歧化酶的活性却增强,进而提高了液泡膜的稳定性和K+/Na+比,表明OsHBP1b具有较强的抗氧化损伤功能[26]。
蒺藜苜蓿(Medicago truncatula)bZIP2和bZIP-26的过量表达可提高植株耐盐性。异源表达大豆GmbZIP44、GmbZIP62与GmbZIP78的转基因拟南芥植株的耐盐性明显增强[10]。玉米HD-Zip转录因子Zmhdz10经脱落酸介导的信号途径正向调控植株的耐盐性,异源表达玉米Zmhdz10的水稻和拟南芥植株的耐盐性均明显增强[27]。在400 mmol/L NaCl处理下,辣椒(Capsicum annuum)HD-Zip转录因子CaHB1的表达明显提高,且异源表达CaHB1的转基因番茄耐盐性得到了较大提高[28]。另外,与野生型烟草相比,刚毛柽柳ThbZIP1转基因烟草的活性氧积累量、细胞死亡数目均降低,且植株保水能力增强,从而提高植株的耐盐性[29]。
3.2 bZIP参与干旱胁迫应答很多植物bZIP家族成员均参与对干旱胁迫的应答。干旱胁迫下苹果叶、根中的16个MdbZIP(MdbZIP 2、8、10、39、46、60、69、72、76、78、79、94、96、98、104和109)均具有差异性的表达,表明以上16个苹果MdbZIP均与干旱胁迫应答相关[8]。干旱处理下,大豆GmbZIP102的表达显著上调;在淹水处理后,GmbZIP102的表达则显著下调,表明GmbZIP102在植物逆境胁迫应答中具有双重作用[30]。干旱处理下,野生型水稻OsbZIP20和OsABA45均上调了大约6倍;OsDERF2基因敲除的水稻植株中,脱落酸响应基因OsbZIP20和OsABA45的表达分别上调25和120倍,表明OsDERF2通过调控其他脱落酸响应基因的表达,进而对脱落酸介导的干旱胁迫应答进行负调控。总之,bZIP家族成员在干旱胁迫下的脱落酸信号通路中也发挥重要作用[31]。
bZIP在一些农作物中的过量表达可显著提高作物耐旱性[23]。水稻OsbZIP71被干旱胁迫强烈诱导,其过量表达时可显著提高转基因水稻的抗旱性[24]。向日葵(Helianthus annuus)Hahb-4受干旱和脱落酸的调控,Hahb-4的转基因拟南芥植株的耐旱性显著增强[32]。另外,异源表达玉米HD-Zip Ⅰ家族Zmhdz10的转基因水稻耐旱性增强[27]。
聚乙二醇(PEG)可模拟干旱胁迫。苎麻(Boehmeria nivea)经聚乙二醇处理24至72 h后,其叶、根中分别有9 281、8 627个基因具有差异性表达,其中bZIP转录因子Comp28477和Comp58004可能与干旱胁迫相关[33]。在PEG处理6 h和24 h后,绿豆(Vigna radiata)bZIP转录因子VrbZIP52的表达显著上调,表明VrbZIP52可能参与植株的干旱胁迫应答[17]。在PEG处理24 h后,黄瓜根组织中CsbZIP6、CsbZIP8、CsbZIP12、CsbZIP15、CsbZIP29、CsbZIP30、CsbZIP44、vbZIP53、CsbZIP55和CsbZIP59共10个CsbZIP表达量均上调,而叶组织中的这些CsbZIP则下调,表明bZIP基因的表达具有组织特异性。另外,在PEG处理后的不同时间(0、3、12和24 h),黄瓜根中的CsbZIP12和CsbZIP44的表达水平呈逐渐上升趋势,而叶中的CsbZIP29、CsbZIP30和CsbZIP44的表达水平则呈逐渐下降趋势,说明bZIP基因的表达具有时间特异性[1]。
3.3 bZIP参与冷胁迫应答低温影响农作物的生长发育和质量,而诸多转录因子参与植物对冷胁迫应答的调控[34]。
较之于野生型拟南芥,冷处理下异源表达小麦TabZIP6以及异源表达茶树(Camellia sinensis)CsbZIP6的转基因拟南芥植株均出现了存活率降低,相对电导率增大,丙二醛含量提高,可溶性糖含量降低的现象,说明TabZIP6与CsbZIP6的表达均降低了转基因拟南芥植株的抗冻性[35]。过量表达OsABF2的水稻植株的耐冷、耐盐、耐旱性均增强[36]。但在水稻的耐冷性试验中发现,OsbZIP52的转基因植株存活率为18%,野生型存活率为78.03%,可见OsbZIP52的表达与水稻的低温耐性呈负相关[37]。
小麦bZIP转录因子TaABL(ABI-like)为水稻OsAREB2和玉米ZmABI5的同源物,TaABL的过量表达提高了小麦的耐寒性[36]。脯氨酸为植物的耐寒渗透调节物,大豆GmbZIP44、GmbZIP62和GmbZIP78可促进脯氨酸的合成,进而增强植物对冷、冻胁迫的耐性。同时,GmbZIP44、GmbZIP62和GmbZIP78可激活其下游基因ERF5、KIN1、CORl5A和COR78的表达,提高植株的耐盐和耐寒性[15]。白菜(Brassica rapa)受冷处理后,27个BrbZIP的表达均显著上调,其中Bra000256、Bra003320、Bra004689、Bra011648、Bra020735和Bra023540可能为冷应答的关键基因[5]。低温处理下,胡萝卜(Daucus carota)根中的bZIP类蛋白Lip(Low temperature-induced protein)的表达上调,从而增强了胡萝卜的抗寒性[38]。另外,番茄的启动子序列分析发现,51个HD-Zip(SlHZ01-51)的启动子中均含有低温响应元件LTRE(Low temperature responsive element),表明这51个番茄HD-Zip均参与植株对冷胁迫的响应[39]。
3.4 bZIP参与渗透胁迫应答脯氨酸脱氢酶1(Proline dehydrogenase 1,PDH1)为脯氨酸降解途径中的第一限速酶。拟南芥种子中ARR18调控蛋白与抑制种子萌发的AtbZIP63相互作用,共同调节脯氨酸脱氢酶1的活性,进而提高植株对低渗胁迫的耐受力[40]。低渗胁迫下,拟南芥bZIP转录因子VIP1(AtbZIP51)在细胞核中快速积累,以响应低渗胁迫[5, 41]。同时,VIP1与bZIP29可形成异源二聚体,该二聚体通过与低渗应答基因CYP707A1、CYP707A3启动子中的低渗响应元件(AGCTGK)结合,共同响应渗透胁迫[42]。使用NaHCO3(50 mmol/L)处理野生大豆(Glycine soja),发现其中一编码HD-Zip蛋白的Gshdz4基因在叶片和根中高表达;而异源表达Gshdz4的拟南芥植株对NaHCO3、KHCO3胁迫的耐受力明显提高,说明Gshdz4参与了植株的渗透胁迫应答[43]。鉴于高盐、干旱和低温均可引起对植物的渗透胁迫,因而bZIP不仅在植物应答高盐、干旱及冷胁迫中担任重要作用,同时对渗透胁迫进行响应(图 2)。

|
| 图 2 植物bZIP类转录因子在非生物胁迫中的调控功能 |
bZIP参与调控某些与ABA途径相关的信号传导通路。在拟南芥种子萌发及开花期间,bZIP转录因子ABI5(ABA insensitive 5)和ABFs(ABRE binding factors)是调控ABA信号的关键因子[44]。拟南芥abi5突变体对ABA敏感性降低,由于ABA抑制种子的萌发,因而ABI5可通过ABA介导的信号传导通路,激活种子中特定基因的表达,进而促进种子的萌发[44]。研究发现,AtbZIP63、AtbZIP3和ABI8可能在ABA介导的葡萄糖转运中发挥重要作用[5, 36]。异源表达番茄SIAREB的拟南芥植株,通过产生AtRd29A、AtCOR47及SICI7-like脱水蛋白,对ABA介导的干旱和盐胁迫进行应答[36]。
植物中的ABF2/AREB1、ABF4/AREB2和ABF3均参与ABA信号的应答调控[44]。水稻OsbZIP52/RISBZ5、OsAREB1/OsABF2、OsABI5/OREB1和OSBZ8通过与靶基因的ABRE元件结合,调控植物ABA响应基因的表达[3]。Oshox22过量表达可提高水稻植株的ABA含量,表明Oshox22调控ABA的生物合成,同时该基因可能通过ABA介导的信号传导途径,调控植株对干旱和盐胁迫的应答[25]。另外,ABA可诱导黄花蒿(Artemisia annua)AabZIP1的表达来激活下游ADS和CYP71AV1的表达,进而调控青蒿素的生物合成[45]。
3.6 bZIP参与机械损伤胁迫应答bZIP可参与植物对机械损伤胁迫的应答。拟南芥AtbZIP29在增生组织中特异表达,其通过与细胞周期调控因子及细胞壁再生相关基因的特异性结合,调控叶片和根分生组织的细胞数量,进而促进组织再生。细胞分裂周期中,bZIP29可与bZIP69相互作用并形成二聚体,从而在侧根的根原基、根冠以及茎尖的分生组织中发挥作用,参与调控机械损伤的胁迫应答[42]。
伤口可强烈诱导向日葵(Helianthus annuus)HD-Zip转录因子HaHB4的表达。较之于对照植株,向日葵HaHB4的转基因拟南芥受伤害和细菌感染时,其茉莉酸和乙烯含量明显提高,水杨酸含量则降低。表明HaHB4通过调控植物激素如乙烯或植物内源生长调节物质茉莉酸和水杨酸的产生,参与机械损伤的胁迫应答,进而降低对植株的伤害[40]。另外。损伤诱导野生型烟草HD-ZipI转录因子NaHD20的表达,但烟草NaHD20基因沉默植株受伤害后,其茉莉酸、乙烯和水杨酸的含量与对照植物相比无明显差异,表明NaHD20在烟草的机械损伤应答中并未影响植物激素的合成,其机制尚未知[46]。
机械等物理损伤会导致树干韧皮部的坏死(Trunk phloem necrosis,TPN),降低天然橡胶的产量。在天然橡胶受到损伤时,miR166及其靶基因HD-ZIP Ⅲ通过调控树干木质部和韧皮部的分化,促进树皮的再生,进而提高橡胶的产量[47]。
4 bZIP参与植物生物胁迫应答bZIP转录因子参与虫害、病原体感染等生物胁迫的应答[48]。水稻OsbZIP1则可能通过水杨酸(Salicylic acid,SA)、茉莉酸(Jasmonic acid,JA)及ABA信号转导通路,增强对稻瘟病菌(Magnaporthe grisea)的抗性[49]。小麦TabZIP1基因可能通过乙烯/茉莉酸甲酯(Methyl jasmonate,MeJA)介导的信号转导通路,增强对丁香假单胞菌(Pseudomonas syringae pv)的抗性[50]。辣椒CabZIP1的转基因拟南芥植株对丁香假单胞菌的抗性增强,其耐盐、耐旱性也均有提高[51]。向日葵(Helianthus annuus)HD-Zip转录因子HaHB10通过增加植株中水杨酸(Salicylic acid,SA)的含量和降低茉莉酸(Jasmonic acid,JA)的含量,对丁香假单胞菌等生物胁迫进行应答;同时,HAHB10可激活下游基因HASEP3和HAFT的表达,促进植株的开花[52]。过量表达HD-Zip转录因子ATHB1的拟南芥突变体植株,对昆虫如桃蚜(Myzus persicae)以及病原菌如白粉菌(Oidium neolycopersici)、霜霉菌(Hyaloperonospora arabidopsidis)的抗性增强,但其保留了对丁香假单胞菌的易感性,说明ATHB1在植物胁迫应答中可能具有双重作用[53-54]。另外,AtbZIP10通过与顺式作用元件G-box和C-box的结合,负向调控拟南芥植株对病原菌及其它胁迫的抗性;拟南芥LSD1(Lesions simulating disease resistance 1)蛋白可抑制细胞死亡,研究发现lsd1基因突变体植株中AtbZIP10的过表达促进细胞的非程序化死亡[55]。
灰翅夜蛾(Spodoptera littoralis)和草地贪夜蛾(Spodoptera frugiperda)的幼虫侵害拟南芥和玉米,导致植株受伤害;较之于野生型植株,向日葵HAHB4的转基因拟南芥和玉米植株上的灰翅夜蛾和草地贪夜蛾的幼虫数量减少,且灰翅夜蛾和草地贪夜蛾幼虫的体重也有所降低,表明向日葵HAHB4增强了转基因拟南芥和玉米的抗虫能力[56]。
5 展望植物bZIP转录因子分布广泛,种类繁多,其在植物的发育、种子成熟,以及胁迫信号转导和病原体的防御中担任重要作用。目前,国内外对bZIP的研究主要集中在模式植物拟南芥、经济作物水稻、大麦和高粱等,其研究方向则集中在逆境胁迫中的调控机制和功能。但由于不同植物中bZIP家族成员的数量不等,且同一家族成员在不同物种中也可能具有不同的功能。因此,尚无法全面揭示植物中某一或某些bZIP的作用机制和功能。
bZIP参与调控种子成熟、休眠、植物发育和衰老等生物学过程,在非生物胁迫如盐害、干旱、冷害、渗透胁迫及机械损伤中均具有重要的作用。同时,bZIP可调控水杨酸、茉莉酸及ABA信号通路,在植株抵抗虫害和病原体感染中也具有不可或缺的功能。本文对bZIP的结构特点、bZIP家族及分类特征、作用机制及其在生物、非生物胁迫应答中的功能等进行了全面的综述。因而,对于深入理解bZIP在植物应答多种生物和非生物胁迫中的复杂调控网络具有指导意义。另外,鉴于bZIP转录因子在植物胁迫应答中的重要功能,运用基因工程手段,在作物中转基因过量表达bZIP或调控特定bZIP的表达水平,可实现作物多重抗逆性及综合品质的提高,进而培育优良作物新品种。
鉴于bZIP生物学功能的多样性和重要性,未来可进一步扩大bZIP转录因子的研究领域,加强对bZIP在更广泛物种中的深入研究,发掘其更多的理论和应用价值[12]。同时,更多地关注bZIP的应答调控途径,通过植株示踪技术揭示bZIP网络调控机制动态过程[57],系统生物学方法对bZIP的上、下游基因及其所构成的调控网络进行深入的探究,揭示bZIP应答环境胁迫的网络调控机制[36, 57]。例如,通过研究根发育途径与盐胁迫调控网络的交叉响应机理,全面阐明盐胁迫影响根系发育的分子机制[58]。总之,对bZIP的分布、分类、结构和功能的全面了解,并实现其研究的全局、动态和立体化,将是未来的研究方向。
| [1] |
Baloglu MC, Eldem V, Hajyzadeh M, et al. Genome-wide analysis of the bZIP transcription factors in cucumber[J]. PLoS One, 2014, 9(4): e96014. DOI:10.1371/journal.pone.0096014 |
| [2] |
Ali Z, Sarwat SS, Karim I, et al. Functions of plant's bZIP transcription factors[J]. Pakistan Journal of Agricultural Sciences, 2016, 53(2): 303-314. DOI:10.21162/PAKJAS |
| [3] |
E ZG, Zhang YP, Zhou JH, et al. Mini review roles of the bZIP gene family in rice[J]. Genet Mol Res, 2014, 13(2): 3025-3036. DOI:10.4238/2014.April.16.11 |
| [4] |
Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290(5499): 2105-2110. DOI:10.1126/science.290.5499.2105 |
| [5] |
Hwang I, Jung HJ, Park JI, et al. Transcriptome analysis of newly classified bZIP transcription factors of Brassica rapa in cold stress response[J]. Genomics, 2014, 104(3): 194-202. |
| [6] |
Nijhawan A, Jain M, Tyagi AK, et al. Genomic survey and gene expression analysis of the basic leucine zipper transcription factor family in rice[J]. Plant Physiology, 2008, 146(2): 333-350. |
| [7] |
Jin Z, Xu W, Liu A. Genomic surveys and expression analysis of bZIP gene family in castor bean (Ricinus communis L.)[J]. Planta, 2014, 239(2): 299-312. DOI:10.1007/s00425-013-1979-9 |
| [8] |
Li YY, Meng D, Li MJ, et al. Genome-wide identification and expression analysis of the bZIP gene family in apple (Malus domestica)[J]. Tree Geneticsa & Genomes, 2016, 12(4): 82. |
| [9] |
Li D, Fu F, Zhang H, et al. Genome-wide systematic characterization of the bZIP transcriptional factor family in tomato (Solanum lycopersicum L.)[J]. BMC Genomics, 2015, 16: 771. DOI:10.1186/s12864-015-1990-6 |
| [10] |
Wang Z, Cheng K, Wan L, et al. Genome-wide analysis of the basic leucine zipper (bZIP) transcription factor gene family in six legume genomes[J]. BMC Genomics, 2015, 16: 1053. DOI:10.1186/s12864-015-2258-x |
| [11] |
Wang XL, Chen X, Yang TB, et al. Genome-wide identification of bZIP family genes Involved in drought and heat stresses in strawberry (Fragaria vesca)[J]. International Journal of Genomics, 2017, 2017: 3981031. |
| [12] |
Liu JY, Chen NN, Chen F, et al. Genome-wide analysis and expression profile of the bZIP transcription factor gene family in grapevine (Vitis vinifera)[J]. BMC Genomics, 2014, 15: 281. DOI:10.1186/1471-2164-15-281 |
| [13] |
Jakoby M, Weisshaar B, Drögelaser W, et al. bZIP transcription factors in Arabidopsis[J]. Trends in Plant Science, 2002, 7(3): 106-111. |
| [14] |
Gibalova A, Steinbachova L, Hafidh S, et al. Characterization of pollen-expressed bZIP protein interactions and the role of ATbZIP18 in the male gametophyte[J]. Plant Reprod, 2017, 30(1): 1-17. |
| [15] |
Liao Y, Zou HF, Wei W, et al. Soybean GmbZIP44, GmbZIP62 and GmbZIP78 genes function as negative regulator of ABA signaling and confer salt and freezing tolerance in transgenic Arabidopsis[J]. Planta, 2008, 228(2): 225-240. DOI:10.1007/s00425-008-0731-3 |
| [16] |
Schutze K, Harter K, Chaban C. Post-translational regulation of plant bZIP factors[J]. Trends in Plant Science, 2008, 13(5): 247-255. |
| [17] |
Wang L, Zhu J, Li X, et al. Salt and drought stress and ABA responses related to bZIP genes from V.radiata and V.angularis[J]. Gene, 2018, 651: 152-160. DOI:10.1016/j.gene.2018.02.005 |
| [18] |
Sornaraj P, Luang S, Lopato S, et al. Basic leucine zipper (bZIP) transcription factors involved in abiotic stresses:A molecular model of a wheat bZIP factor and implications of its structure in function[J]. Biochim Biophys Acta, 2016, 1860(1 Pt A): 46-56. |
| [19] |
Yoshida T, Fujita Y, Maruyama K, et al. Four Arabidopsis AREB/ABF transcription factors function predominantly in gene expression downstream of SnRK2 kinases in abscisic acid signalling in response to osmotic stress[J]. Plant Cell and Environment, 2015, 38(1): 35-49. |
| [20] |
Nie X, Ji X, Liu Y, et al. Elucidation of the specific formation of homo-and heterodimeric forms of ThbZIP1 and its role in stress[J]. International Journal of Molecular Sciences, 2014, 15(6): 10005-10017. DOI:10.3390/ijms150610005 |
| [21] |
Golldack D, Li C, Mohan H, et al. Tolerance to drought and salt stress in plants:Unraveling the signaling networks[J]. Frontiers in Plant Science, 2014, 5: 151. |
| [22] |
Liu J, Srivastava R, Che P, et al. Salt stress in Arabidopsis utilize a signal transduction pathway related to endoplasmic reticulum stress signaling[J]. Plant Journal, 2007, 51(5): 897-909. DOI:10.1111/j.1365-313X.2007.03195.x |
| [23] |
Tang W, Page M, Fei YJ, et al. Overexpression of AtbZIP60deltaC gene alleviates salt-induced oxidative damage in transgenic cell cultures[J]. Plant Molecular Biology Reporter, 2012, 30(5): 1183-1195. DOI:10.1007/s11105-012-0437-3 |
| [24] |
Liu C, Mao B, Ou S, et al. OsbZIP71, a bZIP transcription factor, confers salinity and drought tolerance in rice[J]. Plant Mol Biol, 2014, 84(1/2): 19-36. |
| [25] |
Zhang S, Haider I, Kohlen W, et al. Function of the HD-Zip I gene Oshox22 in ABA-mediated drought and salt tolerances in rice[J]. Plant Molecular Biology, 2012, 80(6): 571-585. DOI:10.1007/s11103-012-9967-1 |
| [26] |
Lakra N, Nutan KK, Das P, et al. A nuclear-localized histone-gene binding protein from rice (OsHBP1b) functions in salinity and drought stress tolerance by maintaining chlorophyll content and improving the antioxidant machinery[J]. Journal of Plant Physiology, 2015, 176: 36-46. DOI:10.1016/j.jplph.2014.11.005 |
| [27] |
Zhao Y, Ma Q, Jin X, et al. A novel maize homeodomain-leucine zipper (HD-Zip) I gene, Zmhdz10, positively regulates drought and salt tolerance in both rice and Arabidopsis[J]. Plant Cell Physiology, 2014, 55(6): 1142-1156. DOI:10.1093/pcp/pcu054 |
| [28] |
Oh SK, Yoon J, Choi GJ, et al. Capsicum annuum homeobox 1(CaHB1) is a nuclear factor that has roles in plant development, salt tolerance, and pathogen defense[J]. Biochemical and Biophysical Research Communications, 2013, 442(1/2): 116-121. |
| [29] |
Kumar J, Singh S, Singh M, et al. Transcriptional regulation of salinity stress in plants:A short review[J]. Plant Gene, 2017, 11: 160-169. DOI:10.1016/j.plgene.2017.04.001 |
| [30] |
Zhang M, Liu Y, Shi H, et al. Evolutionary and expression analyses of soybean basic leucine zipper transcription factor family[J]. BMC Genomics, 2018, 19(1): 159. DOI:10.1186/s12864-018-4511-6 |
| [31] |
Guo Y, Huang R, Duan L, et al. The APETALA2/ethylene-responsive factor transcription factor OsDERF2 negatively modulates drought stress in rice by repressing abscisic acid responsive genes[J]. Journal of Agricultural Science, 2017, 155(6): 1-12. |
| [32] |
Manavella PA, Arce AL, Dezar CA, et al. Cross-talk between ethylene and drought signalling pathways is mediated by the sunflower Hahb-4 transcription factor[J]. Plant Journal, 2006, 48(1): 125-137. DOI:10.1111/tpj.2006.48.issue-1 |
| [33] |
An X, Chen J, Zhang J, et al. Transcriptome profiling and identification of transcription factors in ramie (Boehmeria nivea L.Gaud) in response to PEG treatment, using illumina paired-end sequencing technology[J]. International Journal of Molecular Sciences, 2015, 16(2): 3493-3511. DOI:10.3390/ijms16023493 |
| [34] |
Chinnusamy V, Zhu JH, Zhu JK. Gene regulation during cold acclimation in plants[J]. Physiologia Plantarum, 2006, 126(1): 52-61. |
| [35] |
Cai W, Yang Y, Wang W, et al. Overexpression of a wheat (Tritic-um aestivum L.) bZIP transcription factor gene, TabZIP6, decreased the freezing tolerance of transgenic Arabidopsis seedlings by down-regulating the expression of CBFs[J]. Plant Physiol Biochem, 2018, 124: 100-111. DOI:10.1016/j.plaphy.2018.01.008 |
| [36] |
Banerjee A, Roychoudhury A. Abscisic-acid-dependent basic leucine zipper (bZIP) transcription factors in plant abiotic stress[J]. Protoplasma, 2017, 254(1): 3-16. DOI:10.1007/s00709-015-0920-4 |
| [37] |
Liu C, Wu Y, Wang X. bZIP transcription factor OsbZIP52/RISBZ5:a potential negative regulator of cold and drought stress response in rice[J]. Planta, 2012, 235(6): 1157-1169. DOI:10.1007/s00425-011-1564-z |
| [38] |
Ito K, Kusano T, Tsutsumi KI. A cold-inducible bZIP protein gene in radish root regulated by calcium-and cycloheximide-mediated signals[J]. Plant Science, 1999, 142: 57-65. DOI:10.1016/S0168-9452(98)00250-7 |
| [39] |
Zhang Z, Chen X, Guan X, et al. A genome-wide survey of homeodomain-leucine zipper genes and analysis of cold-responsive HD-Zip I members expression in tomato[J]. Bioscience Biotechnology and Biochemistry, 2014, 78(8): 1337-1349. DOI:10.1080/09168451.2014.923292 |
| [40] |
Veerabagu M, Kirchler T, Elgass K, et al. The interaction of the Arabidopsis response regulator ARR18 with bZIP63 mediates the regulation of PROLINE DEHYDROGENASE expression[J]. Molecular Plant, 2014, 7(10): 1560-1577. DOI:10.1093/mp/ssu074 |
| [41] |
Tsugama D, Liu S, Takano T. The bZIP protein VIP1 is involved in touch responses in Arabidopsis roots[J]. Plant Physiology, 2016, 171(2): 1355-1365. |
| [42] |
Leene JV, Blomme J, Kulkarni SR, et al. Functional characterization of the Arabidopsis transcription factor bZIP29 reveals its role in leaf and root development[J]. Journal of Experimental Botany, 2016, 67(19): 5825-5840. DOI:10.1093/jxb/erw347 |
| [43] |
Cao L, Yu Y, Chen C, et al. A novel Glycine soja homeodomain-leucine zipper (HD-Zip) I gene, Gshdz4, positively regulates bicarbonate tolerance and responds to osmotic stress in Arabidopsis[J]. BMC Plant Biology, 2016, 16(1): 184. |
| [44] |
Hossain MA, Cho JI, Han M, et al. The ABRE-binding bZIP transcription factor OsABF2 is a positive regulator of abiotic stress and ABA signaling in rice[J]. Journal of Plant Physiology, 2010, 167(17): 1512-1520. DOI:10.1016/j.jplph.2010.05.008 |
| [45] |
Zhang F, Fu X, Lv Z, et al. A basic leucine zipper transcription factor, AabZIP1, connects abscisic acid signaling with artemisinin biosynthesis in Artemisia annua[J]. Molecular Plant, 2015, 8(1): 163-175. DOI:10.1016/j.molp.2014.12.004 |
| [46] |
Re DA, Dezar CA, Chan RL, et al. Nicotiana attenuata NaHD20 plays a role in leaf ABA accumulation during water stress, benzylacetone emission from flowers, and the timing of bolting and flower transitions[J]. Journal of Experimental Botany, 2011, 62(1): 155-166. DOI:10.1093/jxb/erq252 |
| [47] |
Lertpanyasampatha M, Viboonjun U, Kongsawadworakul P, et al. Differential expression of microRNAs and their targets reveals a possible dual role in physiological bark disorder in rubber tree[J]. Journal of Plant Physiology, 2014, 171(13): 1117-1126. DOI:10.1016/j.jplph.2014.05.001 |
| [48] |
Amorim LL, Da FDSR, Guida-Santos M, et al. Transcription factors involved in plant resistance to pathogens[J]. Current Protein & Peptide Science, 2017, 18(4): 335-351. |
| [49] |
Meng XB, Zhao WS, Lin RM, et al. Identification of a novel rice bZIP-type transcription factor gene, OsbZIP1, involved in response to infection of Magnaporthe grisea[J]. Plant Molecular Biology Reporter, 2005, 23(3): 301-302. DOI:10.1007/BF02772762 |
| [50] |
Zhang Y, Zhang G, Xia N, et al. Cloning and characterization of a bZIP transcription factor gene in wheat and its expression in response to stripe rust pathogen infection and abiotic stresses[J]. Physiological and Molecular Plant Pathology, 2008, 73(4/5): 88-94. |
| [51] |
Lee SC, Choi HW, Hwang IS, et al. Functional roles of the pepper pathogen-induced bZIP transcription factor, CAbZIP1, in enhanced resistance to pathogen infection and environmental stresses[J]. Planta, 2006, 224(5): 1209-1225. DOI:10.1007/s00425-006-0302-4 |
| [52] |
Dezar CA, Giacomelli JI, Manavella PA, et al. HAHB10, a sunflower HD-Zip Ⅱ transcription factor, participates in the induction of flowering and in the control of phytohormone-mediated responses to biotic stress[J]. Journal of Experimental Botany, 2011, 62(3): 1061-1076. DOI:10.1093/jxb/erq339 |
| [53] |
Rabara RC, Tripathi P, Rushton PJ. The potential of transcription factor-based genetic engineering in improving crop tolerance to drought[J]. OMICS, 2014, 18(10): 601-614. DOI:10.1089/omi.2013.0177 |
| [54] |
Gao D, Appiano M, Huibers RP, et al. Activation tagging of ATHB13 in Arabidopsis thaliana confers broad-spectrum disease resistance[J]. Plant Molecular Biology, 2014, 86(6): 641-653. DOI:10.1007/s11103-014-0253-2 |
| [55] |
Kaminaka H, Näke C, Epple P, et al. bZIP10-LSD1 antagonism modulates basal defense and cell death in Arabidopsis following infection[J]. Embo Journal, 2006, 25(18): 4400-4411. DOI:10.1038/sj.emboj.7601312 |
| [56] |
Manavella PA, Dezar CA, Bonaventure G, et al. HAHB4, a sunflower HD-Zip protein, integrates signals from the jasmonic acid and ethylene pathways during wounding and biotic stress responses[J]. Plant Journal for Cell and Molecular Biology, 2010, 56(3): 376-388. |
| [57] |
Llorca CM, Potschin M, Zentgraf U. bZIPs and WRKYs:two large transcription factor families executing two different functional strategies[J]. Frontiers in Plant Science, 2014, 5: 169. |
| [58] |
Gruber V, Blanchet S, Diet A, et al. Identification of transcription factors involved in root apex responses to salt stress in Medicago truncatula[J]. Mol Genet Genomics, 2009, 281(1): 55-66. DOI:10.1007/s00438-008-0392-8 |