环阿屯醇(Cycloartenol)广泛分布于植物中, 是重要的药效物质, 近年来研究发现它具有明显的抗炎[1-2]、抗氧化[3]、抗肿瘤[4-5]、调节胆固醇[6]等药理活性。此外, 它还是重要的药物中间体和先导化合物, 是植物甾醇、甾体皂苷等化合物半化学合成及生物合成的关键底物。环阿屯醇合酶(Cycloartenol Synthase, 简称CAS)作为合成环阿屯醇的关键调控基因, 也是很多植物甾醇及甾体类物质生物合成途径中的重要环化酶[7]。目前, 已有20多种植物的CAS基因被分离和克隆出来, 如拟南芥[8](Arabidopsis thaliana)、盾叶薯蓣[9](Dioscorea zingiberensis)、光果甘草[10](Glycyrrhiza glabra)、黄芪[11](Astragalus membranaceus)、丹参[12](Salvia miltiorrhiza)和刺五加[13](Eleutherococcus senticosus)等, 并通过异源表达进行功能验证。在生物体内, 三萜类物质和植物甾醇均通过异戊二烯途径合成, 且都源于同一前体物质-2, 3-氧化鲨烯[14]。Liang等[15]研究表明, CAS可与β-AS竞争2, 3-氧化鲨烯, 对CAS基因的表达进行抑制后, 减少了植物甾醇合成, 使2, 3-氧化鲨烯更多的进入到三萜皂苷代谢支流, 从而间接提高了三萜皂苷的产量, 说明CAS不仅是植物甾醇合成过程中的关键酶, 同时又是三萜物质合成的重要调控位点。虽然目前已在CAS基因的分离、纯化、克隆及表达分析上取得了一定的进展, 但是对于CAS的酶结构以及催化2, 3-氧化鲨烯发生环化的机理尚不清楚。因此, 该酶性质及催化机理的研究对次生代谢调控具有重要意义。
CAS属于氧化鲨烯环化酶(OSCs)大家族的成员之一, OSC催化的环化作用是以2, 3-氧化鲨烯为底物经过一系列的质子化、环化、重排和去质子化作用完成的。然而, 由于OSCs分子结构较大且属于膜蛋白, 晶体结构获得困难[16], 目前, 只有来自人、牛和酵母的羊毛甾醇合酶被纯化获得晶体结构, 通过三维晶体结构研究催化机理及活性位点的工作无法开展[17], 从而阻碍了酶进一步的改进及应用。同源模建是根据模板蛋白, 预测目标蛋白的三维结构。对于尚未或者无法获得实验晶体结构的蛋白质, 同源模建无疑是一种重要而有效的辅助手段。近年来, 越来越多的研究者以已解析的晶体结构为模板, 结合生物化学、有机化学和计算机辅助设计等开展OSCs催化活性的研究, 以深入理解OSCs的催化环化/重排反应机理, 产物多样性以及活性功能氨基酸残基。
综上所述, CAS反应机制的研究是近年来一个极富挑战性的难点和热点, 揭示其催化的活性位点及环化的机制, 具有重要的理论意义。同时该酶能竞争三萜合成的共同底物2, 3-氧化鲨烯, 实现对次级代谢支路的调控, 具有十分广阔的应用前景。本研究以罗汉果CAS为对象开展研究, 通过分析序列, 采用同源建模及分子模拟的软件模拟预测CAS三维结构和活性中心, 以及其催化底物2, 3-氧化鲨烯的环化的机制, 旨为酶的改造及其代谢调控奠定基础。
1 材料与方法 1.1 材料 1.1.1 同源建模序列来源及模板选择CAS氨基酸序列为本实验室前期研究所获得[18], 已登录于NCBI数据库, 基因登录号为HQ128566。基于CAS氨基酸序列, 利用Blast程序在蛋白质结构数据库(Protein data bank, PDB)中搜寻与其氨基酸序列一致性大于或等于30%的X射线衍射晶体结构用于三维结构的建模。
1.1.2 受体结构准备上文同源建模得到的CAS酶结构用于下面的分子对接。初始结构用AutoDockTools 1.5.6进行处理, 保存蛋白原有电荷, 并生成pdbqt文件用于对接。
1.1.3 配体结构准备小分子配体2, 3-氧化鲨烯以及碳正离子中间体的三维结构如图 1所示。使用MOPAC程序优化分子结构并计算AM1原子电荷用于后续的分子对接, 然后用AutoDock Tools 1.5.6处理配体结构, 处理后生成相应的pdbqt文件用于对接。
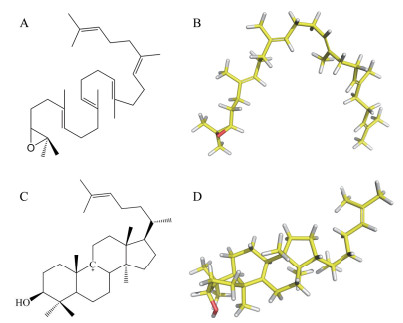
|
| 图 1 2, 3-氧化鲨烯以及碳正离子中间体的分子结构 A和B为2,3-氧化鲨烯分子结构;C和D为碳正离子中间体分子结构,左侧为中间体的二维结构,右侧为中间体的三维结构,以棍棒模型显示,黄色为碳原子,白色为氢原子,红色为氧原子 |
根据获取的模板蛋白三维结构文件以及序列比对文件, 根据序列的相似性推测结构的相似性, 使用Modeller 9.17对CAS进行同源模建, 从而获取目标蛋白CAS的三维结构。所有计算均在MolDesigner分子模拟平台(MolDesigner Molecular Simulation Platform)上完成。
1.2.2 模型评价本文分别用PROCHECK和Verify-3D程序对优化后模型蛋白结构进行评价。Ramacha-ndran plot用于阐述蛋白质或肽立体结构中肽键内α碳原子和羰基碳原子间的键的旋转度对α碳原子和氮原子间键的旋转度, 主要用来指明蛋白质或肽类中氨基酸残基的允许和不允许的构象。Ramachandran plot主要分为三个区域, 允许区(红色区域), 最大允许区(黄色区域), 不允许区(空白区域)。
1.2.3 分子对接分子对接采用AutoDock 4.2.6程序实现。首先确定配体对接位点, 对接口袋中心点坐标为(26.264, 70.645, 14.631), 网格数目分别为40×60×50, 格点间距为0.375埃(Ångström, 符号Å), 对接口袋区域的格点能量采用AutoGrid程序进行计算。对接时, 构象搜索策略采用拉马克遗传算法(Lamarckian Genetic Algorithm, LGA), 关键参数如下:对接次数(Number of GA Runs)设置为100, 种群数目(Population Size)设置为150, 最大迭代次数(Maximum Number of evaluations)设置为25 000 000, 其余参数采用默认值。所有计算均在MolDesigner分子模拟平台(MolDesigner Molecular Simulation Platform)上完成。
2 结果 2.1 同源建模 2.1.1 模板选择通过在PDB数据库中搜寻发现, 在下载的OSCs氨基酸序列中, 仅人的羊毛甾醇合酶1W6J和1W6K能获得一致性大于或等于30%的晶体模板, 也仅这2个蛋白可以进行同源建模。图 2为CAS和2个模板的序列比对结果, 模板覆盖率为86%, 序列一致性为49%。

|
| 图 2 CAS与模板1W6J、1W6K的序列比对 |
CAS模型的Ramachandran plot显示模型中的氨基酸残基如图 3所示, 90.8%的氨基酸位于允许区, 8.7%的氨基酸残基位于最大允许区域, 只有0.5%的氨基酸残基位于扭转角的禁止区域。模型中99.5%的蛋白质残基的二面角都在合理的范围之内, 符合立体化学能量规则。

|
| 图 3 模建CAS酶的Ramachandran plot |
Verify-3D是UCLA的David Eisenberg教授开发的一种基于"穿线"法的模型评价程序。该方法采用3D-1D的打分函数来检测所构建模型与自身氨基酸序列的匹配度关系。80%残基的Verify score大于0.2即认为模型合理。Verify-3D图显示了序列中每个氨基酸的打分。从图 4可知, CAS结构中的92.34%氨基酸残基在Verify-3D中Verify score平均值大于0.2, 模型通过了Verify 3D检测, 表明所建模型质量较好。
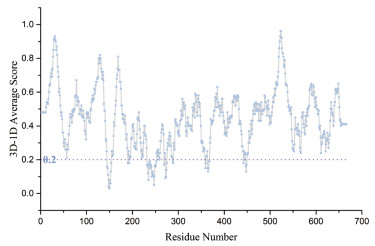
|
| 图 4 CAS的Verify-3D图 |
为进一步讨论建模所得模型的合理性和分析CAS的催化活性中心结构, 本文对1W6J晶体蛋白结构与CAS模型进行了叠合对比。通过CAS模型与1W6J晶体结构的叠合对比分析可得(图 5), CAS酶模型与晶体蛋白间的RMSD为0.246 Å, CAS能够与晶体模板很好重合。晶体模板上的催化中心主要由Asp455、Cys456、Cys533、Tyr503、His232关键残基构成。通过叠合对比, CAS酶上的Asp491、Cys492、Cys570、Tyr540、His265残基与晶体模板的关键残基相互重合。
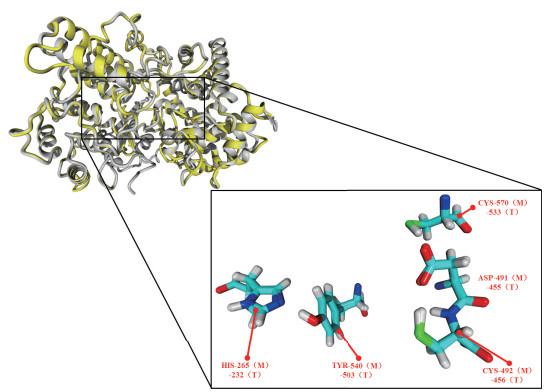
|
| 图 5 模建CAS酶与晶体蛋白1W6J的叠合图示 注:蛋白显示为条带状模型,黄色为CAS酶,灰色为晶体蛋白。催化位点的残基显示为棍棒模型。残基序号后括号中的M代表同源建模所得模型,T代表同源建模所用的模板 |
对接结果表明2, 3-氧化鲨烯分子能够结合到CAS酶的活性空腔, 结合自由能为-11.15 kcal/mol。为进一步探索CAS酶对2, 3-氧化鲨烯的催化机制, 我们对CAS酶和2, 3-氧化鲨烯的相互作用进行了分析。如图 6-A、C所示, 2, 3-氧化鲨烯上的环氧与CAS酶的Asp491羧酸氢原子可形成一个氢键, 键长为1.8 Å, 该氢键引发后续的环化反应。2, 3-氧化鲨烯的疏水链与CAS酶结合空腔中的His265、Pro375、Val376、Phe480、Phe734残基间可形成疏水作用, 疏水作用可进一步加强配体与CAS的结合, 有利于反应物2, 3-氧化鲨烯稳定结合在酶的活性空腔中。
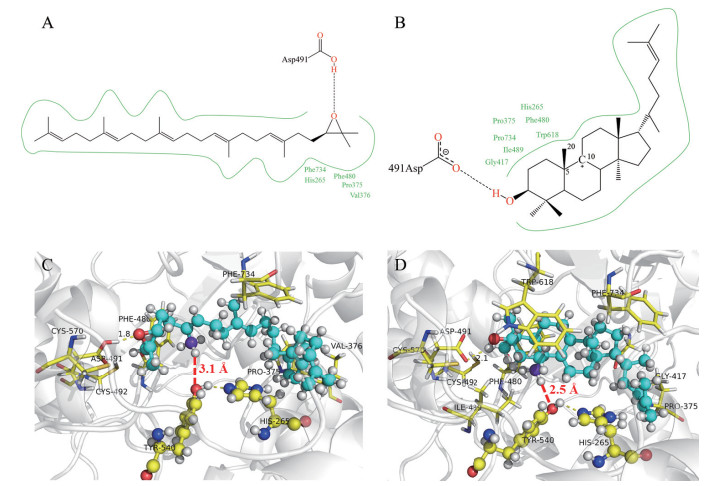
|
| 图 6 小分子与CAS酶的相互作用分析 A和B:2,3-氧化鲨烯、碳正离子中间体与残基二维相互作用模式分析;C和D:2,3-氧化鲨烯、碳正离子中间体与残基三维相互作用模式分析(二维作用模式分析中,绿色曲线以及绿色残基为形成疏水作用残基,黑色虚线为氢键。三维作用模式中,蛋白显示为透明灰色条带状模型,配体为青色棍棒模型,C27显示为蓝紫色球型模型,TYR540和HIS265显示为黄色球棍模型,其余残基为黄色棍棒模型,红色虚线为氢键作用) |
在CAS酶催化2, 3-氧化鲨烯过程中, C27和C11间会形成单键, 进而生成产物环阿屯醇。据此我们推测, 原本环化后的C27碳正离子中间体, 经过碳正离子迁移后形成C11位的碳正离子中间体(如图 6-B所示), 而C27位上的氢被结合位点附近的残基夺走, 进而C27、C11形成单键。在2, 3-氧化鲨烯与CAS酶催化位点的对接结构中, 2, 3-氧化鲨烯能够进入到催化位点, 且Tyr540的羟基氧原子与C27位上的氢原子间距离为3.1 Å(如图 6-C红色虚线所示), 表明Tyr540可能具有接收C27上的氢质子进而催化C27、C11成键的作用。
碳正离子中间体与CAS酶的结合构象中, 中间体C27原子(如图 6-D所示, C27显示为蓝紫色球型)上的一个氢原子与Tyr540的羟基氧原子的距离为2.5 Å(如图 6-D红色虚线所示), 该距离比反应物体系中的距离明显减小, 表明去质子化的Tyr540羟基氧原子可能夺取C27上的氢原子, 使得C27与C11间形成单键生成产物环阿屯醇。
另外, 我们进一步发现CAS酶活性空腔内的Tyr540残基上的羟基氢原子能够与His265的氮原子形成氢键。说明在催化过程中, Tyr540羟基上的氢原子可能转移到His265的氮原子上, 去质子化的Tyr540作为氢质子受体接受2, 3-氧化鲨烯甲基上的氢原子, 从而导致2, 3-氧化鲨烯被催化为环阿屯醇。
2.3 催化机制推测根据上述对接结果, 本文推测CAS酶对2, 3-氧化鲨烯的催化机理主要分三步完成(图 7):(1)Cys492和Cys570残基向Asp491提供氢质子, 2, 3-氧化鲨烯上的环氧基夺取Asp491上的氢质子后断裂, 由于碳正离子-π电子作用(cationic-π interaction)引起四个环的环化反应形成C27正离子中间体; (2)C27正离子中间体经过一系列碳正离子重排, 形成C11正离子中间体; (3)Tyr540残基上的羟基氢原子能够与His265的氮原子形成氢键, 被去质子化的Tyr540夺取C27上的氢质子, 进而C27与C11原子形成单键生成环阿屯醇。
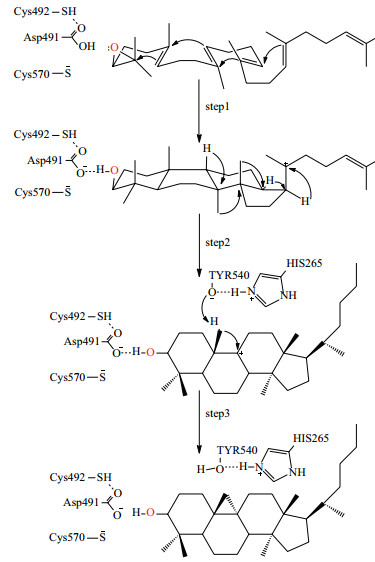
|
| 图 7 2,3-氧化鲨烯形成环阿屯醇的机理图 |
研究蛋白质的结构是了解蛋白质的生物学功能的重要途径。氨基酸序列对蛋白质构象起着决定性的作用, 很多同源性的氨基酸序列就可以决定同一蛋白构象, 这是蛋白质三维结构预测的理论基础[19]。目前研究蛋白质空间结构方法包括X射线晶体衍射技术和核磁共振技术, 均属于生物学实验方法, 它们能够准确测定蛋白三维结构, 但同时也存在许多技术难点, 尤其是膜蛋白晶体结构难以获得, 使得OSCs蛋白方面的研究进展十分缓慢[20]。为了加快新蛋白质的结构测定及其与化合物的相互作用方式, 当前通过PDB蛋白结构信息库中搜索已知结构的同源蛋白质作为模板, 利用同源建模软件对新蛋白进行结构建模是最为常用且有效的一种蛋白质结构预测方法。随着生物信息学的快速发展及其在生物学研究中的突出作用, 蛋白质同源模建技术已经成为一种可靠性高且被广泛认可的蛋白结构预测技术。任何一对氨基酸序列长度大于80的蛋白质, 如果两者的序列同源性超过30%, 则可认为它们具有相似的三维结构, 即蛋白质的基本折叠相同[21]。本研究中所用的环阿屯醇合酶基因CAS的编码区能够编码765个氨基酸, 在PDB数据库中搜索发现蛋白1W6J和1W6K与CAS的氨基酸序列同源性高达49%。因此符合蛋白同源建模的条件, 可以以此二者为模板对CAS的三维结构模型进行预测。
关于OSCs晶体结构及催化机制的研究较少, Thoma等[22]首次报道了人氧化鲨烯环化酶LaS的晶体结构, 发现Asp455、Cys456、Cys533是催化环化形成的活性位点, 而His232则是终止反应的关键。Wu等[23]通过蛋白同源建模发现, 酿酒酵母ERG7的His234可与LaS His232叠合, 后续通过实验验证, 该位点为催化氧化鲨烯环化形成多种不同结构产物的关键位点; Ma等[24]通过对黄瓜中的Bi蛋白进行同源建模比对分析, 推测Leu146和Tyr439为形成不同环化产物的活性位点; Hoshino等[17]也通过与LaS和SHC的同源建模阐述了催化β-AS形成的活性位点。以同源建模为基础, 分子对接则可以找到配体小分子与蛋白受体在其活性区域相结合时能量最低的构象, Major等[25]利用此技术分析了三萜类化合物BPP形成的机制。所以, 利用同源建模、分子对接等计算机软件模拟技术已成为相关氧化鲨烯环化酶的活性位点及催化机制研究的有力工具。本研究用同源建模软件对CAS蛋白结构与其对应底物2, 3-氧化鲨烯和相应碳正离子中间体的相互作用模式进行模拟, 结果表明CAS酶上的Asp491、Cys492、Cys570、Tyr540、His265残基是催化环化的关键氨基酸。这些研究对进一步运用生物实验手段研究其结构和功能, 为设计高活性的酶以及调控次级代谢通路有重要意义。
随着分子生物学研究的突飞猛进, 点突变技术成为一项重要的分子生物学实验技术手段, 在生物和医学领域中的应用非常广泛。通过同源建模和分子对接找到影响催化作用的活性位点氨基酸残基, 可很好地指导突变研究。定点突变技术已广泛地应用于蛋白质改造及结构-功能关系研究中, 如利用点饱和突变技术鉴定蛋白质功能位点, 提高酶比活力, 改善酶热稳定性、底物结合特异性及立体异构特异性等多方面性质[26]。通过定点突变实验, β-AS活性位点氨基酸的鉴别, 催化机理、骨架形成的必须结构单位等研究已经取得了显著进展, 且还获得了几十种具有药用潜力的目标化合物[17]。所以下一步, 通过运用点突变技术对CAS关键活性位点进行定向改造, 可获得具有巨大商业价值的目标产物, 同时通过改善酶活力、扩大催化阈值可为中药化学成分的高效合成提供新技术。
4 结论本文通过同源建模和分子对接技术, 对CAS酶催化2, 3-氧化鲨烯生成环阿屯醇的机制进行了探讨。结果表明, Asp491、Cys492、Cys570、Tyr540、His265是CAS酶的关键催化位点, 其中位于活性空腔顶部的Asp491、Cys492、Cys570通过质子化2, 3-氧化鲨烯上的环氧基来激活催化反应, 而位于活性口袋底部的Tyr540、His265则通过对底物去质子化形成产物来终止反应。此外, 活性空腔内存在大量的疏水氨基酸, 则通过疏水作用稳定反应物、中间体结合在活性空腔。据此推测CAS酶催化机制为:Asp491质子化2, 3-氧化鲨烯引发四个环化反应形成C20正离子中间体, 然后中间体经过一系列的碳正离子重排形成C11正离中间体, 最后通过His265与Tyr540去质子化使得C27与C11原子间形成单键生成产物环阿屯醇。
| [1] |
Akihisa T, Yasukawa K, Yamaura M, et al. Triterpene alcohol and sterol ferulates from rice bran and their anti-inflammatory effects[J]. Journal of Agricultural and Food Chemistry, 2000, 48(6): 2313-2319. DOI:10.1021/jf000135o |
| [2] |
杨秀伟, 韩美华, 钟国跃, 等. 半夏中抑制肿瘤坏死因子-α产生的新脑苷[J]. 中草药, 2008, 39(4): 485-490. DOI:10.3321/j.issn:0253-2670.2008.04.002 |
| [3] |
Islam MS, Yoshida H, Matsuki N, et al. Antioxidant, free radical-scavenging, and NF-kappaB-inhibitory activities of phytosteryl ferulates:structure-activity studies[J]. Journal of Pharmacological Sciences, 2009, 111(4): 328-337. |
| [4] |
Prakash Chaturvedula VS, Zhou BN, Gao Z, et al. New lupane triterpenoids from Solidago canadensis that inhibit the lyase activity of DNA polymerase beta[J]. Bioorganic & Medicinal Chemistry, 2004, 12(23): 6271-6275. |
| [5] |
Sultana S, Alam A, Khan N, et al. Inhibition of benzoyl peroxide and ultraviolet-B radiation induced oxidative stress and tumor promotion markers by cycloartenol in murine skin[J]. Redox Report:Communications in Free Radical Research, 2003, 8(2): 105-112. DOI:10.1179/135100003125001422 |
| [6] |
Fukuoka D, Okahara F, Hashizume K, et al. Triterpene alcohols and sterols from rice bran lower postprandial glucose-dependent insulinotropic polypeptide release and prevent diet-induced obesity in mice[J]. J Appl Physiol(1985), 2014, 117(11): 1337-1348. DOI:10.1152/japplphysiol.00268.2014 |
| [7] |
张忠廉, 罗祖良, 石宏武, 等. 植物药活性成分环阿屯醇药理、生理活性及其研究展望[J]. 中国中药杂志, 2017, 42(3): 433-437. |
| [8] |
Corey EJ, Matsuda SP, Bartel B. Isolation of an Arabidopsis thaliana gene encoding cycloartenol synthase by functional expression in a yeast mutant lacking lanosterol synthase by the use of a chromatographic screen[J]. Proc Natil Acad Sci USA, 1993, 90(24): 11628-11632. DOI:10.1073/pnas.90.24.11628 |
| [9] |
陈迪, 陈永勤, 杨之帆, 等. 盾叶薯蓣环阿屯醇合酶基因克隆与表达[J]. 西北植物学报, 2009, 29(2): 221-228. DOI:10.3321/j.issn:1000-4025.2009.02.002 |
| [10] |
Hayashi H, Hiraoka N, Ikeshiro Y, et al. Molecular cloning and characterization of a cDNA for Glycyrrhiza glabra cycloartenol synthase[J]. Biological & Pharmaceutical Bulletin, 2000, 23(2): 231-234. |
| [11] |
Chen J, Wu XT, Xu YQ, et al. Global transcriptome analysis profiles metabolic pathways in traditional herb Astragalus membranaceus Bge. var. mongolicus(Bge.)Hsiao[J]. BMC Genomics, 2015, 16(S7): 1-20. DOI:10.1186/1471-2164-16-S7-S1 |
| [12] |
李珍, 王东浩, 姚伟, 等. 丹参环阿屯醇合酶基因克隆及表达分析[J]. 西北植物学报, 2013, 33(7): 1285-1291. |
| [13] |
邢朝斌, 龙月红, 吴鹏, 等. 刺五加环阿屯醇合酶基因的克隆及其表达分析[J]. 中草药, 2012, 43(7): 1387-1392. |
| [14] |
罗祖良, 张凯伦, 马小军, 等. 三萜皂苷的合成生物学研究进展[J]. 中草药, 2016, 47(10): 1806-1814. DOI:10.7501/j.issn.0253-2670.2016.10.029 |
| [15] |
Liang Y, Zhao S, Zhang X. Antisense suppression of cycloartenol synthase results in elevated ginsenoside levels in Panax ginseng hairy roots[J]. Plant Molecular Biology Reporter, 2009, 27(3): 298-304. DOI:10.1007/s11105-008-0087-7 |
| [16] |
Schulz-Gasch T, Stahl M. Mechanistic insights into oxidosqualene cyclizations through homology modeling[J]. Journal of Computational Chemistry, 2003, 24(6): 741-753. |
| [17] |
Hoshino T. β-Amyrin biosynthesis:catalytic mechanism and substrate recognition[J]. Organic & Biomolecular Chemistry, 2017, 15(14): 1-55. |
| [18] |
Zhao H, Tang Q, Mo C, et al. Cloning and characterization of squalene synthase and cycloartenol synthase from Siraitia grosvenorii[J]. Acta Pharmaceutica Sinica B, 2017, 7(2): 215-222. DOI:10.1016/j.apsb.2016.06.012 |
| [19] |
邓海游, 贾亚, 张阳. 蛋白质结构预测[J]. 物理学报, 2016, 65(17): 169-179. |
| [20] |
樊盛博, 吴妍洁, 杨兵, 等. 蛋白质结构与相互作用研究新方法-交联质谱技术[J]. 生物化学与生物物理进展, 2014, 41(11): 1109-1125. |
| [21] |
Gunasekaran K, Nussinov R. How different are sttiructurally flexible and rigid binding sites? Sequence and structural features discriminating proteins that do and do not undergo conformational change upon ligand binding[J]. Journal of Molecular Biology, 2007, 365(1): 257-273. |
| [22] |
Thoma R, Schulzgasch T, D'Arcy B, et al. Insight into steroid scaffold formation from the structure of human oxidosqualene cyclase[J]. Nature, 2004, 432(7013): 118-122. DOI:10.1038/nature02993 |
| [23] |
Wu T K, Liu Y T, Chang C H, et al. Site-saturated mutagenesis of histidine 234 of Saccharomyces cerevisiae oxidosqualene-lanosterol cyclase demonstrates dual functions in cyclization and rearrangement reactions[J]. Journal of the American Chemical Society, 2006, 128(19): 6414-6419. DOI:10.1021/ja058782p |
| [24] |
Ma Y, Zhou Y, Ovchinnikov S, et al. New insights into substrate folding preference of plant OSCs[J]. Science Bulletin, 2016(18): 1407-1412. |
| [25] |
Major D T, Weitman M. Electrostatically guided dynamics——the root of fidelity in a promiscuous terpene synthase?[J]. Journal of the American Chemical Society, 2012, 134(47): 19454-19462. DOI:10.1021/ja308295p |
| [26] |
陶苏丹, 刘佳, 陈喜文, 等. 点饱和突变技术及其在蛋白质工程中的应用[J]. 中国生物工程杂志, 2007, 27(8): 82-86. |




