2. 新乡医学院第三附属医院,新乡 453003;
3. 国药集团中国生物技术股份有限公司,北京 100000
2. The Third Affiliated Hospital of Xinxiang Medical University, Xinxiang 4530033;
3. China National Biotec Group Company Limited, Beijing 100000
2013年3月,中国首次报道人感染新型H7N9禽流感病毒病例,引起严重甚至致命呼吸道疾病。随后,疫情迅速席卷中国大陆20多个省、市、自治区,中国香港、中国台湾、马来西亚和加拿大等地陆续报道H7N9禽流感病毒感染病例,截至2019年10月2日,共造成1 568例感染、616人死亡[1]。研究证实H7N9禽流感病毒在第5次和第6次大流行期间,感染家禽和人类且对雪貂和鸡群的致病性增强[2-4]。因此,美国疾病预防和控制中心结合流感风险评估结果,将H7N9亚型禽流感病毒列为高风险病原[5]。流行病学调查表明大部分患者具有活禽接触史,意味着活禽市场和新型H7N9流感爆发存在着紧密的联系[6]。目前,新型H7N9病毒无法通过呼吸道、母婴、血液等方式实现人-人之间的传播[7],但它具有与人类感染相关的基因组成[8],在少量非人源高致病H7N9毒株中,发现神经氨酸酶抑制剂(Neuraminidase-inhibitor,NAI)抗性(NA中的R292K)和哺乳动物适应性突变(如PB2中的E627K和A588V)[9]。此外,H7N9禽流感病毒人-人传播突变动力学模型分析表明,将来H7N9病毒可以获得人-人传播的特征[10]。因此,对H7N9禽流感病毒的研究工作亟需深入开展,以避免给人类健康造成威胁。
NA是流感病毒主要囊膜纤突之一,天然NA蛋白有4个相同的单体组成四聚体,每种单体全长约470个氨基酸,并有胞内区、跨膜区、茎部区及头部区4个区域组成[11]。近来研究成果表明,NA除了在病毒释放中发挥作用,还有助于病毒与细胞糖蛋白唾液酸基团结合,补充血凝素(Hemagglutinin,HA)的受体结合功能,提高NA酶活性,并促进病毒感染[12]。无论是自然突变还是通过遗传修饰获得的NA活性位点突变体,其框架和催化残基都可以在不同程度上改变病毒复制能力、感染性和对抗病毒抑制剂的敏感性[11, 13]。总之,NA在病毒吸附、侵入、释放以及维持与HA的功能平衡等方面起着重要的多功能作用[11]。
本研究以新型H7N9禽流感病毒(安徽株)NA蛋白胞外区片段为研究对象,进行基本理化性质和高级结构等生物信息学分析。为了在E. coli中实现高效表达,选取NA蛋白胞外区序列并进行密码子优化与合成。把构建正确的pET28b-tN9重组载体转化至感受态细胞,将进一步经亲和纯化、SDS-PAGE检测及质谱鉴定的目标蛋白免疫新西兰大白兔,制备多克隆抗体,以期为N9亚型禽流感病毒血清学检测方法的建立奠定基础。
1 材料与方法 1.1 材料原核表达载体pET28b(+)、E. coli DH5α和E. coli BL21(DE3)感受态细胞由新乡学院医学院医学检验技术中心实验室保存;E. coli Rosetta和E. coli Arctic Express(DE3)感受态细胞购自南京钟鼎生物公司;Fast Digest限制性内切酶Nco Ⅰ和Xho Ⅰ购自Thermo Scientific公司;质粒小量制备试剂盒和Solution Ⅰ购自TAKARA公司;DNA凝胶回收试剂盒购自OMEGA BIO-TEK公司;Ni Sepharose 6 FF、Sepharose 4B纯化填料购自GE公司;弗氏完全佐剂、弗氏不完全佐剂由新乡医学院提供;HRP标记山羊抗兔IgG和DAB显色液购自武汉博士德公司;TMB显色液购自碧云天公司;新西兰大白兔购自武汉生物制品研究所实验动物中心;人用H7N9灭活病毒原液(上海株)由上海生物制品研究所陈则研究员馈赠。
1.2 方法 1.2.1 新型H7N9禽流感病毒NA蛋白胞外区片段的生物学信息分析参考相关文献[14],采用EXPASY系统的Protparam程序分析新型H7N9禽流感病毒(安徽株)NA蛋白胞外区片段(truncated N9,tN9)理化特征(https://web.expasy.org/protparam/);使用Protscale程序Kyte & Doolittle算法分析其疏水性(https://web.expasy.org/protscale/);使用SOPMA软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测二级结构;使用PHYRE2 protein fold recognition server(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测全长NA蛋白和NA蛋白胞外区片段三级结构[15],其PDB文件用VMD(Visual Molecular Dynamics)1.9.3软件进行可视化分析[16]。
根据生物信息学分析结果,按照文献方法[17]以GenScript Rare Codon Analysis工具对NA胞外区核苷酸序列(1 290 bp)进行基因密码子优化设计,确认目标序列合框后由南京金斯瑞公司进行合成。
1.2.2 目标基因表达载体的构建目标基因合成时,在5’端添加Nco Ⅰ酶切位点(CCATGG),在3’端删除TAA终止密码子,添加Xho Ⅰ酶切位点(CTCGAG),合成后克隆至T载体。以Nco Ⅰ和Xho Ⅰ双酶切表达载体pET28b和上述T载体,凝胶回收目的序列,以Solution Ι进行连接,将连接产物转化至DH5α感受态细胞,涂布于含卡那霉素的LB平板(终浓度为50 μg/mL),37℃培养16-20 h。以载体通用引物为扩增引物,以重组单菌落为模板,进行PCR鉴定,提取阳性克隆送武汉金开瑞生物工程有限公司进行测序验证,测序正确的表达质粒命名为pET28b-tN9。
1.2.3 目的蛋白的诱导表达、亲和纯化与质谱鉴定将pET28b-tN9分别转化E. coli BL21(DE3)、E. coli Rosetta和E. coli Arctic Express(DE3)感受态细胞,挑取重组单菌落分别于50 mL含卡那霉素的LB液体培养基中培养,当OD值达到0.5时,以0.2 mmol/L的IPTG终浓度进行诱导,E. coli BL21(DE3)、E. coli Rosetta重组菌分别在30℃和37℃条件下诱导5 h,E. coli Arctic Express(DE3)在16℃温度下诱导过夜。离心收集菌体进行超声破碎,分离上清和沉淀,以10% SDS-PAGE分析蛋白的表达情况。
根据上步结果选取其中一种重组菌和诱导条件进行扩大培养,包涵体形式表达的蛋白经变性、复性后,参照相关文献[18]用镍柱亲和层析法纯化目标蛋白,采用不同浓度咪唑溶液进行洗脱,收集流穿液和洗脱液,以10% SDS-PAGE检测蛋白纯化情况。纯化后蛋白浓缩并以SDS-PAGE电泳检测蛋白纯度,将电泳后目标条带切割,送至生工生物工程(上海)股份有限公司进行串联飞行时间质谱(Maldi-tof-tof)检测[19],以进一步验证目标蛋白。
1.2.4 动物免疫与多克隆抗体制备将验证正确的目的蛋白与等体积的弗氏完全佐剂充分混合后,以多点皮下方式免疫新西兰大白兔(400 μg/只)。免疫前经耳缘静脉采血并分离血清,用作阴性对照。每2周免疫1次,之后用相同剂量的重组蛋白与弗氏不完全佐剂配伍,第4次免疫一周后采血大量制备抗血清,并准备纯化。结合文献方法[20],将重组目标蛋白偶联Sepharose 4B制备成抗原亲和纯化层析柱,将所得抗血清与PBS等量混合后缓慢上样,待抗体结合后用甘氨酸缓冲液洗脱,然后在PBS中4℃透析过夜,进行后续多克隆抗体检测。
1.2.5 Western blotting法鉴定多抗特异性采用Western blotting方法检测抗体特异性。将纯化的重组蛋白、pet28a空载体诱导后产物和人用H7N9灭活病毒原液(上海株),经SDS-PAGE电泳后电转至PVDF膜,以1% BSA于37℃封闭1 h,PBST洗涤3次,每次5 min;用制备的多克隆抗体(1:3 000稀释)作为一抗,室温孵育1 h,PBST洗涤3次,每次5 min;加入1:1 500稀释的山羊抗兔HRP-IgG,室温孵育1 h,PBST洗涤3次,每次5 min,DAB显色并拍照留用。
1.2.6 间接ELISA法测定多克隆抗体的效价将重组蛋白用包被液稀释成3 μg/mL,每孔100 μL,4℃包被。次日弃去包被液,洗板3次,每孔加入200 μL封闭液,37℃恒温孵育1 h,用于ELISA检测。制备的抗体按1:500倍比稀释,HRP标记的山羊抗兔IgG稀释度为1:2 000,显色剂为TMB,用酶标仪测定各孔吸光度(A450)值。实验设阴性血清对照,样品A450值≥阴性对照A450值的2.1倍判为阳性。
2 结果 2.1 NA蛋白胞外区片段生物信息学分析结果通过Protparam程序分析,tN9分子式为C2154H3283N623O674S27,分子质量单位为49.6 Ku,理论等电点为6.56,半衰期为30 h(Mammalian)、> 20 h(Yeast)、> 10 h(Escherichia coli),不稳定指数43.49,为不稳定蛋白。应用Protscale构建的疏水图谱显示,多肽链第270位的Asp具有最高的分值1.389,疏水性最强,第292位的Pro具有最低的分值-2.978,亲水性最强,整体来看,该基因编码蛋白为亲水性蛋白。
以SOPMA软件预测该重组蛋白二级结构,其结果表明,α螺旋(Alpha helix,缩写为h)占7.95%;β折叠(Extended strand,缩写为e)占31.82%;β转角(Beta turn,缩写为t)占7.73%;无规则卷曲(Random coil,缩写为c)占52.50%,主要以无规则卷曲、β折叠为主(图 1)。
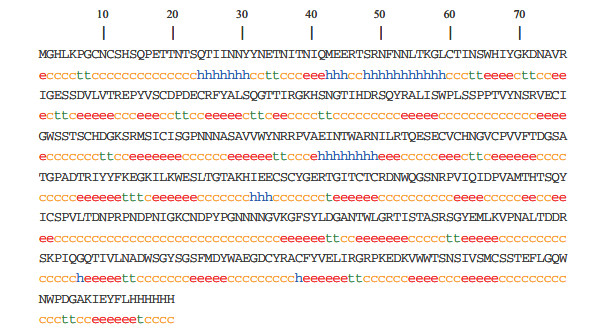
|
| 图 1 SOPMA软件预测的tN9二级结构 |
将截短前后NA蛋白序列提交至PHYRE2服务器自动建模,构建了NA蛋白截断前后的三维结构(图 2-A、图 2-B)。将PDB文件用VMD软件进行处理,并以黄色标记出HIS原子位置,比对分析显示截短前后蛋白质茎部区和头部区结构无显著差异,删除胞内区和跨膜区不会影响NA蛋白免疫原性,HIS tag暴露于空间之外,可以用于后续蛋白纯化(图 2-C、图 2-D)。
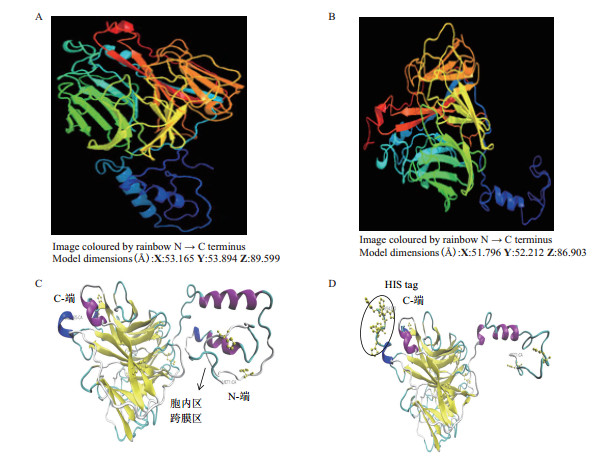
|
| 图 2 NA蛋白截短前(A、C)后(B、D)结构比对 |
密码子偏好性是影响蛋白表达的重要因素之一。经在线稀有密码子分析工具进行密码子优化分析,结果表明tN9编码基因密码子适应指数(Codon adaptation index,CAI)为0.59,GC含量为43.64%,密码子使用频率分布系数CFD为13%。根据大肠杆菌密码子偏好性,对tN9序列稀有密码子进行同义替换后,CAI指数为0.91,达到理想值范围(0.8-1.0),GC含量为58.55%,符合理想值区间(30%-70%),有利于后续蛋白的高效表达。使用序列处理在线工具包(SMS,http://www.bio-soft.net/sms/index.html)对优化前后的序列进行比对(图 3)。

|
| 图 3 tN9基因序列密码子优化前后比对 |
将靶基因与pET28b连接,将连接产物转化E. coli DH5α感受态细胞。挑选重组菌落进行PCR鉴定,凝胶电泳显示在1 500 bp左右出现目标条带,与预期大小相符,阳性质粒测序结果进一步证实重组载体构建成功(图 4)。
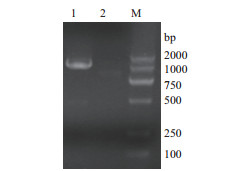
|
| 1:阳性重组菌落;2:阴性重组菌落;M:DL2 000 Marker 图 4 重组菌落PCR鉴定 |
以IPTG诱导3种重组菌后,目标蛋白均有表达,仅在Arictic中有微弱的可溶性表达(图 5泳道16),表达量较低,其余均为包涵体表达形式(图 5)。考虑到后续动物免疫的抗原剂量要求,选择重组E. coli BL21(DE3)为诱导菌株,进一步扩大培养并进行诱导、表达,诱导条件为30℃,诱导时间为5 h。
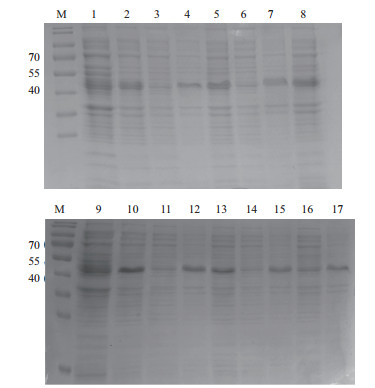
|
| M:蛋白Marker;1-7:分别为Rosetta重组菌诱导前、30℃诱导后、30℃诱导后超声破碎上清、30℃诱导后超声破碎沉淀、37℃诱导后、37℃诱导后超声破碎上清、37℃诱导后超声破碎沉淀;8、16、17:分别为Arctic Express(DE3)重组菌诱导后、诱导后超声破碎上清、诱导后超声破碎沉淀;9-15:分别为BL21(DE3)重组菌诱导前、30℃诱导后、30℃诱导后超声破碎上清、30℃诱导后超声破碎沉淀、37℃诱导后、37℃诱导后超声破碎上清、37℃诱导后超声破碎沉淀 图 5 SDS-PAGE检测重组tN9蛋白的表达 |
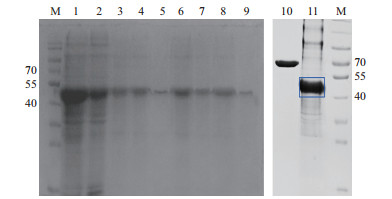
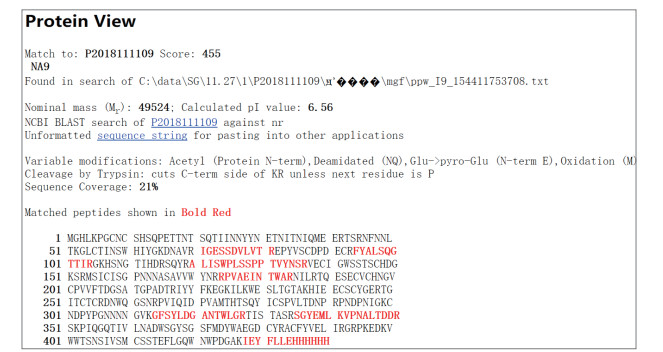
诱导结束后,将离心收获的菌体进行超声破碎,以含8 mol/L尿素的PBS重悬沉淀,使之彻底溶解,并在含4 mol/L尿素、2 mol/L尿素、1 mol/L尿素的Buffer中透析,然后上柱亲和纯化,并用100 mmol/L、150 mmol/L和250 mmol/L的咪唑梯度进行洗脱,收集洗脱液,进行SDS-PAGE鉴定(图 6-A)。混合梯度洗脱样品,以BSA标准品为对照,SDS-PAGE分析其纯度不低于85%。切胶后目标条带(图 6-B)进行串联飞行时间质谱分析,经数据库搜索,得到的蛋白质匹配得分为455,分子质量49 524u,等电点(Isoelectric point,PI)6.56,匹配度21%(图 7)。
|
| 1:流穿液;2:10 mmol/L的咪唑洗脱液;3-5:100 mmol/L的咪唑洗脱液;6-8:150 mmol/L的咪唑洗脱液;9:250 mmol/L的咪唑洗脱液;10:BSA标准品;11:纯化后的目标蛋白;M:蛋白Marker 图 6 SDS-PAGE检测tN9重组蛋白亲和纯化产物 |
|
| 图 7 tN9蛋白数据库搜索结果 |
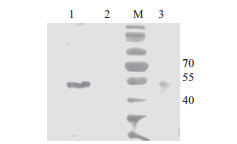
Western blotting检测结果表明,于图 8泳道1和3,50 kD左右位置各出现一条蛋白条带(tN9分子量49.6 kD,全长NA分子量51.8 kD),表明制备的抗体能与经IPTG诱导的重组蛋白和天然新型H7N9禽流感病毒(上海株)发生特异性反应,具有良好的反应原性。
|
| 1:重组蛋白;2:pet28a载体诱导后;M:蛋白Marker;3:人用H7N9灭活病毒原液(上海株) 图 8 Western blotting鉴定tN9多克隆抗体免疫反应性 |
将tN9蛋白免疫兔子制备的抗体进行1:500倍比稀释,间接ELISA法测定抗体效价,结果显示效价为1:25 6000(图 9)。

|
| 图 9 ELISA检测tN9蛋白多克隆抗体效价 |
系统发育分析表明,新型H7N9禽流感病毒是H7N3禽流感病毒、HX(2或11)N9流感病毒和H9N2禽流感病毒的重配株。该病毒HA基因来自鸭源H7N3病毒,NA基因来自于候鸟源HX(2或11)N9病毒,其余的6个基因(PB2、PB1、PA、NP、M和NS)来自H9N2病毒[6]。经过多亚型病毒重配后,赋予了H7N9病毒新的流行特征:对人和哺乳动物具有高致病性,但对包括鸡在内的家禽一般具有低致病性;具有截短的NA蛋白,茎部缺失5个氨基酸;部分人源H7N9病毒分离株在受体结合位点HA中存在天然Q226L突变、PB2蛋白在第627和701位置具有Lys和Asn突变;鸟源H7N9分离株PB2蛋白在第627和701位置保留Glu和Asp,这些新特征对于H7N9禽流感病毒在哺乳动物宿主上呼吸道进行复制是非常重要的[6]。
流感病毒基因易发生抗原漂移、抗原转换,从而可能导致流感爆发,NA基因是流感病毒基因组突变率较高的基因[21]。在一些H7N9临床分离株中发现了与耐药性相关的NA氨基酸突变位点,NA-R292K,研究表明H7N9分离株NA的R292K取代,对奥司他韦和帕拉米韦产生高度耐药性,对扎那米韦产生部分耐药性。而且,耐药突变位点的H7N9病毒株,在人体的病毒复制能力、对小鼠的毒力和在豚鼠间的传播能力,与未发生突变的病毒株相比没有差别[22]。NA作为受体破坏酶,可以切割α 2-3和α 2-6连接唾液酸,释放病毒颗粒[23],为了进一步确定NA在病毒受体结合中的可能作用,对人畜共患H7N9病毒株的N9 NA比对、分析,结果证实N9 NA具有与唾液酸结合的活性血细胞吸附(Hemadsorption,Hb)位点,其不寻常的唾液酸酶位点动力学特征,可增强病毒与唾液酸受体类似物的整体结合能力,尤其是增强和人α 2,6-连接唾液酸相似受体的结合[24]。因此,亟需深入开展对N9蛋白结构突变和受体功能活性的研究。
目前,流感病毒疫苗的有效成分主要基于HA蛋白,研究表明NA可能也具备良好的免疫原性[25]。研究人员制备了基于NA的重组病毒样颗粒疫苗,免疫N1 NA VLPs的雪貂产生高滴度的血清NA抑制抗体(NA-inhibition,NI),可以保护雪貂免受致死剂量的病毒侵袭,这为基于NA疫苗的开发提供了支持[26]。目前,尚无关于N9 NA作为疫苗标靶的报道,试推测N9蛋白可能同样存在病毒特异性抗原表位,诱导产生保护性抗体。总之,NA在病毒侵入、释放、小分子药物作用标靶和免疫原性方面发挥多重重要功效[25],因此,深入研究NA的结构和功能、NA在流感感染和复制周期中的复杂作用,特别是H7N9病毒HA和NA糖蛋白如何各自发挥作用,实现功能平衡以维持病毒适应性等方面具有重要意义[11],然而目前尚无针对N9蛋白的特异性抗体,这一定程度上限制了对N9蛋白的深入研究。
随着技术的进步,人们对流感病毒的研究日益深入,但对HA糖蛋白的研究较多,对NA的报道较少,仅有在酵母[27]、哺乳动物细胞[28]和昆虫细胞[29]等中可溶性表达H5N1病毒和H1N1病毒NA单体和四聚体的报道,对于N9的表达和纯化,Margine等[30]以杆状病毒表达系统表达并纯化NA蛋白,而关于N9基因原核表达及免疫原性的研究鲜有报道。本试验通过基因重组方法,利用pET28b表达tN9蛋白,IPTG诱导后经SDS-PAGE鉴定,基本以包涵体形式存在。吴艳菊等[31]、万润等[21]分别以原核表达系统表达H5N1亚型和H6N6亚型流感病毒的NA,表达蛋白均以包涵体形式存在,这与本试验结果一致。本研究表达的安徽株H7N9流感病毒tN9包涵体经变性、复性后制备的多抗,Western blotting显示其与人用H7N9禽流感病毒(上海株)结合较弱(H7N9禽流感病毒全病毒上样量5 μL),以H7N9禽流感病毒(上海株)作为包被抗原,ELISA检测效价仅为1:2 000(H7N9禽流感病毒全病毒以1:1 000稀释包被,该部分数据未显示),可能的主要原因为:NA蛋白的含量本身不高(无法准确定量H7N9全病毒中的NA含量);以当前的包涵体蛋白变性、复性工艺获得的目标蛋白与天然蛋白可能存在一定的结构和功能差异;上海株H7N9禽流感病毒NA氨基酸存在突变[32](上海株N9是K294,安徽株N9是R294),从而影响了二者之间的结合活性。
4 结论本研究采用生物信息学方法对新型H7N9禽流感病毒NA蛋白胞外区抗原进行预测,删除NA胞质区和跨膜区并对截断后的片段进行原核表达及多克隆抗体制备。纯化的tN9蛋白具有良好的免疫原性,能诱导兔子产生高水平免疫反应,所制备的特异性多克隆抗体具有较高的效价,可用于开展NA蛋白结构和功能研究,也可用于建立H7N9禽流感病毒血清学检测方法,为进一步探讨NA蛋白特性、H7N9病毒对人和动物的感染机制以及新型疫苗的开发提供了条件。
| [1] |
Food and Agriculture Organization of the United Nations. H7N9 situation update[EB/OL]. http://www.fao.org/ag/againfo/programmes/en/empres/H7N9/situation_update.html. (accessed on 02 October 2019).
|
| [2] |
Gong S, Qi F, Li F, et al. Human-derived A/Guangdong/Th005/2017(H7N9)exhibits extremely high replication in the lungs of ferrets and is highly pathogenic in chickens[J]. Viruses, 2019, 11(6): 494. DOI:10.3390/v11060494 |
| [3] |
Pu Z, Luo T, Yang J, et al. Rapid evolving H7N9 avian influenza A viruses pose new challenge[J]. J Infect, 2019, 78(3): 249-59. |
| [4] |
Sun X, Belser JA, Pappas C, et al. Risk assessment of fifth-wave H7N9 influenza a viruses in mammalian models[J]. J Virol, 2019, 93(1): e01740-18. |
| [5] |
Wang GL, Gray GC, Chen JM, et al. Will China's H7N9 control strategy continue to be effective?[J]. Open Forum Infect Dis, 2019, 6(6): ofz258. DOI:10.1093/ofid/ofz258 |
| [6] |
Su W, He H. Hmerqence and pandemic potential of avian influenza A(H7N9) virus[M]// Manjur M shah, Tanveer Beg, Mohammad Mahainood, et al. Microbiology in Agriculture and Human Healt InTech, 2015: 33-44.
|
| [7] |
Wang J, Xu H, Mu C, et al. A study on mother-to-fetus/infant transmission of influenza A(H7N9)virus: Two case reports and a review of literature[J]. Clin Respir J, 2018, 12(11): 2539-2545. DOI:10.1111/crj.12953 |
| [8] |
Pu Z, Xiang D, Li X, et al. Potential pandemic of H7N9 avian influenza a virus in human[J]. Front Cell Infect Microbiol, 2018, 8: 414. DOI:10.3389/fcimb.2018.00414 |
| [9] |
Quan C, Shi W, Yang Y, et al. New threats from H7N9 influenza virus: spread and evolution of high- and low-pathogenicity variants with high genomic diversity in wave five[J]. J Virol, 2018, 92(11): e00301-18. DOI:10.1128/JVI.00301-18 |
| [10] |
Peng J, Yang H, Jiang H, et al. The origin of novel avian influenza A(H7N9)and mutation dynamics for its human-to-human transmissible capacity[J]. PLoS One, 2014, 9(3): e93094. DOI:10.1371/journal.pone.0093094 |
| [11] |
McAuley JL, Gilbertson BP, Trifkovic S, et al. Influenza virus neuraminidase structure and functions[J]. Front Microbiol, 2019, 10: 39. DOI:10.3389/fmicb.2019.00039 |
| [12] |
Wen F, Wan XF. Influenza neuraminidase: underrated role in receptor binding[J]. Trends Microbiol, 2019, 27(6): 477-479. DOI:10.1016/j.tim.2019.03.001 |
| [13] |
Govorkova EA. Consequences of resistance: in vitro fitness, in vivo infectivity, and transmissibility of oseltamivir-resistant influenza A viruses[J]. Inflvenza and Other Respiratory Viruses, 2012, 7(S1): 50-57. |
| [14] |
张耀刚, 曹得萍, 李超群, 等. 细粒棘球绦虫转酮醇酶生物信息学分析[J]. 中国病原生物学杂志, 2017, 12(3): 233-237. |
| [15] |
You H, Gobert GN, Du X, et al. Functional characterisation of Schistosoma japonicum acetylcholinesterase[J]. Parasit Vectors, 2016, 9(1): 328. DOI:10.1186/s13071-016-1615-1 |
| [16] |
Deihim B, Hassanzadeh M, Shafiei-Jandaghi NZ, et al. Characterization of the DNA mismatch repair proteins MutS and MutL in a hypermutator Acinetobacter baumannii[J]. Microb Pathog, 2017, 113: 74-84. DOI:10.1016/j.micpath.2017.10.001 |
| [17] |
刘宏汉, 姜雨婷, 孙琦, 等. 一种新型贻贝足丝抗氧化蛋白的原核重组表达及功能[J]. 中国生物化学与分子生物学报, 2018, 34(11): 1227-1238. |
| [18] |
陈慧芹, 王小平, 罗树红, 等. 羊口疮病毒蛋白ORFV035的表达、纯化和多克隆抗体的制备[J]. 生物技术通报, 2017, 33(7): 150-154. |
| [19] |
Webster J, Oxley D. Protein identification by MALDI-TOF mass spectrometry[J]. Methods Mol Biol, 2012, 800: 227-240. |
| [20] |
周必英, 周泠, 刘美辰, 等. 猪带绦虫TSO45W-4B基因的克隆、表达和抗体制备[J]. 中国寄生虫学与寄生虫病杂志, 2013, 31(5): 372-375. |
| [21] |
万润, 华敏, 龙立书, 等. H6N6亚型禽流感病毒N6基因的原核表达与多克隆抗体制备[J]. 中国畜牧兽医, 2018, 45(11): 2989-2995. |
| [22] |
Hai R, Schmolke M, Leyva-Grado VH, et al. Influenza A(H7N9)virus gains neuraminidase inhibitor resistance without loss of in vivo virulence or transmissibility[J]. Nat Commun, 2013, 4: 2854. DOI:10.1038/ncomms3854 |
| [23] |
Gaymard A, Le Briand N, Frobert E, et al. Functional balance between neuraminidase and haemagglutinin in influenza viruses[J]. Clin Microbiol Infect, 2016, 22(12): 975-983. DOI:10.1016/j.cmi.2016.07.007 |
| [24] |
Benton DJ, Wharton SA, Martin SR, et al. Role of neuraminidase in influenza A(H7N9)virus receptor binding[J]. J Virol, 2017, 91(11): e02293-16. DOI:10.1128/JVI.02293-16 |
| [25] |
Krammer F, Fouchier RAM, Eichelberger MC, et al. NAction! How can neuraminidase-based immunity contribute to better influenza virus vaccines?[J]. MBio, 2018, 9(2): e02332-17. DOI:10.1128/mBio.02332-17 |
| [26] |
Smith GE, Sun X, Bai Y, et al. Neuraminidase-based recombinant virus-like particles protect against lethal avian influenza A(H5N1)virus infection in ferrets[J]. Virology, 2017, 509: 90-97. DOI:10.1016/j.virol.2017.06.006 |
| [27] |
Yongkiettrakul S, Boonyapakron K, Jongkaewwattana A, et al. Avian influenza A/H5N1 neuraminidase expressed in yeast with a functional head domain[J]. J Virol Methods, 2009, 156(1-2): 44-51. DOI:10.1016/j.jviromet.2008.10.025 |
| [28] |
Nivitchanyong T, Yongkiettrakul S, Kramyu J, et al. Enhanced expression of secretable influenza virus neuraminidase in suspension mammalian cells by influenza virus nonstructural protein 1[J]. J Virol Methods, 2011, 178(1-2): 44-51. DOI:10.1016/j.jviromet.2011.08.010 |
| [29] |
Schmidt PM, Attwood RM, Mohr PG, et al. A generic system for the expression and purification of soluble and stable influenza neuraminidase[J]. PLoS One, 2011, 6(2): e16284. DOI:10.1371/journal.pone.0016284 |
| [30] |
Margine I, Palese P, Krammer F. Expression of functional recombinant hemagglutinin and neuraminidase proteins from the novel H7N9 influenza virus using the baculovirus expression system[J]. J Vis Exp, 2013(81): e51112. |
| [31] |
吴艳菊, 张治业, 倪小舒, 等. H5N1亚型禽流感病毒NA和NS1蛋白原核表达及多克隆抗体制备[J]. 黑龙江畜牧兽医, 2017(11): 1-4. |
| [32] |
Wu Y, Bi Y, Vavricka CJ, et al. Characterization of two distinct neuraminidases from avian-origin human-infecting H7N9 influenza viruses[J]. Cell Res, 2013, 23(12): 1347-1355. DOI:10.1038/cr.2013.144 |




