2. 辽宁师范大学生命科学学院,大连 116029;
3. 教育部动物性食品安全保障技术工程研究中心,大连 116600
2. School of Life Science and Biotechnology, Liaoning Normal University, Dalian 116029;
3. Ministry of Education Center for Food Safety of Animal Origin, Dalian 116600
噬菌体最早由Herelle[1-2]和Twort[3]同时发现,Herelle将能够杀灭细菌的病毒命名为噬菌体,并提出了噬菌体用于治疗的思想[4]。而关于噬菌体的研究,通常被认为是现代分子生物学发展的基础[5]。30年代末至40年代初,噬菌体在西医中的治疗应用广泛传播[6],但随着抗生素的大量使用,噬菌体治疗在西方国家遭遇搁浅。然而,在前苏联、波兰和捷克共和国,治疗性噬菌体制剂的开发和使用从未停止过。这些国家积累的大量经验表明,以噬菌体为基础的药物是有效而安全的。如今,这种抗菌药物的大规模生产仍在俄罗斯和格鲁吉亚持续进行[7-8]。在波兰,弗罗茨瓦夫免疫学和实验治疗研究所的一个实验中心,仍然有治疗性噬菌体制剂的生产和使用[9]。
近年来,由于多重耐药菌菌株的广泛出现,使新抗生素在医疗实践中的引入陷入了很大困境[10]。疾病控制中心警告提示,现在是“后抗生素时代”,如果现状持续,有可能某一天我们会面临无药可用的境地[11]。噬菌体作为抗生素替代品的可行性正越来越引起人们的关注[12],因此,噬菌体疗法开始重新进入人们的视线[13],同时噬菌体在人类多重耐药菌性疾病治疗的成功案例,也使医疗工作者深受鼓舞[14-15]。
然而,噬菌体治疗的一个主要问题是其安全性的不确定。对于噬菌体安全性的确定,可以通过快速分析噬菌体基因组的方法来进行,即通过确定其基因组中有无毒力基因,溶源基因及毒力转座子等致病因子来判断。同时,相关动物实验也可以为噬菌体的安全性提供一定的实验佐证。
转录组学(Transcriptome),是指以特殊环境或特殊生理情况为依托,某个细胞、组织以及生物体里面所得RNA个数总数,包括信使RNA(mRNA)、核糖体RNA(rRNA)、转运RNA(tRNA)及其他的非编码RNA(Non- coding RNA)。在生命活动中,不可缺少的就是蛋白质,mRNA是其主要编码形式,所以从狭义上来说,转录组其实是特指细胞能够通过转录实现的所有mRNA编码。Illumina公司的新一代测序仪(包括Genome Analyzer及其升级版HiSeq 2000)利用基于单分子簇的边合成边测序技术(Sequencing by synthesis,SBS)和专有的可逆终止化学反应,可以在短时间内获得大量数据[16]。
本课题组在前期实验研究中,获得两株裂解性噬菌体vBSM-A1和vBSP-A2[17],均能特异性裂解金黄色葡萄球菌,但其作为一种外源物质运用于体内,是否会刺激机体产生中和抗体而降低其治疗效果,甚至对机体的安全造成威胁,这是将其运用于临床治疗必须要解决的问题。本研究采用Ilumina HiSeqTM 2000新一代高通量测序技术,进行了金黄色葡萄球菌噬菌体在小鼠乳腺灌注的安全性分析,对噬菌体处理后的小鼠乳腺组织转录组进行测序,将测序得到的大量Unigene进行GO、和KEGG分类统计,给出功能注释和Pathway注释,并进行了通路上调或者下调的相关分析。这些注释信息的完成及相关分析将为噬菌体治疗的安全性提供基础数据,同时也为噬菌体治疗中给药的剂量及方式提供了理论依据。
1 材料与方法 1.1 材料 1.1.1 实验动物所有动物实验均按照国家实验动物福利准则(中国科技部)进行,并经大连理工大学动物福利与研究伦理委员会(2019-040)批准。昆明小鼠购自大连医科大学(大连)实验动物中心。分笼饲养,正常饲喂。环境温度控制在20-26℃,12 h循环光照。适应性饲养一周后,将母鼠与公鼠按1:1的比例合笼过夜,将母鼠移出单独饲养,让其自然分娩,并哺乳一周左右进行实验[18]。
1.1.2 主要试剂TNF-α ELISA检测试剂盒(购自上海碧云天生物技术有限公司);绵羊鲜血琼脂培养基、麦康凯琼脂、普通琼脂培养基按常规方法配制;磷酸盐缓冲液(PBS);三溴乙醇(2.5%)。酵母提取物(Oxoid),胰蛋白胨(Oxoid),琼脂粉(索莱宝公司),氯化钠(天津天大化学试剂厂)。
1.1.3 主要器材电热恒温培养箱(上海一恒),高速微量离心机(北京鼎昊源),高速冷冻离心机(长沙湘仪),洁净工作台(苏州安泰),电热鼓风干燥箱(上海一恒),恒温培养振荡器(上海智城),立式压力蒸汽灭菌器(上海博讯),移液器若干(Thermo/Dragon/ Eppendorf/Genex Beta等),旋涡混合器(海门其林贝尔),JJ500型电子天平(常熟双杰)。
1.2 方法 1.2.1 噬菌体的分离与培养两株金黄色葡萄球菌噬菌体vBSM-A1(GenBank ID:MK_584893)和vBSP-A2(GenBank ID:MK_656892)[17]均分离自新疆农垦建设兵团134牛场,分离样本为牛场污水[19]。对噬菌体进行扩增,即37℃孵育4-6 h,待菌液变澄清且烧瓶下方出现丝絮状白色沉淀即表明噬菌体富集完成[20]。之后,经CSCl密度梯度超速离心纯化噬菌体悬浮液[21]。通过平板计数确定样品中PFU的确切数量。将噬菌体VBSM-A1和VBSP-A2按照1:1体积配制cocktail。
1.2.2 实验分组及噬菌体灌注将泌乳后10 d的雌鼠随机分为3组,每组8只。实验前2 h将母鼠与乳鼠隔离,腹腔注射三溴乙醇(2.5%)以麻醉,剂量为0.015 mL/g[22]。将麻醉后的小鼠腹部向上固定于体视显微镜下,先用75%酒精对母鼠第4对乳腺及其周围皮肤进行消毒。待皮毛润湿,乳头充分暴露后,左手持镊子轻轻夹持固定住小鼠乳头,右手持50 μL钝性微量进样器,从小鼠第4对乳房的乳头口轻轻插入乳头管内,然后将25 μL噬菌体悬液或PBS注入小鼠乳池内。注射完毕后,令小鼠平躺,防止乳腺导管口处感染,待自然苏醒。每组小鼠给药如表 1(PFU:plaque forming unit,噬菌斑形成单位)。
灌注24 h后,将母鼠脱颈处死,对L4和R4乳腺进行无菌解剖。将左侧乳腺组织剥离剪下一部分经液氮冷却,干冰运输至北京百迈客公司进行转录组检测;另一部分放入10%甲醛中固定,进行组织学切片制作;右侧乳腺组织取下称重后,按1:9(W/V)比例加入无菌的PBS研磨。
匀浆处理完毕后,吸取100 μL进行噬菌体数(PFU)的测定,剩余部分使用ELISA试剂盒(Abbkine)进行细胞因子TNF-α检测[23]。
1.2.4 乳腺组织病理组织切片的制备将切除的乳腺组织浸于Bouin氏液中使其充分固定,将具有代表性的组织切片修剪后放入组织处理盒中,经冲洗、脱水、透明、浸蜡、包埋、切片、脱蜡后进行HE染色[24],然后将其取出,晾干,制成中性树胶封片,在显微镜下观察乳腺组织病理变化。每个样本都检查有无坏死、多形核嗜中性粒细胞炎症(中性粒细胞)和淋巴细胞。
1.2.5 ELISA方法检测乳腺组织促炎因子水平小鼠右侧乳腺组织研磨匀浆处理后,转移到离心管中,4℃、3 000 r/min离心20 min,以去除脂肪组织。将上清液转移到新的离心管中继续4℃、3 000 r/min离心10 min,收集上清液,通过ELISA试剂盒的方法检测乳腺组织匀浆中的TNF-α水平。
1.2.6 转录组测序将乳腺组织在干冰条件下送往北京百迈客生物科技有限公司,实验流程包括RNA样品检测,mRNA富集、反转录,末端修复,3’加A,连接接头,PCR富集,文库质控,上机测序(Illumina HiSeq 2500)。
1.2.7 数据统计分析用Prism 5.0和SPSS19.0对试验数据进行统计分析,利用单因素方差分析(ANOVA)进行两组数据间差异性分析,所得数据以Means±SEM表示。*P < 0.05代表差异显著,**P < 0.01代表差异极显著,P > 0.05代表差异不显著。
2 结果 2.1 小鼠外部形态观察及乳腺组织病理学观察相比于正常组,噬菌体灌注组小鼠未呈现明显的生理体征区别,噬菌体VBSM-A1实验组(T02)和噬菌体cocktail实验组(T03)小鼠状态正常,表现为精神状态良好,毛发略有轻度蓬松,行走正常。
H.E.染色结果显示,健康对照组(T01)的小鼠乳腺腺泡结构完整,排列紧密,无炎性细胞浸润;噬菌体VBSM-A1实验组(T02)和噬菌体cocktail实验组(T03)乳腺腺泡排列较为整齐,局部视野有少量上皮细胞脱落,腺泡间隙及腺泡内有少量炎性浸润(箭头处),乳腺上皮细胞基本完好,未观察到明显的细胞坏死崩解(图 1)。
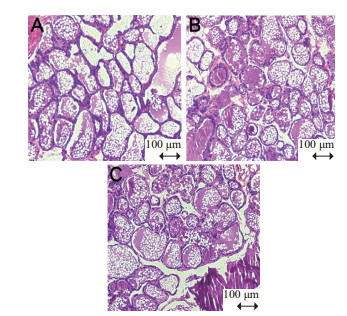
|
| A:健康对照组(T01);B:噬菌体VBSM-A1实验组(T02);C:噬菌体cocktail实验组(T03),下同 图 1 小鼠乳腺组织(HE染色,标尺示100 μm) |
由图 2可知,与健康对照组(T01)相比,噬菌体实验组的TNF-α含量有所增加,而噬菌体cocktail实验组(T03)略高于噬菌体VBSM-A1实验组(T02)。噬菌体cocktail组与对照组相比差异显著(P < 0.05)。由此可知,噬菌体实验组会诱发机体一定的免疫应答,导致炎症因子的上调。
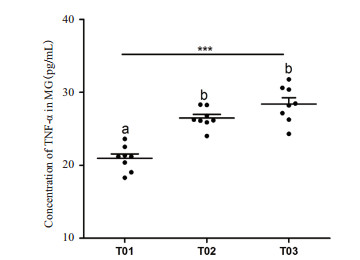
|
| 数据为8个平行实验的平均值±SE。在水平上,不同的小写字母表明各组之间存在显著差异(P < 0.05),下同 图 2 噬菌体处理对TNF-α水平的影响 |
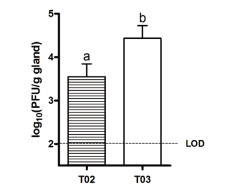
经噬菌体灌注24 h后,在小鼠乳腺组织检测到噬菌体数量如图 3,其中,噬菌体VBSM-A1实验组(T02)组检测到的噬菌斑平均值为3.98×103(103.6)PFU/gland,T03组平均值为检测到的噬菌斑2.56×104(104.4)PFU/gland。
|
| LOD为检出限,即噬菌体能够被检测出的最小极限值 图 3 噬菌体处理组小鼠乳腺组织噬菌体浓度(lg PFU/g) |
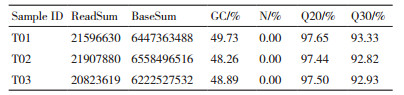
小鼠乳腺组织3组样品的转录组测序,共获得19.23 Gb Clean Data,各样品Clean Data均达到6.41 Gb,质量参数Q20碱基百分比在97.44%以上,Q30碱基百分比在92.82%及以上;过滤后不确定的碱基比例N值为0.00;3组样品的GC含量在48.26%-49.73%之间(表 2)。
分别将各样品的Clean Reads与指定的参考基因组进行序列比对,比对效率从95.35%-95.47%不等。Uniq Mapped Reads的长度最长为39、135和963,最短为37、689和953,比对效率从89.14%-90.61%(表 3)。
表达具有时间和空间特异性,在两个不同条件下,表达水平存在显著差异的基因,称之为差异表达基因。本实验中,差异分组使用”T01_vs_T02_T03”的方式命名。根据两(组)样品之间表达水平的相对高低,差异表达基因可以划分为上调基因(Up-regulated基因)和下调基因(Down-regulated基因)。
对筛选出的差异表达基因做层次聚类分析,将具有相同或相似表达行为的基因进行聚类(图 4)。检测差异表达基因时,需要根据实际情况选取合适的差异表达分析软件(edgeR[25]、DEseq[25]和EBseq[27])。对于有生物学重复的样品的差异表达分析,采用DEseq软件进行差异筛选,将差异倍数(Fold Change)≥2,且FDR < 0.05作为筛选标准。Fold Change表示两样品(组)间表达量的比值,P为差异表达的显著性,对显著性P值进行校正会得到错误发现率FDR值,控制FDR小于一定阈值可以降低差异表达基因的假阳性率。不同的项目可根据项目的特异情况选择合适的指标进行差异筛选。

|
| 横坐标代表样品名称及样品的聚类结果,纵坐标代表差异基因的聚类结果。图中不同的列代表不同的样品,不同的行代表不同的基因。颜色代表了基因在样品中的表达量水平lg(基因+0.000001) 图 4 基因差异表达分析 |
GO数据库由GO组织(Gene ontology consortium)构建于2000年,旨在建立基因及其产物知识的标准词汇体系,适用于各个物种。GO注释系统包含3个主要分支,即生物学过程(Biological process)、细胞组分(Cellular component)和分子功能(Molecular function)。
样品间差异表达基因GO分类统计结果见图 5,横坐标为GO分类,纵坐标左边为基因数目所占百分比,右边为基因数目。此图展示的是在差异表达基因背景和全部基因背景下GO各二级功能的基因富集情况,体现两个背景下各二级功能的地位,具有明显比例差异的二级功能说明差异表达基因与全部基因的富集趋势不同,可以重点分析此功能是否与差异相关。

|
| 横坐标依次为:生物学过程(Biological process)(包括细胞进程,单一有机过程,生物调控,应激反应,代谢过程,多细胞组织过程,发育进程,信号转导,定位,细胞组成及生物起源,多元有机体,免疫系统进程,生殖过程,繁殖,运动,生物附着,生物行为,生物生长,节律进程,生物相位,脱毒过程,突触前进程,细胞凋亡,细胞聚集);细胞组分(Cellular component)(包括细胞组分,细胞,细胞器,细胞膜,膜组分,细胞器组分,大分子复合物,细胞外区域,细胞外区域组分,膜封闭腔,细胞连接,超分子复合物,突触,突触组分,拟核,病毒粒子,病毒粒子组分,其他生物体组分);分子功能(Molecular function)包括(结合,催化活性,信号转导活性,分子转导活性,转运蛋白活性,分子功能调节,核酸结合转录因子活性,结构分子活性,转录因子活性和蛋白结合,电子载体活性,抗氧化活性,翻译调控活性,化学诱导物活性,化学活性,金属伴侣活性,形态发生活性,蛋白质标签,营养物质活性) 图 5 样品间差异表达基因GO分类统计图 |
在生物学过程(Biological process)分支中,相比于PBS对照组(T01),噬菌体实验组(T02、T03)在细胞凋亡(Cell killing),细胞聚集(Cell aggregation)和脱毒过程(Detoxification)均呈现校高的差异基因表达数目,尤其是细胞聚集(Cell aggregation),噬菌体实验组(T02、T03)差异基因表达量比PBS对照组(T01)高2倍还多;突触前进程(Presynaptic process involved in chemical synaptic transmission)则呈现相反的变化趋势,PBS对照组差异基因表达量明显高于噬菌体实验组(T02、T03)。
在细胞组分(Cellular component)分支中,相比PBS对照组(T01),噬菌体实验组(T02、T03)在拟核(Nucleoid)中呈现更多的基因值。有趣的是,在病毒粒子(Virion)和病毒粒子组分(Virion part)这两个基因区域呈现完全相反的结果,在其他有机体(Other organism)及其他有机体组分(Other organism part)只检测到噬菌体实验组(T02、T03)的差异基因表达,其表达量约为200。
在分子功能(Molecular function)分支中,相比于PBS对照组(T01),噬菌体实验组(T02、T03)在抗氧化活性(Antioxidant activity),化学诱导物活性(Chemoattractant activity)相关的差异基因表达呈现出更多的基因数和基因百分数;而在翻译调控活性(Translation regulator activity)相关的差异表达基因,则呈现出相反的趋势,即对照组(T01)呈现更高的基因含量。
2.4.5 差异表达基因KEGG富集分析作为有关Pathway的主要公共数据库[28],KEGG(Kyoto encyclopedia of genes and genomes)提供的整合代谢途径(Pathway)查询,包括碳水化合物、核苷、氨基酸等的代谢及有机物的生物降解,不仅提供了所有可能的代谢途径,而且对催化各步反应的酶进行了全面的注解,包含有氨基酸序列、PDB库的链接等等,是进行生物体内代谢分析、代谢网络研究的强有力工具。采用R包clusterProfiler对差异基因分别进行KEGG通路富集分析。富集分析采用超几何检验方法来寻找与整个基因组背景相比显著富集的KEGG通路。
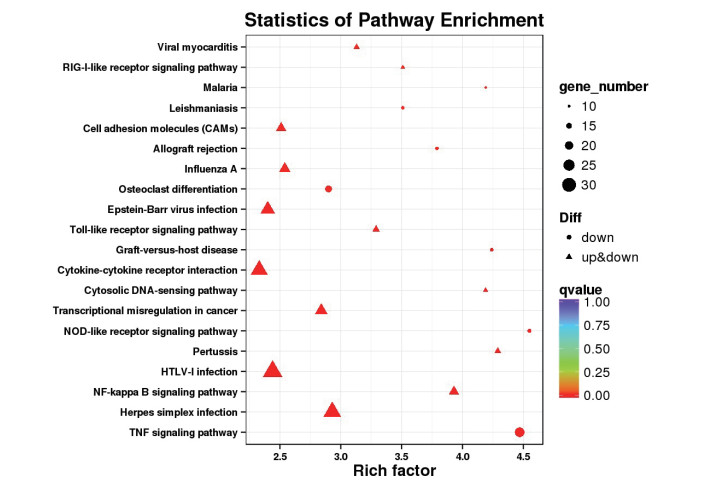
图 6中每一个行代表一个KEGG通路。横坐标为富集因子,表示差异基因中注释到该通路的基因比例与所有基因中注释到该通路的基因比例的比值。富集因子越大,表示差异表达基因在该通路中的富集水平越显著。点的颜色代表q value,点的大小代表注释在该通路中差异基因的数目,点的形状代表该通路中差异基因的类型(如只包含上调基因,只包含下调基因,同时包含上下调基因)。
|
| 横坐标为富集因子;纵坐标依次为:病毒性心肌炎通路,RIG-I-like受体信号通路、疟疾通路、黑热病通路、细胞黏附因子(CAMs),异源排斥反应,甲型流感,破骨细胞分化、EB病毒感染,Toll样受体信号通路,异源宿主排斥反应,细胞因子,细胞因子受体互作,胞质DNA-sensing通路、癌症转录失调,Nod样受体信号通路,百日咳通路,HTLV-I感染,NF-κB信号通路,单纯疱疹病毒通路,TNF信号通路 图 6 差异表达基因KEGG富集分析 |
相比对照组(T01),噬菌体灌注组(T02、T03)中,TNF信号通路富集显著,富集因子接近4.5,只包含下调基因;NF -κB信号通路富集较为显著,富集因子接近4.0,同时包含上下调基因;HTLV-I感染富集因子约为2.5,呈现出最多的差异基因数目,> 30,同时包含上下调基因。
3 讨论近年来,相关致病菌的抗生素耐药性日趋严重,这些多重耐药性菌株已对现代医疗界构成了极大威胁[29-30]。而在畜牧兽医与食品工业领域,亦面临相同的形势。这意味着研发一种经济、有效的抗生素替代物尤为迫切。噬菌体作抗生素替代物或食品抗菌剂使用具有诸多优势,在俄罗斯、格鲁吉亚和波兰等国均有长期且持续的使用历史,其安全性也成为现在研究的重要方向。
噬菌体的发现者Felix d’Herelle最早证实了噬菌体能够在正常人体微生物群中存在。他在人和动物粪便中发现了肠道噬菌体[2]。噬菌体和细菌在同一生态系统中的共存是自然界中常见的现象[30],并且在真核生物、细菌和古细菌分离之前就已经存在了[29]。这些结论表明,在某种程度上,噬菌体是能够和人类安全共存的。
本研究采用Illumina HiSeq高通量测序技术对灌注金黄色葡萄球菌噬菌体的小鼠转录组进行测序,并进行了基因差异表达分析、差异表达基因GO分类和差异表达基因KEGG富集分析。转录组测序产量和比对效率统计显示,共获得19.23 Gb Clean Data,Q30碱基百分比在92.82%及以上;各样品的Clean Reads比对效率从95.35%-95.47%不等。这些数据均表明转录组测序结果真实有效,符合实验分析的标准。
在差异表达基因GO分类分析中显示,噬菌体实验组(T02、T03)在细胞凋亡(Cell killing),细胞聚集(Cell aggregation)和脱毒过程(Detoxification)均呈现校高的差异基因表达数目;在拟核(Nucleoid)中呈现更多的基因值,而在病毒粒子(Virion)和病毒粒子组分(Virion part)这两个基因区域呈现完全相反的结果;在抗氧化活性(Antioxidant activity),化学诱导物活性(Chemoattractant activity)相关的差异基因表达呈现出更多的基因数和基因百分数。在差异表达基因KEGG富集分析中显示,噬菌体灌注组(T02 & T03)中,TNF信号通路富集显著,富集因子接近4.5,只包含下调基因;NF -κB信号通路富集较为显著,富集因子接近4.0,同时包含上下调基因。
这些结果表明,在噬菌体灌注之后,小鼠体内会有一些相关的免疫反应和炎症应答,本实验中的HE染色和TNF-α的测定也得到了相应的结果。噬菌体在乳腺内的PFU检测结果表明,24 h后噬菌体的数量会有较为明显的下降,其PFU从107 PFU/gland下降到103-4 PFU/gland。相比于正常组,噬菌体灌注组小鼠未呈现明显的生理体征区别,其状态正常,表现为精神状态良好,毛发略有轻度蓬松,行走正常。综上所述,尽管噬菌体处理后会有一定的炎症因子上调,但其HE染色和小鼠生理体态以及其他相关数据均表明,炎症趋势轻微,且噬菌体数量会随着时间推移下降,并不会影响小鼠的正常生命体征。
Koen等[31]在实验中发现,相比于攻菌后的噬菌体鸡尾酒治疗组,单一噬菌体鸡尾酒灌注组小鼠(安全对照组)呈现出较低的PFU值,其HE染色结果也更接近于正常小鼠。这也说明在正常小鼠乳腺内,噬菌体数量会有自然的下降,其下降程度与时间成正比。Oliveira等[32]研究发现,将3种裂解性噬菌体-F78E(肌尾噬菌体)、phi F258E(长尾噬菌体)和phi F61E(肌尾噬菌体)分别以口服、鼻喷雾和腹腔注射的方式给药(108 PFU/mL),3 h后,噬菌体在脾脏、肝脏和肺部均能够检测到。它们在脾脏中的浓度分别为3×104、3.5×103和1×102 PFU/mL。在接下来的几个小时内,噬菌体浓度迅速下降,24 h后,噬菌体几乎被清除。
同时,20世纪20年代至30年代的早期研究[33]也表明噬菌体在血液中能够被迅速清除。腹腔注射噬菌体[34]的小鼠血噬菌体滴度(高达1010 PFU/mL)迅速升高,12 h后,滴度下降约103倍,并继续呈软曲线下降,下降趋势因不同类型噬菌体而异。一些噬菌体可以被血液中的红细胞和白细胞吸收[35]。噬菌体颗粒在肝脏会很快失活[32, 36],在脾脏中存活的时间稍长[32, 37],噬菌体抗体的产生会影响噬菌体治疗的效果。此外,从未接受过药物治疗的患者也有一定程度的噬菌体抗体[38]。然而,现有的数据表明,这种抗体的存在与治疗结果的相关性很弱[39]。这些研究结果与本实验结果相似,随着时间的推移,噬菌体在实验动物体内会有下调乃至失活的趋势。
导致噬菌体失活的原因目前尚无定论,戈尔斯基教授领导的一个研究小组最近证实了某些噬菌体颗粒对人类和动物免疫系统的直接影响。结果表明,噬菌体颗粒能降低由细菌或内毒素诱导的中性粒细胞产生的活性氧,抑制T细胞的活化,从而促进移植耐受。这些噬菌体还具有一定程度的抗肿瘤作用[40-41]。研究指出,溃疡性肠黏膜上噬菌体颗粒的浓度显著增加,噬菌体的免疫活性在克罗恩病的病理过程中起一定作用[42]。
另外有人认为,一些噬菌体可以携带“二级”粘附蛋白,这些黏附蛋白与触发DNA注入的装置并没有直接联系。许多假定的二级粘连蛋白含有免疫球蛋白样结构域,这些结构域暴露在噬菌体头部表面、尾部可收缩的覆盖物或噬菌体衣壳的末端[43]。在实验室条件下,这些结构是否能影响噬菌体生长的数据仍然缺失,但在诸如动物体内,乳腺或者肠道中的生态情况下,它们可能具有很大的作用[44]。这些粘附蛋白,可以通过增加噬菌体粒子与感受性宿主细胞相遇后可逆吸收的可能性来实现,从而增加噬菌体的感染率。一方面可以提高噬菌体对宿主菌的侵染效率,从而快速的杀灭病原菌[45],另一方面,也可能会引发机体一定的免疫反应,在开发治疗性噬菌体制剂时,应纵横考虑这些想法,并找到一个平衡点[46]。然而,到目前为止,还没有实验数据来描述天然病毒与免疫系统的相互作用。
迄今为止,噬菌体治疗的严重不良事件还没有任何报道,这证明了噬菌体治疗可以预防耐药细菌感染[47]。与抗生素相反,噬菌体是自我给药和自我限制的,因而耐药性的形成通常较低[48-49]。目前为止,在超过一百年的时间里,没有报告过由噬菌体治疗引起的严重不良事件[50]。然而,仍然需要制定或采取措施来减轻潜在的安全问题。其中一个挑战是噬菌体制剂中必然含有一定量的内毒素或其他有害的细菌成分。而这个问题可以通过使用高纯度的噬菌体制剂来解决[49]。另一个安全问题是细菌溶解可能导致内毒素快速释放[51]。为避免这种情况发生,可以通过基因工程设计非复制噬菌体,使宿主细菌失活而不溶解它们[52]。然而,最近的一项研究发现,噬菌体比β-内酰胺类抗生素能够更快地杀死致病性大肠杆菌,同时,噬菌体释放的内毒素也更少[53]。
对于噬菌体的使用,常见的反对意见之一,是可能出现严重的不良免疫反应(如过敏反应)。但目前的文献中并无描述,使用“噬菌体”和“过敏反应”这两个术语搜索出版物数据库及其变化,并没有发现任何一个病例报告。从大量关于噬菌体使用的历史报告来看,无论给药途径如何,噬菌体治疗后的过敏反应似乎并不常见[54]。“噬菌体”在某些情况下是一种安全有效的抗生素替代品[55]。
4 结论结合本实验结果,鉴于噬菌体治疗会导致一些轻微的炎症反应,以及噬菌体自身数量的下调,可以考虑使用噬菌体和抗生素联合使用的治疗方案。这样不但可以减缓噬菌体可能导致的不良反应,而且还能够降低抗生素的最小使用剂量,从而达到更好的治疗效果。有报道称噬菌体在提高多重耐药细菌对抗生素的敏感性方面有着显著的效果[56]。Chaudhry等[57]的一项研究,报道了噬菌体和抗生素治疗铜绿假单胞菌生物膜的不同策略,同时证明这两种试剂的给药顺序对生物膜根除的结果产生了严重影响。由此可见,利用噬菌体增强抗生素作用的价值是不可低估的。同时,噬菌体的生长优化和纯化也是亟待解决的问题[7]。
如果噬菌体的繁殖率高于其扩散和失活的速率,那么噬菌体将能够有效抑制宿主菌的生长,这被称为噬菌体主动治疗。而在另外一些案例中,为了抑制细菌生长,必须通过人工多次引入大量噬菌体,以保持噬菌体的浓度。例如,在慢性病治疗期间,这种情况被称为噬菌体被动治疗。在治疗稳定的慢性感染过程中,将主动和被动噬菌体疗法进行结合是最可能的方案。这种治疗需要更长的时间,也能够更大概率地治愈患者[58-59]。
目前国内外在噬菌体治疗方面的研究已经如火如荼,但采用转录组测序技术研究噬菌体对动物的影响极少。本实验首次采用转录组测序技术,对噬菌体灌注后的小鼠乳腺组织进行安全性分析,发现实验后小鼠会有轻微炎症反应,但其生命体征未受影响。在未来的实验中,可以考虑抗生素和噬菌体的联合使用,以期达到更好的治疗效果。本实验为噬菌体安全性治疗和噬菌体制剂的发展提供了重要的实验依据,为噬菌体替代或者辅助抗生素的使用奠定了实验基础。
| [1] |
d'Herelle F. Bacteriophage as a treatment in acute medical and surgical infections[J]. Bull N Y Acad Med, 1931, 7(5): 329-348. |
| [2] |
d'Herelle F. On an invisible microbe antagonistic toward dysenteric bacilli: brief note by Mr. F. D'Herelle, presented by Mr. Roux[J]. Research in Microbiology, 2007, 158(7): 553-554. DOI:10.1016/j.resmic.2007.07.005 |
| [3] |
Twort WF. An investigation on the nature of ultramicroscopic viruses[J]. Lancet, 1915, 186(4814): 1241-1243. DOI:10.1016/S0140-6736(01)20383-3 |
| [4] |
Keen EC. Felix d'Herelle and our microbial future[J]. Future Microbiology, 2012(7): 1337-1339. |
| [5] |
Salmond GP, Fineran PC. A century of the phage: past, present and future[J]. Nature Reviews Microbiology, 2015, 13(12): 777-786. DOI:10.1038/nrmicro3564 |
| [6] |
Biswajit B, Sankar A, Paul W, et al. Bacteriophage therapy rescues mice bacteremic from a clinical isolate of vancomycin-resistant Enterococcus faecium[J]. Infection & Immunity, 2002, 70(1): 204-210. |
| [7] |
Jamal M. Bacteriophages: an overview of the control strategies against multiple bacterial infections in different fields[J]. J Basic Microbiol, 2019, 59(2): 123-133. DOI:10.1002/jobm.201800412 |
| [8] |
Hawkey PM, Jones AM. The changing epidemiology of resistance[J]. J Antimicrob Chemother, 2009, 64(S1): 3-10. |
| [9] |
Llanos CA, Citorik RJ, Nickerson KP, et al. Bacteriophage therapy testing against shigella flexneri in a novel human intestinal organoid-derived infection model[J]. Journal of Pediatric Gastroenterology and Nutrition, 2019, 68: 509-516. DOI:10.1097/MPG.0000000000002203 |
| [10] |
Krylov V, Shaburova O, Pleteneva E, et al. Selection of phages and conditions for the safe phage therapy against Pseudomonas aeruginosa infections[J]. Virologica Sinica, 2015, 30(1): 33-44. DOI:10.1007/s12250-014-3546-3 |
| [11] |
Lin DM, Koskella B, Lin HC. Phage therapy: An alternative to antibiotics in the age of multi-drug resistance[J]. World Journal of Gastrointestinal Pharmacology and Therapeutics, 2017, 8(3): 162-173. DOI:10.4292/wjgpt.v8.i3.162 |
| [12] |
Jean-Paul P, Gilbert V, Pieter-Jan C, et al. The magistral phage[J]. Viruses, 2018, 10(2): 64-71. DOI:10.3390/v10020064 |
| [13] |
Doss J, Culbertson K, Hahn D, et al. A review of phage therapy against bacterial pathogens of aquatic and terrestrial organisms[J]. Viruses, 2017, 9(3): 50-60. DOI:10.3390/v9030050 |
| [14] |
Jennes S, Merabishvili M, Soentjens P, et al. Use of bacteriophages in the treatment of colistin-only-sensitive Pseudomonas aeruginosa septicaemia in a patient with acute kidney injury-a case report[J]. Critical Care, 2017, 21(1): 129-131. |
| [15] |
Dedrick RM, Guerrero-Bustamante CA, et al. Engineered bacteriophages for treatment of a patient with a disseminated drug-resistant Mycobacterium abscessus[J]. Nature Medicine, 2019, 25(5): 730-733. |
| [16] |
Quail MA, Kozarewa I, Smith F, et al. A large genome center's improvements to the Illumina sequencing system[J]. Nature Methods, 2008, 5(12): 1005-1010. DOI:10.1038/nmeth.1270 |
| [17] |
Geng HJ, Zou W, et al. Evaluation of phage therapy in the treatment of Staphylococcus aureus-induced mastitis in mice[J]. Folia Microbiol, 2019. DOI:10.1007/s12223-019-00729-9 |
| [18] |
李跃.金黄色葡萄球菌裂解性噬菌体治疗乳房炎的实验研究[D].长春: 吉林大学, 2014. http://d.wanfangdata.com.cn/Conference_8484707.aspx
|
| [19] |
Minsik K, Sangryeol R. Characterization of a T5-like coliphage, SPC35, and differential development of resistance to SPC35 in Salmonella enterica serovar typhimurium and Escherichia coli[J]. Applied & Environmental Microbiology, 2011, 77(6): 2042-2050. |
| [20] |
Bryan D, El-Shibiny A, Hobbs Z, et al. Bacteriophage T4 infection of stationary phase E. coli: life after log from a phage perspective[J]. Frontiers in Microbiology, 2016, 7(9): 1391. |
| [21] |
张建城.溶藻弧菌噬菌体特性及其防控刺参感染的研究[D].大连: 大连理工大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10141-1017243527.htm
|
| [22] |
Tuchscherr LP, Buzzola FR, Alvarez LP, et al. Capsule-negative Staphylococcus aureus induces chronic experimental mastitis in mice[J]. Infection & Immunity, 2005, 73(12): 7932-7937. |
| [23] |
Schmeck B, Moog K, Zahlten J, et al. Streptococcus pneumoniae induced c-Jun-N-terminal kinase- and AP-1 -dependent IL-8 release by lung epithelial BEAS-2B cells[J]. Respiratory Research, 2006, 7(1): 98. |
| [24] |
曲长庆, 邹伟, 张叶军. 一种制作高质量动物组织石蜡切片的方法[J]. 实验室科学, 2018, 21(2): 75-78. DOI:10.3969/j.issn.1672-4305.2018.02.020 |
| [25] |
Robinson MD, Mccarthy DJ, Smyth GK. edgeR: a Bioconductor package for differential expression analysis of digital gene expression dat[J]. Bioinformatics, 2010, 26(1): 139-140. |
| [26] |
Anders SH, Huber W. Differential expression analysis for sequence count data[J]. Genome Biol, 2010, 11(10): R106. DOI:10.1186/gb-2010-11-10-r106 |
| [27] |
Kendziorski NL. EBSeq: An R package for gene and isoform differential expression analysis of RNA-seq data[M], 2014.
|
| [28] |
Shujiro O, Takuji Y, Masami H, et al. KEGG Atlas mapping for global analysis of metabolic pathways[J]. Nucl Acids Res, 2008, 36(S2): 423-426. |
| [29] |
Bamford DH. Do viruses form lineages across different domains of life?[J]. Research in Microbiology, 2003, 154(4): 231-236. DOI:10.1016/S0923-2508(03)00065-2 |
| [30] |
Weinbauer MG. Ecology of prokaryotic viruses[J]. FEMS Microbiology Reviews, 2010, 28(2): 127-181. |
| [31] |
Koen B, Ryan WH, Zachary H, et al. Efficacy and safety of a bovine-associated staphylococcus aureus phage cocktail in a murine model of mastitis[J]. Frontiers in Microbiology, 2017, 8: 2348-2357. DOI:10.3389/fmicb.2017.02348 |
| [32] |
Oliveira A, Sereno R, Nicolau A, et al. The influence of the mode of administration in the dissemination of three coliphages in chickens[J]. Poult Sci, 2009, 88(4): 728-733. |
| [33] |
Sulakvelidze A, Alavidze Z, Morris J. Bacteriophage therapy[J]. Antimicrobial Agents & Chemotherapy, 1934, 2(3858): 1110-1113. |
| [34] |
Uchiyama J, Maeda Y, Takemura I, et al. Blood kinetics of four intraperitoneally administered therapeutic candidate bacteriophages in healthy and neutropenic mice[J]. Microbiology & Immunology, 2010, 53(5): 301-304. |
| [35] |
Bystricky V, Drahos V, Mulczyk M, et al. On the structure of some bacteriophages[J]. Acta Virologica, 1964, 8: 369-372. |
| [36] |
Inchley CJ. The actvity of mouse Kupffer cells following intravenous injection of T4 bacteriophage[J]. Clinical & Experimental Immunology, 1969, 5(1): 173-187. |
| [37] |
Sulakvelidze A. Phage therapy: an attractive option for dealing with antibiotic-resistant bacterial infections[J]. Drug Discovery Today, 2005, 10(12): 807-809. DOI:10.1016/S1359-6446(05)03441-0 |
| [38] |
Slopek S, Weber-Dabrowska B, Dabrowski M, et al. Results of bacteriophage treatment of suppurative bacterial infections in the years 1981-1986[J]. Archivum Immunologiae Et Therapiae Experimentalis, 1987, 35(5): 569-583. |
| [39] |
Letarov AV, Golomidova AK, Tarasyan KK. Ecological basis for rational phage therapy[J]. Acta Naturae, 2010, 2(1): 60-72. DOI:10.32607/20758251-2010-2-1-60-71 |
| [40] |
Miedzybrodzki R, Switalajelen K, Fortuna W, et al. Bacteriophage preparation inhibition of reactive oxygen species generation by endotoxin-stimulated polymorphonuclear leukocytes[J]. Virus Research, 2008, 131(2): 233-242. |
| [41] |
Pajtaszpiasecka E, Rossowska J, Duś D, et al. Bacteriophages support anti-tumor response initiated by DC-based vaccine against murine transplantable colon carcinoma[J]. Immunology Letters, 2008, 116(1): 24-32. DOI:10.1016/j.imlet.2007.11.004 |
| [42] |
Lepage P, Colombet J, Marteau P, et al. Dysbiosis in inflammatory bowel disease: a role for bacteriophages?[J]. Gut, 2008, 57(3): 424-425. DOI:10.1136/gut.2007.134668 |
| [43] |
Fraser JS, Maxwell KL, Davidson AR. Immunoglobulin-like domains on bacteriophage: weapons of modest damage?[J]. Current Opinion in Microbiology, 2007, 10(4): 382-387. DOI:10.1016/j.mib.2007.05.018 |
| [44] |
Letarov A, Manival X, Desplats C, et al. Gpwac of the T4-type bacteriophages: Structure, function, and evolution of a segmented coiled-coil protein that controls viral infectivity[J]. Journal of Bacteriology, 2005, 187(3): 1055-1066. |
| [45] |
Gallet R, Shao Y, Wang IN. High adsorption rate is detrimental to bacteriophage fitness in a biofilm-like environment[J]. BMC Evolutionary Biology, 2009, 9(1): 241-250. |
| [46] |
Mertz L. Battling superbugs: how phage therapy went from obscure to promising[J]. IEEE Pulse, 2019, 10(1): 3-9. |
| [47] |
Mya B, Chelsea B, Kema M, et al. Phage puppet masters of the marine microbial realm[J]. Nature Microbiology, 2018, 3(6): 754-766. |
| [48] |
Örmälä AM, Jalasvuori M. Phage therapy: Should bacterial resistance to phages be a concern, even in the long run?[J]. Bacteriophage, 2013, 3(1): 214-219. |
| [49] |
Loc-Carrillo C, Abedon ST. Pros and cons of phage therapy[J]. Bacteriophage, 2011, 1(2): 111-114. DOI:10.4161/bact.1.2.14590 |
| [50] |
Mölling K. Viren statt antibiotika(in German)[J]. Spektrum der Wissenschaft, 2017, 10(17): 42-49. |
| [51] |
Reindel R, Fiore CR. Phage therapy: considerations and challenges for development[J]. Clinical Infectious Diseases, 2017, 64(11): 1589-1590. DOI:10.1093/cid/cix188 |
| [52] |
Hagens S, Habel A, von Ahsen U, et al. Therapy of experimental Pseudomonas infections with a nonreplicating genetically modified phage[J]. Antimicrobial Agents & Chemotherapy, 2004, 48(10): 3817-3822. |
| [53] |
Dufour N, Delattre RL, Ricard J, et al. The lysis of pathogenic Escherichia coli by bacteriophages releases less endotoxin than by β-Lactams[J]. Clinical Infectious Diseases An Official Publication of the Infectious Diseases Society of America, 2017, 64(11): 1582-1588. DOI:10.1093/cid/cix184 |
| [54] |
Speck P, Smithyman A. Safety and efficacy of phage therapy via the intravenous route[J]. FEMS Microbiology Letters, 2015, 363(3): 242-247. |
| [55] |
Broecker KM, Willy AC. A wake-up call: we need phage therapy now[J]. Viruses, 2018, 10(12): 688-702. DOI:10.3390/v10120688 |
| [56] |
Chan BK, Sistrom M, Wertz JE, et al. Phage selection restores antibiotic sensitivity in MDR Pseudomonas aeruginosa[J]. Scientific Reports, 2016, 6: 267-271. |
| [57] |
Chaudhry WN, Concepción-Acevedo J, Park T, et al. Synergy and order effects of antibiotics and phages in killing Pseudomonas aeruginosa biofilms[J]. PLoS One, 2017, 12(1): e168615. |
| [58] |
Andrzej G, Ryszard M, Jan B, et al. Bacteriophage therapy for the treatment of infections[J]. Current Opinion in Investigational Drugs, 2009, 10(8): 766-774. |
| [59] |
O'Flaherty S, Ross RP, Coffey A. Bacteriophage and their lysins for elimination of infectious bacteria[J]. FEMS Microbiology Reviews, 2010, 33(4): 801-819. |