果实成熟软化一直是采后生物学领域备受关注的热点科学问题,其包括细胞壁的降解、内含物的变化、呼吸速率以及其他的代谢变化等非常复杂的发育调控过程。目前,对果实成熟软化的机制仍不清楚。番茄(Solanum lycopersicum)在我国南北方的种植面积广泛,是茄科作物(番茄、辣椒、茄子、马铃薯等)的重要代表作物,属于典型的呼吸跃变型果实,也是研究果实发育、成熟调控及产后果实品质的理想模式作物[1]。开展番茄果实成熟软化的分子机理研究,找出关键的影响因子,并对其作用及分子机制进行深入研究,不仅丰富了果实采后软化理论研究,而且可为通过生物技术手段来改善果实软化提供技术手段,具有潜在的商业应用价值[2]。
MicroRNAs(miRNAs)几乎参与所有的生物学过程[3-7]。目前,与果实成熟软化相关的大量miRNAs被发现[8-9]。如:番茄miR161、miR173、miR393、miR397、miR398和miR414只有在绿熟期果实中表达量增加;miR159表达量在破色期果实中下降,到红熟期增加;miR156和miR394在成熟期表达量下降,miR396在破色期表达量明显增加;miR828和miR1917表达在红熟期下降[8]。miRNAs不仅在果实成熟各个时期表现出差异表达模式,而且其中一些miRNAs的表达受到乙烯调控,如:番茄miR394、miR414和miR1917的表达受乙烯负调控,而miR156、miR159、miR396、miR482和miRZ6027则为乙烯诱导表达[8-9]。香蕉miR156、miR162、miR171、miR393和miR172等受乙烯诱导表达[10]。上述结果预示miRNAs参与果实成熟过程,但相关的功能研究却鲜有报道。
反向遗传学主要通过超表达或者沉默表达方法实现对基因功能的研究。利用超表达载体转化植物,从而提高miRNA在体内的表达量的方法是一种已被广泛应用于植物miRNA功能研究的成熟方法[11]。由于miRNA小,对miRNA功能沉默的方法一直不能取得令人满意的效果。近年来,短串联靶标模拟技术(STTM)的发现揭示了一种全新的miRNA沉默调控机制,并在miRNA的功能研究中得到应用。STTM是在靶标模拟技术(Target mimic,TM)基础上开发的一种miRNA沉默技术。目前,STTM技术已经应用于多个番茄miRNA功能的研究中,被证明是一种高效的番茄miRNA沉默技术[12-13],但该方法尚未在番茄果实成熟和软化相关研究中得到应用。
本研究利用生物信息学分析,分离鉴定出Sl-miR482前体序列并设计寡核苷酸片段,构建STTM载体,从而为深入阐明Sl-miR482在番茄果实成熟软化中的作用机制奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株和载体大肠杆菌DH5α、农杆菌EHA105、植物表达载体pcamiba-35S来源于玉林师范学院分子生物实验室,克隆载体PEASY- Blunt Clong Kit购于TRANSGENE公司。Heinz和LA1589番茄材料种植于玉林师范学院温室中。
1.1.2 试验试剂及试剂盒DNA聚合酶、限制性内切酶(KpnⅠ、BamhⅠ)、T4连接酶购于Thermo Fisher Scientific公司。质粒提取试剂盒、凝胶回收试剂盒购于Omega公司。
1.2 试验方法 1.2.1 定量PCR采用茎环反转录法合成成熟miRNA的cDNA,在成熟miRNA的3’端设计一条50 nt可自行折叠成茎环结构的特异性RT引物,miR482a_SLR:5’-GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTAGGAA-3’(下划线表示与成熟miRNA3’端反相互补的6个碱基)。设计定量PCR引物(miR482a_SLR_F:5’-GCGGCGT-TccaaTTccacccaT-3’,SLR_R:5’-GTGCAGGGTCCG-AGGT-3’),以cDNA为模板,应用SYBR染料法,对Sl-miR482采用实时定量检测。以U6为内参,U6RT引物及定量PCR引物序列如下:Universal Reverseprimer:5’-GTGCAGGGTCCGAGGTATTC-3’,U6-F:5’-AGACAATTGATGCGTGCGATC-3’,U6-R:5’-GCTGCAACTGCACTACCAAC-3’。按照2-ΔΔCt方法计算基因表达量。
1.2.2 Sl-miR482启动子分析从https://www.solgenomics.net/网站中比对出Sl-miR482序列,选取转录起始位点上游2 000 bp核苷酸序列,利用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对该区域存在的顺式作用元件进行分析。
1.2.3 靶基因预测利用生物信息学网站(http://plantgrn.noble.org/v1_psRNATarget/)对Sl-miR482靶基因进行预测。利用DNAMAN软件进行靶基因同源家族氨基酸序列比对。
1.2.4 STTM-482载体的构建根据Sl-miR482序列,分别设计合成寡核苷酸序列(表 1),以Linker-STTM为模板,进行STTM-482的克隆。进而构建中间载体pEasy-STTM-482。然后以含有35S启动子的pCambia-1300-35S载体作为整体支架,使用KpnⅠ和BamhⅠ双酶切pEasy-STTM-482和pCambia-1300-35S,分别回收目的片段,用T4连接酶将目的基因:载体=3:1进行连接,经生工生物(上海)有限公司测序验证正确,命名为pCambia-35S-STTM-482。
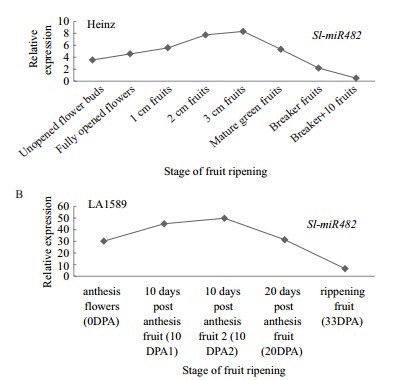
利用Sl-miR482特异性引物对番茄Heinz和LA1589果实发育不同时期中Sl-miR482表达量进行检测,定量PCR结果表明,尽管Heinz和LA1589番茄果实在成熟期长短存在差异,但Sl-miR482的表达量在2种番茄开花期都逐渐上升,到未成熟绿期达到最高峰,随后急剧下降(图 1)。植物基因的时空特异性表达模式,往往预示该基因在特殊的组织或时间内发挥作用。因此,Sl-miR482在果实成熟不同时期的动态表达模式,表明它可能参与调节果实成熟过程,其具体作用及机制有待于进一步阐明。
|
| 图 1 Sl-miR482分别在Heinz和LA1589番茄果实发育时期中的表达模式 |
利用靶基因预测网站(http://plantgrn.noble.org/v1_psRNATarget/)对Sl-miR482靶基因进行预测,结果表明(图 2-A),番茄Solyc11g008140(一种果胶裂解酶基因家族成员,将其命名为Sl-PL13)为其潜在的靶基因。进一步定量PCR结果(图 2-B)表明,在番茄果实发育不同时期,Sl-PL13的表达量在绿熟期后逐渐升高,在破色期达到最高值。此外,在番茄果实成熟阶段,Sl-PL13与Sl-miR482的表达模式呈现一定的此消彼长的反比关系。上述结果预示Sl-miR482可能通过调节Sl-PL13的表达量从而实现对番茄果实成熟的控制。

|
| A:Sl-miR482与Sl-PL13 mRNA 3’-UTR序列配对分析;B:Sl-PL13在果实发育各个时期的表达 图 2 Sl-miR482靶基因预测及靶基因表达分析 |
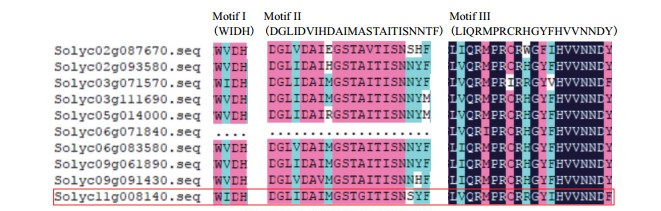
Sl-PL(Solyc03g111690)是一种调节番茄果实软化的果胶裂解酶基因[2, 14]。蛋白序列比对结果表明,Sl-PL13与Sl-PL蛋白序列同源性为55.88%(图 3)。根据Sl-PL蛋白序列信息,本研究利用生物信息学分析从番茄基因组数据库(http://solgenomics.net/)中筛选出10种番茄果胶裂解酶基因家族成员,包括Sl-PL和Sl-PL13在内的所有家族成员都具有3个序列保守的功能区Motif Ⅰ、Motif Ⅱ、Motif Ⅲ(图 4),表明Sl-miR482可能通过果胶裂解酶的活性参与对番茄果实成熟软化的调节。

|
| 图 3 番茄中SlPL13与SlPL氨基酸序列比对 |
|
| 红色边框中为Sl-PL13及其蛋白结构域 图 4 番茄中SlPL家族成员氨基酸序列比对结果 |
选择Sl-miR482转录起始位点上游2 000 bp的DNA序列,通过PlantCARE数据库,对该区域进行基因启动子元件分析。结果(图 5)表明,Sl-miR482启动子中有与过程成熟相关的应答元件[15],包括:6个乙烯应答原件(ERE)、1个茉莉酸甲酯(TGACG-motif)和3个水杨酸等应答原件(TCA-element),表明Sl-miR482在果实成熟过程中发挥着重要的调节作用。

|
| 图 5 Sl-miR482启动子顺式作用元件分析 |
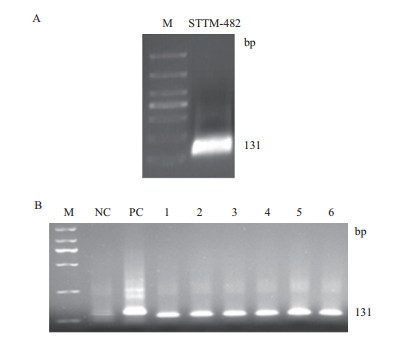
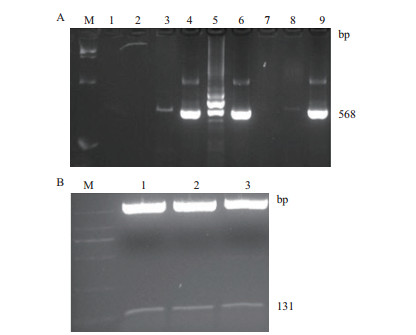
通过接头序列linker-STTM将寡核苷酸片段F-STTM482及R-STTM482连接,得到131 bp长度的STTM-482序列(图 6-A),回收片段,连入pEasy-blunt载体,经菌落PCR及测序验证后,pEasy-blunt-STTM载体构建成功(图 6-B和图 6-C)。利用KpnⅠ和BamhⅠ酶切pCambia-35S及重组质粒pEasy-blunt-STTM,连接后转化,通过菌落PCR验证及酶切验证,pCambia-35S-STTM-482植物表达载体构建成功(图 7-8)。
|
| A:STTM-482的PCR扩增(M:DL2 000分子标记);B:pEasy-blunt-STTM重组子菌落PCR鉴定(M:DL2 000分子标记;NC:阴性对照;PC:阳性对照;1-6:菌落PCR扩增条带) 图 6 pEasy-blunt-STTM重组子菌落PCR鉴定及测序结果 |
|
| A:菌落PCR鉴定(M:DL15 000分子标记);B:pCambia-35S-STTM-482质粒的双酶切验证(M:DL2 000分子标记) 图 7 重组子pCambia-35S-STTM-482的菌落PCR和酶切验证 |

|
| 图 8 STTM-482沉默载体的结构示意图 |
豌豆miR482能够明显增加豌豆结节数目[16];棉花miR482能调节靶基因NBS-LRR的表达影响真菌病原菌感染棉花[17]。2012年,Zuo等[8]发现Sl-miR482在番茄果实成熟软化过程中显示出差异的表达模式,而且Sl-miR482的表达受到乙烯的调节。上述结果表明,miR482不仅参与调控植物生长发育、抗病性等过程,而且在果实成熟软化过程也发挥着重要的作用。此外,茉莉酸甲酯和水杨酸都是果实成熟过程中重要的植物激素,作为应激激素广泛参与调控植物生长发育过程,与果实的成熟衰老有着密切的联系[15]。Sl-miR482启动子中存在茉莉酸甲酯、水杨酸应答元件,进一步表明Sl-miR482参与番茄果实成熟软化过程的调节。
靶基因预测结果显示,在众多可能的靶基因中,由于Sl-PL13与Sl-miR482二者在果实发育期中的表达呈现此消彼长的反比关系。因此,我们认为番茄果胶裂解酶Sl-PL13是Sl-miR482的靶基因,二者的靶向调节关系还有待进一步深入探究。果胶裂解酶别名为果胶酸反式消除酶,已有的研究证实,抑制一种番茄Sl-PL(Solyc03g111690)基因表达,导致果实软化速度明显变慢,但没有影响果实颜色和呈味物质的积累(如酸、糖和香气)[2, 14]。最近几年,PLs(果胶裂解酶基因家族)成为果实成熟软化机制研究的热点和重点。在呼吸跃变型果实和非呼吸跃变型果实中,部分果胶裂解酶基因在成熟果实中表达量达到最高峰,随后逐渐衰减[2, 18-23]。此外,有些PLs基因表达受到内源及外源乙烯的诱导。PLs基因在果实成熟中特异性表达及乙烯诱导表达模式表明,该基因家族在果实成熟过程中促进果肉细胞壁中果胶物质的分解,进而增加聚半乳糖醛酸的溶解性,最终导致果实软化,其具体机制有待于进一步深入阐明。
为探究Sl-miR482对番茄果实成熟软化的功能及调控机制,本研究检测了Sl-miR482在番茄果实发育不同阶段的表达,分析了启动子中顺式作用元件,预测其靶基因为一种果胶酸裂解酶Sl-PL13,并通过STTM技术构建能在植物中表达的pcambia1300-STTM- miR482沉默表达载体,为进一步探索Sl-miR482的功能奠定基础,同时为利用生物技术改善番茄果实成熟提供了候选基因资源。
4 结论筛选出番茄果实成熟软化相关的Sl-miR482,Sl-miR482可能通过抑制果胶酸裂解酶Sl-PL13的活性实现对番茄果实成熟软化的调节。
| [1] |
Wang D, Yeats TH, Uluisik S, et al. Fruit softening: Revisiting the role of pectin[J]. Trends in Plant Science, 2018, 23(4): 302-310. |
| [2] |
Uluisik S, Chapman NH, Smith R, et al. Corrigendum: Genetic improvement of tomato by targeted control of fruit softening[J]. Nature Biotechnology, 2016, 34(10): 1072. |
| [3] |
Liu Z, Li J, Wang L, et al. Repression of callus initiation by the miRNA-directed interaction of auxin-cytokinin in Arabidopsis thaliana[J]. Plant J, 2016, 87(4): 391-402. DOI:10.1111/tpj.13211 |
| [4] |
Zhang Y, Chen X, Yang F, et al. miRNA: A novel link between rice ragged stunt virus and Oryza sativa[J]. Indian Journal of Microbiology, 2016, 56(2): 219-224. DOI:10.1007/s12088-016-0572-x |
| [5] |
Balyan S, Kumar M, Mutum RD, et al. Identification of miRNA-mediated drought responsive multi-tiered regulatory network in drought tolerant rice, Nagina 22[J]. Scientific Reports, 2017, 7(1): 15446. DOI:10.1038/s41598-017-15450-1 |
| [6] |
Taller D, Balint J, Gyula P, et al. Correction: Expansion of capsicum annum fruit is linked to dynamic tissue-specific differential expression of miRNA and siRNA profiles[J]. PLoS One, 2018, 13(8): e0203582. DOI:10.1371/journal.pone.0203582 |
| [7] |
Somyong S, Poopear S, Sunner SK, et al. ACC oxidase and miRNA 159a, and their involvement in fresh fruit bunch yield(FFB)via sex ratio determination in oil palm[J]. Molecular Genetics and Genomics, 2016, 291(3): 1243-1257. DOI:10.1007/s00438-016-1181-4 |
| [8] |
Zuo J, Zhu B, Fu D, et al. Sculpting the maturation, softening and ethylene pathway: the influences of microRNAs on tomato fruits[J]. BMC Genomics, 2012, 13: 7. DOI:10.1186/1471-2164-13-7 |
| [9] |
Gao C, Ju Z, Cao D, et al. MicroRNA profiling analysis throughout tomato fruit development and ripening reveals potential regulatory role of RIN on microRNAs accumulation[J]. Plant Biotechnology Journal, 2015, 13(3): 370-382. DOI:10.1111/pbi.12297 |
| [10] |
Bi F, Meng X, Ma C, et al. Identification of miRNAs involved in fruit ripening in Cavendish bananas by deep sequencing[J]. BMC Genomics, 2015, 16: 776. DOI:10.1186/s12864-015-1995-1 |
| [11] |
Todesco M, Rubio-Somoza I, Paz-Ares J, et al. A collection of target mimics for comprehensive analysis of microRNA function in Arabidopsis thaliana[J]. PLoS Genetics, 2010, 6(7): e1001031. DOI:10.1371/journal.pgen.1001031 |
| [12] |
Kravchik M, Stav R, Belausov E, et al. Functional characterization of microRNA171 family in tomato[J]. Plants, 2019, 8(1): 1-16. |
| [13] |
Jiang N, Meng J, Cui J, et al. Function identification of miR482b, a negative regulator during tomato resistance to Phytophthora infestans[J]. Horticulture Research, 2018, 5: 9. DOI:10.1038/s41438-018-0017-2 |
| [14] |
Yang L, Huang W, Xiong F, et al. Silencing of SlPL, which encodes a pectate lyase in tomato, confers enhanced fruit firmness, prolonged shelf-life and reduced susceptibility to grey mould[J]. Plant Biotechnology Journal, 2017, 15(12): 1544-1555. DOI:10.1111/pbi.12737 |
| [15] |
Bleecker AB, Kende H. Ethylene: a gaseous signal molecule in plants[J]. Annual Review of Cell & Developmental Biology, 2000, 16(1): 1-18. |
| [16] |
Li H, Deng Y, Wu T, et al. Misexpression of miR482, miR1512, and miR1515 increases soybean nodulation[J]. Plant Physiology, 2010, 153(4): 1759-1770. DOI:10.1104/pp.110.156950 |
| [17] |
Shivaprasad PV, Chen HM, Patel K, et al. A microRNA superfamily regulates nucleotide binding site-leucine-rich repeats and other mRNAs[J]. The Plant Cell, 2012, 24(3): 859-874. DOI:10.1105/tpc.111.095380 |
| [18] |
Dominguez-Puigjaner E, Llop I, Vendrell M, et al. A cDNA clone highly expressed in ripe banana fruit shows homology to pectate lyases[J]. Plant Physiology, 1997, 114(3): 1071-1076. DOI:10.1104/pp.114.3.1071 |
| [19] |
Pua EC, Ong CK, Liu P, Liu JZ. Isolation and expression of two pectate lyase genes during fruit ripening of banana(Musa acuminata)[J]. Physiologia Plantarum, 2001, 113: 92-99. DOI:10.1034/j.1399-3054.2001.1130113.x |
| [20] |
Chourasia A, Sane VA, Nath P. Differential expression of pectate lyase during ethylene-induced postharvest softening of mango(Mangifera indica var. Dashehari)[J]. Physiologia Plantarum, 2006, 128: 546-555. DOI:10.1111/j.1399-3054.2006.00752.x |
| [21] |
Medina-Escobar N, Cardenas J, Moyano E, et al. Cloning, molecular characterization and expression pattern of a strawberry ripening-specific cDNA with sequence homology to pectate lyase from higher plants[J]. Plant Mole Biol, 1997, 34(6): 867-877. DOI:10.1023/A:1005847326319 |
| [22] |
Benitez-Burraco A, Blanco-Portales R, Redondo-Nevado J, et al. Cloning and characterization of two ripening-related strawberry(Fragaria x ananassa cv. Chandler)pectate lyase genes[J]. Journal of Experimental Botany, 2003, 54(383): 633-645. DOI:10.1093/jxb/erg065 |
| [23] |
Nunan KJ, Davies C, Robinson SP, et al. Expression patterns of cell wall-modifying enzymes during grape berry development[J]. Planta, 2001, 214(2): 257-264. DOI:10.1007/s004250100609 |





