2. 福建农林大学植物保护学院,福州 350002
2. College of Plant Protection, Fujian Agriculture and Forestry University, Fuzhou 350002
由稻瘟病菌(Magnaporthe oryzae)引起的稻瘟病常常爆发成灾,造成水稻减产高达50%,每年造成的粮食损失足以养活六千万人口,严重威胁着全球粮食安全[1]。稻瘟病菌作为研究寄主-病原真菌相互作用、丝状真菌侵染致病机理的重要模式生物,其侵染水稻的过程经历了分生孢子的萌发、芽管的产生、附着胞和侵入栓的形成、以及侵染菌丝的生长和致病等过程[2],涉及到了复杂的蛋白质翻译后修饰。随着研究的深入,蛋白质精氨酸甲基化越来越受到研究者的青睐[3],且关于催化其发挥作用的蛋白质精氨酸甲基转移酶(Protein arginine methyltransferases,PRMTs)的研究也越来越多。
PRMTs普遍存在于真核生物中,主要催化硫代腺苷甲硫氨酸(S-adenosylmethionine,SAM)上的甲基转移到靶蛋白精氨酸残基末的胍基上,进而调控着DNA修复、信号转导、细胞发育以及癌症发生等过程[3-5]。根据催化形式的不同,PRMTs可以分为4种类型:Type Ⅰ主要催化形成单甲基化(Monomethylated arginine,MMA)和非对称二甲基化(Asymmetric dimethylated arginine,ADMA); Type Ⅱ主要催化形成MMA和对称二甲基化(Symmetric dimethylated arginine,SDMA); Type Ⅲ只催化形成MMA,而Type Ⅳ则催化形成δ-MMA[6-7]。到目前为止,报道最多的是Type I和Type II的PRMTs[4, 6],而鉴定出的3个Type III PRMTs分别来自人类(human)[8]、酿酒酵母(Saccharomyces cerevisiae)[9]和布氏锥虫(Trypanosoma brucei)[10],Type IV PRMT仅在S. cerevisiae[11]中有报道。
PRMT1是Type I中最主要的精氨酸甲基转移酶,在真核生物中是高度保守的。哺乳动物中对PRMT1的研究最为透彻,研究发现人类中的PRMT1是正常胚胎、细胞周期进程、细胞移动和信号转导所必需的[4-5, 12-13]。植物中对PRMT1的研究相对较少,其中拟南芥(Arabidopsis thaliana)中初步鉴定出PRMT1的同源蛋白是AtPRMT1a和AtPRMT1b,这两个蛋白都定位于细胞质和细胞核中,在体外能催化RNA甲基转移酶AtFib2、纤维蛋白和组蛋白H4[14]。此外,AtPRMT1b(PRMT11)能与MBD7(methyl-DNA-binding protein 7)相互作用,但是AtPRMT1a和AtPRMT1b的单突变体或双突变体在长日照条件下都没有任何明显的表型[15]。水稻中PRMT1的同源蛋白OsPRMT1也定位于细胞质和细胞核中,且在水稻的各个组织中都能表达,其中成熟叶片中表达量最高,体外具有甲基转移酶的活性[16]。
在真菌中,酿酒酵母(S. cerevisiae)中的Hmt1p是首次报道的PRMT1同源蛋白,该蛋白定位于细胞核中,虽然并不是酵母生长所必需的,但其催化活性却是Npl3p和Cbp80(cap-binding protein 80)缺失突变体所必需的[17-19]。此外Hmt1p除甲基化组氨酸和非组氨酸蛋白外[17-18, 20-22],还可以甲基化小核糖体蛋白S2(Small ribosomal protein S2,Rps2)[23]。白色念珠菌(Candida albicans)中的CaHmt1与Hmt1p功能类似[24]。近几年丝状真菌中的PRMT1/Hmt1p同源蛋白的研究已取得了一些进展,例如禾谷镰刀菌(Fusarium graminearum)AMT1基因在菌丝生长、应对胁迫反应、产生毒素和侵染植物等过程中都发挥了重要的作用,且该蛋白调控着FgHrp1的甲基化和核运输过程[25]; 粗糙脉孢菌(Neurospora crassa)中AMT-1基因缺失后导致菌丝伸长速率降低,并增强了对麦角固醇生物合成抑制剂伏立康唑的敏感性[26]。与此不同的是,构巢曲霉(Aspergillus nidulans)中RmtA基因缺失后并没有影响该菌的营养生长、有性孢子和无性孢子的产生,但是会导致氧胁迫条件下生长明显减慢[27]。此外,黄曲霉(Aspergillus flavus)RmtA蛋白作为一个全局调控因子,既负调控着孢子的产生,又促进菌核的发育,正向调控着次生代谢过程,控制黄曲霉毒素和其他未知代谢物的产生[28]。
稻瘟病菌(M. oryzae)中含有4个PRMT基因(MoPRMT 1-4),生物信息学分析发现它们均含有保守的甲基转移酶结构域,在丝状真菌中是高度保守的[29],其中MoPRMT1与MoPRMT2、MoPRMT3、MoPRMT4的序列相似性分别为43%、44%和34%,表达谱分析结果表明MoPRMT1在侵染后24 h表达量达到最高峰,而其他阶段表达水平基本一致[29],然而该基因在稻瘟病菌侵染循环过程中发挥的具体作用尚未报道。根据真菌基因的命名规律,参照酵母中同源基因的名称,本研究将编码PRMT1蛋白的基因MGG_04584重新命名为MoHMT1(hnRNP arginine N-methyltransferase),并利用同源重组的基因敲除技术,分析其在稻瘟病菌生长发育和侵染致病过程中的作用,为进一步揭示稻瘟病菌MoHMT1的生物学功能及其参与的信号途径提供基础,也有利于进一步阐明稻瘟病菌的致病分子机理。
1 材料与方法 1.1 材料稻瘟病菌菌株Ku80由美国普渡大学许金荣博士惠赠; 大肠杆菌(Escherichia coli)DH5α、水稻品种CO39均为本实验室所保存; PCR试剂、限制性内切酶、T4 DNA连接酶等均购于大连宝生物公司; 其他常规生化试剂均为国产分析纯; 引物合成(表 1)及DNA测序分析均由华大基因完成。
根据同源重组的原理,以稻瘟病菌菌株Ku80基因组DNA为模板,通过MoHMT1的上下游引物(表 1)分别扩增其上下游片段A和B,然后分别与潮霉素磷酸转移酶基因H片段混合作为模板,利用SOE-PCR技术扩增获得AH和HB片段,进行原生质体的转化[30]。挑选具有潮霉素抗性的转化子,利用PCR的方法筛选基因敲除的阳性转化子。
1.2.2 Mohmt1敲除突变体菌落形态观察和生长速率测定将生长活力大致相同的野生型菌株Ku80、敲除突变体∆Mohmt1-14菌株、∆Mohmt1-19菌株和异位整合突变体Mohmt1-Ect菌株分别挑取大小相等的菌丝块,接种于淀粉酵母固体培养基(15 cm培养皿)的中央,26℃倒置培养,并于接种后的3 d、5 d、7 d、9 d测量菌落直径,第9天对菌落进行拍照。每个样品重复3皿,进行3次独立实验,对数据进行单因素方差分析。
1.2.3 Mohmt1敲除突变体产孢量的统计及形态观察将野生型及突变体相关菌株接种到米糠培养基上直至菌丝长满培养皿(9 cm),然后刮去表面的气生菌丝,26℃开盖光照产孢2 d,用无菌水冲洗收集所有孢子。将孢子悬浮液于三层滤纸过滤后,用血球计数板对孢子数量进行统计。每个样品重复3皿,进行3次独立实验,对数据进行单因素方差分析。
1.2.4 Mohmt1敲除突变体孢子的萌发实验将上述获得的野生型及突变体菌株的孢子悬浮液定容至浓度为1×104 - 2×104个/mL,吸取12 μL滴至Gelbond film疏水表面上,26℃保湿培养,并于培养1 h、2 h、4 h、8 h、12 h后统计孢子的萌发率。每个样品重复5滴,在观察时间段每滴观察3个视野,并进行3次独立实验,对数据进行单因素方差分析。
1.2.5 Mohmt1敲除突变体洋葱表皮侵染实验撕取约4 mm×4 mm大小的洋葱表皮第3层(从外到内)内表皮,置于每孔含有2 mL蒸馏水的24孔培养板孔洞内。调整野生型及突变体菌株的孢子悬浮液的浓度至4×104个/mL,吸取10 μL孢子悬浮液滴至洋葱表皮上,26℃培养,分别于培养24 h和48 h后观察侵染情况。
1.2.6 Mohmt1敲除突变体活体接种实验调整野生型及突变体菌株的孢子悬浮液浓度为1×105个/mL(含0.02% Tween),喷雾接种于水稻品种CO39(三叶期)上,于温室内黑暗保湿处理24 h后光照培养5 d,观察叶片的致病情况,其中叶瘟分级的具体分级标准参考Valent等[31]的方法。本实验独立重复3次。
2 结果 2.1 稻瘟病菌MoHmt1敲除突变体的筛选与鉴定为了明确MoHMT1基因的生物学功能,本研究根据同源重组的原理对MoHMT1基因进行敲除,利用潮霉素磷酸转移酶基因(HPH)重组替换MoHMT1基因完整的ORF片段,并将潮霉素作为筛选标记获得若干候选转化子。首先利用PCR技术对候选的转化子进行DNA水平验证,如图 1-A所示,凡是ORF(表 1 MoHMT1 OF/R)无阳性扩增(约632 bp)且UAH(表 1 MoHMT1 UAF/R)有阳性扩增(约1 450 bp)的转化子即为Mohmt1敲除突变体菌株,本研究将其命名为∆Mohmt1-14和∆Mohmt1-19,而ORF有阳性扩增而UAH无阳性扩增的转化子即为异位整合突变体,命名为Mohmt1-Ect。

|
| A:PCR验证Mohmt1突变体; B:RT-PCR验证Mohmt1突变体; Positive:野生型基因组DNA; Negative:阴性对照H2O 图 1 Mohmt1敲除突变体的筛选鉴定 |
其次,为了确认敲除突变体的可靠性,本研究进一步通过RT-PCR技术对获得的两个敲除突变体和异位整合突变体进行了验证。结果如图 1-B所示,在∆Mohmt1-14和∆Mohmt1-19两个敲除突变体中,几乎检测不到MoHMT1的表达; 而阳性对照和异位整合突变体中检测到MoHMT1的表达。
2.2 MoHMT1基因的缺失导致稻瘟病菌菌丝体生长明显减慢为了明确MoHMT1基因在稻瘟病菌营养生长过程中的作用,本研究对Mohmt1相关突变体进行了分析。结果如图 2所示,在淀粉酵母培养基上,与野生型菌株Ku80相比,∆Mohmt1-14和∆Mohmt1-19突变体菌落明显变小(图 2-A),生长速率明显减慢(图 2-B),且表面的气生菌丝量明显减少变薄(图 2-C),而Mohmt1-Ect则与野生型相比无明显变化。
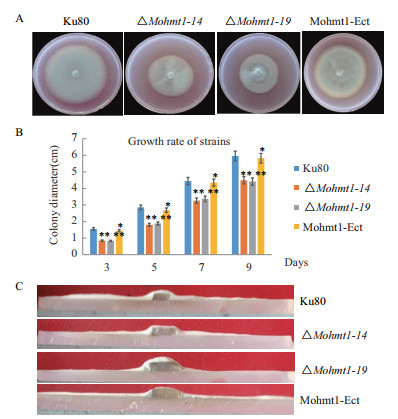
|
| A:Mohmt1敲除突变体的菌落形态; B:Mohmt1敲除突变体的生长速率,*,**分别表示在P < 0.05和P < 0.01水平上差异显著; C:Mohmt1敲除突变体菌落纵切面 图 2 Mohmt1敲除突变体的菌落形态和生长速率 |
本研究继而对Mohmt1相关突变体的产孢量和孢子萌发率进行了统计分析。结果如图 3-A所示,与野生型菌株Ku80相比,∆MoHmt1-14和∆MoHmt1-19的产孢量明显减少,仅为野生型的20%左右,暗示了∆MoHmt1-14和∆MoHmt1-19突变体在产孢量方面存在明显缺陷。然而,尽管产孢量显著下降,但是在4 h时孢子萌发率几乎达到100%,接近野生型的水平(图 3-B),说明突变体的孢子能进行正常地萌发。MoHmt1-Ect则在产孢量和孢子萌发率方面与野生型相比无明显变化。
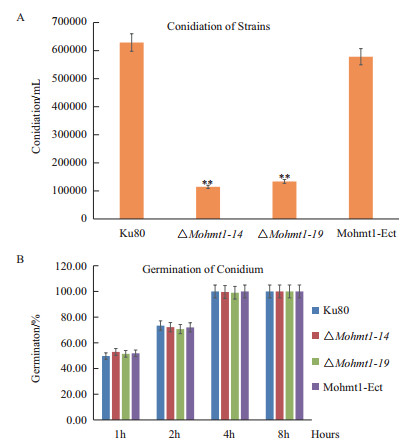
|
| A:Mohmt1敲除突变体的产孢量,**表示在P < 0.01水平上差异显; B:Mohmt1敲除突变体的萌发率 图 3 Mohmt1敲除突变体的产孢量和萌发率统计 |
为了明确MoHmt1缺失突变体在稻瘟病菌侵染过程中发挥的作用,本研究首先进行了洋葱表皮侵染实验。结果如图 4-A所示,在接种到洋葱表皮48 h后,与野生型和异位整合突变体相比,尽管Mohmt1缺失突变体产生的次生菌丝略微减少,但是能成功扩展侵入到邻近的细胞中,暗示了Mohmt1缺失突变体在疏水表面能够进行正常的侵染和扩展。

|
| A:Mohmt1敲除突变体洋葱表皮侵染(48 h),黑色箭头指示次生菌丝; B:Mohmt1敲除突变体水稻叶片侵染 图 4 Mohmt1敲除突变体的洋葱表皮和水稻叶片侵染 |
进一步将Mohmt1相关突变体的孢子悬浮液喷雾接种到水稻叶片上观察其致病性,结果如图 4-B所示,野生型和异位整合突变体形成的病斑几乎连接成片,且叶片枯黄,多为4-5级病斑,而Mohmt1缺失突变体仅仅形成零星的褐色针眼状病斑,病斑数目明显减少,多为1级病斑,偶尔见2级病斑,表明稻瘟病菌Mohmt1缺失突变体对水稻的致病性明显减弱。上述实验表明,MoHMT1基因缺失后尽管孢子能够正常侵入到水稻叶片并进行扩展,但是其对稻瘟病菌的致病性却明显减弱,暗示MoHMT1基因可能在稻瘟病菌的致病过程中发挥了重要的作用。
3 讨论精氨酸的甲基化是真核生物中普遍存在的蛋白质翻译后修饰,其作为转录表观调控因子在pre-mRNA剪接、DNA修复、mRNA转录、细胞信号等过程扮演了重要的角色[3],该修饰过程受到PRMTs的调控。研究表明该家族中最保守的成员PRMT1,在不同丝状真菌中的功能既有保守性又有特异性,例如,禾谷镰刀菌(F. graminearum)和黄曲霉(A. flavus)中AMT1/RmtA参与真菌的生长发育和侵染致病过程,并调控着毒素和其他未知代谢物的产生[25, 28]。而构巢曲霉(A. nidulans)中的RmtA却并不是真菌生长发育和侵染致病所必需的[27],这与酿酒酵母(S. cerevisiae)和白色念珠菌(C. albicans)的研究结果一致[17, 24]。另外,禾谷镰刀菌(F. graminearum)中RmtA基因缺失后对氧胁迫超级敏感[25],而黄曲霉(A. flavus)中RmtA基因缺失后则增强了对氧胁迫的耐受力[28]。这些研究结果为研究稻瘟病菌中MoHmt1(PRMT1的同源蛋白)的功能指明了方向。
鉴于此,本研究根据同源重组的原理获得Mohmt1缺失突变体,并对相关突变体进行表型分析,结果发现MoHMT1缺失后减慢了菌丝生长发育减慢、孢子量降低、对水稻叶片的致病性减弱,然而其具体的调控机制尚未明确,其功能的发挥是否与其催化活性密切相关有待于进一步地研究。
在酿酒酵母(S. cerevisiae)中,Hmt1p能够甲基化Hrp1/Nab2/Npl3等底物蛋白,从而促进其从细胞核中运输出来[18-19, 32],而禾谷镰刀菌(F. graminearum)中AMT1仅仅是核运输FgHrp1而不是FgNab2所必需的[25],暗示了甲基化作用位点在不同的真菌中存在差异。因此稻瘟病中MoHmt1的底物蛋白及其作用位点有哪些,也有待于今后进一步研究。
4 结论从稻瘟病菌(M. oryzae)中鉴定出PRMT1的同源蛋白MoHmt1,根据同源重组的原理获得Mohmt1缺失突变体,对其进行生物学功能分析发现,与野生型相比,Mohmt1缺失突变体的菌落明显变小,生长速率明显减慢,且表面的气生菌丝量明显减少变薄; MoHMT1的缺失也导致产孢量明显下降,仅为野生型的20%左右,但是这些孢子能够正常萌发形成附着胞,并能在洋葱表皮细胞中成功地定殖和扩展; 进一步对水稻叶片进行接种发现,Mohmt1缺失突变体的病斑数量明显少于野生型,病情指数下降,致病性减弱。综上所述,MoHmt1蛋白可能在稻瘟病菌的生长发育和致病过程中发挥了重要的作用。
| [1] |
Talbot NJ. On the trail of a cereal killer:Exploring the biology of Magnaporthe grisea[J]. Annual Review of Microbiology, 2003, 57: 177-202. DOI:10.1146/annurev.micro.57.030502.090957 |
| [2] |
Ebbole DJ. Magnaporthe as a model for understanding host-pathogen interactions[J]. Annual Review of Phytopathology, 2007, 45: 437-456. |
| [3] |
Blanc RS, Richard S. Arginine methylation:the coming of age[J]. Molecular Cell, 2017, 65(1): 8-24. DOI:10.1016/j.molcel.2016.11.003 |
| [4] |
Bedford MT, Clarke SG. Protein arginine methylation in mammals:who, what, and why[J]. Molecular Cell, 2009, 33(1): 1-13. DOI:10.1016/j.molcel.2008.12.013 |
| [5] |
Bedford MT, Richard S. Arginine methylation an emerging regulator of protein function[J]. Molecular Cell, 2005, 18(3): 263-272. DOI:10.1016/j.molcel.2005.04.003 |
| [6] |
Krause CD, Yang ZH, Kim YS, et al. Protein arginine methyltransf-erases:evolution and assessment of their pharmacological and therapeutic potential[J]. Pharmacology Therapeutics, 2007, 113(1): 50-87. |
| [7] |
Bachand F. Protein arginine methyltransferases:from unicellular eukaryotes to humans[J]. Eukaryotic Cell, 2007, 6(6): 889-898. DOI:10.1128/EC.00099-07 |
| [8] |
Zurita-Lopez CI, Sandberg T, Kelly R, et al. Human protein arginine methyltransferase 7(PRMT7)is a type Ⅲ enzyme forming omega-NG-monomethylated arginine residues[J]. The J Biol Chem, 2012, 287(11): 7859-7870. DOI:10.1074/jbc.M111.336271 |
| [9] |
Young BD, Weiss DI, Zurita-Lopez CI, et al. Identification of methylated proteins in the yeast small ribosomal subunit:a role for SPOUT methyltransferases in protein arginine methylation[J]. Biochemistry, 2012, 51(25): 5091-5104. DOI:10.1021/bi300186g |
| [10] |
Fisk JC, Sayegh J, Zurita-Lopez CI, et al. A type Ⅲ protein arginine methyltransferase from the protozoan parasite Trypanosoma brucei[J]. The J Biol Chem, 2009, 284(17): 11590-11600. |
| [11] |
Niewmierzycka A, Clarke S. S-Adenosylmethionine-dependent methylation in Saccharomyces cerevisiae. Identification of a novel protein arginine methyltransferase[J]. The J Biol Chem, 1999, 274(2): 814-824. DOI:10.1074/jbc.274.2.814 |
| [12] |
Ganesh L, Yoshimoto T, Moorthy NC, et al. Protein methyltransferase 2 inhibits NF-kappaB function and promotes apoptosis[J]. Molecular and Cellular Biology, 2006, 26(10): 3864-3874. DOI:10.1128/MCB.26.10.3864-3874.2006 |
| [13] |
Yang Y, Bedford MT. Protein arginine methyltransferases and cancer[J]. Nature Reviews. Cancer, 2013, 13(1): 37-50. DOI:10.1038/nrc3409 |
| [14] |
Yan D, Zhang Y, Niu L, et al. Identification and characterization of two closely related histone H4 arginine 3 methyltransferases in Arabidopsis thaliana[J]. The Biochemical Journal, 2007, 408(1): 113-121. |
| [15] |
Scebba F, De Bastiani M, Bernacchia G, et al. PRMT11:a new Arabidopsis MBD7 protein partner with arginine methyltransferase activity[J]. Plant J, 2007, 52(2): 210-222. DOI:10.1111/j.1365-313X.2007.03238.x |
| [16] |
Ahmad A, Dong Y, Cao X. Characterization of the PRMT gene family in rice reveals conservation of arginine methylation[J]. PLoS One, 2011, 6(8): e22664. DOI:10.1371/journal.pone.0022664 |
| [17] |
Henry MF, Silver PA. A novel methyltransferase(Hmt1p)modifies poly(A)+-RNA-binding proteins[J]. Molecular and Cellular Biology, 1996, 16(7): 3668-3678. DOI:10.1128/MCB.16.7.3668 |
| [18] |
Shen EC, Henry MF, Weiss VH, et al. Arginine methylation facilitates the nuclear export of hnRNP proteins[J]. Genes & Development, 1998, 12(5): 679-691. |
| [19] |
McBride AE, Weiss VH, Kim HK, et al. Analysis of the yeast arginine methyltransferase Hmt1p/Rmt1p and its in vivo function. Cofactor binding and substrate interactions[J]. The J Biol Chem, 2000, 275(5): 3128-3136. |
| [20] |
Kuo MH, Xu XJ, Bolck HA, et al. Functional connection between histone acetyltransferase Gcn5p and methyltransferase Hmt1p[J]. Biochimica et Biophysica Acta, 2009, 1789(5): 395-402. |
| [21] |
Low JK, Wilkins MR. Protein arginine methylation in Saccharomyces cerevisiae[J]. The FEBS Journal, 2012, 279(24): 4423-4443. DOI:10.1111/febs.12039 |
| [22] |
Yagoub D, Hart-Smith G, Moecking J, et al. Yeast proteins Gar1p, Nop1p, Npl3p, Nsr1p, and Rps2p are natively methylated and are substrates of the arginine methyltransferase Hmt1p[J]. Proteomics, 2015, 15(18): 3209-3218. DOI:10.1002/pmic.201500075 |
| [23] |
Lipson RS, Webb KJ, Clarke SG, et al. Rmt1 catalyzes zinc-finger independent arginine methylation of ribosomal protein Rps2 in Saccharomyces cerevisiae[J]. Biochemical and Biophysical Research Communications, 2010, 391(4): 1658-1662. DOI:10.1016/j.bbrc.2009.12.112 |
| [24] |
McBride AE, Zurita-Lopez C, Regis A, et al. Protein arginine methylation in Candida albicans:role in nuclear transport[J]. Eukaryotic Cell, 2007, 6(7): 1119-1129. DOI:10.1128/EC.00074-07 |
| [25] |
Wang G, Wang C, Hou R, et al. The AMT1 arginine methyltransferase gene is important for plant infection and normal hyphal growth in Fusarium graminearum[J]. PLoS One, 2012, 7(5): e38324. DOI:10.1371/journal.pone.0038324 |
| [26] |
Feldman D, Ziv C, Gorovits R, et al. Neurospora crassa protein arginine methyl transferases are involved in growth and development and interact with the NDR kinase COT1[J]. PLoS One, 2013, 8(11): e80756. DOI:10.1371/journal.pone.0080756 |
| [27] |
Bauer I, Graessle S, Loidl P, et al. Novel insights into the functional role of three protein arginine methyltransferases in Aspergillus nidulans[J]. Fungal Genetics and Biology, 2010, 47(6): 551-561. DOI:10.1016/j.fgb.2010.03.006 |
| [28] |
Satterlee T, Cary JW, Calvo AM, et al. RmtA, a putative arginine methyltransferase, regulates secondary metabolism and development in Aspergillus flavus[J]. PLoS One, 2016, 11(5): e0155575. DOI:10.1371/journal.pone.0155575 |
| [29] |
吴立叶, 王国梁, 刘文德. 稻瘟病菌不同生长期蛋白精氨酸甲基转移酶基因的表达分析[J]. 生物技术通报, 2017, 33(6): 104-111. |
| [30] |
Villalba F, Collemare J, Landraud P, et al. Improved gene targeting in Magnaporthe grisea by inactivation of MgKU80 required for non-homologous end joining[J]. Fungal Genetics and Biology, 2008, 45(1): 68-75. DOI:10.1016/j.fgb.2007.06.006 |
| [31] |
Valent B, Farrall L, Chumley FG. Magnaporthe grisea genes for pathogenicity and virulence identified through a series of backcrosses[J]. Genetics, 1991, 127(1): 87-101. |
| [32] |
Green DM, Marfatia KA, Crafton EB, et al. Nab2p is required for poly(A)RNA export in Saccharomyces cerevisiae and is regulated by arginine methylation via Hmt1p[J]. J Biol Chem, 2002, 277(10): 7752-7760. DOI:10.1074/jbc.M110053200 |





