定点突变是研究基因功能与蛋白质结构的常用方法,目前主要使用PCR介导的突变引入技术。PCR即聚合酶链式反应,是一种在体外迅速扩增特定目标DNA片段的分子生物学技术[1-2]。在此基础上发展起来的重叠延伸PCR(Overlap extension PCR,OE-PCR)是基因定点突变的主要方法[3-5]。整个OE-PCR需要4条引物,两轮共3次PCR反应,其繁琐的流程一直为科研工作者所诟病[6-8]。近年来陆续发展出新的定点突变解决方案,主要有滚环扩增法与大引物PCR法[9],据称能简化流程,提高突变效率。也有研究者认为这两种方法各有弊端,如滚环扩增法容易引入新的非预期突变[10],而大引物PCR法始终无法彻底避免对原始基因的扩增[11]。因此,大多数科研工作者仍倾向于使用OE-PCR法制作定点突变,并不断提出优化方案以期提升工作效率。如有人提出平行模板法可以避免原始基因的扩增,于是可在同一个小管内完成对基因突变体的扩增[12]。
结合实际工作经验,我们发现OE-PCR定点突变时效率不稳定,表现为转化之后的克隆形成数较少,克隆阳性率偏低。另一个较为普遍的现象是当把基因从一个载体中酶切出来直接连入另一个载体时,转化形成的克隆数较多,检测阳性率较高(几乎100%)。而克隆一个新基因或制作一个定点突变时,转化形成的克隆数较少,检测阳性率也往往不高。适当增加酶浓度,或延长酶切时间有助于增加克隆形成数和阳性率,但始终无法达到前者的效果。两者的主要区别在于前者酶切PCR产物时切点往往位于DNA末端,而后者酶切质粒时切点位于DNA中间。大多数限制性内切酶都不能作用于DNA顶端序列,因此在设计引物时会在酶切位点外侧增加1-3个“保护碱基”,尽管如此其酶切效率往往只能达到最大值的20%-50%.据此推测靠近PCR产物末端的低效限制性酶切是制约基因克隆和定点突变效率的主要原因。据此,我们提出旁侧引物法以期改善定点突变效率。即将常规OE-PCR上、下游引物匹配位点由基因两端外延50-100 bp,适当增加第二轮PCR产物的长度,使目标基因两侧的酶切位点远离DNA末端,从而提高随后的酶切效率。
周期蛋白依赖性激酶4(Cyclin-dependent kinase 4,CDK4)是细胞周期的重要调节基因[13-14],它与cyclin D1以复合物的形式共同调节细胞周期G1/S转换[15-16]。CDK4_D158N是其重要的激酶活性缺失突变体,常用于CDK4功能研究[17]。以制作CDK4_D158N为例,采用常规引物与旁侧引物法并行OE-PCR操作,发现旁侧引物法极大地提高了基因定点突变的克隆形成数与突变成功率。
1 材料与方法 1.1 材料 1.1.1 菌种与质粒感受态大肠杆菌Trans5α Chemically Competent Cell(CD201-01)购自北京全式金生物技术有限公司;质粒pEGFP_C2(Catalog #6083-1,Clontech),pcDNA3.1(Catalog no. V800-20,Invitrogen)及CDK4基因相关质粒由北京大学生命科学学院张传茂教授馈赠。
1.1.2 试剂2×PCR Reagent(KT207)购自天根生化科技(北京)有限公司;PyrobestTM DNA Polymerase(R005A),DNA Ligation Kit Ver.2.1(6002)购自Takara宝生物工程(大连)有限公司;内切酶EcoR I(R0101V)、Sal I(R0138V)购自NEB纽英伦生物技术北京有限公司;DNA快速回收/纯化试剂盒(NEP013-2)购自北京鼎国昌盛生物技术有限责任公司;质粒小量抽提试剂盒(D0003)购自碧云天生物技术。
1.2 方法 1.2.1 引物设计与合成旁侧引物序列参考Invitrogen中国测序通用引物序列(http://www.thermofisher.com/content/dam/LifeTech/Documents/PDFs/china/common-vector-primer-list.pdf),其他引物采用Primer Premier 5. 0软件设计,相关引物序列见表 1。引物由北京奥科鼎盛生物科技有限公司合成,经PAGE纯化。
扩增目标基因采用高保真PyrobestTM DNA Polymerase,第一轮PCR方案:2 μL 10×buffer,1.2 μL dNTP,0.2 μL primer 1,0.2 μL primer 2,0.2 μL酶,0.1 μL(10 ng)质粒,补水至20 μL;第二轮PCR方案:2 μL 10×buffer,2 μL dNTP,0.2 μL primer 1,0.2 μL primer 2,0.2 μL酶,0.5 μL上游片段回收物,0.5 μL下游片段回收物,补水至20 μL;运行程序皆为:94℃ 5 min;94℃ 30 s,54℃ 30 s,72℃ 1 min,30循环;72℃ 10 min。PCR产物经1%琼脂糖凝胶电泳检测后回收至20 μL ddH2O中。用PCR法检测阳性菌落时选用快速2×PCR Reagent,方案:5 μL 2×Taq,0.1 μL primer 1,0.1 μL primer 2,0.8 μL细菌培养物,补水至10 μL;扩增条件:94℃ 5 min;94℃ 30 s,54℃ 30 s,72℃ 45 s,30循环;72℃ 5 min。
1.2.3 基因克隆与序列分析取5 μL含点突变的目的基因回收物,加2 μL 10×buffer,0.5 μL EcoRI,0.5 μL,Sal I,补水至20 μL,37℃孵育一定时间;取pEGFP_C2质粒1 μg,采取上述同样方式酶切1 h。酶切产物经电泳检测后回收至20 μL ddH2O中。用DNA Ligation Kit Ver.2.1连接:载体0.5 μL,DNA片段4.5 μL,solution I 5 μL混合,16℃孵育30 min。连接产物直接转化感受态大肠杆菌,用卡那霉素固体培养基筛选,结合PCR检测阳性菌落。对相应PCR产物回收或阳性菌落小量提取质粒后进行序列测定,序列测定采用ABI 3730系列测序仪,由北京奥科鼎盛生物科技有限公司完成。
2 结果 2.1 平行模板法无法避免原始基因的扩增平行模板法即选用两个含同一目标基因的不同质粒作为模板,在这两个质粒上分别选一段序列作为引物[18],支持者认为“当两种模板和两条外侧引物同时存在于一个反应管时,可以完全避免对野生型目的基因全长的扩增[12],只有在突变引物存在的情况下才能扩增出产物,因此目标产物一定含有预期定点突变。为检测上述理论,选用pEGFP_C2-CDK4与pcDNA3.1-CDK4两种质粒作为平行模板,选用pcDNA3.1多克隆位点(Multiple cloning site,MCS)上游86 bp处的T7 promoter(pEGFP_C2中无对应序列)作为上游引物,选用pEGFP_C2 MCS下游61 bp处的pEGFP_C3'(pcDNA3.1中无对应序列)作为下游引物。将两种质粒单独作为模板时,都无法扩增出目标产物;将两种质粒混合物作为模板时,很好地扩增出目标产物(图 1-A)。经序列测定,该PCR产物中间为CDK4基因,两端多余序列分别源自pcDNA3.1与pEGFP_C2(图 1-B)。该DNA片段长1 057 bp,恰好为86 bp,909 bp(CDK4基因除终止密码TGA之外的长度),61 bp之和(图 1-C)。可见平行模板法无法避免对原始野生型基因的扩增。
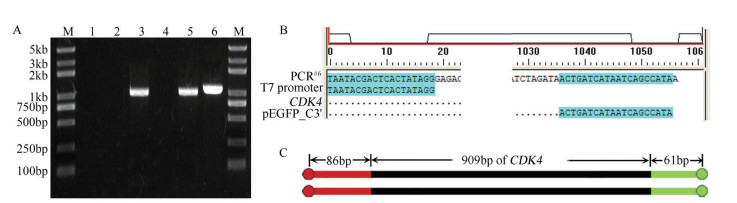
|
| A:1以ddH2O为模板作为负对照;2、3以pcDNA3.1-CDK4为模板;4、5以pEGFP_C2-CDK4为模板;6以pcDNA3.1-CDK4+pEGFP_C2-CDK4混合物为模板;除3以CDK4_A为下游引物,5以CDK4_S为上游引物外,其余上下游引物均为T7 promoter/pEGFP_C3',其他成分一致;M:DNA marker。B:6号管扩增产物序列比对图;C:6号管扩增产物构成示意图 图 1 平行模板法仍能扩增原始基因 |
为验证旁侧引物法的优点,以在pEGFP_C2-CDK4基础上制作其活性缺失突变体CDK4_D158N为例,采用常规末端引物与旁侧引物进行并行OE-PCR操作。常规末端引物分别为匹配CDK4基因两端序列的CDK4_S(含EcoR I位点与3个保护碱基)与CDK4_A(含Sal I位点与2个保护碱基);旁侧引物pEGFP_C5'位于Eco RI位点上游75 bp,旁侧引物pEGFP_C3'位于Sal I位点下游61 bp(图 2-A)。

|
| A:相关引物匹配位置示意图;B:第一轮PCR结果;a以CDK4_S/ D158N_A为引物,b以D158N_S/CDK4_A为引物,c以pEGFP_C5'/ D158N_A为引物,d以D158N_S/pEGFP_C3'为引物,以等量ddH2O代替引物作为负对照;五者皆以pEGFP_C2-CDK4为模板,其他成分亦一致。C:第二轮PCR结果:1、2分别以片段a、b为模板,3以片段a、b混合物为模板,4以质粒pEGFP_C2-CDK4为模板作为正对照;1-4皆以CDK4_S/CDK4_A为引物。5、6分别以片段c、d为模板,7以片段c,d混合物为模板,8以质粒pEGFP_C2-CDK4为模板作为正对照;5-8皆以pEGFP_C5'/ pEGFP_C3'为引物,其他成分一致。M:DNA marker 图 2 OE-PCR扩增含点突变的目标基因 |
第一轮PCR产物经电泳检测,得到各自所需的4个片段(图 2-B)。将a+b组合,c+d组合进行第二轮PCR,两者皆得到相应的目标产物(图 2-C)。且电泳结果显示采用旁侧引物时各PCR产物都略大于常规末端引物下的产物,与预期相符。
2.3 突变体的克隆与鉴定为检测不用酶切时间对常规末端引物法的影响,对上述3号产物(图 2-C)的双酶切设置1 h与4 h两个时间组,以对比其效率差异。电泳结果显示不同酶切作用时间下产物大小基本一致,看不出三者的明显区别(图 3-A)。由于此时识别位点位于DNA末端,双酶切只会使其缩短11 bp,故难以通过电泳结果判断酶切作用是否充分。
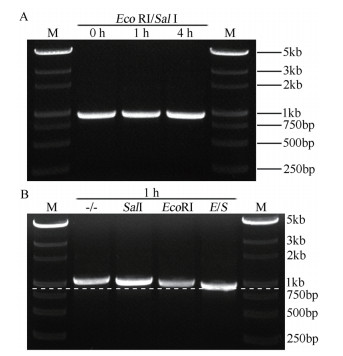
|
| A:常规末端引物PCR产物经Eco R I/Sal I酶切不用时间下的电泳检测结果;B:旁侧引物PCR产物经EcoR I与Sal I以不同组合模式酶切1 h下的电泳检测结果。虚线用以辅助区分不同酶切产物的大小差异。M:DNA marker 图 3 第二轮PCR及酶切结果 |
为检测旁侧引物法下切除两端多余序列后对终产物大小的影响,对7号产物(图 2-C)用Eco RI与Sal I以不同的组合方式进行酶切。考虑到此时识别位点位于DNA中间,酶切效率高,故仅设置一个短时间组。电泳检测发现4种酶切组合下的产物大小不同,其中双酶切产物最小,且条带单一(图 3-B),说明该产物已被充分酶切。
将上述双酶切产物连入载体后经转化、培养以筛选目标突变体。常规末端引物下酶切1 h组只长出3个菌落,经PCR检测,其中仅一个阳性克隆(图 4-A),阳性率仅33%。常规末端引物下酶切4 h组长出数十个菌落,任意挑取10个菌落进行PCR检测,得到7个阳性克隆(图 4-B),阳性率达70%。旁侧引物双酶切1 h组长出数百个菌落,任意挑取10个菌落进行PCR检测,发现全部都是阳性克隆(图 4-C),阳性率100%;对这10个阳性克隆提取质粒进行序列测定,结果显示全部都是pEGFP_C2-CDK4_D158N,即突变成功率为100%。
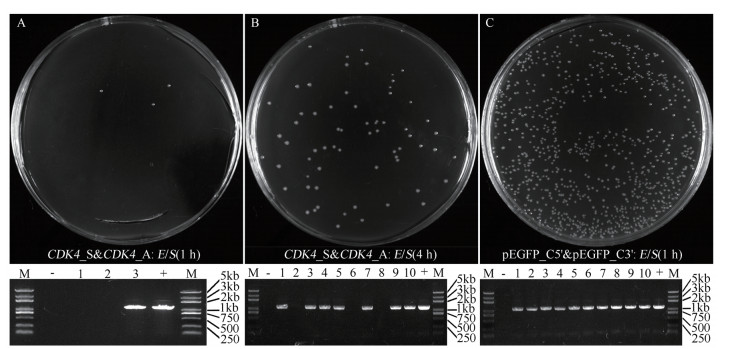
|
| A-C:上图显示各组过夜平板培养基上的成型菌落,下图显示对应菌落的PCR检测结果。检测所用引物皆为pEGFP_C5'/ pEGFP_C3',以ddH2O为模板作为负对照,以质粒pEGFP_C2-CDK4为模板作为正对照,1-10为菌落编号。M:DNA marker;目标产物1 073 bp 图 4 不同方案下的平板筛选及菌落检 |
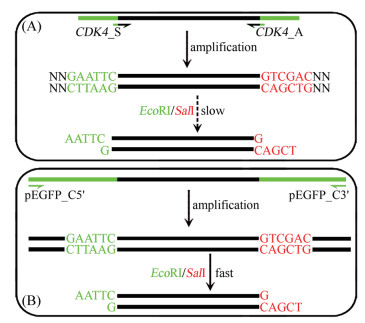
由于OE-PCR定点突变程序繁琐,很多科研工作者都试图提出优化方案,平行模板法即其中之一。然而,我们发现平行模板法并不能避免对原始野生型基因的扩增。结合具体实例,并不难分析其原理。由T7 promoter与pEGFP_C3'引物各自完全延伸的单链DNA虽然在CDK4区域互补,但是没有配对的3'-OH末端,无法继续延伸。而PCR反应中往往会有部分未完全延伸的产物,如果两者恰好在各自的CDK4序列内部中止延伸,那么这两条单链DNA不只互补,且各有一个配对后的3'-OH末端。因此可以互为模板继续延伸,形成一条“杂交”DNA双链。其上游序列源自pcDNA3.1,中部为CDK4序列,下游序列源自pEGFP_C2。该“杂交”DNA双链可以作为后续循环的模板使目标基因CDK4得以扩增(图 5)。因此,试图以平行模板法在一个小管内完成OE-PCR所有扩增反应的构思是不成立的。
Ⅱ型限制性内切酶识别DNA的特定序列并切开双链DNA,若酶切位点靠近DNA末端,则酶切效率受到不同程度的影响[19-20]。在设计引物时,往往在酶切位点外侧添加1-3个“保护碱基”以保证PCR产物的酶切效率。尽管如此,其酶切效率有时也只能达到最大值的20%-50%(图 6-A)。低效酶切也成为基因克隆尤其是PCR定点突变的重要制约因素。有两种方法可提高此时的酶切效率,一是增加引物中“保护碱基”数目,但相应地会增加实验费用,且非配对碱基数的增加还会降低PCR成功率;二是增加酶切反应的酶量或(和)延长酶切作用时间,却会增加内切酶出现星号活性的风险。选用旁侧引物可以很好地解决上述问题,且可以避免诸多不利因素。旁侧引物位于酶切位点外侧50 bp-100 bp,此时对PCR产物酶切近似于甚至优于从质粒中切取基因片段(质粒的超螺旋结构会影响酶切效率),极大地提高酶切反应的效率(图 6-B),由此缩短反应时间,增加克隆形成数及阳性率。
|
| A:采用末端引物CDK4_S/CDK4_A导致酶切位点靠近DNA末端,随后的酶切效率较低;B:采用旁侧引物pEGFP_C5'/ pEGFP_C3'使酶切位点位于DNA片段中间,随后的酶切效率较高 图 6 旁侧引物法提高PCR产物酶切效率原理示意图 |
除此之外,旁侧引物法还有以下两个优点,一是双酶切产物与非酶切或单酶切产物大小有一定差异,可以通过电泳结果直接判断酶切是否充分(图 3-B)。另外,当突变位点靠近基因两端时,采用常规末端引物会使第一轮PCR产物之一偏小,不便于检测及回收。此时采用旁侧引物可以适当增加DNA片段长度,便于后续操作。旁侧引物源于质粒载体上的一段序列,只要插入该载体的基因,都可以之制作定点突变,不会过多增加实验成本。
平行模板法虽无法避免原始基因的扩增,但是其扩增效率明显低于同一个质粒上、下游引物对目标基因的扩增。因此,我们认为仍可利用两个平行质粒分别作为第一轮PCR上下游片段的扩增模板,以减少定点突变过程中原始质粒对第二轮PCR的“污染”。如果在第一轮PCR时选用两种不同的平行质粒,除非两者同时污染各自的PCR产物,否则第二轮PCR不会扩增出原始基因,这样就极大地减少了原始质粒对后续操作的污染。
4 结论本研究论证了平行模板法简化OE-PCR定点突变流程的思路不能成立。我们认为制约OE-PCR定点突变效率的核心因素是靠近DNA末端识别位点的低效限制性酶切。采用旁侧引物法可提高PCR产物的酶切效率,且便于通过电泳监测酶切效果,既节省工作时间,又能大幅提高OE-PCR定点突变的成功率。
| [1] |
Saiki RK, Scharf S, Faloona F, et al. Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia[J]. Science, 1985, 230(4732): 1350-1354. DOI:10.1126/science.2999980 |
| [2] |
Mullis K, Faloona F, Scharf S, et al. Specific enzymatic amplification of DNA in vitro:the polymerase chain reaction[J]. Cold Spring Harbor Symposia on Quantitative Biology, 1986, 51(Pt 1): 263-273. |
| [3] |
Kammann M, Laufs J, Schell J, et al. Rapid insertional mutagenesis of DNA by polymerase chain reaction(PCR)[J]. Nucleic Acids Res, 1989, 17(13): 5404. DOI:10.1093/nar/17.13.5404 |
| [4] |
Lu Y, Xiao S, Yuan M, et al. Using overlap-extension PCR technique to fusing genes for constructing recombinant plasmids[J]. Journal of Basic Microbiology, 2018, 58(3): 273-276. DOI:10.1002/jobm.201700455 |
| [5] |
Hussain H, Chong NF. Combined overlap extension PCR method for improved site directed mutagenesis[J]. BioMed Research International, 2016, 8041532. |
| [6] |
雒丽娜, 王盛, 王玉炯. 利用重叠延伸PCR技术进行定点突变研究[J]. 安徽农业科学, 2012(10): 5779-5781. DOI:10.3969/j.issn.0517-6611.2012.10.022 |
| [7] |
徐书景, 张跃灵, 张妍, 等. 改进重叠延伸PCR技术构建定点双突变[J]. 中国生物工程杂志, 2010(10): 49-54. |
| [8] |
谢振华, 史小军, 蔡国平. 在单个PCR管内快捷完成定点突变[J]. 中国生物化学与分子生物学报, 2006(8): 681-684. DOI:10.3969/j.issn.1007-7626.2006.08.014 |
| [9] |
赵松子, 沈向群. 用滚环扩增与大引物PCR法高效构建定点突变序列[J]. 江西农业学报, 2009(8): 7-8. DOI:10.3969/j.issn.1001-8581.2009.08.003 |
| [10] |
张碧乾, 邓会群, 宫彩霞, 等. 大引物PCR定点突变方法的改进[J]. 湖北大学学报:自然科学版, 2009(1): 79-81. |
| [11] |
谢振华, 史小军. 快捷的定点突变效率近100%的大引物PCR方法(英文)[J]. 生物化学与生物物理进展, 2009, 36(11): 1490-1494. |
| [12] |
莫秋华, 徐湘民, 钟雄霖, 等. 一种改进的大引物PCR定点诱变方法[J]. 中华医学遗传学杂志, 2002(1): 72-75. DOI:10.3760/j.issn:1003-9406.2002.01.020 |
| [13] |
Gao A, Sun T, Ma G, et al. LEM4 confers tamoxifen resistance to breast cancer cells by activating cyclin D-CDK4/6-Rb and ERalpha pathway[J]. Nature Communications, 2018, 9(1): 4180. DOI:10.1038/s41467-018-06309-8 |
| [14] |
Li Z, Razavi P, Li Q, et al. Loss of the FAT1 Tumor suppressor promotes resistance to CDK4/6 inhibitors via the Hippo pathway[J]. Cancer Cell, 2018, 34(6): 893-905. DOI:10.1016/j.ccell.2018.11.006 |
| [15] |
Zhong S, Zhu J, Li Y, et al. Butylene fipronil induces apoptosis in PC12 murine nervous cells via activation of p16-CDK4/6-cyclin D1 and mitochondrial apoptotic pathway[J]. Journal of Biochemical and Molecular Toxicology, 2018. |
| [16] |
Lee Y, Dominy JE, Choi YJ, et al. Cyclin D1-CDK4 controls glucose metabolism independently of cell cycle progression[J]. Nature, 2014, 510(7506): 547-551. DOI:10.1038/nature13267 |
| [17] |
Ruas M, Gregory F, Jones R, et al. CDK4 and CDK6 delay senescence by kinase-dependent and p16INK4a-independent mechanisms[J]. Molecular and Cellular Biology, 2007, 27(12): 4273-4282. DOI:10.1128/MCB.02286-06 |
| [18] |
Upender M, Raj L, Weir M. Megaprimer method for in vitro mutagenesis using parallel templates[J]. Biotechniques, 1995, 18(1): 29-30, 32. |
| [19] |
Maschmann A, Kounovsky-Shafer KL. Determination of restriction enzyme activity when cutting DNA labeled with the TOTO dye family[J]. Nucleosides Nucleotides Nucleic Acids, 2017, 36(6): 406-417. DOI:10.1080/15257770.2017.1300665 |
| [20] |
Chaudhary VK, Shrivastava N, Verma V, et al. Rapid restriction enzyme-free cloning of PCR products:a high-throughput method applicable for library construction[J]. PLoS One, 2014, 9(10): e111538. DOI:10.1371/journal.pone.0111538 |






