洋葱(Allium cepa L.)是百合科葱属植物,是世界上重要的蔬菜之一,年产量位居第二位,仅次于番茄。在中国,洋葱是主要的出口创汇蔬菜,由于其重要的营养和保健价值,越来越受到人们的欢迎,市场需求不断提高。洋葱起源于中亚,种质资源严重匮乏。随着对其品种特性的要求越来越高,改良洋葱品种的综合性状显得尤为重要。
洋葱杂交种具有很强的杂种优势,高产、整齐度高、商品性好[1]。为了提高品种的杂种优势,需要高度纯合亲本自交系。由于洋葱是典型的异花授粉作物,具有自交衰退现象,而且又是两年生植物,通过常规育种手段很难实现高度纯合这个目标[2]。洋葱自交系选育通常是通过多代自交分离纯合而成,需要6-10年,不仅周期长、费工、费时,而且育种效率较低。单倍体加倍获得的双单倍体具有纯合速度快、用于选择的群体小和隐性基因也能表达的优点,是拓展种质资源的高效方案,并能够为选育洋葱杂交种奠定材料基础[3-4]。与传统选育洋葱自交系相比,双单倍体系的选育时间短,仅需两年就能获得完全纯合稳定的自交系[5]。由于双单倍体群体中的每个个体都是完全纯合同质的,可作为重要性状的遗传分析、分子标记及数量性状研究的理想材料。
分子标记在遗传多样分析、重要性状的辅助选择、基因定位等方面具有重要的应用意义,直接反映了个体间DNA的遗传多态性。前人利用RFLP、SSR等分子标记在黄瓜、甜瓜、马铃薯等多种蔬菜作物上鉴定单倍体诱导培养再生植株的纯合性和杂合性[6-11]。在洋葱中,由于开发的标记较少,至今相关研究开展有限,主要以RAPD标记对单倍体诱导培养的再生植株与其供体亲本进行比较分析,未见有利用稳定性更高的共显性标记进行纯合性鉴定的报道[12-13]。
本研究通过离体雌核发育途径培养诱导洋葱未受精花蕾产生单倍体/双单倍体再生植株,利用共显性的雄性不育基因分子标记对再生植株进行检测,从而鉴定再生植株的来源与纯合性,以期为有效利用洋葱单倍体奠定基础,为我国洋葱种质资源创新、杂交种选育、基因工程等相关研究提供重要材料支撑。
1 材料与方法 1.1 材料选择引自日本泷井种苗株式会社(Takii Seed)的3个中日照类型洋葱杂交种“阿盾”(ATON)、“地球”(EARTH)和“大宝”(TABAO),种植于山东省农业科学院蔬菜花卉研究所试验基地。鳞茎收获后在适宜的条件下贮存,当年10月底栽种鳞茎。开花期选择直径为2.5-3.5 mm的未开放花蕾接种在B5培养基(含2 mg/L 2,4-D、2 mg/L 6-BA和100 g/L蔗糖)上进行诱导培养,获得再生植株[14]。
1.2 方法 1.2.1 再生植株倍性鉴定方法利用流式细胞仪对再生植株进行倍性鉴定。取10 mg新鲜叶片,加入0.5 mL的Partec HR-A溶液(分离细胞核的柠檬酸缓冲液)研磨。将样品通过30 μmol/L PartecCelltricsTM微孔膜过滤到测试管中,加0.5 mL PartecHR-B溶液(DAPI溶液)于样品中,利用德国Partec PA流式细胞仪测定植株细胞DNA的相对含量。并绘出DNA曲线图。将已知倍性的二倍体洋葱的相对荧光强度为参照标准确定供试样品的倍性水平。
1.2.2 DNA提取选取洋葱再生植株新鲜叶片,迅速放入液氮中冷冻,于-80℃保存备用。使用天根生化科技(北京)有限公司生产的快捷型植物基因组DNA提取系统(DP321)提取洋葱的基因组DNA,0.8%琼脂糖凝胶电泳及分光光度计检测DNA的浓度和质量。
1.2.3 再生植株的分子标记检测对获得的再生植株利用洋葱细胞核育性基因的分子标记DNF-566和RNS-357[15]对源于杂交种“阿盾”的再生植株进行纯合性检测。利用多重PCR标记ACSKP1[16]对源于杂交种“大宝”、“地球”的再生植株进行纯合性检测,标记所用引物(表 1)由上海生工生物工程股份有限公司合成。
PCR扩增体系总体积为25 μL,包括rTaq聚合酶(5 U/μL)0.2 μL、10×PCR buffer 2.5 μL、dNTP mix(2.5 mmol/L)2 μL、MgCl2(25 mmol/L)1.5 μL、基因组DNA模板2 μL、0.5 μmol/L上下游引物各1 μL、50 ng/L模板DNA 1 μL,加ddH2O补至25 μL。PCR产物经1.0%琼脂糖凝胶电泳检测。根据再生植株和供体二倍体原始材料在Ms基因位点扩增片段的差异进行纯合性分析。
2 结果 2.1 未受精花蕾在诱导培养基中的发育与植株再生将直径为2.5-3.5 mm的未开放洋葱花蕾接种在添加2 mg/L 2,4-D、2 mg/L 6-BA和100 g/L蔗糖的B5培养基上进行诱导培养,2-3 d后花蕾在培养皿中可正常开放,5-6 d后花药败育,子房开始膨大。50 d后,有体细胞胚从子房中长出,并且具有完整的植株结构。3-4个月后胚达到最多,5个月以后几乎不会出现。将所获得的雌核发育胚转移至不含激素的含有30 g/L蔗糖的B5培养基中,经过5 d左右发育成正常植株(图 1)。

|
| A:接种在培养基上的洋葱花蕾;B:花蕾在培养基上开放;C:雌核发育胚出现;D:再生植株 图 1 未受精洋葱花蕾在诱导培养基中的发育过程 |
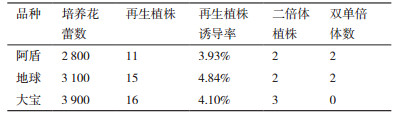
以洋葱正常二倍体叶片的细胞悬浮液为对照,将待测试再生植株叶片的细胞悬浮液在流式细胞仪上进行检测,结果发现二倍体植株在相对荧光强度140处出现分离峰(图 2-A),据此判定分离峰出现在相对荧光强度70处的为单倍体(图 2-B)。结果表明,通过离体诱导培养洋葱未受精花蕾,可以获得单倍体再生植株,“阿盾”、“地球”、“大宝”3个品种的单倍体诱导率分别为3.93%、4.84%和4.10%。同时也获得了7株二倍体,其中4株为双单倍体(表 2)。
|
| A:二倍体植株;B:单倍体植株 图 2 洋葱二倍体和单倍体植株的DNA含量分布图 |
利用天根生化科技(北京)有限公司生产的快捷型植物基因组提取试剂盒提取洋葱再生植株基因组总DNA。随机选取10个DNA样本,每个样本取5 μL DNA与2 μL 6×Loading buffer混匀,经0.8%琼脂糖凝胶电泳检测,显示提取的DNA样品在点样孔处没有明显残留,说明多糖、蛋白质等杂质含量较低。电泳条带清晰无降解现象且基本位于同一位置。在紫外可见分光光度计上检测OD260/OD280值在1.8-2.0之间,表明提取的DNA纯度较高,可以用于下一步PCR扩增试验。
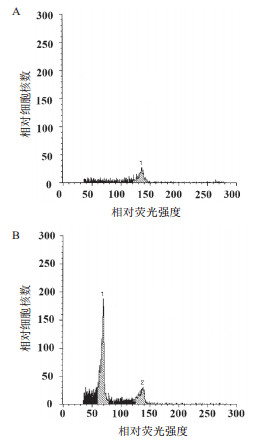
2.4 再生植株的分子标记鉴定为了鉴定再生植株是否为纯合体,利用DNF-566和RNS-357标记对源于杂交种“阿盾”的12株再生植株的基因组DNA进行PCR扩增(图 3)。发现有8株材料(1-5、8、11-12)扩增出一条357 bp的条带,表明这8株材料在Ms位点的基因型为纯合的msms。4株材料(6、7、9-10)扩增出一条566 bp的条带,表明这4株材料在Ms位点的基因型为纯合的MsMs。

|
| M:Marker DL2000;1-12:来源于杂交种“阿盾”的不同单株 图 3 DNF-566和RNS-357标记在部分雌核发育诱导再生植株材料中的扩增结果 |
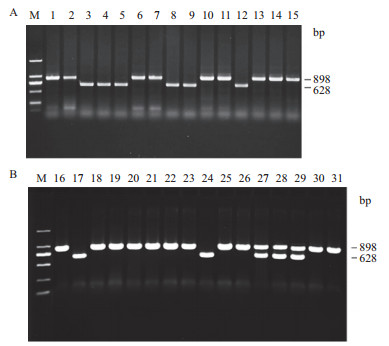
利用AcSKP1标记对源于杂交种“地球”的15株再生植株的基因组DNA进行PCR扩增(图 4)。有9株只扩增出898 bp的条带,6株只扩增出628 bp的条带,说明检测的所有材料在Ms位点的基因型均为纯合的。来源于“大宝”的16株再生植株中,有11株(16、18-23、25-26、30-31)扩增出898 bp条带,2株(17、24)扩增出628 bp条带,表明这13株材料在Ms位点的基因型是纯合的。有3株(27-29)扩增出了898和628 bp 2条片段,扩增后表现出了与供体材料相同的带型,表明这3株则是杂合的二倍体,可能由子房壁或其他体细胞发育而来。检测的再生植株供体材料来源于3个杂交种,在Ms位点的基因型是杂合的(Msms),而检测的材料绝大多数是纯合的,说明这些再生植株源于雌配子体,是通过雌核发育途径诱导培养得到的。
|
| A:“地球”;B:“大宝”;M:Marker DL2000;1-15:“地球”再生植株单株;16-31:“大宝”再生植株单株 图 4 AcSKP1标记在源于杂交种“地球”和“大宝”的雌核发育诱导再生植株材料中的扩增结果 |
洋葱基因组庞大(15 290 Mb)[17],并且重复序列多[18],GC含量仅为32%[19],分子标记研究相对落后,因此,在洋葱分子标记鉴定再生植株纯合性研究方面鲜见报道。Javornik等[13]利用RAPD标记检测离体雌核发育再生植株,与供体亲本相比,总共扩增的4 600个RAPD片段在诱导率高的品系中未发现变异,而在诱导率较低的品系中,检测到2个RAPD片段的存在微小变化。但是RAPD标记一般是显性遗传(极少数是共显性遗传的),这样对扩增产物的记录就可记为“有/无”,这也意味着不能鉴别杂合体和纯合体。Yang等[15]开发出了一对与显性等位基因Ms连锁的SCAR标记DNF-566和与隐性等位基因ms共分离的SCAR标记RNS-357。以此标记为基础,Huo等[16]开发出共显性的多重PCR分子标记AcSKP1,只需经过一次简单的PCR经琼脂糖凝胶电泳即可直接准确鉴定Ms位点的基因型(MsMs、Msms、msms)。相对于RAPD标记,SCAR标记所用引物较长且引物序列与模板DNA完全互补,可在严谨条件下进行扩增,因此,结果稳定性好、可重复性强。基于上述优点,应用洋葱雄性不育基因Ms位点的标记鉴定诱导再生植株的纯合性,这也是该标记在单倍体鉴定应用中的首次报道。
洋葱杂交种在Ms位点的基因型为杂合的Msms,通过雌核发育途径诱导培养获得植株,如果为配子体起源,不管其为单倍体或双单倍体,分子标记位点并未发生改变,应该只具有亲本的Ms或ms,即为纯合的MsMs或msms。如果为体细胞起源,应该具有和供体亲本相同的杂合基因型Msms。本研究中鉴定的43株再生植株中有40株是在Ms位点是纯合的,说明这40株是起源于雌配子体的纯合植株,其余3株是杂合的,表明其来源于供体亲本的体细胞组织。本研究通过流式细胞仪和洋葱雄性不育基因Ms位点分子标记对每个离体雌核诱导再生植株进行了鉴定,获得了4株双单倍体,确认为自然加倍而成的双单倍体。这个结果与前人[20-22]报道10%左右的自然加倍率的结果基本一致。由于本试验是离体雌核发育途径诱导洋葱通过胚状体发育而成的再生植株,没有经过愈伤组织诱导阶段,因此在遗传上具有相对的稳定性。获得的双单倍体再经过细胞质类型鉴定,可以作为候选不育系、保持系或父本系,也可为建立高密度的遗传连锁图奠定材料基础[23]。
4 结论洋葱雄性不育基因Ms位点的分子标记不仅可以作为鉴定洋葱细胞核育性的分子标记,而且可以用于洋葱单倍体再生植株与供体杂交种亲本的同源性与纯合型分析,与流式细胞仪结合可以快速准确鉴定出双单倍体。
| [1] |
Foschi M, Martínez L, Ponce MT, et al. Doble haploides, una estrategia biotecnológica para el mejoramiento genético en cebolla(Allium cepa L.)[J]. Horticultura Argentina, 2009, 28(66): 40-48. |
| [2] |
Fayos O, Valles MP, Claver AG, et al. Doubled haploid production from Spanish onion(Allium cepa L.)germplasm:embryogenesis induction, plant regeneration and chromosome doubling[J]. Front Plant Sci, 2015, 6: 1-11. |
| [3] |
Baldwin S, Revanna R, Pither-Joyce M, et al. Genetic analyses of bolting in bulb onion(Allium cepa L.)[J]. Theoretical and Applied Genetics, 2014, 127(3): 535-547. DOI:10.1007/s00122-013-2232-4 |
| [4] |
Khosa JS, Lee R, Bräuning S, et al. Doubled haploid 'CUDH2107'as a reference for bulb onion(Allium cepa L.)research:development of a transcriptome catalogue and identification of transcripts associated with male fertility[J]. PLoS One, 2016, 11(11): e0166568. DOI:10.1371/journal.pone.0166568 |
| [5] |
Hyde PT, Earle ED, Mutschler MA. Doubled haploid onion(Allium cepa L.)lines and their impact on hybrid performance[J]. HortScience, 2012, 47(12): 1690-1695. DOI:10.21273/HORTSCI.47.12.1690 |
| [6] |
Asadi A, Zebarjadi A, Abdollahi MR, et al. Assessment of different anther culture approaches to produce doubled haploids in cucumber(Cucumis sativus L.)[J]. Euphytica, 2018, 214(11): 216. DOI:10.1007/s10681-018-2297-x |
| [7] |
Meyer R, Salamini F, Uhrig H. Isolation and characterization of potato diploid clones generating a high frequency of monohaploid or homozygous diploid androgenetic plants[J]. Theoretical and Applied Genetics, 1993, 85(6): 905-912. |
| [8] |
Chani E, Veilleux RE, Boluarte-Medina T. Improved androgenesis of interspecific potato and efficiency of SSR markers to identify homozygous regenerants[J]. Plant Cell, Tissue and Organ Culture, 2000, 60(2): 101-112. |
| [9] |
Diao WP, Jia YY, Song H, et al. Efficient embryo induction in cucumber ovary culture and homozygous identification of the regenetants using SSR markers[J]. Scientia Horticulturae, 2009, 119(3): 246-251. DOI:10.1016/j.scienta.2008.08.016 |
| [10] |
Gonzalo MJ, Claveria E, Monforte AJ, et al. Parthenogenic haploids in melon:generation and molecular characterization of a doubled haploid line population[J]. Journal of the American Society for Horticultural Science, 2011, 136(2): 145-154. DOI:10.21273/JASHS.136.2.145 |
| [11] |
Malik AA, Li C, Zhang SX, et al. Efficiency of SSR markers for determining the origin of melon plantlets derived through unfertilized ovary culture[J]. Horticultural Science, 2011, 38(1): 27-34. |
| [12] |
Campion B, Bohanec B, Javornik B. Gynogenic lines of onion(Allium cepa L.):evidence of their homozygosity[J]. Theoretical and Applied Genetics, 1995, 91(4): 598-602. DOI:10.1007/BF00223285 |
| [13] |
Javornik B, Bohanec B, Campion B. Second cycle gynogenesis in onion, Allium cepa L., and genetic analysis of the plants[J]. Plant Breeding, 1998, 117(3): 275-278. |
| [14] |
刘冰江, 缪军, 霍雨猛, 等. 离体雌核发育诱导洋葱单倍体与植株再生[J]. 园艺学报, 2012, 39(11): 2265-2270. |
| [15] |
Yang YY, Huo YM, Miao J, et al. Identification of two SCAR markers co-segregated with the dominant Ms and recessive ms alleles in onion(Allium cepa L.)[J]. Euphytica, 2013, 190(2): 267-277. DOI:10.1007/s10681-012-0842-6 |
| [16] |
Huo YM, Liu BJ, Yang YY, et al. AcSKP1, a multiplex PCR-based co-dominant marker in complete linkage disequilibrium with the male-fertility restoration(Ms)locus, and its application in open-pollinated populations of onion[J]. Euphytica, 2015, 204(3): 711-722. DOI:10.1007/s10681-015-1374-7 |
| [17] |
Labani RM, Elkington TT. Nuclear DNA variation in the genus Allium L. (Liliaceae)[J]. Heredity, 1987, 59(1): 119-128. DOI:10.1038/hdy.1987.103 |
| [18] |
Arumuganathan K, Earle ED. Nuclear DNA content of some important plant species[J]. Plant Molecular Biology Reporter, 1991, 9(3): 208-218. DOI:10.1007/BF02672069 |
| [19] |
Stack SM, Comings DE. The chromosomes and DNA of Allium cepa[J]. Chromosoma(Berlin), 1979, 70(2): 161-181. |
| [20] |
Bohanec B. Doubled haploid onions//Rabinowitch HD, Currah L. Allium crop science: recent advances[M]. Wallinford: CAB International, 2002: 145-157.
|
| [21] |
Duangjit J, Bohanec B, Chan AP, et al. Transcriptome sequencing to produce SNP-based genetic maps of onion[J]. Theoretical and Applied Genetics, 2013, 126(8): 2093-2101. DOI:10.1007/s00122-013-2121-x |
| [22] |
Mathapati GB, Kalia P, Islam S, et al. Influence of culture media and their compositions on haploid induction in indian short day onion[J]. Proceedings of the National Academy of Sciences, India Section B:Biological Sciences, 2019, 89(2): 739-746. DOI:10.1007/s40011-018-0990-0 |
| [23] |
McCallum J, Baldwin S, Macknight R, et al. Molecular mapping of genes and QTL:progress to date and development of new population resources for NGS genetics[M]. Springer, Cham: The Allium Genomes, 2018: 181-196.
|