在自然环境中,植物常常会遭遇各种不利环境,从而导致植物的生长受阻、产量减少、生态价值和经济价值受损,这些不利环境包括生物胁迫和非生物胁迫[1]。生物胁迫有病原体感染、食草动物吸食和人为破坏等;非生物胁迫主要包括干旱胁迫、渗透胁迫或盐胁迫、极端温度(高温、低温)等。随着全球气候的变化,这些非生物胁迫对植物的影响日益加剧,并造成全球主要农作物的减产,而全球气候变化趋势将导致非生物胁迫出现的频率增加。因此,深入研究植物在非生物胁迫下的响应机制(包括信号感知,传递以及转录调控和应答基因的表达),培育抗逆植物新品种,保证全球粮食供给和农林业的有效生产,对于人类生活环境的改善和生活质量的提高有极大的促进作用。
为了在不利环境下正常生长,植物在生理、生化和分子水平上进化出了一系列复杂的响应机制,包括信号的感知、依赖ABA通道和ABA独立通道的信号转导、胁迫诱导基因的表达及进一步激活生理和代谢反应[2]。植物在非生物胁迫响应中的基因产物分为两类:第一类由一些调控下游信号转导及胁迫应答基因表达的蛋白因子组成,称为调节蛋白[3],包括转录因子(NAC、MYB、MYC、WRKY、bZIP和HB等)、蛋白激酶和磷酸激酶等;第二类在植物对非生物胁迫的适应过程中具有直接作用,称为功能蛋白,包括小分子渗透物质(如脯氨酸、甜菜碱和可溶性糖)、晚期胚胎发育丰富(LEA)蛋白、脱水素、保护酶(如SOD、CAT和POD)及水通道蛋白等,这些蛋白质可以通过维持植物细胞膨压、清除活性氧和保护细胞内生物大分子的结构来缓冲各种非生物胁迫对植物细胞的损伤[4]。
转录因子(Transcription factors,TFs)是一种通过与DNA或其他蛋白质复合物的特定序列结合来调控基因表达的蛋白质[5],而转录调控是指在基因表达的过程中,转录因子与胁迫应答基因上游的顺式作用元件相结合从而激活或者抑制基因的表达,转录调控分为依赖ABA通道和ABA独立通道[6]。植物在受到外界干旱、高盐等非生物胁迫时,会引起ABA生物合成的增加,从而激活MYB、MYC、NAC、bZIP等转录因子与下游的相应的胁迫应答基因RD22、Gly、RD29B和RD20A的启动子区特定序列的顺式作用元件MYBRS、MYCRS和ABRE(核心序列为ACGTGGC)结合,从而激活胁迫应答基因的表达;在ABA独立通道中,植物细胞感受到环境刺激后将信号转导至转录因子NAC、DREB2,激活了NAC、DREB2转录因子与相关胁迫应答基因ERD1和RD29A的启动子区特定序列的顺式作用元件DRE/CRT结合,从而激活胁迫应答基因的表达。
植物特异性转录因子NAC是来自矮牵牛的NAM和来自拟南芥的ATAF1、ATAF2和CUC2的首字母缩写,随着植物全基因组测序和EST序列数量的不断增加,在许多物种中都发现了NAC基因,模式植物拟南芥和水稻中分别有117、151个NAC转录因子[7],苹果有180个[8]、香蕉有167个[9]、大豆有152个[10]、沙棘有177个[3]。NAC转录因子不仅参与植物的生长发育(如侧根的发育、花的发育、次生壁的形成及植物激素信号转导等[11]),而且在植物响应非生物胁迫如干旱、盐碱、水涝及低温等过程中都起着重要的调节作用。本文就NAC转录因子的结构特征、作用机理及其在植物非生物胁迫中的功能研究进展进行综述,为NAC相关的基因研究以及植物的抗逆新品种培育提供理论依据。
1 NAC转录因子的结构特征NAC蛋白是最大的植物特异性转录因子家族,其在植物各个发育阶段和不同组织中表达,参与各种生长和发育过程以及调节植物对环境胁迫的反应[12]。NAC家族共有的脱氧核苷酸序列被定义为NAC域,NAC域位于NAC蛋白的N端,全长约150个氨基酸,是高度同源的区域,由A、B、C、D、E 5个亚结构域构成(图 1)。NAC域中的各个亚结构比对表明亚结构域A、C、D是严格保守的,B和E不保守,NAC结构域包含特定的DNA结合结构域(DNA binding domain,DBD),DBD包含在60个氨基酸的区域内,其可能折叠成特异性结合CaMV 35S启动子的螺旋-转角-螺旋基序,B和E亚区域中序列的多样性与功能、发育阶段、组织特异性的多样性有关,转录激活区(Transcriptional activation region,TAR)位于NAC蛋白的C端[13]。
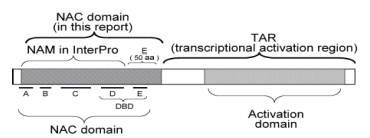
|
| NAC家族基因的NAC结构域位于N端,如图中矩形条纹区域,实线表示子域A、B、C、D和E,NAC域:AtNAM的DNA结合域在子域D和E内,矩形阴影区域表示TAR区(transcriptional activation region),位于NAC结构的C端 图 1 NAC转录因子结构示意图 |
植物在生长发育过程中面临着各种生物和非生物胁迫,严重影响其自身的生长发育和有机物积累,因此,许多学者致力于植物响应非生物胁迫的调控机制研究。随着全球气候变暖,干旱、盐碱和极端温度成为阻碍植物生长和产量的主要因素,本文主要综述NAC转录因子在干旱胁迫、盐胁迫和极端温度胁迫中的调控作用的研究进展。
2.1 NAC转录因子在干旱胁迫中的作用干旱是全球气候变化的直接后果之一,日益加剧的全球气候变暖对农林业生产构成严重威胁。全球气候变暖导致干旱频率增加,严重影响了粮食作物的生存和产量。干旱对于植物生长的影响显著,轻度的干旱降低产量,严重的干旱会引起植物永久萎焉而导致颗粒无收。研究表明,NAC转录因子在干旱胁迫中差异表达,是干旱胁迫关键基因之一。植物NAC转录因子通过4种途径来提高植物的耐旱性,一是通过结合响应干旱胁迫的关键转录因子的顺式作用元件,从而激活相关转录因子的表达来提高植物的耐旱性,如MYB、DREB、WRKY等;二是通过增加植物渗透调节物的含量如脯氨酸、甜菜碱等来维持植物体内渗透平衡,防止蛋白质变性,从而提高植物在干旱胁迫下的存活率;三是通过抑制植物细胞内水分外流以保持较高的相对含水量;四是调节植物的干旱胁迫相关的应答基因的表达来减少胁迫对植物的损伤。
大田干旱条件下,小麦叶片和小麦籽粒的23个TaNAC基因中有7个具有叶片特异性表达、5个具有籽粒特异性表达[14];从菜豆基因组数据库中鉴定出的86种NAC蛋白中22个与干旱相关,MBS是一种顺式作用元件,为参与干旱诱导的MYB结合位点,TsApx6(Thellungiella salsuginea)参与干旱胁迫反应,其启动子区中含有MBS元素[15],与干旱相关的菜豆NAC基因中有16个基因含有MBS顺式作用元件,表明NAC基因参与了菜豆的干旱响应[16]。通过病毒诱导的基因沉默抑制NAC转录因子基因SlJUB1的表达,显著降低了番茄耐旱性,同时引起离子渗漏的增加,过氧化氢水平的升高和各种干旱响应基因表达的降低;拟南芥AtJUB1的过量表达增加了番茄的耐旱性,干旱期间叶片相对含水量较高,过氧化氢水平降低;干旱会引起SlJUB1的表达量升高,直接结合番茄中SlDREB1、SlDREB2和SlDELLA的启动子,从而激活DELLA和应激相关基因DREB2和DREB1的表达,增加了植物的抗旱性[17]。由此可见,JUB1正向调节番茄抗旱性。同时,在拟南芥中已经证明JUB1通过影响植物激素生物合成/信号传导和ROS信号传导中涉及的多种相互关联的细胞途径来限制植物生长并增强对非生物胁迫的耐受性,AtJUB1直接抑制对GA和BR生物合成至关重要的基因(分别为GA3ox1和DWF4)[18],同时它直接激活编码DELLA的基因GAI和RGL1,从而导致DELLA蛋白的积累。此外,AtJUB1直接靶向激活拟南芥响应干旱胁迫的关键转录因子基因DREB2A的表达。将辽宁碱蓬SlNAC1转入拟南芥发现,在干旱胁迫下,转基因拟南芥的成活率较高,转基因植株的水分损失率低于野生型(WT)和对照组(V)植株,成活率高于WT和V植株,表明SlNAC1能增强转基因植物的耐旱性[12]。过表达MusaNAC042的转基因香蕉植株在干旱胁迫下,脯氨酸含量升高,丙二醛含量降低,且在相同的干旱胁迫水平下,转基因植株的相对含水量高于对照植株,说明其具有较强的抗旱性[19]。另外,在干旱胁迫下过表达MusaNAC042可引起9个应激相关基因的表达上调,包括CBF/DREB、5个WRKY和3个LEA基因。干旱胁迫下,过表达ONAC022(N22oe)的转基因水稻植株较野生型(WT)植株耐旱性强,成活率高,生长性能好,蒸腾速率降低,气孔率降低,脯氨酸和可溶性糖含量增加,且ONAC022在转基因水稻中的过表达可以通过ABA依赖通路调控[20]。干旱胁迫下转TaNAC67植株的细胞膜稳定性(CMS)均高于野生型(WT)和对照组,表明CMS的增强是由TaNAC67的过表达引起的,此外,研究发现,TaNAC67突变体在水分胁迫下干旱胁迫应答基因DREB2A、ABI1和ABI2的表达显著且持续上调,DREB2A是参与干旱反应的重要调控因子,它的显著上调增加了下游干旱胁迫应答基因的表达,增强了植物对干旱胁迫的耐受性,ABI1和ABI2参与ABA信号转导的调控,其高表达水平会引起受ABI1和ABI2控制的基因表达上调,并增强植物依赖ABA通路的干旱胁迫响应能力[21]。
2.2 NAC转录因子在渗透胁迫和盐胁迫中的作用盐胁迫是一种非常普遍的环境胁迫,它直接影响植物的生长、发育、分布区域和产量。盐胁迫不仅会引起多种生理变化,如植物失水、黄化和萎蔫[22],还会引起植物体内的生化变化,一些大分子物质如蛋白质和脂类被破坏和水解,导致代谢紊乱,甚至引起植物死亡。此外,盐胁迫下活性氧的过度积累加速了植物萎蔫[23]。盐胁迫诱导的转录因子包括MYB、bZIP、NAC和AP2/ERF等,还有一些植物激素在盐胁迫响应过程中也起着重要的调节作用,如赤霉酸(GA)、茉莉酸(JA)、脱落酸(ABA)和乙烯等[24-25]。NAC转录因子响应盐胁迫主要通过以下几方面:一是通过维持细胞内Na+和K+浓度增强植物的耐盐性;二是通过激活胁迫应答相关基因的表达如LEA、SOD、POD、HRG、ABA和乙烯等保持细胞内环境的相对稳态;三是促进小分子渗透调节物(甜菜碱、脯氨酸、可溶性糖等)的积累从而保护细胞结构的稳定性;四是调节气孔关闭来平衡植物体内的水分状态。
在盐胁迫条件下,苹果MdNAC047直接与乙烯启动子结合并激活其转录,增加了乙烯反应基因的表达,从而增强了苹果对盐胁迫的耐受性[26]。在高盐条件下,ATAF1转基因水稻种子的发芽率高于野生型(WT)水稻种子发芽率,表现出较好的耐盐性,研究发现,ATAF1在水稻中的过表达提高了盐胁迫诱导基因OsLEA3、OsSalT1和OsPM1的转录水平,OsLEA3的积累能维持细胞的结构和功能,以促进植物恢复,OsSalT调控细胞内Na+的浓度,防止细胞受损,并且这3个基因的启动子分别包含5、7和6个ATAF1结合基序,说明ATAF1通过结合OsLEA3、OsSalT1和OsPM1的启动子并激活它们的表达来改善转基因水稻耐盐性[27]。在盐胁迫处理下,ONAC022过表达(N22oe)的转基因水稻与WT相比表现出更强的耐盐性,根和芽中积累的Na+较少,叶片水分散失率降低,蒸腾速率降低,气孔闭合率增加,游离脯氨酸和可溶性糖积累增加,在过表达ONAC009和ONAC058的转基因水稻中也观察到类似的现象[28-29]。此外,N22oe植物的根系较大,说明ONAC022可能在胁迫条件下调节根系,从而提高胁迫耐受能力,与野生型相比表现出生长发育迟缓,这可能是由于胁迫耐受与正常生长发育之间能量的重新分配,从而提高植物在盐胁迫下的存活率[20]。从杨树中克隆NAC13,使用过表达和基因敲除技术得到转基因杨树,过表达NAC13转基因杨树在盐胁迫下,气孔会收缩甚至闭合,脯氨酸含量增加,胁迫相关基因(包括SOD、POD、HRG、ABA和GA相关基因)的差异表达增强了杨树的耐盐性,此外,过表达NAC13突变体的根系比野生型(WT)发达,具有更好的形态学性状,基因沉默的突变体增加了盐胁迫敏感性,证明NAC13在盐胁迫反应中具有重要的调节作用[30]。小麦中的TaNAC29受盐胁迫诱导,盐胁迫处理下,过表达TaNAC29的转基因拟南芥与野生型(WT)相比,积累了较少的H2O2,SOD、POD、APX和CAT活性均显著提高。此外,转基因组应激相关基因(如RD29A、RD29B、KIN)转录水平明显升高,TaNAC29通过增强抗氧化系统,清除活性氧,减少膜损伤,调控应激相关基因的诱导,增强植物耐盐性[31]。柠条的两种NAC转录因子基因CiNAC3和CiNAC4对盐胁迫有调控作用。CiNAC3和CiNAC4的转基因拟南芥耐盐性明显增强,与野生型相比,CiNAC4转基因株系的侧根数显著增加[32]。研究发现,大部分过表达TaNAC67转基因拟南芥的叶绿素含量和可变荧光与最大荧光的比值Fv/Fm优于对照组,表明TaNAC67转基因品系在重度盐胁迫条件下具有更强的光合能力,能够保证更多的有机物积累并提高植物的耐盐性[21]。辽宁碱蓬的SlNAC1能够被盐胁迫诱导,用NaCl溶液处理拟南芥幼苗,野生型和对照组植株大部分死亡,成活率较低(Wt为17%,V为17%),但转基因拟南芥N1和N2系的39%和35%继续生长,可见过表达SlNAC1可以提高植物的耐盐性[12]。
2.3 NAC转录因子在极端温度中的作用极端温度对植物造成生理胁迫,导致减产甚至个体死亡[33]。植物面对的极端温度胁迫主要有极端高温引起的热胁迫和极端低温引起的冷胁迫,如夏季的高温、春季植物萌发后的倒春寒以及冬季冻土之前的寒冷,热胁迫和冷胁迫已成为限制全球作物生产的主要环境因素。植物在极端温度胁迫下,会发生生理和生化水平的一系列变化,使活性氧大量累积,对核酸、蛋白质和脂质造成损害[34]。
2.3.1 NAC转录因子在低温胁迫中的作用低温可引起细胞膜冻结,并通过渗透、氧化和其他胁迫直接或间接地抑制植物的生长,造成不可逆转的损害,严重影响作物产量和质量[35-36]。在低温条件下,植物表现出多种生理生化反应,包括钙含量的增加,活性氧(ROS)的累积、膜脂成分的变化以及形成低温保护蛋白、可溶性糖和氨基酸等低温保护代谢物[37]。此外,低温会诱导植物一系列冷应激基因,这些基因可编码亲水可溶性多肽,增强抗氧化酶的活性,通过修复膜系统和稳定细胞渗透势来改变自身生理生化特性,从而改善植物的抗冻性[38]。
冷信号通路是ICE-CBF转录调控级联[39],CBF转录因子可调节某些冷应激基因启动子中CRT/脱水反应元件(DRE)的表达和功能,CBF表达诱导因子1(ICE1)是一种类似MYC的bHLH转录激活因子。香蕉转录因子MaNAC1是MaICE1的直接靶基因,MaICE1磷酸化和冷应激可以增强MaICE1与MaNAC1启动子结合的能力。低温胁迫下,MaNAC1通过与ICE1-CBF信号通路相互作用提高香蕉果实的耐寒性。辽宁碱蓬的转录因子SlNAC1参与了冷应激响应的调控,过表达SlNAC1的转基因拟南芥在冷胁迫下的成活率显著高于野生型和对照组,说明SlNAC1可增强植物的抗寒性[12]。过表达TaNAC67的转基因拟南芥对严重冰冻胁迫的耐受性强于野生型[21]。梅花中的15个NAC家族基因在低温胁迫下的表达表明其中10个表达均上调,另外还有5个成员的表达受到明显抑制,证明NAC转录因子对冷胁迫具有调控作用[40]。
低温胁迫不仅通过诱导NAC转录因子的表达正向提高植物的耐寒性,也会通过抑制或者下调NAC转录因子的表达或者负调控来减弱植物的耐寒性,如在水稻中,低温胁迫降低了ONAC095的表达,ONAC095-SRDX植株表现出严重的萎萎现象,存活率较低,电解质渗漏率较高,低温诱导基因表达下调,超氧化物歧化酶和过氧化氢酶的活性降低,丙二醛含量增加,植株积累过量的过氧化氢和超氧阴离子,说明ONAC095-SRDX植株体内活性氧的清除失衡,造成植物细胞的毒害[41],致使转基因水稻的耐寒性降低[42]。苹果MdNAC029可特异性结合MdCBF1和MdCBF4启动子,直接抑制MdCBF1和MdCBF4的表达,且过表达MdNAC029降低了苹果愈伤组织和拟南芥的耐寒性,因此,MdNAC029作为负调节因子通过依赖CBF方式调节植物耐寒性[43]。
虽然有关NAC转录因子对耐寒性的调节相关的研究已有报道,但是研究对象很有限,NAC转录因子是冷应激转录网络中潜在的重要调控基因,更多植物的耐寒性调控网络有待于进一步研究。
2.3.2 NAC转录因子在高温胁迫中的作用在植物中,高温引起的热胁迫通常被定义为温度足够高、在足够长的时间内不可逆地损害植物的功能或发育[44]。高温胁迫对植物生长、发育、繁殖和产量产生不利影响,植物各个组织都容易受到高温损伤。高温引起的直接损伤包括蛋白质变性、聚集,膜脂流动性增加,间接损伤包括叶绿体和线粒体酶的失活、蛋白质合成的抑制、蛋白质降解、膜完整性的丧失和细胞骨架结构的破坏等[45]。在热胁迫下,植物通过维持膜稳定性,清除活性氧物质,诱导分子伴侣信号传导和转录激活热激蛋白(Heat shock proteins,HSPs)的表达来提高植物自身的耐热性[46]。
拟南芥NAC019过表达植株比野生型耐热性更强,热因子因子(Heat shock factors,HSFs)是HSPs积累的主要机制。热应激还可调控RCF2的表达,RCF2与NAC019相互作用并使其去磷酸化,RCF2不仅对NAC019的翻译后修饰具有重要作用,同时还参与转录和转录后水平的表达,除HSFA7a转录量减少外,热应激反应基因在NAC019过表达植株中表达水平升高,由此可见,NAC019在拟南芥响应热胁迫中有重要的调控作用[47]。将小麦TaNAC2转入拟南芥,转基因和野生型幼苗同时置于38℃ 90 min,野生型和转基因植株下胚轴较短,在45℃处理2 h后植株均死亡,当两组幼苗第一次在38℃预处理90 min后再暴露于45℃环境中,转基因TaNAC2L拟南芥下胚轴比野生型植株长,表明TaNAC2L转录因子能够提高拟南芥的耐热性。进一步研究表明,高温胁迫下,转基因拟南芥中有6个热应激诱导基因表达水平高于野生型,说明TaNAC2L的过表达可通过上调下游的应激反应基因来增强植物耐热性[48]。
程序性细胞死亡(Programmed cell death,PCD)是生物体中严格调控的适应过程,在植物中,细胞的有序破坏是由多种内源性和环境信号诱导的,植物通过PCD途径将受损细胞和组织清除,从而实现营养物质和资源的有效循环和促进[49]。在极端高温胁迫条件下,植物可能无法维持整个植物器官。因此,牺牲受损的植物组织将有利于维持植物的主要部分(如分生组织和再生器官)。NAC转录因子基因NTL4过表达拟南芥在高温胁迫下通过依赖ABA途径和ABA独立途径进行信号传递,引起细胞内H2O2的积累,H2O2通过正反馈调节诱导NTL4的表达,ROS反过来诱导NTL4转录和NTL4加工,NTL4和ROS构成一个正反馈回路,在高温下引起ROS急剧增加,将受损组织中的营养物和代谢物再循环到分生组织或新形成的叶子中,引起局部PCD,从而增强植物的存活率[50]。
由上述研究可见,NAC转录因子在改善植物对温度胁迫耐受方面具有潜在的应用价值。然而,关于其在高温胁迫响应中作用的研究仍然很有限,相关的信号传递和转录调控网络需要进一步研究来阐明。
3 总结与展望NAC转录因子在非生物胁迫响应过程中有重要的调节作用。研究发现,NAC转录因子往往不是单一的增强植物对某一种非生物胁迫的耐受性,而是协同调控多个非生物胁迫网络。如辽东碱蓬的转录因子SlNAC1不仅可以增强转基因拟南芥的耐旱性,而且对寒冷和高盐胁迫的耐受性也显著增强[12];水稻转录因子ONAC095起着抗旱负调控抗寒正调控的作用[41];香蕉转录因子MusaNAC042对高盐度和干旱胁迫的耐受性都有正向调控作用[20];西瓜中几种NAC转录因子响应多种非生物胁迫及生长发育,如亚家族IV NAC可能在维持干旱或盐条件下的水状态、水和代谢物传导以及向果实的易位方面发挥作用。与之相反,大多数ClNAC对低温逆境的快速和消极反应可能与对冷应激的敏感性有关,ClNAC的表达与非生物胁迫和激素(脱落酸和茉莉酸)的调控网络之间存在互作关系[51]。由此可见,NAC转录因子响应非生物胁迫的网络非常复杂,不仅与不同家族转录因子之间存在级联互作,还与信号传递、保护酶、各种胁迫诱导基因以及植物激素互作共同调控植物对非生物胁迫的综合耐受性。
虽然植物NAC转录因子能够提高植物对于各种非生物胁迫的耐受性,但是也存在一些不容忽视的问题。首先,利用基因手段使NAC转录因子过表达突变体引起的消极影响,如转基因植株矮化,产量减少,开花延迟等[52];第二,过表达的转基因植株同时提高了一种或几种非生物胁迫的耐受性,但对生物胁迫产生不利影响;第三,目前,关于NAC转录因子在非生物胁迫中的作用的研究对象主要是模式植物、主要农作物或经济林树种,对于其他具有很高生态价值和经济价值的林木中NAC转录因子的研究很有限,且国内外学者关于NAC转录因子的极端温度胁迫的研究报道较少。中国西北、东北、以及陕北地区冬季温度低,早晚温差大,倒春寒都会引起低温胁迫,提高植物的耐寒性不仅能够提高植物的成活率节约造林成本还能增加景观树种丰富度。基于以上问题,应该综合深入研究NAC转录因子应对非生物胁迫的基因调控网络,明确其抗逆调控机制,将过表达该基因的消极影响降到最小,实现经济利益、生态利益最大化的农林生产,对于更好地解决全球粮食短缺和人口不断增长的问题以及提高农林作物的经济价值和生态价值具有重要意义。
| [1] |
Zhu J. Abiotic stress signaling and responses in plants[J]. Cell, 2016, 167(2): 313-324. DOI:10.1016/j.cell.2016.08.029 |
| [2] |
Pérez-Clemente RM, Vives V, Zandalinas SI, et al. Biotechnological approaches to study plant responses to stress[J]. BioMed Research International, 2013, 2013(1): 654120. |
| [3] |
Ye GS, Ma YH, Feng ZP, et al. Transcriptomic analysis of drought stress responses of sea buckthorn(Hippophae rhamnoides subsp. sinensis)by RNA-Seq[J]. PLoS One, 2018, 13(8): e202213. |
| [4] |
Shinozaki K, Yamaguchishinozaki K. Gene networks involved in drought stress response and tolerance[J]. J Exp Bot, 2007, 58(2): 221. |
| [5] |
Mun B, Lee S, Park E, et al. Analysis of transcription factors among differentially expressed genes induced by drought stress in Populus davidiana[J]. 3 Biotech, 2017, 7(3): 209. |
| [6] |
Banerjee A, Roychoudhury A. Abscisic-acid-dependent basic leucine zipper(bZIP)transcription factors in plant abiotic stress[J]. Protoplasma, 2017, 254(1): 3-16. DOI:10.1007/s00709-015-0920-4 |
| [7] |
王春雨, 张茜. 植物NAC转录因子功能研究进展[J]. 生物技术通报, 2018, 34(11): 14-20. |
| [8] |
Su H, Zhang S, Yuan X, et al. Genome-wide analysis and identification of stress-responsive genes of the NAM-ATAF1, 2-CUC2 transcription factor family in apple[J]. Plant Physiology & Biochemistry, 2013, 71(2): 11-21. |
| [9] |
Cenci A, Guignon V, Roux N, et al. Genomic analysis of NAC transcription factors in banana(Musa acuminata)and definition of NAC orthologous groups for monocots and dicots[J]. Plant Mole Biol, 2014, 85(1/2): 63-80. |
| [10] |
Le DT, Nishiyama R, Watanabe Y, et al. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J]. DNA Research, 2011, 18(4): 263-276. DOI:10.1093/dnares/dsr015 |
| [11] |
Mao C, Lu S, Lv B, et al. A rice NAC transcription factor promotes leaf senescence via ABA biosynthesis[J]. Plant Physiology, 2017, 174(3): 1747-1763. DOI:10.1104/pp.17.00542 |
| [12] |
Li XL, Yang X, Hu YX, et al. A novel NAC transcription factor from Suaeda liaotungensis K. enhanced transgenic Arabidopsis drought, salt, and cold stress tolerance[J]. Plant Cell Reports, 2014, 33(5): 767-778. DOI:10.1007/s00299-014-1602-y |
| [13] |
Swati P, Pranav Pankaj S, Srivastava PS, et al. NAC proteins:regulation and role in stress tolerance[J]. Trends in Plant Science, 2012, 17(6): 369-381. DOI:10.1016/j.tplants.2012.02.004 |
| [14] |
Guerin C, Roche J, Allard V, et al. Genome-wide analysis, expansion and expression of the NAC family under drought and heat stresses in bread wheat(T. aestivum L.)[J]. PLoS One, 2019, 14(3): e213390. |
| [15] |
Li Z, Zhang J, Li J, et al. The functional and regulatory mechanisms of the thellungiella salsuginea ascorbate peroxidase 6(TsAPX6)in response to salinity and water deficit stresses[J]. PLoS One, 2016, 11(4): e154042. |
| [16] |
Wu J, Wang L, Wang S. Comprehensive analysis and discovery of drought-related NAC transcription factors in common bean[J]. BMC Plant Biol, 2016, 16(1): 193. |
| [17] |
Thirumalaikumar VP, Devkar V, Mehterov N, et al. NAC transcription factor JUNGBRUNNEN1 enhances drought tolerance in tomato[J]. Plant Biotechnol J, 2018, 16(2): 354-366. DOI:10.1111/pbi.12776 |
| [18] |
Shahnejat-Bushehri S, Tarkowska D, Sakuraba Y, et al. Arabidopsis NAC transcription factor JUB1 regulates GA/BR metabolism and signalling[J]. Nature Plants, 2016, 2(3): 16013. DOI:10.1038/nplants.2016.13 |
| [19] |
Tak H, Negi S, Ganapathi TR. Banana NAC transcription factor MusaNAC042 is positively associated with drought and salinity tolerance[J]. Protoplasma, 2017, 254(2): 803-816. DOI:10.1007/s00709-016-0991-x |
| [20] |
Hong YR, Zhang HJ, Huang L, et al. Overexpression of a stress-responsive NAC transcription factor gene ONAC022 improves drought and salt tolerance in rice[J]. Front Plant Sci, 2016, 7: 4. DOI:10.3389/fpls.2016.00004 |
| [21] |
Mao X, Chen S, Li A, et al. Novel NAC transcription factor TaNAC67 confers enhanced multi-abiotic stress tolerances in Arabidopsis[J]. PLoS One, 2014, 9(1): e84359. DOI:10.1371/journal.pone.0084359 |
| [22] |
Parida AK, Das AB. Salt tolerance and salinity effects on plants:a review[J]. Ecotoxicology and Environmental Safety, 2005, 60(3): 324-349. DOI:10.1016/j.ecoenv.2004.06.010 |
| [23] |
Jiang Z, Zhu S, Ye R, et al. Relationship between NaCl- and H2O2-induced cytosolic Ca2+ increases in response to stress in Arabidopsis[J]. PLoS One, 2013, 8(10): e76130. DOI:10.1371/journal.pone.0076130 |
| [24] |
Shu K, Qi Y, Chen F, et al. Salt stress represses soybean seed germination by negatively regulating GA biosynthesis while positively mediating ABA biosynthesis[J]. Front Plant Sci, 2017, 8: 1372. DOI:10.3389/fpls.2017.01372 |
| [25] |
Dou L, He K, Higaki T, et al. Ethylene signaling modulates cortical microtubule reassembly in response to salt stress[J]. Plant Physiology, 2018, 176(3): 1124-2017. |
| [26] |
An JP, Yao JF, Xu RR, et al. An apple NAC transcription factor enhances salt stress tolerance by modulating the ethylene response[J]. Physiol Plant, 2018, 164(3): 279-289. |
| [27] |
Liu Y, Sun J, Wu Y. Arabidopsis ATAF1 enhances the tolerance to salt stress and ABA in transgenic rice[J]. Journal of Plant Research, 2016, 129(5): 955-962. DOI:10.1007/s10265-016-0833-0 |
| [28] |
Xu C, Yaofeng W, Bo L, et al. The NAC family transcription factor OsNAP confers abiotic stress response through the ABA pathway[J]. Plant & Cell Physiology, 2014, 55(3): 604. |
| [29] |
Song S, Chen Y, Chen J, et al. Physiological mechanisms underlying OsNAC5-dependent tolerance of rice plants to abiotic stress[J]. Planta, 2011, 234(2): 331-345. DOI:10.1007/s00425-011-1403-2 |
| [30] |
Zhang X, Cheng Z, Zhao K, et al. Functional characterization of poplar NAC13 gene in salt tolerance[J]. Plant Science, 2019, 281: 1-8. DOI:10.1016/j.plantsci.2019.01.003 |
| [31] |
Xu Z, Gongbuzhaxi, Wang C, et al. Wheat NAC transcription factor TaNAC29 is involved in response to salt stress[J]. Plant Physiology and Biochemistry, 2015, 96: 356-363. DOI:10.1016/j.plaphy.2015.08.013 |
| [32] |
Han X, Feng Z, Xing D, et al. Two NAC transcription factors from Caragana intermedia altered salt tolerance of the transgenic Arabidopsis[J]. BMC Plant Biology, 2015, 15(1): 208. |
| [33] |
Dowd WW, King FA, Denny MW. Thermal variation, thermal extremes and the physiological performance of individuals[J]. Journal of Experimental Biology, 2015, 218(Pt12): 1956. |
| [34] |
Sarvajeet Singh G, Narendra T. Reactive oxygen species and antioxidant machinery in abiotic stress tolerance in crop plants[J]. Plant Physiol Biochem, 2010, 48(12): 909-930. DOI:10.1016/j.plaphy.2010.08.016 |
| [35] |
Yuan P, Yang T, Poovaiah BW. Calcium signaling-mediated plant response to cold stress[J]. International Journal of Molecular Sciences, 2018, 19(12): 3896. DOI:10.3390/ijms19123896 |
| [36] |
Kazemi-Shahandashti SS, Maali-Amiri R. Global insights of protein responses to cold stress in plants:Signaling, defence, and degradation[J]. Journal of Plant Physiology, 2018, 226: 123. DOI:10.1016/j.jplph.2018.03.022 |
| [37] |
Ruelland E, Vaultier MN, Zachowski A, et al. Chapter 2 cold signalling and cold acclimation in plants[J]. Advances in Botanical Research, 2009, 49: 35-150. DOI:10.1016/S0065-2296(08)00602-2 |
| [38] |
Shi Y, Ding Y, Yang S. Molecular regulation of CBF signaling in cold acclimation[J]. Trends in Plant Science, 2018, 23(7): S314931008. |
| [39] |
Knight MR, Heather K. Low-temperature perception leading to gene expression and cold tolerance in higher plants[J]. New Phytologist, 2012, 195(4): 737-751. DOI:10.1111/j.1469-8137.2012.04239.x |
| [40] |
Zhuo X, Zheng T, Zhang Z, et al. Genome-wide analysis of the NAC transcription factor gene family reveals differential expression patterns and cold-stress responses in the woody plant Prunus mume[J]. Genes, 2018, 9(10): 494. DOI:10.3390/genes9100494 |
| [41] |
You J, Chan Z. ROS regulation during abiotic stress responses in crop plants[J]. Front Plant Sci, 2015, 6(4): 690-695. |
| [42] |
Huang L, Hong Y, Zhang H, et al. Rice NAC transcription factor ONAC095 plays opposite roles in drought and cold stress tolerance[J]. BMC Plant Biology, 2016, 16(1): 203. |
| [43] |
An J, Li R, Qu F, et al. An apple NAC transcription factor negati-vely regulates cold tolerance via CBF-dependent pathway[J]. Journal of Plant Physiology, 2018, 221: 74-80. DOI:10.1016/j.jplph.2017.12.009 |
| [44] |
Chauhan H, Khurana N, Tyagi AK, et al. Identification and characterization of high temperature stress responsive genes in bread wheat(Triticum aestivum L.)and their regulation at various stages of development[J]. Plant Mole Biol, 2011, 75(1/2): 35-51. |
| [45] |
Smertenko A, Draber P, Viklicky V, et al. Heat stress affects the organization of microtubules and cell division in Nicotiana tabacum cells[J]. Plant Cell & Environment, 2010, 20(12): 1534-1542. |
| [46] |
Wahid A, Gelani S, Ashraf M, et al. Heat tolerance in plants:An overview[J]. Environmental & Experimental Botany, 2007, 61(3): 199-223. |
| [47] |
Guan Q, Yue X, Zeng H, et al. The protein phosphatase RCF2 and its interacting partner NAC019 are critical for heat stress - responsive gene regulation and thermotolerance in Arabidopsis[J]. Plant Cell, 2014, 26(1): 438-453. DOI:10.1105/tpc.113.118927 |
| [48] |
Guo W, Zhang J, Zhang N, et al. The wheat NAC transcription factor TaNAC2L is regulated at the transcriptional and post-translational levels and promotes heat stress tolerance in transgenic Arabidopsis[J]. PLoS One, 2015, 10(8): e135667. |
| [49] |
De Pinto MC, Locato V, De GL. Redox regulation in plant programmed cell death[J]. Plant Cell & Environment, 2012, 35(2): 234-244. |
| [50] |
Lee S, Lee H, Huh SU, et al. The Arabidopsis NAC transcription factor NTL4 participates in a positive feedback loop that induces programmed cell death under heat stress conditions[J]. Plant Science, 2014, 227: 76-83. DOI:10.1016/j.plantsci.2014.07.003 |
| [51] |
Lv X, Lan S, Guy KM, et al. Global expressions landscape of NAC transcription factor family and their responses to abiotic stresses in Citrullus lanatus[J]. Scientific Reports, 2016, 6: 30574. DOI:10.1038/srep30574 |
| [52] |
Shao H, Wang H, Tang X. NAC transcription factors in plant multiple abiotic stress responses:progress and prospects[J]. Front Plant Sci, 2015, 6: 902. |




