2. 贵州大学动物科学学院,贵阳 550025
2. College of Animal Sciences, Guizhou University, Guiyang 550025
热休克蛋白(Heatshock protein,HSP)是生物体在不良因素作用下产生的一组特殊应激蛋白[1],在分子伴侣活动、细胞分裂、信号传导及转录和翻译等过程具有调控作用[2]。此外,HSP还参与肿瘤及病毒感染调控[3],在疾病的发生中扮演重要角色。HSP70作为HSP家族的主要成员之一,是生物体中普遍存在一种高度保守的蛋白[4-5],由于其应激反应最为敏感,以及在医学临床中发挥的重要作用[6]使得HSP70成为HSP家族中研究最为深入的一种。大量研究表明,HSP70可通过介入信号转导通路来影响机体功能。如TGF-β信号在肿瘤发生早期具有抑制细胞增生的作用,过表达HSP70后对TGF-β信号通路有着明显的抑制作用[7]。除此之外,HSP70家族可以作为细胞应激的强效缓冲系统,使得肿瘤细胞很大程度上依赖这一缓冲系统生存[8]。HSPA8属于HSP70家族组成型表达的同源蛋白[9],是该家族的主要管家蛋白质,占细胞总蛋白质含量的1%[10],正常情况下低表达或不表达,应激状态下细胞内HSP编码基因被激活进行表达[11],参与细胞内蛋白的正确折叠、移位及降解等过程[12-13],维持细胞的正常形态及功能,保证细胞的稳定性。目前关于HSPA8基因及其表达产物在机体发展过程中的作用机制尚不完全清楚,需进一步深入研究。本研究通过克隆人HSPA8基因CDS区,运用在线软件对HSPA8基因序列进行生物信息学分析,构建pEGFP-C1-HSPA8重组真核表达载体,转染HEK-293T细胞,观察其亚细胞定位情况,旨在为进一步研究HSPA8的胞内功能奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株、载体及细胞pMD-19-T载体、pEGFP-C1载体分别购自TaKaRa公司和BioVector公司;JM109感受态细胞、人前列腺癌细胞和HEK-293T细胞由贵州大学高原山地动物遗传育种与繁殖教育部重点实验室保存。
1.1.2 试剂与仪器Trizol Reagent;反转录试剂盒、质粒提取物试剂盒(康为);无内毒素质粒提取试剂盒(QIAGEN);胶回收试剂盒、2×Taq PCR Master mix(康为);限制性内切酶BamH I、Kpn I及T4 DNA ligase连接酶(TaKaRa);琼脂粉、胰蛋白胨、氨苄青霉素(OXOID);DMEM细胞基础培养基、胎牛血清(FBS)、胰酶、OPti-MEM®培养基(gibco);Lipofectamine3000转染试剂盒(ThermoFisher公司);倒置荧光显微镜(Nikon)。
1.2 方法 1.2.1 引物设计根据GenBank数据库登录的人HSPA8(NM_006597.5)基因序列,利用premer pre-mer 5.0软件设计扩增HSPA8基因特异性引物,上游引物HSPA8-F:5'-GGGGTACCTCCTACAC CC CAG-CAACCATG-3',下游引物HSPA8-R:5'-CGGGATC-CGCTACATCTACACTTGGTTG GC TTA-3',上下游分别加入酶切位点Kpn I和BamH,预期扩增片段大小为1 980 bp,送上海英俊生物技术有限公司合成。
1.2.2 HSPA8基因的克隆 1.2.2.1 总RNA的提取与目的基因的扩增在超净工作台内,以人前列腺癌细胞为材料,按照Trizol说明书提取总RNA,根据康为逆转录试剂盒说明书进行反转录,以逆转录的cDNA为模板进行PCR反应。反应体系为20 µL,采用2×Taq PCR Master Mix试剂进行PCR扩增DNA目的片段,优化的PCR反应体系为cDNA模板1 µL,上下游引物各1 µL,2×Taq PCR Master Mix 10 µL,RNase-Free Water 7 µL。反应程序为:95℃预变性5 min;94℃变性30 s,63℃退火30 s,72℃延伸1 min 30 s,35个循环;72℃终延伸5 min。扩增产物用1%的琼脂糖凝胶电泳检测,如检测结果均正确,送上海英俊生物技术有限公司测序,检测其准确性,用于下一步实验。
1.2.2.2 pMD-19-HSPA8亚克隆载体的构建将PCR胶回收产物按照说明书连接pMD19-T载体,连接体系10 µL:PCR产物4 µL,p MD19-T 1 µL,Solution I 5 µL,在16℃条件下连接过夜或更长。将连接产物转化至大肠杆菌JM109感受态细胞,挑选单菌落进行扩大培养,12 h后进行菌液PCR鉴定,对鉴定为阳性的菌液进行质粒提取,利用双酶切进一步鉴定其正确性,用于下一步实验。双酶切体系为20 µL:质粒6 µL、Buffer 2 µL、限制性内切酶Kpn I和BamH I各1 µL、ddH2O 10 µL,37℃水浴锅酶切3 h,琼脂糖凝胶电泳检测酶切结果,菌液PCR鉴定和双酶切鉴定均正确的质粒,装样送上海英俊生物技术有限公司进行测序。
1.2.3 HSPA8基因的生物信息学分析利用生物信息学在线软件ProtParam、SOPMA及SWISS-MODE对HSPA8基因理化性质分析、二级结构和三级结构进行预测分析,同时利用GenBank中登录的人和动物HSPA8基因序列绘制遗传进化树,并运用在线软件STRING、PSORT Ⅱ Prediction对HSPA8相互作用蛋白和HSPA8蛋白亚细胞定位进行预测分析。
1.2.4 pEGFP-C1-HSPA8重组真核表达载体的构建以构建的T克隆载体为模板,采用Kpn I、BamH I两种限制性内切酶分别酶切pMD-19-HSPA8和pEGFP-C1两质粒,1%琼脂糖凝胶电泳检测,分别胶回收目的片段,将pEGFP-C1与pMD-19-HSPA8目的片段的胶回收产物经T4 DNA ligase 16℃过夜连接,采用含卡那霉素的LB琼脂平板筛选阳性菌落,构建pEGFP-C1-HSPA8重组真核表达载体。(构建方法同2.2亚克隆载体的构建)。
1.2.5 pEGFP-C1-HSPA8真核表达载体在HEK-293T细胞中的表达及亚细胞定位将培养至对数期的HEK-293T细胞接种于8孔细胞培养板进行培养,待细胞汇合度达到80%左右进行转染,以pEGFP-C1空载体为对照组,pEGFP-C1-HSPA8为试验组,采用脂质体转染法进行转染,置于37℃含5% CO2的细胞培养箱培养36 h后进行亚细胞定位实验。具体参照赵佳福等[14]在研究从江香猪亚细胞定位情况中实验步骤(固定、破膜、染核在暗室操作)。
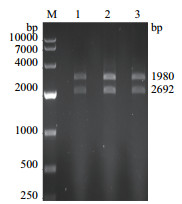
2 结果 2.1 pMD-19-HSPA8亚克隆载体鉴定结果挑取单菌落,进行菌液PCR鉴定,利用1%的琼脂糖凝胶电泳检测其正确性,结果显示,在1、2、3泳道出现1 980 bp目的条带,见图 1;提取对应菌株的质粒DNA,采用Kpn I、BamH I两种酶进行双酶切鉴定,结果显示在1、2、3泳道出现1 980 bp和2 692 bp两条亮带,结果见图 2,表明对应的菌株已成功将目的基因克隆进载体pMD-19中。

|
| 图 1 PCR扩增凝胶电泳检测结果 |
|
| 图 2 双酶切凝胶电泳检测结果 |
利用在线软件(https://web.expasy.org/protparam/)对HSPA8蛋白理化性质进行预测,结果显示人HSPA8基因的CDS区全长为1 941 bp,分子式为C3111H4998N860O994S17,蛋白的分子量为70.89 kD,共编码646个氨基酸,其中含量较高的氨基酸有赖氨酸(8.4%)、甘氨酸(8.2%)和丙氨酸(7.7%),含量较少的是组氨酸(1.1%)、胱氨酸(0.6%)和色氨酸(0.3%);带正电荷的残基总数(精氨酸+赖氨酸)82个,带负电的残基总数(天冬氨酸+谷氨酸)95个,其在哺乳动物体外网织红细胞中的半衰期为30 h,在酵母体内大于20 h,大肠杆菌体内大于10 h,理论等电点为5.37,预测HSPA8蛋白属于亲水性蛋白。
2.2.2 HSPA8蛋白二级结构分析结果在线软件SOPMA对HSPA8蛋白二级结构的预测结果显示:HSPA8蛋白二级结构丰富,其中α-螺旋占41.64%,延伸链占18.42%,β-转角占7.74%,无规则卷曲占32.20%(图 3)。

|
| 图 3 HSPA8蛋白二级结构预测 |
在线软件SWISS-MODEL对HSPA8蛋白的三级结构预测结果见图 4,该蛋白中α-螺旋和无规卷曲所占比列最高,与二级结构分析结果相符。

|
| 图 4 HSPA8蛋白三级结构预测 |
运用生物信息学软件MEGA6.0绘制人与其他动物HSPA8基因的遗传进化树。由图 5可知,人与黑猩猩的HSPA8基因位于同一分支,遗传距离最近;而与虹鳟鱼的HSPA8基因位于不同分支,且遗传距离最远。

|
| 图 5 人与其他动物HSPA8基因系统进化树 |
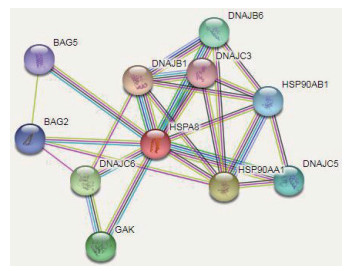
通过STRING在线软件分析与HSPA8相互作用蛋白,结果显示:HSPA8蛋白与BAG2、BAG5、DNAJB1、DNAJB6、DNAJC3、DNAJC5、DNAJC6、GAK、HSP90A1A和HSP90AB1等10种蛋白具有蛋白间相互作用(图 6)。
|
| 图 6 与HSPA8相互作用的蛋白网络 |
利用在线软件PSORT Ⅱ Prediction对HSPA8蛋白的亚细胞定位,结果显示:HSPA8蛋白的细胞定位在细胞质中占65.2%,细胞核占30.4%,内质网占4.4%。
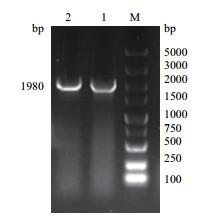
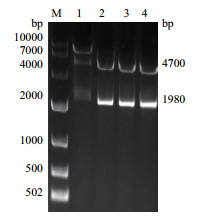
2.3 pEGFP-C1-HSPA8重组质粒鉴定结果经酶切、连接、转化后,挑取单菌落,进行菌液PCR鉴定,结果如图 7,在泳道1-2出现1 980 bp的亮带,提取对应泳道质粒进行双酶切鉴定(图 8),2个酶切产物检测均在1 980 bp和4 700 bp左右出现单一亮带,表明pEGFP-C1-HSPA8真核表达载体已构建成功。
|
| 图 7 菌液PCR凝胶电泳检测结果 |
|
| 图 8 双酶切凝胶电泳检测结果 |
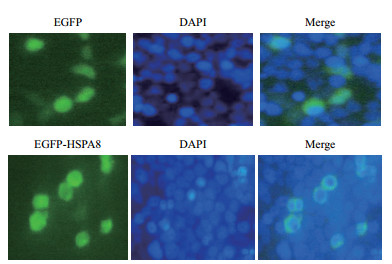
通过荧光共定位检测发现(图 9),HSPA8蛋白在细胞质与细胞核中均有表达,均匀分布于整个细胞。
|
| 图 9 瞬时转染 HEK-293T 细胞荧光显微镜观察 |
大量研究表明,HSPA8与诸多细胞功能存在联系。在哺乳动物细胞质和细胞核中,伴侣辅助因子通过调节HSPA8活性来调控ATP的结合与水解,如抗凋亡蛋白BAG-1依赖HSP40加速刺激HSPA8对ATP的水解,推动ATP与ADP之间的循环[14],HSPA8的另一主要细胞功能是参与蛋白质折叠,不仅结合未折叠的新合成蛋白质,还与各种蛋白质聚集体相互作用并减少其形成[15]。此外,HSPA8在细胞蛋白质降解过程中发挥重要作用,如在ACT(肌动蛋白),CRYA(晶状体蛋白)或HIS2H2AC / H2A进行泛素化及其随后的蛋白酶体降解,都需要HSPA8的参与[16]。随着HSPA8的深入研究,发现HSPA8蛋白与多种疾病蛋白存在相互作用,如博尔纳病毒蛋白X、乳头瘤病毒蛋白L229和流感病毒蛋白M1等,这些相互作用与其核质穿梭特性相结合,使HSPA8分子伴侣在病毒感染过程中发挥关键作用。α-突触核蛋白在退化的多巴胺神经元中积累是帕金森病(Parkinson’s disease,PD)的主要致病蛋白,而HSPA8能有效地分离α-突触核蛋白,并促进解聚成无毒的α-突触核蛋白单体[17]。甲基转移酶METTL21A能够甲基化HSPA8蛋白,且改变HSPA8对帕金森病相关蛋白α-突触核蛋白的单体和纤维形式的亲和力,而HSPA8的C末端尾部中的靶赖氨酸的突变消除了它们的甲基化[18]。因此HSPA8活性对预防PD病理学至关重要。近年一些研究报道,HSPA8对肿瘤与癌细胞的扩散与抑制存在一定联系,HSPA8能够抑制肿瘤细胞生长并改变细胞活力[19],这对于癌症治疗的药物靶标具有重要意义。如山楂酸(Maslinic acid,MA)是一种天然五环三萜,对各种类型的癌细胞都具有细胞毒活性,MA能够通过下调Panc-28细胞中的HSPA8诱导自噬[20]。
本研究克隆人HSPA8基因CDS区,序列比对分析发现人与黑猩猩的同源性最高(99%),这与HSPA8基因遗传进化树结果相一致,说明人与黑猩猩亲源关系最近。HSPA8蛋白分析发现,HSPA8蛋白存在N-末端核苷酸结合结构域(Nucleotide-binding domain,NBD)和C-末端底物结合结构域(Substrate-binding domain,SBD),两个结构域是变构偶联的,NBD负责结合和水解ATP,SBD与底物蛋白结合;HSPA8蛋白相互作用预测显示,与之作用的蛋白共10种,主要包括J蛋白家族与BAG家族。BAG家族多个成员作为HSPA8伴侣蛋白,促进HSP70和HSC70蛋白释放ADP的核苷酸交换因子(Nucleotide exchange factor,NEF),从而触发底物蛋白释放,通过与HSPA8的核苷酸结合结构域的结合介导核苷酸释放,与底物结构域结合介导底物释放[21]。如BAG1和BAG3的BAG结构域和HSPA8 NBD之间的相互作用介导核苷酸的释放,从而加速HSPA8中ADP释放[22]。J蛋白家族成员主要通过J结构域与HSPA8结合。在预测的蛋白中共有4个J蛋白家族成员,值得注意的是DNAJC6在早发性PD中存在突变[23],是否暗示DNAJC6与HSPA8相互作用对PD具有重要调控作用。除了预测的成员外,在线人类孟德尔遗传(Online mendelian inheritance in man,OMIM)数据库中发现,HSPA8还与其他大量的J蛋白家族成员存在相互作用,如DNAJB2、DNAJC21和DNAJC7等;蛋白亚细胞定位预测显示,人HSPA8蛋白主要定位在细胞质(65.2%),通过HSPA8蛋白与EGFP融合表达,发现该蛋白均匀分布于整个细胞,这与预测结果存在差异,但该结果与Rashedan等[24]研究一致。进一步通过癌症基因组(The cancer genome atlas,TCGA)计划数据库发现,人HSPA8蛋白主要分布在细胞膜与细胞核,这与本实验结果相符。同时在TCGA数据库中发现HSPA8与DNAJC21、HDJC9、DNAJB12和DNAJB14存在相互作用,其中DNAJC21和HDJC9主要定位在细胞核,通过与HSPA8相互作用来维持细胞的正常形态与功能[25-26];DNAJB12和DNAJB14是内质网中的跨膜蛋白,其作为HSPA8蛋白的伴侣蛋白,可将HSPA8招募到内质网膜,对膜蛋白中错误折叠的蛋白进行降解[27]。这些研究表明HSPA8通过定位至细胞核与细胞膜来行使功能,进一步验证HSPA8蛋白定位于细胞膜与细胞核。此外,研究发现,HSPA8蛋白在热诱导的应激过程中被抑制,HSPA8蛋白被限制在细胞核内[28],表明HSPA8可在细胞核与细胞质间穿梭,该过程受何种蛋白调控,以及限制在核内行使的功能还需进一步研究。本研究成功克隆人HSPA8基因CDS区,生物信息学分析和亚细胞定位结果为今后深入研究该基因的生物学功能及全面了解HSPA8在细胞内的作用奠定基础。
4 结论成功克隆了人HSPA8基因CDS区,并对序列进行生物信息学分析。构建了pEGFP-HSPA8真核表达载体,亚细胞定位研究发现HSPA8-GFP融合蛋白均匀地分布在293-T细胞中。
| [1] |
胡晓燕, 曲音波. 热休克蛋白的研究进展[J]. 生命科学, 1999(S1): 55-59. |
| [2] |
Stricher F, Macri C, Ruff M, et al. HSPA8/HSC70 chaperone protein:structure, function, and chemical targeting[J]. Autophagy, 2013, 9(12): 1937-1954. DOI:10.4161/auto.26448 |
| [3] |
尚进才, 王亚洲, 高岩, 等. 热休克蛋白的研究进展[J]. 医学理论与实践, 2010, 23(5): 527-529. DOI:10.3969/j.issn.1001-7585.2010.05.013 |
| [4] |
唐雪峰, 李建柱, 赵存真, 等. 热休克蛋白70生物学功能及在家禽组织中的表达[J]. 畜牧与兽医, 2018, 50(4): 144-148. |
| [5] |
耿俊楠. HSPA1A和HSPA8在分娩中的功能研究[D].武汉: 华中农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10504-1016156282.htm
|
| [6] |
刘嘉敏, 赵元莙. HSP70的研究进展及其在生物医学中的应用[J]. 教育教学论坛, 2017, 65(50): 61-62. DOI:10.3969/j.issn.1674-9324.2017.50.028 |
| [7] |
李一豪. HSP70在TGF-β信号转导中的作用[D].保定: 河北大学, 2011. http://cdmd.cnki.com.cn/article/cdmd-10075-1011290856.htm
|
| [8] |
刘宗亮, 张仁梅, 孟庆国. 以热休克蛋白HSP70为靶点的药物研究进展[J]. 中国药科大学学报, 2017, 48(4): 416-424. |
| [9] |
Kampinga HH, Hageman J, Vos MJ, et al. Guidelines for the nomenclature of the human heat shock proteins[J]. Cell Stress & Chaperones, 2009, 14(1): 105-111. |
| [10] |
Stricher F, Macri C, Ruff M, et al. HSPA8/HSC70 chaperone protein[J]. Autophagy, 2013, 9(12): 1937-1954. DOI:10.4161/auto.26448 |
| [11] |
徐小娜, 曲彦. 热休克蛋白70重组腺病毒载体的构建及鉴定[J]. 青岛大学医学院学报, 2008, 44(2): 162-164. |
| [12] |
Chappell TG, Welch WJ, Schlossman DM, et al. Uncoating ATPase is a member of the 70 kilodalton family of stress proteins[J]. Cell, 1986, 45(1): 3-13. DOI:10.1016/0092-8674(86)90532-5 |
| [13] |
Thulasiraman V, Yang CF, Frydman J. In vivo newly translated polypeptides are sequestered in a protected folding environment[J]. Embo Journal, 1999, 18(1): 85-95. DOI:10.1093/emboj/18.1.85 |
| [14] |
Lüders J, Demand J, Papp O, et al. Distinct isoforms of the cofactor BAG-1 differentially affect Hsc70 chaperone function[J]. Journal of Biological Chemistry, 2000, 275(20): 14817-14823. DOI:10.1074/jbc.275.20.14817 |
| [15] |
Strickland E, Qu BH, Millen L, et al. The molecular chaperone hsc70 assists the in vitro folding of the n-terminal nucleotide-binding domain of the cystic fibrosis transmembrane conductance regulator[J]. Journal of Biological Chemistry, 1997, 272(41): 25421-25424. DOI:10.1074/jbc.272.41.25421 |
| [16] |
Stancovski I. Ubiquitin-dependent degradation of certain protein substrates in vitro requires the molecular chaperone Hsc70[J]. Journal of Biological Chemistry, 1997, 272(14): 9002-9010. DOI:10.1074/jbc.272.14.9002 |
| [17] |
Sala G, Marinig D, Riva C, et al. Rotenone down-regulates HSPA8/hsc70 chaperone protein in vitro:A new possible toxic mechanism contributing to Parkinson's disease[J]. Neurotoxicology, 2016, 54: 161-169. DOI:10.1016/j.neuro.2016.04.018 |
| [18] |
Jakobsson ME, Moen A, Bousset L, et al. Identification and characterization of a novel human methyltransferase modulating Hsp70 protein function through lysine methylation[J]. Journal of Biological Chemistry, 2013, 288(39): 27752-27763. DOI:10.1074/jbc.M113.483248 |
| [19] |
Nirdé P, Derocq D, Maynadier M, et al. Heat shock cognate 70 protein secretion as a new growth arrest signal for cancer cells[J]. Oncogene, 2010, 29(1): 117-127. DOI:10.1038/onc.2009.311 |
| [20] |
Tian Y, Xu H, Farooq AA, et al. Maslinic acid induces autophagy by down-regulating HSPA8 in pancreatic cancer cells[J]. Phytotherapy Research, 2018, 32(7): 1320-1331. DOI:10.1002/ptr.6064 |
| [21] |
Lee DW, Kim SJ, Oh YJ, et al. Arabidopsis BAG1 functions as a cofactor in Hsc70-mediated proteasomal degradation of unimported plastid proteins[J]. Molecular Plant, 2016, 9(10): 1428-1431. DOI:10.1016/j.molp.2016.06.005 |
| [22] |
Rauch JN, Zuiderweg ERP, Gestwicki JE. Non-canonical interactions between heat shock cognate protein 70(Hsc70)and Bcl2-associated anthanogene(BAG)co-chaperones are important for client release[J]. Journal of Biological Chemistry, 2016, 291(38): 19848-19857. DOI:10.1074/jbc.M116.742502 |
| [23] |
Olgiati S, Quadri M, Fang M, et al. DNAJC6 mutations associated with early-onset Parkinson's disease[J]. Annals of Neurology, 2016, 79(2): 244-256. DOI:10.1002/ana.24553 |
| [24] |
Rashedan M. Characterization of the localization of heat-shock proteins, HSPA8 and HSPA5, in mammalian cells[J]. Dissertations & Theses- Gradworks, 2013, 36(2): 58-64. |
| [25] |
Han C, Chen T, Li N, et al. HDJC9, a novel human type C DnaJ/HSP40 member interacts with and cochaperones HSP70 through the J domain[J]. Biochemical and Biophysical Research Communications, 2007, 353(2): 280-285. DOI:10.1016/j.bbrc.2006.12.013 |
| [26] |
Tummala H, Walne AJ, Williams M, et al. DNAJC21 mutations link a cancer-prone bone marrow failure syndrome to corruption in 60S ribosome subunit maturation[J]. American Journal of Human Genetics, 2016, 99(1): 115-124. DOI:10.1016/j.ajhg.2016.05.002 |
| [27] |
Yo-Hei Y, Taiji K, Shuku M, et al. A novel ER J-protein DNAJB12 accelerates ER-associated degradation of membrane proteins including CFTR[J]. Cell Structure and Function, 2010, 35(2): 107-116. DOI:10.1247/csf.10023 |
| [28] |
Kodiha M. Stress inhibits nucleocytoplasmic shuttling of heat shock protein hsc70[J]. Am J Physiol Cell Physion, 2005, 289(4): C1034-C1041. DOI:10.1152/ajpcell.00590.2004 |




