2. 昆山市畜牧兽医站,昆山 215300;
3. 江苏京海禽业集团有限公司,海门 226100
2. Animal Husbandry and Veterinary Station of Kunshan City, Kunshan 215300;
3. Jiangsu Jinghai Poultry Group Co. Ltd, Haimen 226100
鸡球虫病是由一种或多种球虫侵入鸡的肠道上皮细胞而引起的急性流行性寄生虫病,其中以寄生于盲肠的柔嫩艾美尔球虫(E. tenella)是最为严重的球虫病之一[1-2]。据统计每年全球因球虫病造成的经济损失达30多亿美元[2]。目前球虫病的预防主要通过严格饲养管理、添加抗球虫药物和接种活虫疫苗来实现[3]。药物预防和活疫苗接种会造成药物残留和引发养殖风险,不能从根本上解决问题[4]。在分子水平上寻找与E. tenella感染相关的差异基因或主效基因,通过遗传选育培育抗病品种将是解决这一问题的有效途径。Chen等[5]通过转录组分析发现E. tenella的苹果酸脱氢酶高表达(EtMDH)可能与E. tenella的发育和侵袭过程中的耐药性有关。Guo等[6]通过鸡E.tenella体内感染4.5 d盲肠上皮细胞感染组和非感染组的转录组基因芯片杂交分析发现,上调或下调的差异表达基因参与了免疫应答、细胞凋亡、免疫球蛋白的生成等过程。Xu等[7]和Zhou等[8]分别对鸡盲肠微生物16S rRNA基因高变区进行高通量测序以探讨盲肠微生物种类和组成。
京海黄鸡是经国家遗传资源委审定的优质肉鸡新品种,具有小型、优质、早熟、抗逆等突出特点。本试验以京海黄鸡为试验材料,采用RNA-seq技术筛选E. tenella感染和非感染组盲肠组织差异表达基因,旨为后续进一步分析差异基因在抵抗球虫感染过程中相互作用的分子机制,以及研究与球虫感染相关联差异表达基因的遗传标记,培育球虫病抗性新品种或新品系提供一定的参考依据。
1 材料与方法 1.1 材料随机选择体重接近的健康半同胞1日龄京海黄鸡母雏鸡(购自江苏省海门京海黄鸡资源场)12只,在无球虫环境中用无抗饲料隔离笼养至30日龄。试鸡经粪便检测均无球虫卵囊感染后,饲养于经汽油喷灯火焰消毒的无球虫单笼中,随机均分为2组,感染组每只鸡经口注射2.5万个E. tenella孢子化卵囊,对照组则接种等量生理盐水。感染后第7天,选取感染组盲肠病变记分最高的3只鸡以及对照组3个个体。按照Trizol RNA提取试剂盒说明书,分别提取6个样品的总RNA,感染组记为JS1-JS3,非感染组记为JC1-JC3。经浓度、纯度、完整性检测合格,且无降解、无污染的总RNA用于转录组测序。
用于感染的E. tenella孢子化卵囊由扬州大学兽医学院寄生虫教研室,经非球虫疫苗免疫的健康鸡体内传代一次后收集获得(方法参考戴国俊等[9]),于4℃保存备用。
1.2 方法 1.2.1 cDNA文库构建及Illumina测序检测合格的6个盲肠组织总RNA送至北京诺禾致源生物信息科技有限公司(干冰保存)进行文库构建及转录组测序。通过带有Oligo(dT)的磁珠富集、纯化mRNA。以mRNA为模板合成cDNA。按照标准的Illumina建库流程对每个测序样本的cDNA构建小片段测序文库。采用2×100 pair-end模式在Illumina HiSeq2000测序平台进行测序。
1.2.2 原始数据处理与分析测序得到的原始图像数据文件经碱基识别分析转化为原始测序序列(Raw reads)。为了保证数据分析的精准度,测定Q20、GC含量和重复序列水平等质量参数,去除带接头、低质量、N(N表示无法确定碱基信息)比例大于10%的测序序列(Reads)后得到的序列(Clean reads)用于后续分析。从基因组网站下载(ftp://ftp.ensembl.org/pub/release-75/fasta/gallus gallus/dna/)参考基因组和基因注释模型。运用Bowtie v2.0.6软件构建参考基因组指数[10],并用TopHat v2.0.9软件[11]将上述clean reads比对到参考基因组。采用HTSeq软件统计各样品定位到基因组的reads的数量[12]。
1.2.3 差异表达基因的筛选差异基因的表达量通过RPKM(Reads per kilo bases per million reads)均一化处理后,用DESeq2软件筛选感染和对照组的差异表达基因[13],筛选的标准为:FC(fold change)≥2且padj(校正后的P值) < 0.05。P值的矫正参考Benjamini-Hochberg所建方法[14]。
1.2.4 差异表达基因的GO和KEGG富集分析采用GOseq进行功能富集分析[15],将校正的P值小于0.05的差异表达基因显著富集到各GO分类(term)。采用KOBAS(2.0)软件进行通路富集分析[16],差异表达基因经与KEGG数据库比对,统计各个KEGG通路各层级的差异表达基因数量,并确定差异表达基因主要参与的代谢途径和信号通路。
1.2.5 差异表达基因的qRT-PCR验证随机选取10个差异表达基因,使用NCBI在线引物设计软件Primer-Blast设计引物(上下游引物跨越外显子-外显子边界),基因名称及引物序列等信息见表 1,其中YWHAZ和TBP内参基因根据文献[17]选择。按照SYBR Green(中国大连生物公司)和ABI 7500 SDS系统(Applied Biosystems,USA)提供的qRT-PCR方法进行RNA-seq结果的可靠性验证,每个样品重复3次。qRT-PCR反应体系与条件参考Liu等[18]的方法,相对表达量采用2-ΔΔCt法计算。
接种后1-3 d,各组鸡无特殊症状且与感染前无差异;第4天,感染组鸡开始排血粪;第5天排血粪最多;第6天排血粪开始减少。病鸡精神萎靡,废食,肛门周围羽毛污秽,呈共济失调,两翅下垂,出现麻痹、痉挛等症状,感染组试验期内未出现个体死亡情况。第7天剖检病鸡,部分见盲肠肿胀发黑,形成肠芯。非感染对照组试验鸡精神状态良好,生长状态和正常饲养鸡无差异。
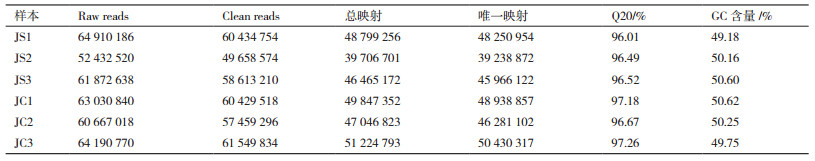
2.2 RNA转录组测序数据检测质量转录本测序结果及质量参数见表 2。由表可见,每个样本100个碱基(bp)长度的raw reads的数量在52 432 520到64 910 184之间,说明丰度高;GC碱基的百分含量在49.18%-50.62%之间,说明各样本的GC含量较为一致;质量值Q20在96%以上,表明RNA-seq测序数据的数量和质量均较高,可以用于后续分析。为了排除接头和低质量reads对转录信息分析的干扰,6个样本去除带接头、低质量和N(N表示无法确定碱基信息)比例大于10%的reads后得到34.8 Gb的clean reads。共有80%的clean reads比对到鸡参考基因组上,其中78%是有唯一比对位置的reads。
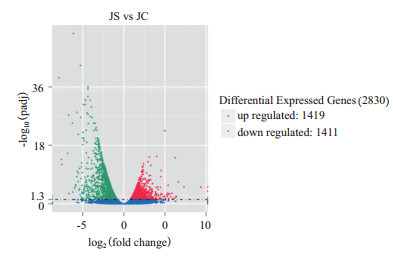
从差异倍数(Fold change)和显著水平(P value)两个水平进行差异表达基因的评估筛选,将差异基因的表达倍数在2倍以上,显著水平padj < 0.05确定为表达有显著差异基因时,由图 1可见,E. tenella感染组和非感对照组共有2 830个差异表达基因,其中表达上调的基因有1 419个,下调的基因有1 411个。当差异基因的表达倍数在5倍以上,显著水平仍为padj < 0.05时,筛选到的差异表达基因共有57个,其中表达上调的基因有23个,下调的基因有34个。
|
| 红点、绿点分别表示显著上调和下调的差异基因(padj < 0.05);蓝点表示没有达显著水平的基因(padj >0.05) 图 1 感染组与对照组差异表达基因火山图 |
GO分析表明,共有83.25%(2 356个)差异基因获得GO功能注释,差异表达基因富集的GO terms共有11 236个,其中显著富集的有545个,涉及生物过程(Biological process),细胞成分(Cellular component),分子功能(Molecular function)3大类。图 2为前30条显著富集的GO terms。由图可见,差异基因显著富集的与生物过程有关的GO terms数量最多,达21个,其中富集最多的是细胞交流(GO:0007154),富集最少的是血管发育(GO:0001568)。其次为与细胞成分有关的terms有8个,差异基因富集最多的是膜(GO:0016020),富集最少的是线粒体内膜(GO:0005743),在分子功能方面,差异表达基因显著富集的Term只有1个,其功能与氧化还原酶活性(GO:0016491)有关。
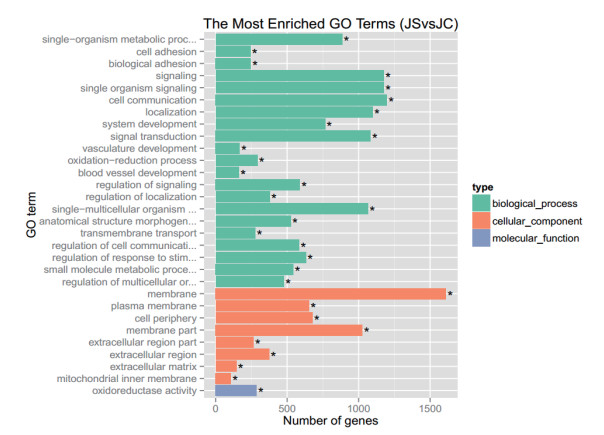
|
| *指被显著富集的GO功能类别(校正P值<0.05) 图 2 显著富集的前30个GO类别 |
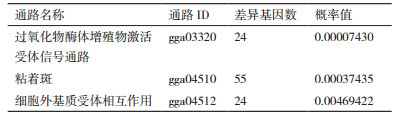
KEGG差异表达基因显著富集的前20条通路见图 3。其中最为显著的通路有3条,分别是过氧化物酶体增殖物激活受体信号通路(PPAR signaling pathway,ID:gga03320)、黏着斑通路(Focal adhesion,ID:gga04510)和细胞外基质受体相互作用(ECM-receptor interaction,ID:gga04512),富集的差异基因分别有24、55和24个,3个通路中与免疫反应有关的基因有血管生成素样蛋白4(ANGPTL4),长链脂酰辅酶A合成酶5(ACSL5),血管内皮细胞生长因子C(VEGFC),促分裂素原活化蛋白激酶10(MAKP10)和白细胞分化抗原分化簇44(CD44)等,相关信息见表 3。
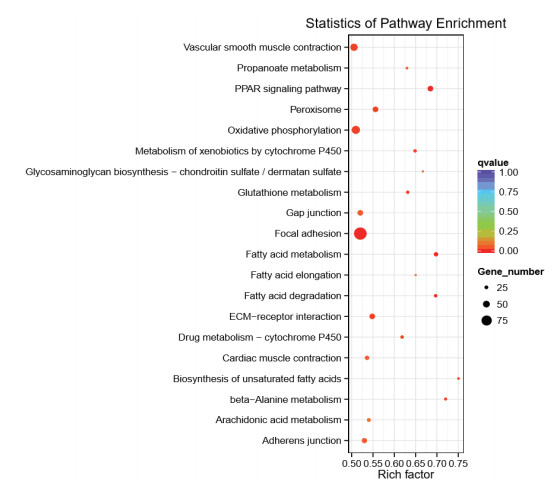
|
| 圆点的大小表示通路中差异表达基因的数量。点的颜色对应不同的q值(校正后的P值<0.05) 图 3 前20位显著富集的功能性KEGG通路 |
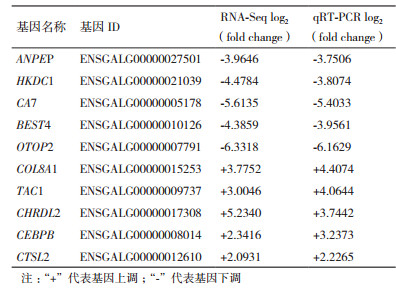
在不同倍性(2-7倍)改变的差异基因中挑选上调和下调的差异基因各5个,以YWHAZ和TBP作为内参[17],运用荧光定量PCR验证RNA-seq结果的可靠性。10个差异表达基因验证结果见表 4,所选10差异表达基因2种不同检测方法的上调与下调倍数经相关分析,相关系数达到了0.988,相关极显著(P < 0.000),决定系数R2=0.975。荧光定量测定数据进一步验证了转录组测序结果的可靠性。
鸡球虫病是一种严重危害养殖业的流行性急性寄生虫病,给养殖业造成巨大的经济损失[2]。传统预防球虫的方法存在很大的弊端,从分子水平上寻找球虫感染有关的基因,用于抗病育种,提高群体的免疫抗性十分重要。本研究对鸡E. tenella感染组和非感对照组盲肠组织的转录组文库进行分析,发现存在2 830个差异基因,其中上调基因1 419个,基因下调1 411个;与球虫感染相关的免疫基因包括ANGPTL4、ACSL5、VEGFC、CD44和MAKP10等。主要通路有过氧化物酶体通路、黏着斑通路、细胞外基质受体相互作用,研究结果为从分子应答过程和免疫应答机制方面研究E. tenella的感染提供了依据。
过氧化物酶体增殖物激活受体(PPARS)属于细胞核受体超家族,主要功能是将环境、营养或炎症等刺激转化为细胞内信号,从而发挥重要的信使作用[19]。Daynes等[20]研究表明PPARS对调节免疫反应非常重要,包括调节树突状细胞和T细胞因子的产生和淋巴细胞的增殖。此外,Gou等[21]表明PPARS在细胞凋亡与肿瘤的发生发展中起重要的调节作用。该通路中的差异表达基因ANGPTL4是ANGPTL家族重要成员之一,它可以调节血管通透性和活性氧水平,促进血管生成和伤口愈合[22-23]。研究表明,ANGPTL4可增强内皮细胞间的连接,维持血管完整性,控制炎症反应[24]。此外,Georgiadi等[25]研究发现ANGPTL4过表达在小鼠动脉粥样硬化发展中具有抗炎作用。ACSL5是一种线粒体定位酶,参与细胞发育、凋亡以及肿瘤的发生[26]。有研究表明ACSL5可能在狼疮样小鼠模型的免疫功能障碍中起重要作用[27]。
黏着斑通路是细胞间或者细胞与细胞外基质之间连接的纽带,控制细胞的生长、分化、扩散、黏附、迁移及凋亡等行为[28],鸡感染E. tenella后,盲肠组织会发生严重损伤,引发宿主一系列的生理反应以抵抗球虫对肠道的破坏,此过程涉及到盲肠上皮屏障的紧缩,其原因可能是通过粘着斑通路加强上皮细胞和细胞外基质之间的相互作用来完成[29]。该通路上的一个差异表达基因VEGFC作为重要的淋巴管生长因子,主要参与淋巴管的生成,在动物的免疫防御以及肿瘤转移中起重要的作用[30]。有研究表明,VEGFC能够促进人类多种恶性肿瘤中淋巴系统的生成和转移[31],Küchler等[32]研究表明,斑马鱼淋巴系统的生长发育依赖于VEGFC的表达。本研究发现E. tenella感染组中VEGFC的表达量显著上调,可能参与了鸡淋巴系统的生成以增强免疫防御。该通路的另一个差异表达基因MAKP10(JNK3)在哺乳动物中具有重要作用,它是哺乳动物体内丝裂原活化蛋白激酶家族的重要一员,且JNK3主要在脑、心脏、睾丸等组织中大量表达[33],Ying等[34]研究表明MAPK10基因在多种癌细胞中低表达,具有抑制肿瘤的发生和发展的功能。但MAPK10基因在禽类中它的主要表达组织及在禽类免疫中的作用尚不明确,有待进一步研究。
细胞外基质(ECM)是由结构大分子和功能大分子组成的复杂混合物,在维持细胞、组织的结构和发挥细胞、组织功能中起重要作用[35]。研究证明,ECM是通过多种跨膜分子(主要是整合素)直接或间接控制细胞活动,如细胞的粘附、迁移、分化、增殖和凋亡[36]。本研究在该通路上筛选到的与免疫有关的其中一个差异表达基因为CD44。CD44是透明质酸受体同时也是细胞表面的一种糖蛋白,参与细胞与细胞的相互作用,细胞粘附、迁移和侵袭[37]。DeGrendele等[38]研究表明T细胞与抗体结合可诱导CD44-HA的生成,促进了炎症部位淋巴细胞的外渗,具有增强免疫功能的作用。Protin等[39]通过对CD44缺陷小鼠和正常组小鼠进行生长发育分析,表明CD44在成年动物的胸腺和周围淋巴结的转运中起重要作用。本研究在鸡柔嫩艾美尔球虫感染和非感染的盲肠组织转录组测序差异表达基因分析中发现CD44的表达差异显著,可能该基因在鸡球虫的感染中有重要作用。
4 结论本研究采用RNA-seq技术对鸡E. tenella感染组与对照组组织的转录组进行测序分析,共有2 830个差异表达基因,其中表达上调的基因有1 419个,下调的基因有1 411个。GO分析表明,共有83.25%(2 356个)差异基因获得GO功能注释。KEGG通路富集分析发现,差异基因显著富集的信号通路有黏着斑、细胞外基质-受体相互作用、过氧化物酶体增殖物激活受体。这些通路中的差异基因有ANGPTL4、ACSL5、VEGFC、CD44和MAKP10,这些基因在宿主柔嫩艾美耳球虫感染过程中发挥重要作用。
| [1] |
林雨鑫, 张菁菁, 戴国俊, 等. 京海黄鸡杂交配套系亲本对柔嫩艾美耳球虫的敏感性[J]. 中国兽医学报, 2015, 35(7): 1074-1078. |
| [2] |
钱根林, 李健, 樊彦红, 等. 鸡球虫疫苗研究进展[J]. 上海畜牧兽医通讯, 2013(2): 28-29. DOI:10.3969/j.issn.1000-7725.2013.02.012 |
| [3] |
王诗琴, 辛世杰, 王晓慧, 等. IL-6基因启动子区单核苷酸多态对京海黄鸡柔嫩艾美尔球虫抗性指标的影响[J]. 扬州大学学报:农业与生命科学版, 2018, 39(1): 36-41. |
| [4] |
Cong F, Liu X L, Han Z, et al. Transcriptome analysis of chicken kidney tissues following coronavirus infectious bronchitis virus infection[J]. BMC Genomics, 2013, 14: 743-756. DOI:10.1186/1471-2164-14-743 |
| [5] |
Chen T, Huang B, Zhao Q, et al. Molecular characterization and functional analysis of Eimeria tenella malate dehydrogenase[J]. Parasitology Research, 2018, 117(1): 1-11. |
| [6] |
Guo A, Cai J, Gong W, et al. Transcriptome analysis in chicken cecal epithelia upon infection by Eimeria tenella in vivo[J]. PLoS One, 2013, 8(5): e64236. DOI:10.1371/journal.pone.0064236 |
| [7] |
Xu Y, Yang H, Zhang L, et al. High-throughput sequencing technology to reveal the composition and function of cecal microbiota in Dagu chicken[J]. BMC Microbiology, 2016, 16(1): 259. DOI:10.1186/s12866-016-0877-2 |
| [8] |
Zhou X, Jiang X, Yang C, et al. Cecal microbiota of Tibetan Chickens from five geographic regions were determined by 16S rRNA sequencing[J]. Microbiologyopen, 2016, 5(5): 753-762. DOI:10.1002/mbo3.367 |
| [9] |
戴国俊, 孙明明, 张菁菁, 等. 鸡柔嫩艾美尔球虫病抗性主成分分析评估模型的建立[J]. 畜牧兽医学报, 2015, 46(3): 467-475. |
| [10] |
Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [11] |
Trapnell C, Pachter L, Salzberg SL. TopHat:discovering splice junctions with RNA-Seq[J]. Bioinformatics, 2009, 25(9): 1105-1111. DOI:10.1093/bioinformatics/btp120 |
| [12] |
Anders S, Pyl PT, Huber W. HTSeq-a Python framework to work with high-throughput sequencing data[J]. Bioinformatics, 2015, 31(2): 166-169. |
| [13] |
Love MI, Simon A, Vladislav K, et al. RNA-Seq workflow:gene-level exploratory analysis and differential expression[J]. F1000Res, 2015, 4: 1070. DOI:10.12688/f1000research.7035.1 |
| [14] |
Benjamini Y, Hochberg Y. Controlling the false discovery rate:a practical and powerful approach to multiple testing[J]. J Royal Statistical Soc Series B, 1995, 57: 289-300. |
| [15] |
Young MD, Wakefield MJ, Smyth GK, et al. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biol, 2010, 11(2): R14. DOI:10.1186/gb-2010-11-2-r14 |
| [16] |
Mao X, Cai T, Olyarchuk JG, et al. Automated genome annotation and pathway identification using the KEGG Orthology(KO)as a controlled vocabulary[J]. Bioinformatics, 2005, 21(19): 3787-3793. DOI:10.1093/bioinformatics/bti430 |
| [17] |
Yue H, Lei XW, Yang FL, et al. Reference gene selection for normalization of PCR analysis in chicken embryo fibroblast infected with H5N1 AIV[J]. Virol Sin, 2010, 25(6): 425-431. DOI:10.1007/s12250-010-3114-4 |
| [18] |
Liu Z, Ji T, Yin L, et al. Transcriptome sequencing analysis reveals the regulation of the hypopharyngeal glands in the Honey Bee, Apis mellifera carnica Pollmann[J]. PLoS One, 2013, 8(12): e81001. DOI:10.1371/journal.pone.0081001 |
| [19] |
王静.鞘脂与PPAR相互作用及其对癌细胞凋亡调控的影响[D].北京: 中国农业大学, 2004. http://cdmd.cnki.com.cn/article/cdmd-10019-2004077750.htm
|
| [20] |
Daynes RA, Jones DC. Emerging roles of ppars in inflammation and immunity[J]. Nature Reviews Immunology, 2002, 2(10): 748-759. DOI:10.1038/nri912 |
| [21] |
Gou Q, Gong X, Jin L, et al. Peroxisome proliferator-activated receptors(PPARs)are potential drug targets for cancer therapy[J]. Oncotarget, 2017, 8(36): 60704-60709. |
| [22] |
Zhu P, Goh YY, Chin HFA, et al. Angiopoietin-like 4:a decade of research[J]. Biosci Rep, 2012, 32(3): 211-219. DOI:10.1042/BSR20110102 |
| [23] |
Shafik NM, Mohamed DA, Bedder AE, et al. Significance of tissue expression and serum levels of angiopoietin-like protein 4 in breast cancer progression:link to NF-κB /P65 activity and pro-Inflammatory cytokines[J]. Asian Pac J Cancer Prev, 2015, 16(18): 8579-8587. |
| [24] |
Bouleti C, Mathivet T, Coqueran B, et al. Protective effects of angiopoietin-like 4 on cerebrovascular and functional damages in ischaemic stroke[J]. European Heart Journal, 2013, 34(47): 3657-3668. DOI:10.1093/eurheartj/eht153 |
| [25] |
Georgiadi A, Wang Y, Stienstra R, et al. Overexpression of angiopoietin-like protein 4 protects against atherosclerosis development[J]. Arteriosclerosis, Thrombosis, and Vascular Biology, 2013, 33(7): 1529-1537. DOI:10.1161/ATVBAHA.113.301698 |
| [26] |
Klaus C, Kaemmerer E, Reinartz A, et al. TP53 status regulates ACSL5-induced expression of mitochondrial mortalin in enterocytes and colorectal adenocarcinomas[J]. Cell & Tissue Research, 2014, 357(1): 267-278. |
| [27] |
Catalá-Rabasa A, Dorothy N, Mario SJ, et al. High ACSL5 transcript levels associate with systemic lupus erythematosus and apoptosis in jurkat T lymphocytes and peripheral blood cells[J]. PLoS One, 2011, 6(12): e28591. DOI:10.1371/journal.pone.0028591 |
| [28] |
张冠华, 智发朝. 黏着斑的结构、功能及在肿瘤转移中作用[J]. 现代消化及介入诊疗, 2015(2): 174-177. DOI:10.3969/j.issn.1672-2159.2015.02.038 |
| [29] |
Gay CG, Pastoret PP, Pinard-van DL. Animal genomics for animal health preface[J]. Developments in Biologicals, 2008, 132(4): 13. |
| [30] |
牛虹, 周浩本, 刘怀民, 等. 微小RNA-507靶向调控VEGFC表达及对肝癌细胞生物学行为和PI3K/Akt通路的影响[J]. 临床肿瘤学杂志, 2018, 23(8): 673-679. DOI:10.3969/j.issn.1009-0460.2018.08.001 |
| [31] |
Chaudary N, Milosevic M, Hill RP. Suppression of vascular endothelial growth factor receptor 3(VEGFR3)and vascular endothelial growth factor C(VEGFC)inhibits hypoxia-induced lymph node metastases in cervix cancer[J]. Gynecologic Oncology, 2011, 123(2): 393-400. DOI:10.1016/j.ygyno.2011.07.006 |
| [32] |
Küchler AM, Gjini E, Peterson-Maduro J, et al. Development of the zebrafish lymphatic system requires vegfc signaling[J]. Current Biology, 2006, 16(12): 1244-1248. DOI:10.1016/j.cub.2006.05.026 |
| [33] |
汤丽平.候选抑癌基因MAPK10在原发性肝细胞肝癌中表达的表观遗传学调控机制及临床意义研究[D].重庆: 重庆医科大学, 2012.
|
| [34] |
Ying J, Li H, Cui Y, et al. Epigenetic disruption of two proapoptotic genes MAPK10/JNK3 and PTPN13/FAP-1 in multiple lymphomas and carcinomas through hypermethylation of a common bidirectional promoter[J]. Leukemia, 2006, 20(6): 1173-1175. DOI:10.1038/sj.leu.2404193 |
| [35] |
Lee HJ, Jang M, Kim H, et al. Comparative transcriptome analysis of adipose tissues reveals that ECM-receptor interaction is involved in the depot-specific adipogenesis in cattle[J]. PLoS One, 2013, 8(6): e66267. DOI:10.1371/journal.pone.0066267 |
| [36] |
Zhang H, Ye J, Weng X, et al. Comparative transcriptome analysis reveals that the extracellular matrix receptor interaction contributes to the venous metastases of hepatocellular carcinoma[J]. Cancer Genetics, 2015, 208(10): 482-491. DOI:10.1016/j.cancergen.2015.06.002 |
| [37] |
Ponta H, Sherman L, Herrlich PA. CD44:from adhesion molecules to signaling regulators[J]. Nat Rev Mol Cell Biol, 2003, 4: 33-45. DOI:10.1038/nrm1004 |
| [38] |
DeGrendele HC, Estess P, Siegelman MH, et al. Requirement for CD44 in activated T cell extravasation into an inflammatory site[J]. Science, 1997, 278(5338): 672-675. DOI:10.1126/science.278.5338.672 |
| [39] |
Protin U, Schweighoffer T, Jochum W, et al. CD44-deficient mice develop normally with changes in subpopulations and recirculation of lymphocyte subsets[J]. Journal of Immunology, 1999, 163(9): 4917-4923. |