2. 中国农业科学院生物技术研究所, 北京 100081
2. Biotechnology Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081
几丁质又名甲壳素、甲壳质,是一种由N-乙酰葡糖胺通过β-(1,4)糖苷键连接形成的多糖。广泛分布于海产品虾、蟹的外壳,真菌和藻类等植物的细胞壁中,同时也是节肢动物,如大多数昆虫的外骨骼的重要组成成分[1]。在自然界中,几丁质是继纤维素之后的第二大含量丰富的多糖物质[2]。该物质由Henri Braconnot于1811年首次在真菌中发现[3],但该物质几乎不溶于常用溶剂,从而严重限制了其商业应用。几丁质经几丁质脱乙酰酶(Chitin deacetylase,CDA)水解可得到易溶于酸的壳聚糖[4]。壳聚糖作为几丁质的衍生物,同属于大分子多糖聚合物,同时是自然界中发现的唯一碱性多糖,具有极大的应用价值和商业前景。
在工业上,壳聚糖可用来制备水凝胶、纤维、纳米粒子等物质;根据其成膜性则可使纺织材料达到耐水洗、抗皱、抗静电等效果[5]。在医学上,由于其具有较好的生物相容性,生物降解性和无毒性[6],常被用来制作动脉支架、药物缓释微胶囊等材料[7]。作为天然食品添加剂,对肉制品[8]、果蔬制品[9]和海产品[10]等有显著的保鲜作用。此外,还具有澄清果汁[11]、延缓淀粉老化和增加面包持水性[12]等功效,从而改善食品风味和质感。根据其与金属离子结合能力和絮凝作用,可有效处理环境中的污水,同时易被降解不会造成二次污染[13]。其良好的持水性和成膜性,也使之在化妆品行业中有一定的应用[14]。
目前工业生产中主要利用浓碱(40% NaOH)和高温长时间处理法脱去虾蟹外壳中几丁质的乙酰基团来制备壳聚糖[15]。该过程不仅会造成浓碱的大量浪费,产生的工业废水也会造成严重的环境污染。且该过程不易控制,生成的壳聚糖为脱乙酰度不同的混合物且质量不稳定。反应过程耗时长、耗能高[16],增加了生产成本。而利用几丁质脱乙酰酶脱去几丁质的乙酰基团是一种绿色高效的、条件可控的方法,该方法具有高度特异性,可定向得到所需降解产物,避免意外降解底物糖链的其它基团[17]。
几丁质脱乙酰酶是指能够脱去底物上乙酰基团的酶,目前研究的脱乙酰酶的主要作用底物有几丁质、壳聚糖、壳寡糖等,底物不同,酶的种类,作用方式往往不同。本文通过搜集已发表的包括真菌、细菌及动物等来源的脱乙酰酶基因序列,通过NCBI网站(www.ncbi.nlm.nih.gov)进行BLAST分析,对含有特定CE4超家族、spore_pdaA超家族、uraD_N-term-dom超家族结构域序列的酶进行归纳。下面将从这一类酶的催化机制,反应条件,金属离子的影响等方面进行阐述。
1 几丁质脱乙酰酶的催化机制 1.1 CE4超家族CAZy数据库(http://www.cazy.org/)中的碳水化合物脂酶4(Carbohydrate esterase 4,CE4)超家族,主要包括几丁质脱乙酰酶,细菌肽聚糖N-乙酰葡糖氨脱乙酰酶以及木聚糖脱乙酰酶等,这类酶可以移除乙酰基团从而将N-乙酰-D-葡萄糖胺(GlcNAc)转变为氨基葡萄糖(GlcN),该家族成员一般包含NodB同源结构域和His-His-Asp金属离子结合三联体结构,属于利用酸碱催化的金属酶[18]。
1.1.1 CE4超家族水解底物特性近年来,关于该类几丁质脱乙酰酶的研究主要集中在其底物的识别特性。2017年,挪威生命科学大学Liu等[19]从Aspergillus nidulans FGSC A4中鉴定出一种几丁质脱乙酰酶AnCDA(GenBank EAA66447),该酶对(GlcNAc)2有单端脱乙酰作用,对(GlcNAc)3-6则具有完全脱乙酰作用,对α-几丁质、β-几丁质也有一定活性,但对于GlcNAc无活性。同年,日本大学Hirano等[20]从Shewanella baltica ATCC BAA-1091中筛选出的SbCODs(GenBank ABN60929.1),与AnC-DA具有相似的催化机制,即SbCODs对(GlcNAc)2的还原端进行脱乙酰反应,形成GlcNAc-GlcN,对GlcNAc无活性,但对(GlcNAc)3-4均有脱乙酰活性。另外来自于真菌灰盖鬼伞(Coprinopsis cinerea)的几丁质脱乙酰酶CDA1(GenBank EAU83261.2)和CDA2(GenBank EAU83234.2)[21]也对GlcNAc均无反应,对(GlcNAc)2和40%的脱乙酰壳聚糖有一定的脱乙酰作用,且当几丁质粉末和胶体几丁质作为底物时无酶活。但这两种酶对乙二醇几丁质(Glycol chitin)均有较高酶活。海洋节细菌属(Arthrobacter)的几丁质脱乙酰酶ArCE4A(GenBank LT630322)[22]对GlcNAc、(GlcNAc)2、α-几丁质和β-几丁质几乎无活性,但对部分乙酰化的壳聚糖有一定的脱乙酰活性,且对乙酰木聚糖具有较高活性,脱乙酰度可达18.9%。同样,来自Pestalotiopsis sp.的几丁质脱乙酰酶PesCDA(GenBank KY024221)[23]对胶体几丁质有轻微的脱乙酰作用,且该酶脱乙酰效果随着底物乙酰度的增加而增加。当以(GlcNAc)2-6作为底物进行脱乙酰反应时发现,该酶对于(GlcNAc)n(n > 3)时存在脱乙酰效果,但最终产物仍会保留3个乙酰基团。与之相同的的是,根霉菌Rhizopus circinans分泌的几丁质脱乙酰酶RcCDA(GenBank EU086737)[24],也仅对聚合度≥3的低聚物具有脱乙酰活性,这也与其它毛霉菌来源的脱乙酰酶相符。
然而与上述几种酶都不同的是,来自于酿酒酵母(Saccharomyces cerevisiae)的几丁质脱乙酰酶ScCDA2[25]除了可作用于(GlcNAc)2-6外,还可对胶质几丁质、α-几丁质和β-几丁质3种均产生一定的脱乙酰作用。
来源于Vibrio Cholerae的VcCOD(GenBank WP_130350642)[26]通过对底物(GlcNAc)1-6测试发现该酶不能脱去GlcNAc中的乙酰基团,底物为(GlcNAc)2时表现出最大酶活,且酶活随着链长的增加酶活逐渐降低。同样,Pacheco等[27]通过对来自胶孢炭疽菌的几丁质脱乙酰酶研究发现,通过降低底物链长有助于脱乙酰反应,另外在酸性介质中提高溶解度和降低底物结晶度有促进脱乙酰效果。但ArCE4A在底物(GlcNAc)3-5中却随着聚合度的增加呈现酶活递增趋势,这与上述两种酶对糖链长度的偏好完全相反。
柄孢霉(Podospora anserina)中含有一种具有两个几丁质结合结构域(Chitin binding domain,CBD)的几丁质脱乙酰酶PaCDA(GenBank CAP60162),Hossbach等[28]通过研究去除一个或全部几丁质结合结构域的PaCDA发现,不溶于水的胶质壳五糖作为底物时含有CBDs的PaCDA比完全删除该结构域的PaCDA有更好的脱乙酰效果。表明CBDs有助于酶与不溶的底物进行反应。此外,该酶易作用于高度乙酰化的壳聚糖高聚物,但无法使底物完全脱乙酰化。
1.1.2 CE4超家族结构信息通过对上述CE4超家族的CDA酶进行氨基酸多序列比对发现,该家族的酶在His-His-Asp三连体结构上显示出高度保守性,并均含有H-D催化活性部位。此外,每个CDA均含有CE4超家族保守基序motif(1-5)(图 1)。由此可见,今后在进行新酶鉴定和分子改造时可加强对这些区域的对比与研究。

|
| 黄色区域为脱乙酰酶保守序列; 红色箭头为His-His-Asp三联体Zn2+离子结合位点; 绿色三角代表具有催化作用的氨基酸; 蓝色横线区域为CE4超级家族保守基序(Motif 1-5) 图 1 蛋白序列同源性及活性位点分析 |
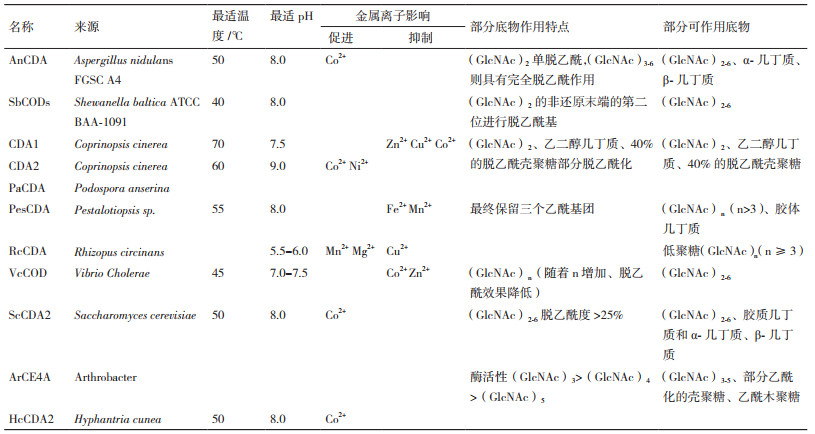
此外,通过使用SWISS-MODEL[29-33]对CDA1、CDA2、PesCDA、PaCDA进行蛋白质三级结构同源建模,再进一步利用PyMOL(www.pymol.org)进行三级结构比对发现,这几种酶的三级结构均有较高的吻合度。通过AnCDA的晶体结构与RcCDA同源建模的结果进行对比发现,虽然序列相似性不高,但其结构相似性很高。同时,这几种酶的三级结构分析也揭示了这几种酶均含有该家族所特有的(α/β)8折叠桶结构和6个loop区(图 2)。
|
| 图 2 CE4超家族脱乙酰酶蛋白同源结构模型 |
spore_pdaA超家族是多糖脱乙酰酶中的一个超家族。该家族的酶主要存在于细菌孢子壁上,主要作用于N-乙酰肽聚糖和几丁寡糖。PdaA是枯草芽孢杆菌中的一种脱乙酰酶,该酶与CE4家族的NodB结构域有较高同源性,对于该酶的报道多集中于N-乙酰胞壁酸的脱乙酰作用,其最适反应pH值为7.0,对于几丁质的脱乙酰作用较少,但其与CE4家族较高的同源性也使其作为今后CDA潜在研究对象[34]。
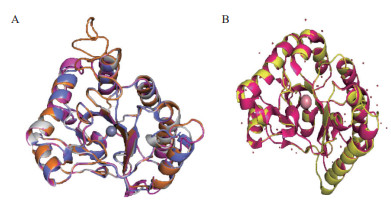
1.2.2 spore_pdaA超家族结构信息通过蛋白表面凹陷中的保守残基推断该家族蛋白含有一个与β/α桶状结构相关的折叠,与CE4家族相比,其蛋白表面凹陷中特有的Lys和两个Arg残基提供的阳离子也使其能够更好地结合聚合度低于3的N-乙酰氨基葡萄糖低聚物。通过已报道的X-射线观测和电子云密度可知GlcNAc与该酶结合时会将一些水分子从活性部位替换出来,同时根据蛋白与底物N-乙酰氨基葡萄糖的结合特点,揭露了其与CE4家族相似的Asp和其它His残基的催化作用(图 3)[34-35]。
|
| 红色区为α螺旋、蓝色区为β折叠 图 3 spore_pdaA超家族蛋白表面氨基酸残基分析 |
uraD是一种尿酸盐分解代谢蛋白,参与尿囊酸盐分解形成尿囊酸反应的最后一步,且通过序列比对发现该类蛋白与几丁质脱乙酰酶家族具有同源性。
通过海洋基因组文库筛选出的几丁质脱乙酰酶rCdaYJ[36],通过生信分析发现该酶属于uraD_N-term-dom超家族。进一步实验发现该酶可脱去对硝基乙酰苯胺上的乙酰基团,但是目前未发现该家族酶蛋白能水解几丁质中的乙酰基团。
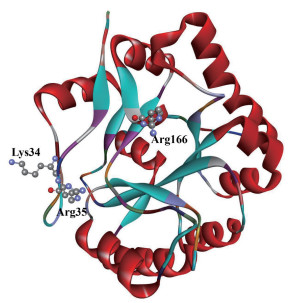
1.3.2 uraD_N-term-dom超家族结构信息通过对uraD家族puuE蛋白与CE4家族多种酶对比研究发现,蛋白质和蛋白质抑制剂复合物的晶体结构与多糖脱乙酰酶β/α桶状结构具有总体相似性(图 4),但在低聚物结合和活性部位几何结构上存在显著差异。例如,存在于CE4家族中的His-His-Asp金属离子结合结构在puuE中被Glu-His-Trp代替,此外,uraD家族在CE4家族第一个β/α桶状结构前多折叠的一个该结构为其结合小分子底物提供便利[37]。

|
| 图 4 uraD_N-term-dom超家族puuE蛋白二级结构分析 |
本文中提及的几丁质脱乙酰酶多数在50℃左右达到最大酶活。例如,AnCDA、ScCDA2、HcCDA2[38]三者最适温度均为50℃。而SbCODs、VcCOD两种酶的最适温度稍低,分别为40℃、45℃。PesCDA、CDA2、CDA1则有较高最适反应温度,分别为55、60和70℃。
此外,对于pH值而言,多数酶偏爱弱碱性环境。如AnCDA、SbCODs、PesCDA、ScCDA2和HcCDA2 5种酶的最适pH值均为8.0。此外,CDA1、CDA2最适pH值分别为7.5、9.0,VcCOD根据不同体系的缓冲溶液,其pH值在7.0-7.5之间。但是RcCDA却在偏酸性的环境中发挥最大活性,其最适pH值为5.5-6.0。其中AnCDA、ScCDA2、HcCDA2三种酶有着相同的最适温度和pH值。
除了CE4家族的脱乙酰酶外,分别来自spore_pdaA家族和uraD超家族的PdaA和rCdaYJ的最适pH值分别为7.0和7.4。其中rCdaYJ最适温度仅为28℃,这可能是因为rCdaYJ是海洋基因组文库来源的酶,与其所处环境有关。
3 金属离子催化作用某些情况下金属离子的催化作用对于酶发挥活性具有非常重要的作用,但是不同的酶对于金属离子的反应不同。较为常见的起促进作用的金属离子是Co2+,如AnCDA、CDA2、ScCDA2、HcCDA2以及毛柄金钱菌(Flammulina velutipes)来源的脱乙酰酶均在Co2+存在的体系中表现为酶活提高,但是VcCOD、CDA1表现出截然相反的现象,Co2+对这两个酶有抑制作用,尽管CDA1与CDA2来源于同一生物体。此外,在有些酶中Mn2+、Mg2+也可起到酶活促进作用。而Cu2+、Zn2+则在部分酶中展现出酶活抑制作用。有趣的是,ScCDA2结构上存在锌离子结合保守结构域,但在Co2+离子存在时展现出最大活力。
4 脱乙酰机制目前针对脱乙酰酶的研究中脱乙酰机制研究是一个重点方向,然而就当前研究而言,众多脱乙酰酶在脱乙酰基过程中有着不尽相同的脱乙酰产物,且没有标准的脱乙酰路径。目前,脱乙酰反应中几乎所有酶唯一一致的方面就是均从非还原末端开始对几丁寡糖进行脱乙酰反应。但对于水解几丁二糖,一般生成GlcNAc-GlcN。对于除单糖、二糖之外的N-乙酰低聚糖,不同的酶会与糖链上不同位置的N-乙酰基发生水解反应。如对于N-乙酰四糖,PesCDA仅能将其水解为AADA(A代表GlcNAc,D代表GlcN),同样的VcCOD也仅能将N-乙酰四糖脱去单个乙酰基最终生成ADAA单一产物,而SCCDA2则可通过三步水解反应在第一次生成DAAA、ADAA和AADA的基础上,进一步水解产生DDAA、DADA和ADDA,最终得到DDDA。
5 总结与展望几丁质脱乙酰酶主要来源于真菌和海洋生物,其最适温度范围为40-70℃,最适温度集中在50℃左右,是一类可耐受较高温度的酶。这类酶的最适pH值集中在8.0附近,最适pH值范围为5.5-9.0,大多数酶在碱性条件下活性较高。而Co2+则是最常见的增强酶活的金属离子,且多数酶均容易受到金属离子的影响。在底物识别方面,几乎均无法使GlcNAc脱除乙酰基,少数酶可脱去(GlcNAc)2非还原端的乙酰基团,大多数酶易识别(GlcNAc)3-6,而α-几丁质和β-几丁质等难溶于水的高聚合度多糖,由于其疏水性导致多数酶无法对其产生酶活反应,详见表 1。
自然环境中的微生物资源丰富,蕴藏着巨大的基因资源,如Whitman等[39]认为土壤、海底淤泥及地表下微生物丰度分别为2.6×1029、3.5×1030及(0.25-2.5)×1030,这些微生物中约有99%是未培养微生物,其中可能包含了很多具有综合性质优良的未知酶蛋白。随着宏基因组学和基因测序技术的发展,目前已有大量的来源于不同自然环境的宏基因组数据可在EBI metagenomics、iMicrobe、IMG/M等公开基因组数据库中下载。如利用hmmsearch从CAM_PROJ_GOS单个宏基因组数据集中就可检索到3 423条几丁质脱乙酰酶的基因(E-value值小于0.000 1)。这些数据也表明了在宏基因数据库中蕴藏了大量的基因资源,而这些基因资源“大数据”将是未来几年分子生物学研究的热点。
随着节能、环保、高效的工业生产理念深入人心,高效脱除几丁质中乙酰基团的酶制剂必定被市场需求。目前国内外研究中几丁质脱乙酰酶主要来源于真菌,而海洋生物中虾蟹类生物含有大量几丁质及壳聚糖,因此在海洋基因组文库中筛选脱乙酰酶,目的性更强,更具有针对性。另外该酶目前主要存在对多糖的脱乙酰度较低,对于晶体几丁质等疏水性多糖难以发挥作用等不足之处,故今后应加强在脱乙酰酶分子结合结构域上进行分析改造,并加强构建稳定高效的工程菌株从而弥补真菌等生物体自身表达量偏低的缺陷。目前国内外部分机构已检测到某些几丁质脱乙酰酶可定点脱去特定位置的乙酰基团,但这种现象的发生机理却尚不明确,进一步对几丁质脱乙酰酶水解机理的研究将有助于定向获得目的脱乙酰产物。我国每年有大量的虾蟹外壳等物质被直接废弃,开发几丁质脱乙酰酶对废弃物二次利用具有极高的价值,随着对几丁质脱乙酰酶的深入研究,今后必定可通过多种生物原料开发出高质量的壳聚糖副产物。
| [1] |
Rinaudo M. Chitin and chitosan: Properties and applications[J]. Progress in Polymer Science, 2006, 31(7): 603-632. DOI:10.1016/j.progpolymsci.2006.06.001 |
| [2] |
Dash M, Chiellini F, Ottenbrite RM, et al. Chitosan-A versatile semi-synthetic polymer in biomedical applications[J]. Progress in Polymer Science, 2011, 36(8): 981-1014. DOI:10.1016/j.progpolymsci.2011.02.001 |
| [3] |
Muzzarelli RA, Boudrant J, Meyer D, et al. Current views on fungal chitin/chitosan, human chitinases, food preservation, glucans, pectins and inulin: A tribute to Henri Braconnot, precursor of the carbohydrate polymers science, on the chitin bicentennial[J]. Carbohydra Polym, 2012, 87(2): 995-1012. DOI:10.1016/j.carbpol.2011.09.063 |
| [4] |
Pillai CKS, Paul W, Sharma CP. Chitin and chitosan polymers: Chemistry, solubility and fiber formation[J]. Progress in Polymer Science, 2009, 34(7): 641-678. DOI:10.1016/j.progpolymsci.2009.04.001 |
| [5] |
Enescu D. Use of chitosan in surface modification of textile materials[J]. Romanian Biotechnological Letters, 2008, 13(6): 4037-4048. |
| [6] |
Anitha A, Sowmya S, Kumar PTS, et al. Chitin and chitosan in selected biomedical applications[J]. Progress in Polymer Science, 2014, 39(9): 1644-1667. DOI:10.1016/j.progpolymsci.2014.02.008 |
| [7] |
Cheung RCF, Ng TB, Wong JH, et al. Chitosan: An update on potential biomedical and pharmaceutical applications[J]. Marine Drugs, 2015, 13(8): 5156-5186. DOI:10.3390/md13085156 |
| [8] |
Cui H, Yuan L, Lin L. Novel chitosan film embedded with liposome-encapsulated phage for biocontrol of Escherichia coli O157: H7 in beef[J]. Carbohydra Polym, 2017, 177: 156-164. DOI:10.1016/j.carbpol.2017.08.137 |
| [9] |
Batista RA, Perez Espitia PJ, Jullyana DSSQ, et al. Hydrogel as an alternative structure for food packaging systems[J]. Carbohydra Polym, 2019, 205: 106-116. DOI:10.1016/j.carbpol.2018.10.006 |
| [10] |
Olatunde OO, Benjakul S. Natural preservatives for extending the shelf-life of seafood: A revisit[J]. Comprehensive Reviews in Food Science and Food Safety, 2018, 17(6): 1595-1612. DOI:10.1111/1541-4337.12390 |
| [11] |
Rungsardthong V, Wonputtanakul N, Kongpien N, et al. Application of fungal chitosan for clarification of apple juice[J]. Process Biochemistry, 2006, 41(3): 589-593. DOI:10.1016/j.procbio.2005.08.003 |
| [12] |
No HK, Meyers SP, Prinyawiwatkul W, et al. Applications of chitosan for improvement of quality and shelf life of foods: A review[J]. Journal of Food Science, 2007, 72(5): R87-R100. DOI:10.1111/j.1750-3841.2007.00383.x |
| [13] |
Ngah WSW, Teong LC, Hanafiah MKM. Adsorption of dyes and heavy metal ions by chitosan composites: A review[J]. Carbohydra Polym, 2011, 83(4): 1446-1456. DOI:10.1016/j.carbpol.2010.11.004 |
| [14] |
Aranaz I, Acosta N, Civera C, et al. Cosmetics and cosmeceutical applications of chitin, chitosan and their derivatives[J]. Polymers, 2018, 10(2): E213. |
| [15] |
Kumar M. A review of chitin and chitosan applications[J]. Reactive & Functional Polymers, 2000, 46(1): 1-27. |
| [16] |
Abdou ES, Nagy KSA, Elsabee MZ. Extraction and characterization of chitin and chitosan from local sources[J]. Bioresource Technology, 2008, 99(5): 1359-1367. DOI:10.1016/j.biortech.2007.01.051 |
| [17] |
Tokuyasu K, Ono H, Ohnishikameyama M, et al. Deacetylation of chitin oligosaccharides of dp 2-4 by chitin deacetylase from Colletotrichum lindemuthianum[J]. Carbohydrate Research, 1997, 303(3): 353-358. DOI:10.1016/S0008-6215(97)00166-3 |
| [18] |
Blair DE, Schuttelkopf AW, Macrae JI, et al. Structure and metal-dependent mechanism of peptidoglycan deacetylase, a streptococcal virulence factor[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(43): 15429-15434. DOI:10.1073/pnas.0504339102 |
| [19] |
Liu Z, Gay LM, Tuveng TR, et al. Structure and function of a broadspecificity chitin deacetylase from Aspergillus nidulans FGSC A4[J]. Scientific Reports, 2017, 7(1): 1746. DOI:10.1038/s41598-017-02043-1 |
| [20] |
Hirano T, Shiraishi H, Ikejima M, et al. Chitin oligosaccharide deacetylase from Shewanella baltica ATCC BAA-1091[J]. Bioscience Biotechnology and Biochemistry, 2017, 81(3): 547-550. DOI:10.1080/09168451.2016.1254529 |
| [21] |
Wang Y, Niu X, Guo X, et al. Heterologous expression, characteri-zation and possible functions of the chitin deacetylases, Cda1 and Cda2, from mushroom Coprinopsis cinerea[J]. Glycobiology, 2018, 28(5): 318-332. DOI:10.1093/glycob/cwy007 |
| [22] |
Tuveng TR, Rothweiler U, Udatha G, et al. Structure and function of a CE4 deacetylase isolated from a marine environment[J]. PLoS One, 2017, 12(11): 15. DOI:10.1371/journal.pone.0187544 |
| [23] |
Cord-Landwehr S, Melcher RLJ, Kolkenbrock S, et al. A chitin deacetylase from the endophytic fungus Pestalotiopsis sp efficiently inactivates the elicitor activity of chitin oligomers in rice cells[J]. Scientific Reports, 2016, 6: 38018. DOI:10.1038/srep38018 |
| [24] |
Gauthier C, Clerisse F, Dommes J, et al. Characterization and cloning of chitin deacetylases from Rhizopus circinans[J]. Protein Expression and Purification, 2008, 59(1): 127-137. DOI:10.1016/j.pep.2008.01.013 |
| [25] |
Zhu XY, Zhao Y, Zhang HD, et al. Characterization of the specific mode of action of a chitin deacetylase and separation of the partially acetylated chitosan oligosaccharides[J]. Marine Drugs, 2019, 17(2): E74. DOI:10.3390/md17020074 |
| [26] |
Li X, Wang LX, Wang X, et al. The chitin catabolic cascade in the marine bacterium Vibrio cholerae: Characterization of a unique chitin oligosaccharide deacetylase[J]. Glycobiology, 2007, 17(12): 1377-1387. DOI:10.1093/glycob/cwm096 |
| [27] |
Pacheco N, Trombotto S, David L, et al. Activity of chitin deacetylase from Colletotrichum gloeosporioides on chitinous substrates[J]. Carbohydra Polym, 2013, 96(1): 227-232. DOI:10.1016/j.carbpol.2013.03.051 |
| [28] |
Hossbach J, Busswinkel F, Kranz A, et al. A chitin deacetylase of Podospora anserina has two functional chitin binding domains and a unique mode of action[J]. Carbohydra Polym, 2018, 183: 1-10. DOI:10.1016/j.carbpol.2017.11.015 |
| [29] |
Guex N, Peitsch MC, Schwede T. Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer: A historical perspective[J]. Electrophoresis, 2009, 30: S162-S173. DOI:10.1002/elps.200900140 |
| [30] |
Bertoni M, Kiefer F, Biasini M, et al. Modeling protein quaternary structure of homo- and hetero-oligomers beyond binary interactions by homology[J]. Scientific Reports, 2017, 7: 10480. DOI:10.1038/s41598-017-09654-8 |
| [31] |
Bienert S, Waterhouse A, De Beer TP, et al. The SWISS-MODEL Repository-new features and functionality[J]. Nucleic Acids Research, 2017, 45(D1): D313-D319. DOI:10.1093/nar/gkw1132 |
| [32] |
Waterhouse A, Bertoni M, Bienert S, et al. SWISS-MODEL: homology modelling of protein structures and complexes[J]. Nucleic Acids Research, 2018, 46(W1): W296-W303. DOI:10.1093/nar/gky427 |
| [33] |
Benkert P, Biasini M, Schwede T. Toward the estimation of the absolute quality of individual protein structure models[J]. Bioinformatics, 2011, 27(3): 343-350. DOI:10.1093/bioinformatics/btq662 |
| [34] |
Fukushima T, Kitajima T, Sekiguchi J. A polysaccharide deacetylase homologue, PdaA, in Bacillus subtilis acts as an N-acetylmuramic acid deacetylase in vitro[J]. Journal of Bacteriology, 2005, 187(4): 1287-1292. DOI:10.1128/JB.187.4.1287-1292.2005 |
| [35] |
Blair DE, Van Aalten DMF. Structures of Bacillus subtilis PdaA, a family 4 carbohydrate esterase, and a complex with N-acetyl-glucosamine[J]. FEBS Letters, 2004, 570(1-3): 13-19. DOI:10.1016/j.febslet.2004.06.013 |
| [36] |
Liu JL, Jia ZJ, Li S, et al. Identification and characterization of a chitin deacetylase from a metagenomic library of deep-sea sediments of the Arctic Ocean[J]. Gene, 2016, 590(1): 79-84. DOI:10.1016/j.gene.2016.06.007 |
| [37] |
Ramazzina I, Cendron L, Folli C, et al. Logical identification of an allantoinase analog(puuE)recruited from polysaccharide deacetylases[J]. Journal of Biological Chemistry, 2008, 283(34): 23295-23304. DOI:10.1074/jbc.M801195200 |
| [38] |
Yan X, Zhao D, Guo W, et al. Cloning, expression and enzymatic characterization of chitin deacetylases from Hyphantria cunea[J]. Scientia Agricultura Sinica, 2017, 50(5): 849-858. |
| [39] |
Whitman WB, Coleman DC, Wiebe WJ. Prokaryotes: The unseen majority[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583. DOI:10.1073/pnas.95.12.6578 |