铁是所有生命所必需的元素之一,它是细胞内多种重要蛋白或酶的关键组分或辅因子,在呼吸、光合、生物固氮以及活性氧和活性氮代谢等多种生命活动中起重要作用。尽管铁是地球上丰度仅次于铝的第二大金属元素,但在大部分生境中主要以极难溶的Fe3+形式存在,很难被生物直接吸收利用。为了满足对铁元素的需求,细菌通过进化获得了多种转运系统来摄取环境中的铁[1]。例如,除了通过Feo等转运系统直接吸收可溶性的Fe2+外,细菌采取合成分泌铁载体吸收低可溶性Fe3+的方式更加普遍,一些动物致病菌还可以利用宿主来源的血红素作为自身铁源。
根瘤菌是一类能够与豆科植物结瘤固氮的革兰氏阴性细菌的总称。作为兼性共生菌,根瘤菌的生活周期包括自生和共生两个阶段。其中,大部分时期根瘤菌在土壤中营腐生生活,在此期间它们需要同其他土壤微生物就包括铁在内的各类营养展开激烈竞争。条件适宜情况下,根瘤菌通过根毛或侧根与主根间的缝隙侵染豆科植物的根部皮层细胞,形成固氮器官根瘤[2]。在根瘤侵染细胞中,根瘤菌被来源于植物质膜的共生体膜所包被,进一步增殖分化形成类菌体(Bacteroid)。类菌体通过消耗植物提供的碳源生成ATP和还原力驱动固氮,并将其反馈给植物供其生长利用。根瘤菌在共生固氮阶段对铁的需求相比自生阶段会极大提高,这是由于类菌体高效固氮需要表达大量固氮酶以及在低氧条件下维持呼吸作用的氢化酶、细胞色素等含铁蛋白[3]。其中,每分子固氮酶复合体含34个铁原子,而它们占全部可溶蛋白的比例可高达约30%(总蛋白的11%)[4-5]。与此同时,根瘤中的植物细胞需要合成大量豆血红蛋白来维持类菌体固氮所需低氧环境,同样对铁有着极大的需求。事实上,据报道土壤中缺乏铁会对根瘤菌和豆科植物的共生产生负面影响,例如根瘤菌缺铁会导致其竞争结瘤能力降低,而植物缺铁抑制结瘤的起始过程以及根瘤的发育[6-8]。目前,人们对根瘤内铁的分配和转运机制还缺乏清晰的认识。
从分类角度看,目前已知的根瘤菌分布在α-和β-变形菌纲近20个属的200多个种,系统发育多样性极高[9]。除了少数核心共生基因相对保守外,没有任何基因是根瘤菌所共有且特有的[10-11]。基因组上的巨大差异也导致了根瘤菌在生长、代谢、遗传调控及共生结瘤等方面表现出极大多样性[10]。近年来,针对根瘤菌铁代谢的研究主要集中在豌豆根瘤菌(Rhizobium leguminosarum)、苜蓿中华根瘤菌(Sinorhizobium meliloti)和日本慢生根瘤菌(Bradyrhizobium japonicum)这3个属于α-变形菌纲的模式物种。其中,前两者属于根瘤菌科(Rhizobiaceae),而后者属于慢生根瘤菌科(Bradyrhizobiaceae),它们的祖先大约在5亿年前分化[12],因而它们在铁转运代谢及相关调控方面已表现出显著差异。本文将总结近年来有关根瘤菌铁转运代谢及其调控方面的研究进展,分析归纳出可能的工作模型以及未来仍待解决的问题,为后续研究者提供借鉴。
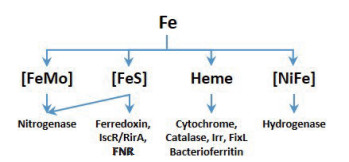
1 根瘤菌中铁的存在形式及生理功能细胞中的铁一般仅有少部分以游离铁离子形式存在,大部分则以配体或辅因子的形式与蛋白结合,发挥活性调节、电子传递等作用。根瘤菌中的含铁蛋白除了在其他细菌中普遍存在的铁氧还原蛋白、细胞色素、过氧化氢酶、储铁蛋白以及多种转录调控蛋白外,还有其共生时特异表达的固氮酶、氢化酶以及高亲氧活性呼吸链蛋白组分等(图 1)。在这些含铁蛋白中,血红素和铁硫簇是最常见的两种铁的存在形式,其中铁硫簇又可分为[2Fe2S]和[4Fe4S]等不同类型。在固氮酶复合体中,除铁蛋白结合的[4Fe-4S]簇外,钼铁蛋白中的α亚基和β亚基分别与[Mo-7Fe-9S-高柠檬酸]和[8Fe-7S]结合,后两种特殊的辅因子均由特定的nif基因负责装配[4, 13]。部分根瘤菌编码的氢化酶(Hydrogenase)可以氧化共生固氮过程中产生的氢气合成ATP以避免能量的浪费,该酶的催化活性依赖于hyp操纵子负责合成的[NiFe]辅因子[14-15]。
|
| Nitrogenase:固氮酶;hydrogenase:氢化酶;cytochrome:细胞色素;ferredoxin:铁氧还蛋白;catalase:过氧化氢酶;bacterioferritin:细菌贮铁蛋白;IscR/RirA/FNR/Irr/FixL:转录调控蛋白 图 1 根瘤菌中铁存在的主要形式及参与合成的蛋白 |
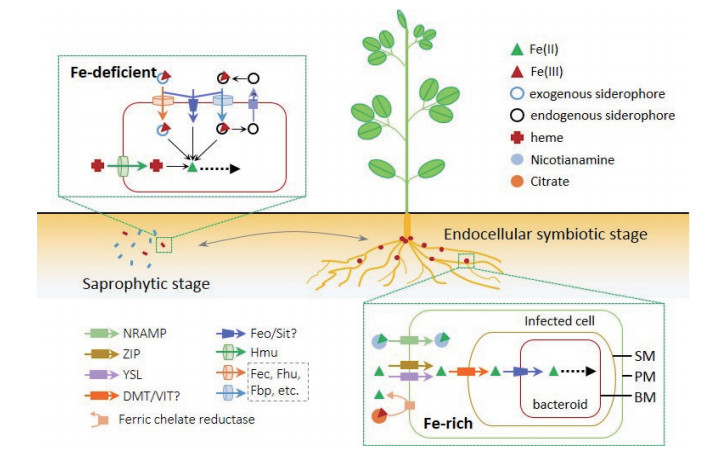
基于实验室纯培养条件下的研究表明,根瘤菌可以利用Fe2+、Fe3+和血红素等不同形式的铁作为自身铁源。在自然条件下,根瘤菌很可能依据其在自生和共生不同阶段所处环境中铁的存在形式采用不同策略获得铁(图 2)。
|
| 在土壤中营腐生生活时,大部分根瘤菌通常处于铁饥饿状态,主要利用内源和外源铁载体以及TonB依赖的Fec、Fhu、Fbp等转运系统吸收Fe3+;对于一些不编码定位于细胞内膜上铁载体转运系统的Bradyrhizobium菌株,它们首先通过外膜受体蛋白和TonB/ExbB/ExbD供能系统将Fe3+-铁载体复合物泵入周质空间,然后利用铁还原酶将螯合态Fe3+还原为Fe2+并释放,再通过Feo系统将Fe2+吸收到胞质内。在根瘤衰老破裂时期,根瘤菌可以通过同样依赖于TonB的Hmu系统摄取豆血红蛋白降解释放的血红素。在正常根瘤中,被侵染的宿主细胞可能通过NRAMP、ZIP、YSL等系统吸收由木质部传输而来的Fe2+及其复合物,胞质中的低氧低pH环境条件可以维持铁离子以可溶性Fe2+存在,因而根瘤菌在共生阶段可能处于相对富铁的状态。Fe2+可以通过DMT系统跨过共生体膜进入共生小体,然后由根瘤菌编码的Feo或Sit转运系统直接吸收。PM:plasma membrane(侵染细胞质膜);SM:symbiosome membrane(共生体膜);BM:bacteroid membrane(类菌体膜) 图 2 假定的根瘤菌铁吸收机制模型 |
当环境中缺铁时,通过合成和分泌铁载体再通过依赖于TonB的转运系统摄取Fe3+-铁载体复合物是在细菌中最常见的铁吸收策略[16]。铁载体是一类具有高特异性Fe3+螯合能力的低分子量(约300-1 500 Da)有机化合物,尽管其分子结构多样,但所包含金属结合基团的变化却比较有限。根据铁螯合基团的不同,常见的铁载体主要被划分为儿茶酚型、异羟肟酸酯型、α-羟基羧酸型以及复合型几类,其他类型的金属离子亲和基团如恶唑啉、噻唑啉、羟基吡啶和β-羟基酸等也偶有发现[17]。根据生物合成过程中非核糖体肽合成酶(Nonribosomal peptide synthetase,NRPS)参与与否,铁载体又可以分为NRPS依赖型或非NRPS依赖型两种[18-19]。
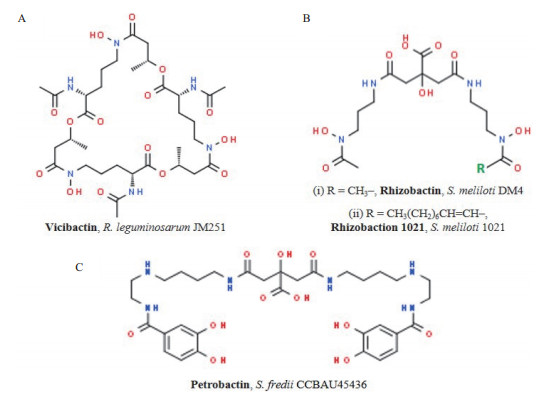
迄今为止,已有3种根瘤菌内源性铁载体获得了结构鉴定(图 3-A-B):(1)R. leguminosarum JM251等菌株合成的三羟肟酸型铁载体vicibactin;(2)S. meliloti DM1菌株合成的α-羟基羧酸盐型铁载体rhizobactin;(3)S. meliloti 1021菌株合成的含柠檬酸盐衍生物的二氢肟酸盐型铁载体rhizobactin 1021[20-22]。其中,vicibactin的合成依赖于NRPS,而后两者不依赖于NRPS。不同种根瘤菌甚至同种根瘤菌的不同菌株所合成铁载体通常具有较大差异,如R. leguminosarum IARI 102和Rhizobium sp(cow-pea)RA-1并非合成vicibactin,而是合成结构尚不清楚的儿茶酚类铁载体[23-24]。
|
| 目前已知3种由根瘤菌合成的铁载体vicibactin、rhizobabtin、rhizobactin 1021已从相应菌株培养液中分离纯化并进行结构鉴定。Petrobactin的分子结构及生物合成途径已在炭疽杆菌(Bacillus anthraci)中获得解析,该铁载体由asbABCDEF操纵子负责合成,S. fredii CCBAU45436中含该基因簇的的同源基因 图 3 不同根瘤菌合成的几种铁载体的分子结构 |
从遗传角度讲,不同根瘤菌铁载体合成方面的差异是相关基因差异所导致。例如,R. leguminosarum中vicibactin的合成由vbsGSO/vbsADL/vbsC/vbsP四个操纵子编码的酶负责,S. meliloti中rhizobactin的合成则由rhbABCDEF操纵子负责,而rhizobactin 1021可能还需要rhbG的参与[18, 21, 25-26]。在根瘤菌中,负责铁载体合成的基因经常位于质粒或染色体的基因岛,其种属分布十分不保守,具有显著的水平基因转移特征。在S. fredii的某些菌株中,预测到的铁载体合成基因簇与炭疽杆菌中asbABCDEF同源(蛋白序列相似性31%-47%),该基因簇负责合成与炭疽杆菌致病性密切相关的原儿茶酚型铁载体petrobactin(图 3-C)[27]。
包括根瘤菌在内的绝大部分革兰氏阴性细菌都通过依赖于TonB的转运系统(TonB-dependent transporters,TBDTs)摄取Fe3+-铁载体复合物,该系统通常由外膜受体通道蛋白和内膜上的TonB/ExbB/ExbD供能系统及ABC转运子3部分组成[28-29]。分泌到胞外的铁载体螯合Fe3+后,可以与外膜受体蛋白以不消耗能量的方式特异性结合,然后由质子动力提供能量并通过TonB/ExbB/ExbD复合物传递给外膜受体使其发生构象变化,将Fe3+-铁载体复合物转运到周质空间[29]。随后,周质空间内的Fe3+-铁载体复合物进一步与ABC转运子中的底物结合蛋白结合,在ATP水解酶驱动下通过内膜上透性酶转运至胞内[29]。
在许多已测序的根瘤菌中,除了针对内源铁载体的外膜受体蛋白编码基因外,通常还存在额外的外膜受体编码基因。这可能意味着在根瘤菌中还普遍存在着利用外源铁载体吸收铁的机制,而这很可能对根瘤菌在土壤中的竞争存活是有利的。有意思的是,B. japonicum USDA110基因组中没有发现负责内源性铁载体合成的基因,但却编码至少3个外膜铁载体受体蛋白(EntR、FegA、FhuE)以及完整的TonB/ExbB/ExbD供能系统[30]。遗传证据表明,该菌株可以利用这3种外膜受体通过TonB依赖的方式分别特异地转运desferrioxamine、ferrichrome和enterobactin等外源铁载体[30]。
当Fe3+-铁载体复合物被吸收到细胞中后,Fe3+通常以被还原成Fe2+的形式释放,这一还原反应由铁还原酶催化完成[29]。目前仅在少数细菌中鉴定的铁还原酶几乎都是胞质可溶的且它们的催化活性依赖于核黄素[31-32]。此外,某些铁还原酶则以铁硫簇作为其辅酶,如FhuF[33]。目前,在根瘤菌中唯一研究较为清楚的铁还原酶是由B. japonicum编码的FrcB,其编码基因与编码铁载体外膜受体的pyoR同源基因位于同一操纵子内[34]。FrcB是定位于内膜的二聚体蛋白且其稳定性严格依赖于其特定保守组氨酸残基与血红素的结合,这一特征与真核生物中的铁还原酶一致[35]。在大肠杆菌中的异源表达以及体外酶活测定实验均证实FrcB具有铁还原酶活性,且在缺铁条件下该蛋白以Irr依赖性方式被强烈诱导表达[34]。然而,最近的一项研究发现缺失frcB基因的B. japonicum菌株仍可以利用外源铁载体吸收铁供自身生长,这意味着该菌还编码其他铁还原酶参与铁载体依赖的铁吸收[30]。
2.1.2 血红素的摄取通过血红素转运及降解获得铁曾被认为是动物致病性细菌所特有的铁吸收方式。直到1997年,Noya等[36]发现根瘤菌及其他一些非致病性细菌同样具备在缺铁条件下利用血红素以及血红蛋白作为铁源进行生长的能力。随后,人们在B. japonicum、R. leguminosarum和S. meliloti几种模式根瘤菌中相继鉴定了相对较为保守的血红素转运相关的基因[37-40]。与铁载体的转运十分相似,根瘤菌对血红素的转运同样依赖于TonB/ExbB/ExbD供能复合体以及特异性识别血红素的外膜受体蛋白[29, 38]。HmuR和ShmR分别是由B. japonicum和S. meliloti编码的外膜血红素受体蛋白,它们对于根瘤菌吸收血红素是必需的[40-41]。尽管两者蛋白相似性仅为23%,但它们都含有典型的血红素受体FRAP/NPNL基序。在R. leguminosarum中,存在与ShmR蛋白序列一致性高达65%的同源蛋白编码基因。血红素在TonB/ExbB/ExbD驱动下经外膜受体蛋白进入周质空间后,会进一步由hmuTUV编码的ABC转运子将其泵入胞质中[37-38]。值得注意的是,B. japonicum和S. meliloti中hmuTUV缺失突变并不会导致血红素利用能力的完全丧失,推测它们还编码其他类型的血红素转运系统[38]。进入胞质中的血红素既可以被根瘤菌直接利用,也可以通过氧化途径降解卟啉环释放出Fe2+[42]。
2.1.3 Fe2+的吸收转运虽然铁一般仅在酸性且厌氧环境中才以Fe2+形式存在,但由于其相对于Fe3+高可溶性和可以直接通过外膜进入周质空间的特点,而被认为是细菌利用铁的首选形式。因此,大部分细菌都保留了直接吸收Fe2+的能力。目前,在细菌中鉴定的Fe2+转运系统主要是Feo转运系统,此外还发现Yfe转运系统、Efe转运系统、Sit转运系统等也具有Fe2+转运能力[43]。
在根瘤菌中,研究较多的Fe2+转运系统是由B. japonicum USDA110菌株中feoAB基因所编码的Feo系统。同位素标记实验表明,Feo转运系统的功能失活不仅会导致该菌株无法有效吸收Fe2+,而且Fe3+的吸收转运也会被抑制,但血红素的吸收不受影响[44]。因此,研究者认为该菌株对Fe3+的吸收是首先在胞外或周质空间将Fe3+还原为Fe2+,然后再进一步通过Feo系统转运至细胞内的方式实现的。Sinorhizobium和Rhizobium属的根瘤菌基因组中不含feoAB同源基因,但都编码另一种在其他细菌中被证实可以非特异地转运Fe2+的Sit转运系统[45-46]。尽管S. meliloti编码的Sit转运系统主要功能是转运Mn2+,但同时也具有一定的Fe2+转运能力,并且调控该系统表达的调控蛋白Mur的活性除了受到Mn2+调节外,也部分受到Fe2+的影响[46]。
2.2 类菌体吸收铁的方式如前所述,根瘤菌在共生阶段分化形成的类菌体需要合成大量固氮酶、氢化酶以及细胞色素c等与固氮密切相关的含铁蛋白,因此铁是与共生固氮效率密切相关的因素之一。Slatni等[47]研究发现菜豆根瘤中铁的含量与其固氮效率呈正相关。在此情况下,类菌体面临怎样的铁营养条件,采用何种策略吸收铁成为人们所关注的问题。
根瘤中存在大量豆血红蛋白,使人们曾一度怀疑根瘤菌是否在共生阶段以血红素作为自身所需的铁源。然而,考虑到根瘤菌在侵染细胞内被共生体膜所包被,并不与豆血红蛋白直接接触,而游离血红素的浓度通常又非常低,上述猜测的可能性大大降低。另有大量遗传学研究表明,根瘤菌在自生条件下吸收血红素所用的转运系统缺失对共生固氮没有显著影响,不是共生所必需的[37-38]。因此,目前普遍认为根瘤菌通过吸收血红素获得铁的机制很可能主要发生在根瘤衰老阶段[3]。
尽管尚缺乏充分的直接证据,已有的研究表明类菌体很可能主要以吸收Fe2+的方式获得铁。一方面,在侵染细胞中类菌体被共生体膜所包被形成共生小体,共生体膜外偏酸和低氧的植物胞质环境有利于维持铁离子以Fe2+形式存在。放射性同位素实验表明,从大豆根瘤中分离纯化的共生小体可以吸收Fe2+和Fe3+,但吸收Fe2+速率明显快于Fe3+[48-49]。其中,Fe2+的转运很可能由定位于共生体膜上的NRAMP家族转运子GmDMT1实现,尽管该转运子同时还具备Zn2+、Mn2+等其他二价金属离子转运能力[50]。共生体膜与类菌体之间的共生周质空间内pH相比于植物细胞质更低(可以达到4.5-5),并且可以检测到较强的铁还原酶活性,更进一步确保类菌体胞外微环境中可以存在较高浓度的Fe2+[49, 51]。另一方面,在几种典型的模式根瘤菌中,仅转运Fe2+的Feo系统或Sit系统缺失则导致显著的共生缺陷[44, 46, 52],而负责铁载体或血红素转运的基因突变则不影响共生表型[26, 37-38, 40, 42, 53-55]。以上证据除了证明类菌体很可能以Fe2+方式吸收铁之外,还强烈暗示侵染细胞中很可能处于相对富铁的状态。支持这一猜想的最新证据来自于S. fredii,其中铁响应调节蛋白RirA缺失会导致共生固氮能力降低,而该蛋白已被证实在高铁条件下会抑制TonB依赖的铁吸收[56]。
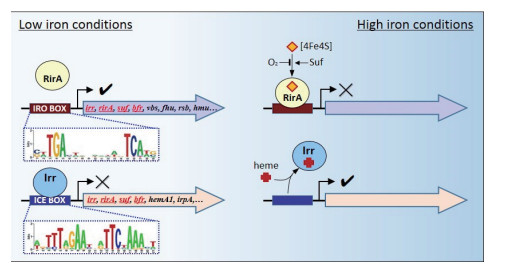
3 根瘤菌胞内铁稳态的调控尽管铁具有极其重要的生物学功能,但如果细胞中存在过量的铁则容易引发芬顿反应生成活性氧类物质,进而造成毒害作用[57]。因此,细胞内自由铁离子的浓度必须控制在较低的水平。与其他细菌相一致,根瘤菌内铁离子稳态通过以下4种途径实现:(1)调控转运相关基因的表达;(2)利用铁储藏蛋白将铁转化为非自由状态;(3)依据环境中铁的多寡选择性表达依赖或非依赖于铁的酶类;(4)调控铁外排通道蛋白的表达。在大肠杆菌等模式物种中,铁代谢相关基因主要受全局性铁代谢调控蛋白Fur调控[58]。虽然大部分根瘤菌也都编码Fur同源蛋白,但它们主要负责调控与锰离子转运相关的基因,因而被重新命名为Mur[41, 59]。实际上,根瘤菌中铁稳态的实现主要依赖于另外两种调控蛋白Irr和RirA所介导的调控(图 4)[58]。
Irr属于Fur调控蛋白家族,该蛋白最早被发现在B. japonicum中调控血红素合成途径[60],而现已被证实是广泛分布于α-变形菌纲根瘤菌和非根瘤菌中响应低铁条件的全局转录调控因子[61]。基于基因芯片的转录组分析发现,B. japonicum中大多数受外源铁调节的基因在转录水平上受到Irr调控[62]。其中,与铁吸收以及血红素降解相关的基因受正调控,而与铁的利用、贮藏以及外排相关的基因如bfr则受负调控。然而,在R. leguminosarum和S. meliloti中的研究却仅发现Irr作为抑制子发挥作用,负调控下游靶基因的表达[61, 63]。基于体外蛋白-DNA互作以及定点突变等分析的结果表明,B. japonicum中Irr与受其正调控和负调控的基因启动子区均存在结合作用,因此Irr可能除了抑制基因表达外,在某些情况下还扮演激活子角色[62, 64-65]。基于序列比对分析,人们发现在许多受Irr调控的基因启动子区存在被命名为ICE(Iron control element)的保守顺式作用元件[37, 66]。
非常有趣的是,Irr与Fur、Zur、Nur、Mur等其他Fur家族成员不同,它并非直接与特定金属离子结合进而激活自身DNA结合活性,而是通过与血红素结合间接感知细胞内铁的含量。在B. japonicum中,不结合血红素的Irr结构较稳定,而当其与血红素结合时则被降解[67]。现有研究表明,Irr的降解依赖于其自身所包含的两个血红素结合域HRM(Heme-regulatory motif)和HXH(Histidine-x-histidine)结构域[68-69]。由R. leguminosarum编码的Irr缺少HRM结构域,而HXH结构域与血红素结合只对其DNA结合活性有影响,但并不会导致蛋白的降解[70]。
除了Irr之外,属于Rhizobiaceae科的细菌还普遍编码属于Rrf2家族的铁响应转录调控蛋白RirA[59, 71-75]。与Irr恰巧相反,RirA在高铁条件下负调控许多与铁载体合成转运以及血红素吸收相关的基因[76]。与此相一致,RirA缺失突变体通常表现出在高铁培养基中生长迟缓的表型,且对H2O2更加敏感[73, 75]。基于转录组和蛋白质组学的研究发现,S. meliloti和R. leguminosarum中受RirA调控的基因除了参与铁吸收之外,还涉及硫代谢、能量代谢以及胞外多糖合成等途径[71, 73]。通过生物信息学方法,Rodionov和Yeoman等在受RirA调控的基因启动子区发现了保守的IRO调控元件(Iron-Rhodo-box)[59, 72]。与其他Rrf2家族蛋白类似,RirA蛋白序列中含有可以与铁硫簇结合的保守Cys残基。最新研究表明,虽然RirA可以与[2Fe2S]和[4Fe4S]两种铁硫簇都发生结合,但仅在与[4Fe4S]结合时具备较强的DNA结合活性[77]。因此,RirA的调节活性除了依赖于细胞内铁的浓度外,还受到硫元素以及氧气含量的调节[77]。
在同时含Irr和RirA的根瘤菌中,一些下游基因通常同时受到两者的协同调控,其中包括编码这两种调控蛋白的基因本身,这种严密的协同调控可能有利于细胞对自身铁营养状态做出快速响应[59, 63, 78]。除此之外,受RirA和Irr调控的基因还编码一些菌株特异性的局部铁代谢调节蛋白,如HmuP、RhrA和RpoI等。在S. meliloti中,HmuP的表达受RirA负调控,而其本身对于shmR的表达是必需的[79]。然而,在缺少RirA的B. japonicum中,HmuP则通过与Irr协同作用激活hmuR操纵子的表达[80]。RhrA是属于AraC家族的转录调控因子,在S. meliloti中该蛋白可以激活负责rhizobactin 1021合成的rhbABCDEF操纵子[26, 81]。在R. leguminosarum中,负责合成vicibaction的vbs基因启动子依赖于特殊的σ因子RpoI起始转录[25]。
4 结语铁在根瘤菌中参与许多重要蛋白的合成,尤其在共生固氮过程中发挥极其重要的作用。基于大量自生条件下的研究,人们发现根瘤菌可利用多种转运系统吸收不同形式的铁,并通过Irr和RirA等调节蛋白介导的精密调控实现细胞铁稳态。然而,共生阶段根瘤菌在宿主细胞内通过何种方式获取铁以及如何与宿主协调实现铁营养的平衡至今仍没有清晰答案。根瘤菌遗传多样性以及铁转运系统的冗余客观上为此方面研究造成了困难,进一步的研究需针对特定菌株构建遗传背景更加清晰的突变体展开。相应地,铁在根瘤内的转运代谢途径同时也需要从宿主植物方面展开。近年来日益发展成熟的同步辐射X光吸收谱(X-ray absorption fine spectroscopy,XAFS)可以实现原位金属离子分布及化学形态的检测,应用此技术或可在上述方面取得突破性进展。鉴于铁广泛参与了与共生固氮密切相关的物质、能量以及信号分子代谢,如何通过改变根瘤菌铁代谢调控共生固氮效率也是未来值得研究的方向。
| [1] |
Ratledge C, Dover LG. Iron metabolism in pathogenic bacteria[J]. Annual Reviews in Microbiology, 2000, 54(1): 881-941. DOI:10.1146/annurev.micro.54.1.881 |
| [2] |
Oldroyd GE, Murray JD, Poole PS, et al. The rules of engagement in the legume-rhizobial symbiosis[J]. Annual Review of Genetics, 2011, 45: 119-144. DOI:10.1146/annurev-genet-110410-132549 |
| [3] |
Brear EM, Day DA, Smith PM. Iron:an essential micronutrient for the legume-rhizobium symbiosis[J]. Frontiers in Plant Science, 2013, 4: 359. |
| [4] |
Dixon R, Kahn D. Genetic regulation of biological nitrogen fixation[J]. Nat Rev Microbiol, 2004, 2(8): 621-631. DOI:10.1038/nrmicro954 |
| [5] |
Verma D, Nadler KD. The Rhizobium-legume symbiosis:the host's point of view[M]. Plant gene research: genes involved in microbe-plant interactions. Berlin:Springer-Verlag, 1984.
|
| [6] |
O'Hara GW, Dilworth MJ, Boonkerd N, et al. Iron-deficiency specifically limits nodule development in peanut inoculated with Bradyrhizobium sp.[J]. New Phytologist, 1988, 108(1): 51-57. DOI:10.1111/j.1469-8137.1988.tb00203.x |
| [7] |
Tang C, Robson AD, Dilworth MJ. The role of iron in nodulation and nitrogen fixation in Lupinus angustifolius L[J]. New Phytologist, 1990, 114(2): 173-182. DOI:10.1111/j.1469-8137.1990.tb00388.x |
| [8] |
Battistoni F, Platero R, Noya F, et al. Intracellular Fe content influences nodulation competitiveness of Sinorhizobium meliloti strains as inocula of alfalfa[J]. Soil Biology and Biochemistry, 2002, 34(5): 593-597. DOI:10.1016/S0038-0717(01)00215-2 |
| [9] |
Shamseldin A, Abdelkhalek A, Sadowsky MJ. Recent changes to the classification of symbiotic, nitrogen-fixing, legume-associating bacteria:a review[J]. Symbiosis, 2017, 71(2): 91-109. DOI:10.1007/s13199-016-0462-3 |
| [10] |
Masson-Boivin C, Giraud E, Perret X, et al. Establishing nitrogen-fixing symbiosis with legumes:how many rhizobium recipes?[J]. Trends in Microbiology, 2009, 17(10): 458-466. DOI:10.1016/j.tim.2009.07.004 |
| [11] |
Tian CF, Zhou YJ, Zhang YM, et al. Comparative genomics of rhizobia nodulating soybean suggests extensive recruitment of lineage-specific genes in adaptations[J]. Proceedings of the National Academy of Sciences, 2012, 109(22): 8629-8634. DOI:10.1073/pnas.1120436109 |
| [12] |
Turner SL, Young JPW. The glutamine synthetases of rhizobia:phylogenetics and evolutionary implications[J]. Molecular Biology and Evolution, 2000, 17(2): 309-319. DOI:10.1093/oxfordjournals.molbev.a026311 |
| [13] |
Curatti L, Rubio LM. Challenges to develop nitrogen-fixing cereals by direct nif-gene transfer[J]. Plant Science, 2014, 225: 130-137. DOI:10.1016/j.plantsci.2014.06.003 |
| [14] |
Manyani H, Rey L, Palacios JM, et al. Gene products of the hupGHIJ operon are involved in maturation of the iron-sulfur subunit of the [NiFe] hydrogenase from Rhizobium leguminosarum bv. viciae[J]. J Bacteriol, 2005, 187(20): 7018-7026. DOI:10.1128/JB.187.20.7018-7026.2005 |
| [15] |
Cecilia B, Belén B, Juan I, et al. Diversity and evolution of hydrogenase systems in rhizobia[J]. Applied & Environmental Microbiology, 2002, 68(10): 4915-4924. |
| [16] |
Ratledge C, Dover LG. Iron metabolism in pathogenic bacteria[J]. Annual Reviews in Microbiology, 2000, 54(1): 881-941. DOI:10.1146/annurev.micro.54.1.881 |
| [17] |
Raymond KN, Dertz EA. Biochemical and physical properties of siderophores[M]. Iron Transport in Bacteria. Washington DC: ASM Press, 2004.
|
| [18] |
Challis GL. A widely distributed bacterial pathway for siderophore biosynthesis independent of nonribosomal peptide synthetases[J]. Chembiochem, 2005, 8(13): 601-611. |
| [19] |
Donadio S, Monciardini P, Sosio M. Polyketide synthases and nonribosomal peptide synthetases:the emerging view from bacterial genomics[J]. Natural Product Reports, 2007, 24(5): 1073-1109. DOI:10.1039/b514050c |
| [20] |
Persmark M, Pittman P, Buyer JS, et al. Isolation and structure of rhizobactin 1021, a siderophore from the alfalfa symbiont Rhizobium meliloti 1021[J]. Cheminform, 1993, 24(39): 3950-3956. |
| [21] |
Dilworth MJ, Carson KC, Giles RGF, et al. Rhizobium leguminosarum bv. viciae produces a novel cyclic trihydroxamate siderophore, vicibactin[J]. Microbiology, 1998, 144(3): 781-791. |
| [22] |
Smith MJ, Shoolery JN, Schwyn B, et al. Rhizobactin, a structurally novel siderophore from Rhizobium meliloti[J]. Journal of the American Chemical Society, 1985, 107(6): 1739-1743. DOI:10.1021/ja00292a047 |
| [23] |
Modi M, Shah KS, Modi VV. Isolation and characterisation of catechol-like siderophore from cowpea Rhizobium RA-1[J]. Archives of Microbiology, 1985, 141(2): 156-158. |
| [24] |
Patel HN, Chakraborty RN, Desai S B. Isolation and partial characterization of phenolate siderophore from Rhizobium leguminosarum IARI 102[J]. FEMS Microbiology Letters, 2010, 56(2): 131-134. |
| [25] |
Carter RA, Worsley PS, Sawers G, et al. The vbs genes that direct synthesis of the siderophore vicibactin in Rhizobium leguminosarum:their expression in other genera requires ECF sigma factor RpoI[J]. Molecular Microbiology, 2002, 44(5): 1153-1166. DOI:10.1046/j.1365-2958.2002.02951.x |
| [26] |
Lynch D, O'Brien J, Welch T, et al. Genetic organization of the region encoding regulation, biosynthesis, and transport of rhizobactin 1021, a siderophore produced by Sinorhizobium meliloti[J]. J Bacteriol, 2001, 183(8): 2576. DOI:10.1128/JB.183.8.2576-2585.2001 |
| [27] |
Pfleger BF, Lee JY, Somu RV, et al. Characterization and analysis of early enzymes for petrobactin biosynthesis in Bacillus anthracis[J]. Biochemistry, 2007, 46(13): 4147-4157. DOI:10.1021/bi6023995 |
| [28] |
Postle K, Larsen RA. TonB-dependent energy transduction between outer and cytoplasmic membranes[J]. Biometals, 2007, 20(3-4): 453-465. DOI:10.1007/s10534-006-9071-6 |
| [29] |
Noinaj N, Guillier M, Barnard TJ, et al. TonB-dependent transporters:regulation, structure, and function[J]. Annual Review of Microbiology, 2010, 64: 43-60. DOI:10.1146/annurev.micro.112408.134247 |
| [30] |
Chatterjee A, O'Brian MR. Rapid evolution of a bacterial iron acquisition system[J]. Molecular Microbiology, 2018, 108(1): 90-100. |
| [31] |
Vojtech S, Spanning RJMV, Igor K. Ferric reductase A is essential for effective iron acquisition in Paracoccus denitrificans[J]. Microbiology, 2009, 155(4): 1294-1301. DOI:10.1099/mic.0.022715-0 |
| [32] |
Wang S, Wu Y, Outten FW. Fur and the novel regulator YqjI control transcription of the ferric reductase gene yqjH in Escherichia coli[J]. J Bacteriol, 2011, 193(2): 563-574. DOI:10.1128/JB.01062-10 |
| [33] |
Matzanke BF, Stefan A, Volker S, et al. FhuF, part of a siderophore-reductase system[J]. Biochemistry, 2004, 43(5): 1386. DOI:10.1021/bi0357661 |
| [34] |
Small SK, O'Brian MR. The Bradyrhizobium japonicum frcB gene encodes a diheme ferric reductase[J]. J Bacteriol, 2011, 193(16): 4088-4094. DOI:10.1128/JB.05064-11 |
| [35] |
Mckie A. The role of Dcytb in iron metabolism:an update[J]. Biochem Soc Trans, 2008, 36(6): 1239-1241. DOI:10.1042/BST0361239 |
| [36] |
Noya F, Arias A, Fabiano E. Heme compounds as iron sources for nonpathogenic Rhizobium bacteria[J]. J Bacteriol, 1997, 179(9): 3076-3078. DOI:10.1128/jb.179.9.3076-3078.1997 |
| [37] |
Nienaber A, Hennecke H, Fischer HM. Discovery of a haem uptake system in the soil bacterium Bradyrhizobium japonicum[J]. Molecular Microbiology, 2001, 41(4): 787-800. |
| [38] |
Wexler M, Yeoman KH, Stevens JB, et al. The Rhizobium leguminosarum tonB gene is required for the uptake of siderophore and haem as sources of iron[J]. Molecular Microbiology, 2001, 41(4): 801-816. |
| [39] |
Battistoni F, Platero R, Duran R, et al. Identification of an iron-regulated, hemin-binding outer membrane protein in Sinorhizobium meliloti[J]. Applied and Environmental Microbiology, 2002, 68(12): 5877-5881. DOI:10.1128/AEM.68.12.5877-5881.2002 |
| [40] |
Vanesa A, O'Brian MR, Elena F. ShmR is essential for utilization of heme as a nutritional iron source in Sinorhizobium meliloti[J]. Applied & Environmental Microbiology, 2008, 74(20): 6473-6475. |
| [41] |
Expert D, O'Brian MR. Molecular aspects of iron metabolism in pathogenic and symbiotic plant-microbe associations[M]. Springerbriefs in Molecular Science, 2012.
|
| [42] |
Sumant P, O'Brian MR. The hmuQ and hmuD genes from Bradyrhizobium japonicum encode heme-degrading enzymes[J]. J Bacteriol, 2006, 188(18): 6476. DOI:10.1128/JB.00737-06 |
| [43] |
Hantke K. Is the bacterial ferrous iron transporter FeoB a living fossil?[J]. Trends in microbiology, 2003, 11(5): 192-195. DOI:10.1016/S0966-842X(03)00100-8 |
| [44] |
Sankari S, O'Brian MR. The Bradyrhizobium japonicum ferrous iron transporter FeoAB is required for ferric iron utilization in free-living aerobic cells and for symbiosis[J]. Journal of Biological Chemistry, 2016, 291(30): 15653. DOI:10.1074/jbc.M116.734129 |
| [45] |
Mourad S, Simon L, Dozois CM. A SitABCD homologue from an avian pathogenic Escherichia coli strain mediates transport of iron and manganese and resistance to hydrogen peroxide[J]. Microbiology, 2006, 152(3): 745-758. DOI:10.1099/mic.0.28682-0 |
| [46] |
Davies BW, Walker GC. Disruption of sitA compromises Sinorhi-zobium meliloti for manganese uptake required for protection aga-inst oxidative stress[J]. J Bacteriol, 2007, 189(5): 2101-2109. DOI:10.1128/JB.01377-06 |
| [47] |
Slatni T, Krouma A, Aydi S, et al. Growth, nitrogen fixation and ammonium assimilation in common bean (Phaseolus vulgaris L) subjected to iron deficiency[J]. Plant and Soil, 2008, 312(1-2): 49-57. DOI:10.1007/s11104-007-9481-4 |
| [48] |
Moreau S, Meyer J, Puppo A. Uptake of iron by symbiosomes and bacteroids from soybean nodules[J]. FEBS Letters, 1995, 361(2-3): 225-228. DOI:10.1016/0014-5793(95)00155-3 |
| [49] |
Levier K, Day DA, Guerinot ML. Iron uptake by symbiosomes from soybean root nodules[J]. Plant Physiology, 1996, 111(3): 893-900. DOI:10.1104/pp.111.3.893 |
| [50] |
Kaiser BN, Moreau S, Castelli J, et al. The soybean NRAMP homologue, GmDMT1, is a symbiotic divalent metal transporter capable of ferrous iron transport[J]. The Plant Journal, 2003, 35(3): 295-304. DOI:10.1046/j.1365-313X.2003.01802.x |
| [51] |
Pierre O, Engler G, Hopkins J, et al. Peribacteroid space acidification:a marker of mature bacteroid functioning in Medicago truncatula nodules[J]. Plant, Cell & Environment, 2013, 36(11): 2059-2070. |
| [52] |
Gill Jr PR, Neilands JB. Cloning a genomic region required for a high-affinity iron-uptake system in Rhizobium meliloti 1021[J]. Molecular Microbiology, 1989, 3(9): 1183-1189. DOI:10.1111/j.1365-2958.1989.tb00268.x |
| [53] |
Fabiano E, Gill PR, Noya F, et al. Siderophore-mediated iron acquisition mutants in Rhizobium meliloti 242 and its effect on the nodulation kinetic of alfalfa nodules[J]. Symbiosis, 1995, 19: 197-211. |
| [54] |
Cuív PÓ, Keogh D, Clarke P, et al. The hmuUV genes of Sinorhizobium meliloti 2011 encode the permease and ATPase components of an ABC transport system for the utilization of both haem and the hydroxamate siderophores, ferrichrome and ferrioxamine B[J]. Molecular Microbiology, 2008, 70(5): 1261-1273. DOI:10.1111/j.1365-2958.2008.06479.x |
| [55] |
Chao T, Becker A, Buhrmester J, et al. The Sinorhizobium meliloti fur gene regulates, with dependence on Mn (Ⅱ), transcription of the sitABCD operon, encoding a metal-type transporter[J]. J Bacteriol, 2004, 186(11): 3609-3620. DOI:10.1128/JB.186.11.3609-3620.2004 |
| [56] |
Crespo-Rivas JC, Navarro-Gómez P, Alias-Villegas C, et al. Sinorhizobium fredii HH103 RirA is required for oxidative stress resistance and efficient symbiosis with soybean[J]. International Journal of Molecular Sciences, 2019, 20(3): 787. DOI:10.3390/ijms20030787 |
| [57] |
Papanikolaou G, Pantopoulos K. Iron metabolism and toxicity[J]. Toxicology and Applied Pharmacology, 2005, 202(2): 199-211. DOI:10.1016/j.taap.2004.06.021 |
| [58] |
Johnston AW, Todd JD, Curson AR, et al. Living without Fur:the subtlety and complexity of iron-responsive gene regulation in the symbiotic bacterium Rhizobium and other α-proteobacteria[J]. Biometals, 2007, 20(3-4): 501-511. DOI:10.1007/s10534-007-9085-8 |
| [59] |
Rodionov DA, Gelfand MS, Todd JD, et al. Computational reconstruction of iron-and manganese-responsive transcriptional networks in α-proteobacteria[J]. PLoS Comput Biol, 2006, 2(12): e163. DOI:10.1371/journal.pcbi.0020163 |
| [60] |
Hamza I, Chauhan S, Hassett R, et al. The bacterial Irr protein is required for coordination of heme biosynthesis with iron availability[J]. Journal of Biological Chemistry, 1998, 273(34): 21669. DOI:10.1074/jbc.273.34.21669 |
| [61] |
O'Brian MR. Perception and homeostatic control of iron in the Rhizobia and related bacteria[J]. Annual Review of Microbiology, 2015, 69: 229-245. DOI:10.1146/annurev-micro-091014-104432 |
| [62] |
Jianhua Y, Indu S, Andrea L, et al. Bradyrhizobium japonicum senses iron through the status of haem to regulate iron homeostasis and metabolism[J]. Molecular Microbiology, 2010, 60(2): 427-437. |
| [63] |
Todd JD, Sawers G, Rodionov DA, et al. The Rhizobium leguminosarum regulator IrrA affects the transcription of a wide range of genes in response to Fe availability[J]. Molecular Genetics & Genomics, 2006, 275(6): 564-577. |
| [64] |
Rudolph G, Semini G, Hauser F, et al. The Iron control element, acting in positive and negative control of iron-regulated Bradyrhizobium japonicum genes, is a target for the Irr protein[J]. J Bacteriol, 2006, 188(2): 733. DOI:10.1128/JB.188.2.733-744.2006 |
| [65] |
Small SK, Puri SI, O'Brian MR. Positive control of ferric siderophore receptor gene expression by the Irr protein in Bradyrhizobium japonicum[J]. J Bacteriol, 2009, 191(5): 1361-1368. DOI:10.1128/JB.01571-08 |
| [66] |
Gesine R, Geo S, Felix H, et al. The Iron control element, acting in positive and negative control of iron-regulated Bradyrhizobium japonicum genes, is a target for the Irr protein[J]. J Bacteriol, 2006, 188(2): 733. DOI:10.1128/JB.188.2.733-744.2006 |
| [67] |
Qi Z, Hamza I, O'Brian MR. Heme is an effector molecule for iron-dependent degradation of the bacterial iron response regulator (Irr) protein[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(23): 13056-13061. DOI:10.1073/pnas.96.23.13056 |
| [68] |
Jianhua Y, Koichiro I, O'Brian MR. Two heme binding sites are involved in the regulated degradation of the bacterial iron response regulator (Irr) protein[J]. Journal of Biological Chemistry, 2005, 280(9): 7671-7676. DOI:10.1074/jbc.M411664200 |
| [69] |
Ishikawa H, Nakagaki M, Ai B, et al. Unusual heme binding in the bacterial iron response regulator protein (Irr):spectral characterization of heme binding to heme regulatory motif[J]. Biochemistry, 2011, 50(6): 1016-1022. DOI:10.1021/bi101895r |
| [70] |
Chloe S, White GF, Todd JD, et al. Heme-responsive DNA binding by the global iron regulator Irr from Rhizobium leguminosarum[J]. Journal of Biological Chemistry, 2010, 285(21): 16023-16031. DOI:10.1074/jbc.M109.067215 |
| [71] |
Todd JD, Wexler M, Sawers G, et al. RirA, an iron-responsive reg-ulator in the symbiotic bacterium Rhizobium leguminosarum[J]. Microbiology, 2002, 148(12): 4059-4071. DOI:10.1099/00221287-148-12-4059 |
| [72] |
Yeoman KH, Curson AR, Todd JD, et al. Evidence that the Rhizobium regulatory protein RirA binds to cis-acting iron-responsive operators (IROs) at promoters of some Fe-regulated genes[J]. Microbiology, 2004, 150(12): 4065-4074. DOI:10.1099/mic.0.27419-0 |
| [73] |
Chao T, Buhrmester J, Hansmeier N, et al. Role of the regulatory gene rirA in the transcriptional response of Sinorhizobium meliloti to iron limitation[J]. Applied and Environmental Microbiology, 2005, 71(10): 5969-5982. DOI:10.1128/AEM.71.10.5969-5982.2005 |
| [74] |
Battisti JM, Smitherman LS, Sappington KN, et al. Transcriptional regulation of the heme binding protein gene family of Bartonella quintana is accomplished by a novel promoter element and iron response regulator[J]. Infection & Immunity, 2007, 75(9): 4373-4385. |
| [75] |
Ngok-Ngam P, Ruangkiattikul N, Mahavihakanont A, et al. Roles of Agrobacterium tumefaciens RirA in iron regulation, oxidative stress response, and virulence[J]. J Bacteriol, 2009, 191(7): 2083-2090. DOI:10.1128/JB.01380-08 |
| [76] |
Viguier C, Cuív PÓ, Clarke P, et al. RirA is the iron response regulator of the rhizobactin 1021 biosynthesis and transport genes in Sinorhizobium meliloti 2011[J]. FEMS Microbiology Letters, 2005, 246(2): 235-242. DOI:10.1016/j.femsle.2005.04.012 |
| [77] |
Martinez MTP, Martinez AB, Crack JC, et al. Sensing iron availa-bility via the fragile [4Fe-4S] cluster of the bacterial transcrip-tional repressor RirA[J]. Chemical Science, 2017, 8(12): 8451-8463. DOI:10.1039/C7SC02801F |
| [78] |
Costa D, Amarelle V, Valverde C, et al. The Irr and RirA proteins participate in a complex regulatory circuit and act in concert to modulate bacterioferritin expression in Ensifer meliloti 1021[J]. Applied & Environmental Microbiology, 2017, 83(16): 817-895. |
| [79] |
Amarelle V, Koziol U, Rosconi F, et al. A new small regulatory protein, HmuP, modulates haemin acquisition in Sinorhizobium meliloti[J]. Microbiology, 2010, 156(6): 1873-1882. DOI:10.1099/mic.0.037713-0 |
| [80] |
Rosalba EH, O'Brian MR. HmuP is a coactivator of Irr-dependent expression of heme utilization genes in Bradyrhizobium japonicum[J]. J Bacteriol, 2012, 194(12): 3137-3143. DOI:10.1128/JB.00071-12 |
| [81] |
ó Cuív P, Clarke P, Lynch D, et al. Identification of rhtX and fptX, novel genes encoding proteins that show homology and function in the utilization of the siderophores rhizobactin 1021 by Sinorhizobium meliloti and pyochelin by Pseudomonas aeruginosa, Respectively[J]. J Bacteriol, 2004, 186(10): 2996. DOI:10.1128/JB.186.10.2996-3005.2004 |