microRNA(miRNA)普遍存在于真核生物细胞内, 是一类长度为21-24 nt的非编码RNA, 通过剪切互补的靶基因mRNA或抑制靶基因的翻译来行使其生物学功能[1-2]。miRNA不仅调控了植物的生长发育过程, 而且在植物抗逆、抗病的过程中发挥着关键的作用, 主要是通过调控相关内源基因的表达使植物对外界胁迫作出响应[3]。miR393通过下调生长素受体基因TIR1和AFB的表达阻断生长素信号的传导途径, 提高拟南芥对细菌性病害的抗性[4]。miR169通过调控靶基因NF-YA5(Nuclear factor YA5), 在拟南芥抗旱中起着重要的作用[5]。随着测序技术的飞速发展, 大量新的miRNA被发现。目前miRBase 22.1(http://www.mirbase.org/)版本已经收录了38 589个miRNA, 然而大多数miRNA的生物学功能尚不明确。miRNA的功能解析, 最关键的是鉴定出miRNA的靶基因。miRNA靶基因的鉴定常用的方法有Northern blot[6]、Western Blot[7]、降解组测序(Degradome sequencing)[8]、MirTrap、RACE(Rapid amplication of cDNA ends)[9]和荧光素酶报告系统[10-11]。利用生物信息学预测miRNA靶基因, 不同的方法和不同的参数条件都可能会得到不同的靶基因。因此, 对预测的靶基因进行实验验证是必需的[12-13]。鉴于这一验证过程的复杂性、耗时性, 建立一种准确且快速地验证miRNA靶基因的方法对研究miRNA的生物学功能具有非常重要的意义。基因的瞬时表达技术为miRNA靶基因的快速验证提供了支撑。在植物中, 目前大多数靶基因验证的瞬时表达系统都是基于农杆菌介导的烟草瞬时表达体系, 主要是将miRNA与带有GFP(或YFP)的候选靶基因同时转染到烟草叶片中, 通过荧光强弱来验证靶基因[10]。水稻是单子叶植物, 运用烟草瞬时表达系统验证水稻miRNA的靶基因不够严谨, 毕竟烟草细胞不同于水稻细胞。GFP发出荧光需要短波光的激发, 而短波光对植物细胞有毒害作用。因此, 在水稻原生质体内, 利用化学发光的荧光素酶LUC来验证水稻miRNA靶基因更具有优势。
miR169是植物体内比较保守的一个大家族, miR169o可以被干旱胁迫诱导表达, 而过表达miR169o则增强了植物对干旱的耐受性[14]。此外, 过表达miR169o能促进水稻的生长, 降低水稻对白叶枯病的抗病性[15]。前期通过qRT-PCR和5'RACE证明了靶基因LOC_Os03g48970.1(后文简称48970)受miR169o的抑制作用[16]。因此, 本实验以miR169o及其靶基因48970的对应关系为参照, 系统研究了在农杆菌介导的烟草和PEG4000介导的水稻原生质体两种瞬时表达体系下miRNA的动态变化, 获得了各自水稻靶基因验证的最适体系, 建立了水稻miRNA靶基因验证的快速检测系统, 以期对水稻miRNA功能的解析提供技术支持。
1 材料与方法 1.1 材料NEB限制性内切酶、T4 DNA连接酶; 天根生物科技公司质粒提取试剂盒; Invitrogen公司TRIzol试剂; 诺唯赞公司cDNA转录酶和实时定量PCR试剂盒; 日本Yakult公司纤维素酶R-10和离析酶R-10;pUC19-nLUC, pCAMBIA1300载体, 农杆菌GV3101, 水稻日本晴, 本氏烟均为实验室所保存。大肠杆菌感受态为全式金Trans1-T1 Phage Resistant; 抗生素及浓度:氨苄青霉素100 μg/mL, 卡那霉素50 μg/mL, 庆大霉素50 μg/mL; 荧光活体成像仪为BERTHOLD TECHNOLOGIES LB 985 NightSHADE; Promega Luminometor荧光活性定量仪; ABI 7500实时定量PCR仪。
所用引物由北京六合华大基因科技有限公司合成, 引物序列见表 1。
采用酶解法制备原生质体[17]。日本晴种子于1/2 MS培养基(2.21 g/L MS, 20 g/L Sugar, 3‰植物凝胶)中28℃培养10 d-12 d, 将幼苗茎部切成0.5 mm大小, 立即放入有酶解液(0.6 mol/L甘露醇, 1.3% Cellulose, 0.75% Macerozyme, 20 mmol/L MES pH5.7, 55℃加热10 min, 冷却后加入0.1% BSA, 2 mmol/L CaCl2, 5 mmol/L β-巯基乙醇, 50 μg/mL羧苄, 过滤除菌)的三角瓶中置于28℃, 70 r/min的摇床中酶解6 h-8 h。35 μm尼龙筛网过滤酶解液, W5溶液(9.0 g/L NaCl, 18.4 g/L CaCl2, 0.37 g/L KCl, 0.9 g/L Glucose, 0.3 g/L MES, KOH调pH至5.7, 灭菌)反复冲洗残渣, 离心(3 200 r/min, 5 min)收集原生质体, 弃上清。
1.2.2 原生质体转化适量W5溶液重悬原生质体至浓度在(2-5)×106个细胞/mL, 100 μL原生质中加入4 μg质粒, 混匀后加入110 μL 40% PEG溶液(4 mL配方:1.6 g PEG, 400 μL 1 mol/L CaCl2, 1 333.3 μL 0.6 mol/L甘露醇, 加灭菌水定容至4 mL), 混匀后室温孵育15 min, 加入1 mL W5, 颠倒混匀, 3 200 r/min, 离心5 min, 弃上清, 再加入1 mL W5, 3 200 r/min, 离心5 min, 弃上清, 加入1 mL W5溶液, 静置培养。
1.2.3 原生质体总RNA的提取参照Trizol说明书, 稍作改进提取RNA, 加入500 μL Trizol, 剧烈震荡, 室温放置2-3 min, 加入200 μL氯仿, 颠倒混匀, 室温放置5 min, 4℃, 12 000 r/min离心15 min, 转移上清, 加入等体积的异丙醇, 室温放置10 min, 4℃, 12 000 r/min离心10 min, 弃上清, 75%乙醇洗涤沉淀, 最后用30 μL RNase-free的ddH2O溶解。
1.2.4 cDNA的反转录及实时荧光定量PCR按诺唯赞公司的反转录试剂盒说明进行去基因组和反转录, 反转时加入0.5 μL 10 μmol/L的颈环引物。参照诺唯赞AceQ qPCR SYBR Green Master试剂盒进行qRT-PCR检测。miR169o相对表达量按照2-ΔΔCt法计算, 其中ΔΔCt =(Ct样品-CtU6)TimeX -(Ct对照样品-CtU6)Time12, TimeX表示任意时间点, Ct为荧光阈值。
1.2.5 LUC活性的检测原生质体转化后不同时间点取样, 取150 μL样品, 剧烈震荡, 加入至不透明96孔板中, 加入0.75 μL LUC底物, 利用Luminometer检测LUC的相对活性。取500 μL样品至24孔板中, 加入2.5 μL LUC底物, 利用CCD成像检测荧光。
1.2.6 农杆菌介导的烟草瞬时表达[18]将pCAMBIA1300-miR169o, pCAMBIA1300-LUC, pCAMBIA1300-LUC-48970, pCAMBIA1300-LUC-48970m3等质粒分别导入到农杆菌GV3101, 涂布在LB固体培养基上(卡那霉素50 μg/mL+庆大霉素50 μg/mL), PCR验证筛选阳性转化子。烟草培养条件, 每周期光照16 h, 25℃, 黑暗8 h, 22℃, 注射一个月左右的烟草。在注射前2 d, 从平板上挑取单菌落摇菌, 28℃过夜培养, 然后按照1:1 000的比例转接到新的培养基中。5 000 r/min, 22℃, 离心5 min收集菌体。用含有10 mmol/L MgCl2, 10 mmol/L MES, 150 μmol/L乙酰丁香酮, pH5.7的转化缓冲液重悬菌体至终浓度为OD600=1.0, 室温静置3 h后, 按照各处理将两种农杆菌按体积1:1混合, 注射烟草叶片。不同时间取样, 观察LUC活性以及检测miR169o的表达量。
2 结果 2.1 相关载体的构建 2.1.1 miR169o过表达载体的构建PCR扩增得到444 bp的miR169o前体基因(MIR169o), 经BamH I和PstI酶切后插入同样酶切的pUC19-nLUC载体上, 得到实验所需的miR169o过表达载体pUC19-miR169o(图 1-A)。pUC19-miR169o与pCAMBIA-1300-nLUC分别用SacI和PstI消化后连接, 将MIR-169o插入pCAMBIA1300载体中, 得到pCAMBIA-1300-miR169o(图 1-B)。

|
| 图 1 pUC19-miR169o(A)和pCAMBIA1300-miR169o(B)载体图 |
分别将pUC19-nLUC和pCAMBIA1300-nLUC载体中的nLUC片段替换为全长的LUC片段, 同时引入SacI、SalI、PstI等酶切位点, 分别得到适用于水稻原生质体系统和烟草瞬时表达系统的pUC19-LUC(图 2-A)和pCAMBIA1300-LUC载体(图 2-B)。
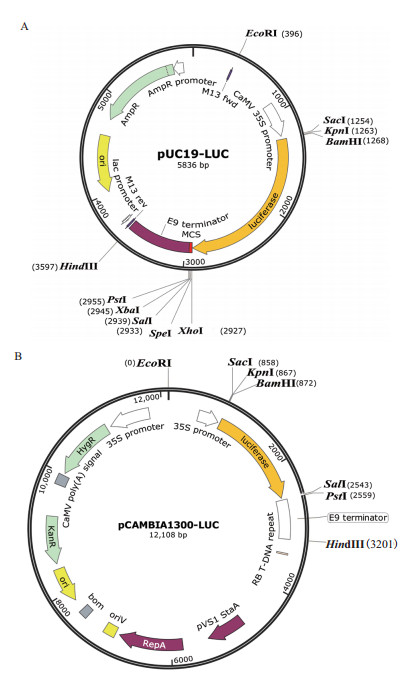
|
| 图 2 pUC19-LUC(A)和pCAMBIA1300-LUC(B)载体图 |
分别从RGAP(Rice Genome Annotation Project)和miRBase数据库下载LOC_Os03g48970.1的CDS序列和miR169o的成熟序列。通过psRNA Target(http://plantgrn.noble.org/psRNATarget/)分析, 发现miR169o结合在48970的3' UTR区(碱基位置为1 237-1 256)。我们将这20个碱基的结合序列插入到LUC基因的3' UTR区(图 3-A), 得到实验所需的pUC19-LUC-48970和pCAMBIA1300-LUC-48970。miRNA与靶基因通过碱基互补配对方式结合, 而结合的核心区域位于miRNA第9-11个碱基[19]。将CTA 3个碱基插入靶基因, 插入位置正好对应于miRNA第10-11个碱基中间, 使得miR169o无法剪切48970m3。将48970m3序列插入到LUC基因3' UTR中, 得到对miR169o不敏感的48970m3表达载体(图 3-B), 即pUC19-LUC-48970m3或pCAMBIA1300-LUC-48970m3。
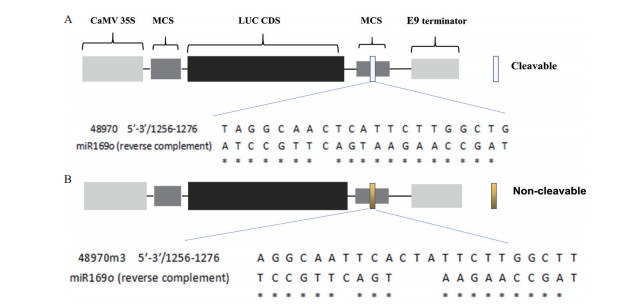
|
| 图 3 LUC-48970(A)和LUC-48970m3(B)载体示意图 |
将pUC19-miR169o分别与pUC19-LUC, pUC19-LUC-48970和pUC19-LUC-48970m3共转化水稻原生质体。利用荧光活体成像仪和Luminometor检测转化12 h、18 h、24 h及36 h后LUC活性, 发现在24 h时, 荧光亮度最大。在3组组合中, pUC19-miR169o与pUC19-LUC-48970共转化后, 荧光活性最弱(图 4-A和4-B), 表明miR169o可以抑制靶基因LOC_Os03g48970.1的转录表达, 而水稻原生质体系统可以用于水稻miRNA靶基因的检测。此外, 利用茎环qRT-PCR检测了转化后不同时间点miR169o的表达水平, 在转化后12-36 h之间miR169o的水平与表达时间具有正相关性, 12 h表达量最低, 而在36 h的表达量最高(图 4-C)。综合荧光检测效果和miR169o的表达水平, 建议miRNA靶基因验证的恰当检测时间在水稻原生质体转化后24-36 h之间。
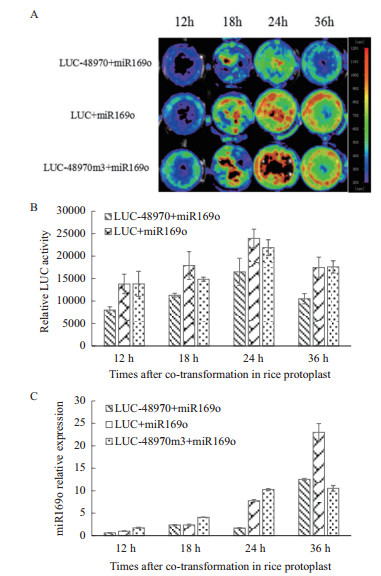
|
| A:荧光强度的荧光活体成像检测。红色斑点中心银灰色是荧光最强区; B:荧光强度的Luminometor检测; C:水稻原生质体转化后成熟miR169o的动态水平检测 图 4 水稻原生质体表达系统中的靶基因验证 |
将pCAMBIA1300-miR169o过表达载体、pCAMBIA1300-LUC-48970和pCAMBIA1300-LUC-48970m3融合载体, 分别导入农杆菌中, 共注射烟草叶片进行共表达。在注射烟草48 h、72 h和96 h后检测LUC活性, 发现在过表达72 h的荧光强度最高(图 5-A); miR169o和48970共表达中的LUC活性明显弱于miR169o和LUC, miR169o和48970m3共表达中的LUC活性, 表明烟草瞬时表达系统可以用于水稻miRNA的靶基因验证。此外, 在24 h、48 h、72 h和96 h四个时间点分别取样, 以24 h LUC与miR169o共注射处理作为对照, 以EF1为内参, 通过茎环qRT-PCR检测成熟miR169o的动态水平。结果表明注射农杆菌24-96 h后, 成熟miR169o的表达水平逐渐增高, 在48 h时, miR169o的表达水平已经提高到20倍左右(图 5-B)。因此, 综合荧光强度测定和成熟miR169o的水平, 建议miRNA靶基因验证的恰当检测时间在烟草瞬时表达后48-72 h之间。
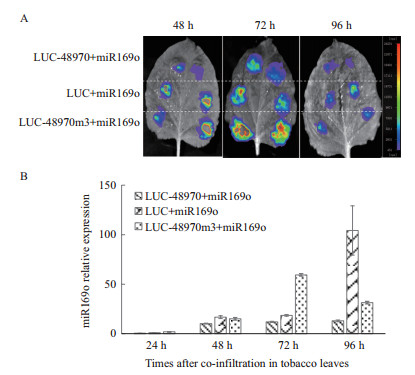
|
| A:农杆菌注射烟草后不同时间点荧光亮度的检测。红色斑点中心银灰色区域是荧光太强, 超过仪器检测的最高值; B:农杆菌注射烟草后不同时间点miR169o表达量的检测 图 5 烟草瞬时表达系统中miR169o的靶基因验证 |
在miRNA靶基因验证实验中, 前人更多地关注靶基因的表达, 而鲜有关注miRNA的动态表达。如Li等[20]共表达了artificial miRNA与其对应的靶基因, 通过Western Blot分析靶基因编码产物的表达差异; Martinho等[21]过表达了miRNA, 检测了转录水平上内源靶基因的差异性表达。而本研究测定了在共表达后不同时间点LUC活性和miRNA表达水平, 从而可以动态反映miR169o对48970的特异调控过程。
无论是在水稻原生质体系统还是在烟草瞬时表达系统中, miR169o的表达水平总体上都会随着时间的延长而呈现一定的上升。三种处理组合间miR169o的表达水平虽然存在着一定程度的差异, 但都是在miR169o和LUC-48970组合中miR169o的表达水平较低, 而其它两种组合中miR169o水平相对较高。相较于其它组合, miR169o和LUC-48970组合中的LUC活性明显降低, 表明靶基因的转录被明显地抑制。因此, 本系统中miRNAs水平的差异对检测结果并没有造成影响, 究其表达水平差异的具体原因则尚不明确。
随着测序技术的发展, 已经发现了大量的水稻miRNA在应对生物胁迫和非生物胁迫中发挥重要作用, 而在解析这些miRNA调控机制的过程中最重要的内容是明确miRNA的靶基因及其功能。本文建立的靶基因验证系统, 可以为miRNA靶基因的筛选、验证提供极大的便利条件, 节省时间和实验成本, 为大批量的miRNA功能的解析提供技术支撑。当然, 由于miRNA和LUC融合基因都处于动态的表达过程中, 完全的均一化各处理中miRNA和LUC融合基因的转化效率和表达量是比较困难的, 因此, 利用本系统进行筛选验证, 并结合其它靶基因验证方法精确定位靶位点才是miRNA靶基因验证的完美解决手段。
4 结论本研究确立了适合水稻miRNA靶基因验证的系统。在水稻原生质体转化体系中, 恰当的检测时间点在转化后24-36 h; 在烟草瞬时表达体系中, 恰当的检测时间点在共注射后48-72 h。
| [1] |
Bartel DP. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281-297. DOI:10.1016/S0092-8674(04)00045-5 |
| [2] |
Debat HJ, Ducasse DA. Plant microRNAs:recent advances and future challenges[J]. Plant Molecular Biology Reporter, 2014, 32(6): 1257-1269. DOI:10.1007/s11105-014-0727-z |
| [3] |
Shukla LI, Chinnusamy V, Sunkar R. The role of microRNAs and other endogenous small RNAs in plant stress responses[J]. Biochimica Et Biophysica Acta, 2008, 1779(11): 743-748. DOI:10.1016/j.bbagrm.2008.04.004 |
| [4] |
Navarro L, Dunoyer P, Jay F, et al. A plant miRNA contributes to antibacterial resistance by repressing auxin signaling[J]. Science, 2006, 312(5772): 436-439. DOI:10.1126/science.1126088 |
| [5] |
Li WX, Oono Y, Zhu J, et al. The Arabidopsis NFYA5 transcription factor is regulated transcriptionally and posttranscriptionally to promote drought resistance[J]. Plant Cell, 2008, 20(8): 2238-2251. DOI:10.1105/tpc.108.059444 |
| [6] |
Llave C, Xie Z, Kasschau KD, et al. Cleavage of scarecrow-like mRNA targets directed by a class of Arabidopsis miRNA[J]. Science, 2002, 297(5589): 2053-2056. DOI:10.1126/science.1076311 |
| [7] |
Li S, Liu L, Zhuang X, et al. MicroRNAs inhibit the translation of target mRNAs on the endoplasmic reticulum in Arabidopsis[J]. Cell, 2013, 153(3): 562-574. DOI:10.1016/j.cell.2013.04.005 |
| [8] |
Thomson DW, Bracken CP, Goodall GJ. Experimental strategies for microRNA target identification[J]. Nucleic Acids Research, 2011, 39(16): 6845-6853. DOI:10.1093/nar/gkr330 |
| [9] |
Song C, Jia Q, Fang J, et al. Computational identification of citrus microRNAs and target analysis in citrus expressed sequence tags[J]. Plant Biology, 2010, 12(6): 927-934. DOI:10.1111/j.1438-8677.2009.00300.x |
| [10] |
Li Y, Zhao SL, Li JL, et al. Osa-miR169 negatively regulates rice immunity against the blast fungus Magnaporthe oryzae[J]. Front Plant Sci, 2017, 8(6): 2. |
| [11] |
Boutla A, Delidakis C, Tabler M. Developmental defects by antisense-mediated inactivation of micro-RNAs 2 and 13 in Drosophila and the identification of putative target genes[J]. Nucleic Acids Research, 2003, 31(17): 4973-4980. DOI:10.1093/nar/gkg707 |
| [12] |
Baek D, Villén J, Shin C, et al. The impact of microRNAs on protein output[J]. Nature, 2008, 455(7209): 64-71. DOI:10.1038/nature07242 |
| [13] |
Sethupathy P, Megraw M, Hatzigeorgiou AG. A guide through present computational approaches for the identification of mammalian microRNA targets[J]. Nature Methods, 2006, 3(11): 881-886. DOI:10.1038/nmeth954 |
| [14] |
陈禹彤, 陈华民, 余超, 等. 水稻miR169o及其靶基因OsNF-YAs对缺水胁迫的早期表达模式[J]. 生物技术通报, 2015, 31(8): 76-81. |
| [15] |
Yu C, Chen YT, Cao YQ, et al. Overexpression of miR169o, an overlapping microRNA in response to both nitrogen limitation and bacterial infection, promotes nitrogen use efficiency and susceptibility to bacterial blight in rice[J]. Plant and Cell Physiology, 2018, 59(6): 1234-1247. DOI:10.1093/pcp/pcy060 |
| [16] |
余超.水稻对白叶枯病菌侵染和低氮胁迫反应的共调控因子鉴定[D].北京: 中国农业科学院, 2014. http://cdmd.cnki.com.cn/Article/CDMD-82101-1014326788.htm
|
| [17] |
Chen SB, Tao LZ, Zeng LR, et al. A highly efficient transient protoplast system for analyzing defence gene expression and protein-protein interactions in rice[J]. Molecular Plant Pathology, 2006, 7(5): 417-427. DOI:10.1111/j.1364-3703.2006.00346.x |
| [18] |
Chen HM, Zou Y, Shang YL, et al. Firefly luciferase complementation imaging assay for protein-protein interactions in plants[J]. Plant Physiology, 2008, 146(2): 368-376. DOI:10.1104/pp.107.111740 |
| [19] |
Schwab R, Palatnik JF, Riester M, et al. Specific effects of microRNAs on the plant transcriptome[J]. Developmental Cell, 2005, 8(4): 517-527. DOI:10.1016/j.devcel.2005.01.018 |
| [20] |
Li JF, Chung HS, Niu YJ, et al. Comprehensive protein-based artificial microRNA screens for effective gene silencing in plants[J]. Plant Cell, 2013, 25(5): 1507-1522. DOI:10.1105/tpc.113.112235 |
| [21] |
Martinho C, Confraria A, Elias CA, et al. Dissection of miRNA pathways using Arabidopsis Mesophyll protoplasts[J]. Molecular Plant, 2015, 8(2): 261-275. DOI:10.1016/j.molp.2014.10.003 |





