氮是植物生长发育所必需的主要营养元素之一,然而自然界中大量的氮气必须被还原为硝态氮或者氨态氮后才能被植物吸收利用。在自然界氮素还原中,生物固氮占据主导地位,它是指分子态氮气在生物体内被还原成氨的过程。目前发现只有原核生物能够进行生物固氮[1]。在生物固氮中,根瘤菌与豆科植物形成的共生固氮是目前已知效率最高且被研究最为广泛的一种。然而,根瘤菌的宿主范围主要限于豆科植物,这是限制其被广泛应用于非豆科植物(如禾本科作物)的瓶颈[2]。因此,扩大根瘤菌宿主范围乃至实现非固氮植物的结瘤及固氮是目前农业可持续发展和养分高效利用的关键途径之一。
在根瘤菌-豆科植物共生固氮中,豆科植物形成的根瘤是根瘤菌进行生物固氮的场所。根瘤菌与豆科植物共生固氮包括两个相对独立且密不可分的途径,即根瘤菌入侵和根瘤发育[3-4]。植物根部释放出的类黄酮化合物被匹配的根瘤菌感知,并激活nodD基因表达,诱导合成并释放结瘤因子[5]。这些结瘤因子进而被植物结瘤因子受体蛋白复合物识别,如百脉根中的NFR1/NFR5(Nod factor receptor 1 /Nod factor receptor 5)与共生受体激酶SymRK(Symbiosis Receptor Kinase)形成的复合物,激活下游共生信号通路[6]。近年来,与共生受体激酶有关的分子调控机制得到广泛研究,如Nick4(NFR5-interacting cytoplasmic kinase 4)能够和NFR5相互作用,连接了结瘤因子信号识别和根瘤器官发生的信号通路[7]。小G蛋白ROP6(Rho-like GTPase 6)能够与NFR5互作调控侵染线的生长和根瘤发生[8]。百脉根中的类二氢黄酮醇蛋白DFL1(Dihydroflavonol-4-reductase-like protein 1)能够与NFR5互作,揭示信号通路可能通过负反馈方式直接调控类黄酮的合成[9]。共生信号的激活最终可以引起细胞内钙离子内流[10]。DMI1(Does not Make Infections 1)作为核膜上的离子通道,能够通过控制离子出入调节共生信号[11]。细胞核内的钙离子振荡被CCaMK(Calcium- and Calmodulin-dependent protein Kinase)蛋白激酶解码,CCaMK通过磷酸化CYCLOPS,CYCLOPS作为能够结合DNA的转录激活子,能够与DELLA蛋白、NSP1(Nodulation signaling pathway 1)蛋白、NSP2(Nodulation signaling pathway 2)蛋白等形成转录复合物共同激活共生相关基因的表达[12-14]。根瘤菌对豆科植物类黄酮类化合物的响应具有特异性,并且豆科植物相应根瘤菌结瘤因子也具有一定特异性。因此,豆科植物分泌的类黄酮类化合物以及根瘤菌结瘤因子的结构差异是调控根瘤菌宿主范围的一个关键。根瘤菌侵染宿主植物后,根瘤菌沿侵染线到达并定殖于根部皮层细胞,诱导根瘤原基发育。根据形态结构,根瘤大致分为定型根瘤和不定型根瘤。定型根瘤的根瘤原基起始于外皮层细胞,而不定型根瘤的根瘤原基起始于内皮层细胞[15]。在根瘤共生细胞中,根瘤菌分化形成能够固氮的类菌体[16],从而将空气中氮气还原为氨供植物吸收利用。
从进化上推测,根瘤菌-豆科植物互作起源于病原菌-植物的病原反应(Pathogenesis),作为一种外来微生物,根瘤菌入侵仍然会激发植物一定的免疫反应。如在共生互作早期,植物一些免疫应答基因表达量上调,这些防御反应在随后的共生互作中被逐渐降低或者抑制。而作为宿主,植物也进化出相应机制来抑制自身免疫反应进而实现与根瘤菌互惠共生。如苜蓿NAD1(Nodules with Activated Defence)、DNF2(Defective in Nitrogen Fixation 2)等蛋白的缺失特异激发了根瘤中免疫反应,进而导致根瘤菌定殖和类菌体发育中的终止[17-19]。根瘤菌和病原细菌与植物的互作方式是相似的,即不论是共生体(如根瘤菌)还是病原体(如病原假单胞杆菌)抑制植物免疫反应的策略也有可能大致类似。那么植物是如何识别和区分根瘤菌与病原菌,又是如何调控自身免疫反应来“邀请”根瘤菌而不是病原菌的入侵和定殖?总之,宿主防御反应与共生反应的平衡对于根瘤菌入侵和根瘤发生是至关重要的。我们将从根瘤菌MAMP、根瘤菌三型效应蛋白以及根瘤菌定殖等3个方面论述根瘤发生过程中宿主和根瘤菌对植物免疫反应的调控。
1 根瘤菌MAMP在共生互作中作用植物可以通过模式识别受体(Pattern Recog-nition Receptor,PRR)感知微生物相关分子模式(Microbe-Associated Molecular Patterns,MAMP)并激发免疫反应,被称为PTI反应(Pattern-triggered im-munity,模式触发的免疫反应)[20]。该免疫反应在抵御病原菌入侵和调控植物周围环境中微生物种类和组合起重要作用。而微生物MAMP和植物PRR的多样化组合正是调控与不同微生物互作的一个关键因素[21]。根瘤菌是一种典型的革兰氏阴性菌。革兰氏阴性菌细胞壁分为外壁层和内壁层。外壁层最外层是脂多糖,内壁层含有一层结构疏松的肽聚糖。那么根瘤菌和病原菌表面具有的相同的分子模式是怎样被植物识别的;在根瘤发生和共生建立的过程中,豆科植物产生的免疫反应又是如何被根瘤菌和植物调控的?
在拟南芥中,细菌鞭毛蛋白(flagellin或flg22,代表N端保守的22个氨基酸肽段)被LRR(Leucine-rich repeat)受体激酶FLS2(Flagellin Sensing 2)识别[22]。另一个LRR受体激酶BAK1(BRI1-associated receptor kinase)作为细菌鞭毛蛋白的共受体介导了免疫反应的激活[23]。flgII-28,细菌鞭毛蛋白上另一个激发免疫反应的肽段,能够被茄科植物的FLS3(Flagellin-Sensing 3)识别并激发免疫反应[24]。同样,根瘤菌,如百脉根中慢生型根瘤菌(Mesorhizobium loti),接种植物后也激发百脉根免疫反应,这暗示根瘤菌也可能有激发免疫反应的MAMP。被病原菌flg22处理后的百脉根与根瘤菌共生识别大大降低,最终导致根瘤数目减少和根瘤固氮酶活降低,然而在百脉根根瘤发生后,flg22处理并没有抑制根瘤数目和固氮酶活,这表明植物PTI反应特异抑制根瘤菌入侵。然而从M. loti纯化的鞭毛蛋白并不能诱导百脉根的免疫反应。通过对比flg22序列发现,根瘤菌的鞭毛蛋白氨基酸序列发生变异,这些突变可能是植物识别和激发植物免疫反应的关键[25]。因此,这可以从进化上解释根瘤菌可以通过鞭毛蛋白序列的突变逃避植物FLS2介导免疫反应的抑制。
此外,根瘤菌还有很多多糖类的MAMP,如结瘤因子、脂多糖、胞外多糖和肽聚糖等。在拟南芥中受体激酶CERK1(Chitin elicitor receptor kinase 1)和受体蛋白LYM1/LYM3以及水稻中的两个LysM-containing蛋白LYP4和LYP6共同介导细菌细胞壁肽聚糖PGN介导的免疫反应[26-27]。但是目前根瘤菌的细胞壁组成成分肽聚糖有何作用仍未知。
十字花科植物中的Lectin S-domain受体激酶LORE介导了病原菌脂多糖引起的免疫反应[28]。而水稻中的OsCERK1蛋白也与脂多糖介导的免疫反应有关[29]。那么根瘤菌的脂多糖在共生中具有什么功能?据报道苜蓿根瘤菌脂多糖突变体Rm6963与苜蓿结瘤时间延迟并且与野生型根瘤菌相比结瘤竞争能力降低[30],这表明根瘤菌脂多糖在共生中起正调控作用。但是脂多糖在根瘤菌与豆科植物的分子对话中如何发挥正向功能,植物中的哪些蛋白参与了这个过程,这些问题仍然需要我们去探索。
真菌细胞壁组成成分几丁质是一种被广泛研究的MAMP。在拟南芥中,AtCERK1和AtLYK5形成的受体复合物共同介导几丁质引起的免疫反应[31-32]。根瘤菌结瘤因子的化学结构式是脂壳修饰的短链几丁质[33-34]。且结瘤因子受体,如百脉根LjNFR1/LjNFR5和苜蓿MtLYK3/MtNFP,与几丁质受体具有较高同源性[35-39]。在本氏烟草中过表达LjNFR1/LjNFR5或者MtLYK3/MtNFP可以激发叶片细胞程序性死亡[40],这暗示共生信号通路在进化上与几丁质激发的免疫信号通路具有相关性。最近研究还发现,除了在共生反应中起关键作用,结瘤因子还可以诱导细菌鞭毛蛋白受体FLS2的降解,进而抑制MAMP激发的植物免疫反应,拟南芥AtLYK3在介导结瘤因子抑制植物免疫中起关键作用[41]。那么,豆科植物中AtLYK3的同源蛋白是否存在抑制植物免疫反应的功能还有待于研究。
除此之外,有证据表明根瘤菌产生的胞外多糖(Exopolysaccharides,EPS)也参与了根瘤共生。根瘤菌能够产生胞外多糖,并分泌到胞外。这个特性显示出它有可能和结瘤因子一样,是第一批接触到宿主的分子模式[42]。在百脉根中EPS的LysM受体EPR3能够区分匹配和不匹配的根瘤菌的胞外多糖。EPS介导的EPR3识别通路促进了根皮层细胞内的侵染以及根和根瘤菌的有效互作。使用根瘤菌胞外多糖突变体R7AexoB接种野生型百脉根和epr3突变体显示EPR3可能存在共受体[43]。
在MAMP触发的植物PTI反应中,对于MAMP的识别通常是由多个受体蛋白形成的复合物共同进行的。AtFLS2能够和AtBAK1形成共受体复合物共同介导flg22引起的免疫反应;AtCERK1能够和AtLYK5形成共受体复合物共同介导几丁质引起的免疫反应;水稻中的OsCERK1能够和OsCEBiP形成共受体复合物介导几丁质引起的免疫反应;AtCERK1能够和AtLYM1和AtLYM3形成共受体复合物介导PGN引起的免疫反应。而在结瘤因子介导的共生途径中,NFR1能够和NFR5形成复合物共同识别并介导下游的免疫反应[44-46]。因此,共生过程中根瘤菌其他分子模式的识别也有可能是通过共受体复合物介导的。
由于根瘤菌中的很多分子模式与病原菌分子模式结构非常相似,并且在已鉴定的与免疫相关的苜蓿突变体nad1中很多受体类蛋白表达上调,但是哪些分子模式被这些受体类蛋白识别激发免疫反应影响共生过程仍然未知[17-18]。在根瘤菌和植物互相识别的过程中,植物是如何区分匹配的根瘤菌以及不匹配的根瘤菌和病原菌,根瘤菌又是如何利用和调整自身的分子模式以帮助自己与植物的互作过程,这些还存在很多未知。在未来,我们或许可以通过各种技术综合鉴定出这些在共生过程中发挥着重要作用的分子模式以及其相应的受体蛋白,为人们更好的利用这个识别模式奠定基础。
2 根瘤菌释放的效应蛋白触发的免疫反应的调节植物类受体激酶和类受体蛋白识别病原菌的MAMP后触发免疫反应,试图杀死病原菌。病原菌则释放效应蛋白进入植物体内,通常靶定植物PTI反应中的各个组分阻止免疫反应的产生,以便让自己顺利侵入。而植物也进化出识别效应蛋白的R(Resistance)蛋白。R蛋白能够特异识别病原菌释放的效应蛋白,从而激活免疫反应(Effctor-triggered Immunity,ETI)[47]。
Flor[48]提出gene-for-gene假说,即高度特异的R蛋白和效应蛋白一对一地直接结合。如水稻的Pi-ta R蛋白能够直接结合水稻稻瘟病菌Magnaporthe grisea的AvrPita蛋白[49]。Guard model则证明了R蛋白也能够被间接地激活。即R蛋白不检测效应蛋白的直接行为,而是识别R蛋白复合物中宿主蛋白的异常[50]。如效应蛋白AvrRPM1诱导RIN4(RPM1-INTERACTING PROTEIN 4)的磷酸化,RIN4能够与RPM1互作,RPM1感知到RIN4的变化后激起免疫反应[51]。有时R蛋白保护的宿主蛋白是植物为病原菌设置的“诱饵”,即植物进化出与效应蛋白靶标结构类似的蛋白。丁香假单胞杆菌的效应蛋白AvrPphB能够靶标和分裂PBS1(AVRPPHB SUSCEPTIBLE 1),激活R蛋白RPS5[52-53]。
在豆科植物与根瘤菌的互作中,宿主植物需要对根瘤数目和发育、根瘤菌定殖、根瘤菌固氮效率以及与根瘤菌之间的营养交换等进行精细的调控。而根瘤菌作为外来入侵的细菌,可以向植物细胞内分泌效应蛋白来调控与植物的共生互作。在宿主应对病原微生物的反应中,病原菌通过Ⅲ型分泌系统分泌的效应蛋白主要是抑制宿主的免疫反应。除少部分根瘤菌(如苜蓿根瘤菌)外,大部分根瘤菌基因组中都含有编码Ⅲ型分泌系统的各个基因[54]。然而,根瘤菌Ⅲ型效应蛋白是否也参与调控共生互作中的免疫反应大都未知。目前被研究的几个Ⅲ型效应蛋白在共生中主要起正调控作用,然而也有实验证明根瘤菌Ⅲ型效应蛋白在共生互作中起负调控作用。如在植物病原菌Ralstonia中转入根瘤菌的共生质粒,赋予它形成无效瘤的能力。在经过几轮的重复竞争性接种后,根瘤菌能够通过自发地抑制三型分泌系统从而获得形成更多共生根瘤的能力[55]。
那么目前被鉴定功能的效应蛋白主要也是来自根瘤菌Ⅲ型分泌系统,但是大部分在共生中具有正调控作用,这可能与根瘤菌在漫长的进化过程中作出的改变有关。根瘤菌分泌的蛋白被命名为NOPs(Nodulation outer proteins)[56]。根瘤菌NGR234中NopM是IpaH效应蛋白家族的成员。NopM是E3泛素连接酶,在烟草中能够抑制MAMP触发的ROS的产生。NopM也能抑制MAPK途径。在NGR234与Lablab purpureus的共生中,NopM能够增加根瘤的数量[57]。NopL作为一个具有多个磷酸化位点的根瘤菌特异的效应蛋白,能够抑制百脉根中PR基因的表达。在NGR234与Flemingia congesta的共生中增加结瘤数量,在NGR234与菜豆的共生中延迟根瘤衰老[58-60]。NopP是根瘤菌特异的效应蛋白,在NGR234与Tephrosia vogelii和Flemingia congesta的共生中增加结瘤数量[61]。NopT是C58半胱氨酸蛋白酶,在NGR234与灰毛豆共生中有正向调控的作用[62-63]。NopC是HH103的一个效应蛋白,在HH103与大豆的互作中有正向调控的作用[64]。
通过上述的描述,可以看出根瘤菌分泌的效应蛋白很多在根瘤菌与豆科植物的共生中起正向调控作用,但也有部分效应蛋白在根瘤菌与不同的豆科植物的互作中具有不同的作用[65]。根瘤菌的三型分泌系统能够分泌出很多效应蛋白,但是负向调控共生的效应蛋白还有很多未被鉴定。由于病原菌的效应蛋白可以靶标到免疫反应过程中的各个组分,因此根瘤菌效应蛋白靶标的研究能够为人们了解共生通路的免疫调控奠定基础。
3 根瘤菌定殖过程中免疫反应的调节根瘤主要有两种形态,如大豆、百脉根等产生的定型根瘤和苜蓿等产生的不定型根瘤[15](图 1)。二者其中一个区别在于根瘤原基起始的根部的皮层细胞不同。不定型根瘤的细胞分区发生于内皮层细胞和中柱鞘,定型根瘤的细胞通常发生于中皮层或者外皮层细胞[66-67]。不定型根瘤的形态通常是圆柱状或者分叉状,并且根瘤细胞根据根瘤菌侵染状态不同可大致分为4个区域:Ⅰ区为根瘤顶端的分生组织;Ⅱ区为从侵染线释放根瘤菌侵染植物细胞的侵染区;Ⅱ-Ⅲ区为过渡区;Ⅲ区为类菌体固氮的区域;Ⅳ区的类菌体正在衰老,为衰老区。相比较而言,定型根瘤没有一个持续的分生组织,形态趋近于球形,没有类似不定型根瘤的分区[68-69]。
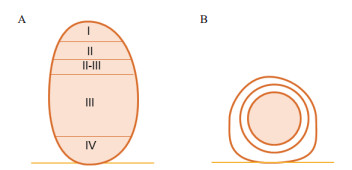
|
| 图 1 苜蓿产生的不定型根瘤(A)和百脉根产生的定型根瘤(B) |
定形根瘤与不定型根瘤除了根瘤形态不同(不定型根瘤具有根瘤分区)外,最大的区别是大部分不定型根瘤中的根瘤菌会经历末端分化:细胞膨大,基因组加倍,膜的修饰以及繁殖能力的丧失等[70]。目前认为是根瘤中一类特异表达的抗菌肽(NCRs,Nodule-specific cysteine rich peptides)所控制的[71]。苜蓿根瘤类菌体形态的苜蓿中华根瘤菌需要BacA降低植物NCR肽诱导的膜透化作用和NCR肽的杀菌能力,而体外NCR肽诱导根瘤菌使其具有类菌体特性则不依赖于BacA蛋白[72]。这表明豆科植物在和根瘤菌的博弈过程中使用了免疫这一武器而且双方都对这一免疫反应进行了调节。
但是由于在根瘤中存在免疫反应抑制的机制,我们无法直接检测到是否有类似PTI或者其他免疫反应在根瘤发育中有重要的功能。NAD1,SymCRK(Symbiotic Cysteine-rich Receptor Kinase),DNF2以及RSD(Regulator of symbiosome differentiation)等基因和突变体的鉴定为研究根瘤中的免疫反应的调节提供了材料[17-19, 73-74]。Yu等[75]利用nad1突变体揭示了在根瘤发育过程中NADPH氧化酶RbohBCD以及CDPK5(Calcium-Dependent Protein Kinase5)蛋白的重要功能。在nad1突变体中敲除RBOHB,C,D以及CDPK5后能够使突变体根瘤回复成具有正常功能的根瘤。而在野生型中过表达CDPK5-VK会使根瘤产生坏死。由此可以看出,CDPK-Rboh复合物产生的活性氧能够破坏根瘤发育过程中的免疫和共生平衡。在nad1或者symcrk突变体中受体类激酶蛋白上调,而CDPK和Rboh引发的活性氧爆发是PTI反应中一个典型的胞内反应。因此这似乎给予我们PTI反应参与了根瘤发育过程的信号。当NAD1、DNF2、SymCRK和RSD等抑制根瘤发育不同阶段免疫反应的任一个基因缺失后,根瘤便会表现出过度的免疫反应,导致根瘤菌定殖和类菌体发育受阻,根瘤坏死[76]。
4 总结与展望病原菌与植物的“斗智斗勇”主要表现在两个层次:MAMP触发的MTI反应和效应蛋白触发的ETI反应(图 2)。根瘤菌与豆科植物的互惠互利的共生模式与病原菌与植物的互作方式类似但也存在差异。在共生互作中,豆科植物供给根瘤菌碳源使其繁殖生长,而根瘤菌则提供给植物氮源。但豆科植物会在满足自己氮源需求的基础上尽量限制根瘤菌获取的能源,而根瘤菌则尽可能获取更多能源和避免氮气还原中的能量消耗。因此二者之间会互相“抵制”又互相“包容”。但是根瘤菌与植物互作的研究由于根瘤自身的发育过程以及相对紧凑的根瘤结构而受到一定限制,因此我们可以以病原菌的研究为基础探究植物根瘤内的免疫反应调控。但是由于根瘤内存在许多根瘤特异表达的基因,并且根瘤菌与植物的互作模式与病原菌仍存在很大差异,因此我们需要辩证性地参考植物病原菌的研究。
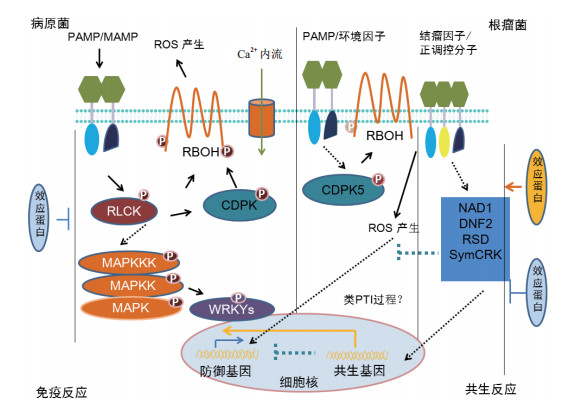
|
| MAMP:微生物相关分子模式,Microbia-Associated Molecular Patterns;RLCK:胞质类受体激酶,Receptor-Like Cytoplasmic Kinases;RBOH:呼吸爆发氧化酶同系物,Respiratory Burst Oxidase Homolog;CDPK:钙依赖蛋白激酶,Calcium-Dependent Protein Kinase;NAD1:防御反应激活的根瘤基因,NODULES WITH ACTIVATED DEFENSE;DNF2:固氮缺陷基因,Defective in Nitrogen Fixation 2;SymCRK:共生相关富含半胱氨酸的受体激酶,Symbiotic Cysteine-rich Receptor Kinase;RSD:共生体分化调控基因,Regulator of Symbiosome Differentiation 图 2 根瘤发育过程中植物对自身先天免疫反应的调控 |
就MTI而言,根瘤菌作为革兰氏阴性菌,其分子模式与病原菌有相同之处,但又有其自身的特点,因此植物如何识别和区分病原菌以及根瘤菌,并与后者建立互利共生的过程是目前国际上的研究热点。植物如何调控哪些根毛被侵染,当根瘤菌进入根毛后,根瘤菌的繁殖速度是否受到抑制,这些过程是否与免疫反应相关还需进一步研究。
而当根瘤菌进入豆科植物根瘤后,植物与根瘤菌的“博弈”也与病原菌和植物的斗争有一定的差异。模式植物苜蓿的根瘤为不定型根瘤,其根瘤具有严格的功能分区,而百脉根的根瘤为定型根瘤。目前鉴定的突变体nad1、symcrk等苜蓿突变体产生坏死的根瘤,而当这些基因在百脉根根瘤中缺失后,并未产生明显表型[77]。这种现象可能与苜蓿根瘤中类菌体的末端分化有关。这可能也表明在苜蓿等产生不定型根瘤的植物中存在着对根瘤菌更严谨的调控,而这种调控与植物的免疫反应相关。但是对于根瘤菌调控植物免疫反应的研究仍较少。因此不定型根瘤和定型根瘤对于根瘤菌免疫反应相关的调控具有差异,那么哪些基因参与了这个调控过程以及这个差异是否直接影响根瘤菌的固氮效率还有待探究。并且当结瘤起始后,能够抑制免疫反应的根瘤菌中的蛋白又是如何被激活,在这些蛋白缺失之后植物是否识别根瘤菌为病原菌从而启动免疫反应仍然未知。
至于根瘤菌释放的效应蛋白,多数似乎在共生过程中具有促进结瘤固氮的正向调控作用。令人疑惑的是,病原菌大部分依赖Ⅲ型分泌系统释放的效应蛋白诱导植物的免疫反应,而在根瘤菌中,Ⅲ型分泌系统只在部分根瘤菌中存在,而苜蓿中华根瘤菌中没有Ⅲ型分泌系统,只有Ⅳ型分泌系统。那么通过Ⅳ型分泌系统释放的效应蛋白在共生中的功能是怎样的?并且这些效应蛋白在植物中的靶标以及工作方式如何仍是我们的盲区。因此,在根瘤发育过程中各种精细而又严密的调控仍然需要科学家们的研究阐释。
| [1] |
Dixon R, Kahn D. Genetic regulation of biological nitrogen fixation[J]. Nat Rev Microbiol, 2004, 2(8): 621-631. DOI:10.1038/nrmicro954 |
| [2] |
王二涛. 植物-根瘤菌共生固氮[J]. 中国基础科学, 2016, 18(1): 21-27. DOI:10.3969/j.issn.1009-2412.2016.01.004 |
| [3] |
Oldroyd GED, Downie JA. Coordinating nodule morphogenesis with rhizobial infection in legumes[J]. Annu Rev Plant Biol, 2008, 59(1): 519-546. DOI:10.1146/annurev.arplant.59.032607.092839 |
| [4] |
Oldroyd GED, Murray JD, Poole PS, et al. The rules of engagement in the legume-rhizobial symbiosis[J]. Annu Rev Genet, 2011, 45(1): 119-144. DOI:10.1146/annurev-genet-110410-132549 |
| [5] |
Broughton WJ, Perret X. Genealogy of legume-Rhizobium symbioses[J]. Curr Opin Plant Biol, 1999, 2(4): 305-311. DOI:10.1016/S1369-5266(99)80054-5 |
| [6] |
Cao Y, Halane MK, Gassmann W, et al. The role of plant innate immunity in the legume-Rhizobium symbiosis[J]. Annu Rev Plant Biol, 2017, 68(1): 535. DOI:10.1146/annurev-arplant-042916-041030 |
| [7] |
Wong JEMM, Nadzieja M, Madsen LH, et al. A Lotus japonicus cytoplasmic kinase connects Nod factor perception by the NFR5 LysM receptor to nodulation[J]. Proc Natl Acad Sci USA, 2019, 116(28): 14339-14348. DOI:10.1073/pnas.1815425116 |
| [8] |
Ke D, Fang Q, Chen C, et al. The small GTPase ROP6 interacts with NFR5 and is involved in nodule formation in Lotus japonicus[J]. Plant Physiol, 2012, 159(1): 131-143. DOI:10.1104/pp.112.197269 |
| [9] |
Duan L, Pei J, et al. A Dihydroflavonol-4-Reductase-Like protein interacts with NFR5 and regulates rhizobial infection in Lotus japonicus[J]. Mol Plant Microbe Interact, 2019, 32(4): 401-412. DOI:10.1094/MPMI-04-18-0104-R |
| [10] |
Morieri G, Martinez EA, Jarynowski A, et al. Host-specific Nod-factors associated with Medicago truncatula nodule infection differentially induce calcium influx and calcium spiking in root hairs[J]. New Phytol, 2013, 200(3): 656-662. DOI:10.1111/nph.12475 |
| [11] |
Peiter E, Sun J, Heckmann AB, et al. The Medicago truncatula DMI1 protein modulates cytosolic calcium signaling[J]. Plant Physiol, 2007, 145(1): 192-203. |
| [12] |
Singh S, Parniske M. Activation of calcium- and calmodulin-dependent protein kinase (CCaMK), the central regulator of plant root endosymbiosis[J]. Curr Opin Plant Biol, 2012, 15(4): 444-453. DOI:10.1016/j.pbi.2012.04.002 |
| [13] |
Singh S, Katzer K, Lambert J, et al. CYCLOPS, a DNA-binding transcriptional activator, orchestrates symbiotic root nodule development[J]. Cell Host Microbe, 2014, 15(2): 139-152. DOI:10.1016/j.chom.2014.01.011 |
| [14] |
Jin Y, Liu H, Luo D, et al. DELLA proteins are common components of symbiotic rhizobial and mycorrhizal signaling pathways[J]. Nat Commun, 2016, 7: 12433. DOI:10.1038/ncomms12433 |
| [15] |
Brewin NJ. Development of the legume root nodule[J]. Annu Rev Cell Biol, 1991, 7: 191-226. DOI:10.1146/annurev.cb.07.110191.001203 |
| [16] |
Marsh JF, Rakocevic A, Mitra RM, et al. Medicago truncatula NIN is essential for rhizobial-independent nodule organogenesis induced by autoactive calcium/calmodulin-dependent protein kinase[J]. Plant Physiol, 2007, 144(1): 324-335. |
| [17] |
Wang C, Yu H, et al. NODULES WITH ACTIVATED DEFENSE 1 is required for maintenance of rhizobial endosymbiosis in Medicago truncatula[J]. New Phytol, 2016, 212(1): 176-191. DOI:10.1111/nph.14017 |
| [18] |
Domonkos Á, Kovács S, Gombár A, et al. NAD1 controls defense-like responses in Medicago truncatula symbiotic nitrogen fixing nodules following rhizobial colonization in a BacA-independent manner[J]. Genes, 2017, 8(12): 387. DOI:10.3390/genes8120387 |
| [19] |
Bourcy M, Brocard L, Pislariu CI, et al. Medicago truncatula DNF2 is a PI-PLC-XD-containing protein required for bacteroid persistence and prevention of nodule early senescence and defense-like reactions[J]. New Phytol, 2013, 197(4): 1250-1261. DOI:10.1111/nph.12091 |
| [20] |
Hacquard S, Spaepen S, Garrido-Oter R, et al. Interplay between innate immunity and the plant microbiota[J]. Annu Rev Phytopathol, 2017, 4(55): 565-589. |
| [21] |
Yu X, Feng B, He P, et al. From chaos to harmony:responses and signaling upon microbial pattern recognition[J]. Annu Rev Phytopathol, 2017, 55(1): 109-137. DOI:10.1146/annurev-phyto-080516-035649 |
| [22] |
Chinchilla D, Zipfel C, Robatzek S, et al. A flagellin-induced complex of the receptor FLS2 and BAK1 initiates plant defence[J]. Nature, 2007, 448(7152): 497-500. DOI:10.1038/nature05999 |
| [23] |
Zipfel C, Kunze G, Chinchilla D, et al. Perception of the bacterial PAMP EF-Tu by the receptor EFR restricts agrobacterium-mediated transformation[J]. Cell, 2006, 125(4): 749-60. DOI:10.1016/j.cell.2006.03.037 |
| [24] |
Hind SR, Strickler SR, Boyle PC, et al. Tomato receptor FLAGELLIN-SENSING 3 binds flgⅡ-28 and activates the plant immune system[J]. Nat Plants, 2016, 2(9): 16128. DOI:10.1038/nplants.2016.128 |
| [25] |
Lopez-Gomez M, Sandal N, Stougaard J, et al. Interplay of flg22-induced defence responses and nodulation in Lotus japonicus[J]. J Exp Bot, 2012, 63(1): 393-401. DOI:10.1093/jxb/err291 |
| [26] |
Willmann R, Lajunen HM, Erbs G, et al. Arabidopsis lysin-motif proteins LYM1 LYM3 CERK1 mediate bacterial peptidoglycan sensing and immunity to bacterial infection[J]. Proc Natl Acad Sci USA, 2011, 108(49): 19824-19829. DOI:10.1073/pnas.1112862108 |
| [27] |
Liu B, Li JF, Ao Y, et al. OsLYP4 and OsLYP6 play critical roles in rice defense signal transduction[J]. Plant Signal Behav, 2013, 8(2): e22980. DOI:10.4161/psb.22980 |
| [28] |
Ranf S, Gisch N, Schäffer M, et al. A lectin S-domain receptor kinase mediates lipopolysaccharide sensing in Arabidopsis thaliana[J]. Nat Immunol, 2015, 16(4): 426-433. DOI:10.1038/ni.3124 |
| [29] |
Desaki Y, Kouzai Y, Ninomiya Y, et al. OsCERK1 plays a crucial role in the lipopolysaccharide-induced immune response of rice[J]. New Phytol, 2018, 217(3): 1042-1049. DOI:10.1111/nph.14941 |
| [30] |
Lagares A, Caetano-Anollés G, Niehaus K, et al. A Rhizobium meliloti lipopolysaccharide mutant altered in competitiveness for nodulation of alfalfa[J]. J Bacteriol, 1992, 174(18): 5941-52. DOI:10.1128/jb.174.18.5941-5952.1992 |
| [31] |
Cao Y, Liang Y, Tanaka K, et al. The kinase LYK5 is a major chitin receptor in Arabidopsis and forms a chitin-induced complex with related kinase CERK1[J]. ELife, 2014, 3: e03766. DOI:10.7554/eLife.03766 |
| [32] |
Liu T, Liu Z, et al. Chitin-induced dimerization activates a plant immune receptor[J]. Science, 2012, 336(6085): 1160-1164. DOI:10.1126/science.1218867 |
| [33] |
D'Haeze W, Holsters M. Nod factor structures, responses, and perception during initiation of nodule development[J]. Glycobiology, 2002, 12(6): 79R-105R. DOI:10.1093/glycob/12.6.79R |
| [34] |
Younes I, Rinaudo M. Chitin and chitosan preparation from marine sources. structure, properties and applications[J]. Mar Drugs, 2015, 13(3): 1133-1174. DOI:10.3390/md13031133 |
| [35] |
Amor BB, Shaw SL, Oldroyd GED, et al. The NFP locus of Medicago truncatula controls an early step of Nod factor signal transduction upstream of a rapid calcium flux and root hair deformation[J]. Plant J, 2003, 34(4): 495-506. DOI:10.1046/j.1365-313X.2003.01743.x |
| [36] |
Limpens E, Franken C, Smit P, et al. LysM domain receptor kinases regulating rhizobial Nod factor-induced infection[J]. Science, 2003, 302(5645): 630-633. DOI:10.1126/science.1090074 |
| [37] |
Madsen EB, Madsen LH, Radutoiu S, et al. A receptor kinase gene of the LysM type is involved in legume perception of rhizobial signals[J]. Nature, 2003, 425(6958): 637-640. DOI:10.1038/nature02045 |
| [38] |
Radutoiu S, Madsen LH, Madsen EB, et al. Plant recognition of symbiotic bacteria requires two LysM receptor-like kinases[J]. Nature, 2003, 425(6958): 585-592. DOI:10.1038/nature02039 |
| [39] |
Smit P, Limpens E, Geurts R, et al. Medicago LYK3, an entry receptor in rhizobial nodulation factor signaling[J]. Plant Physiol, 2007, 145(1): 183-191. |
| [40] |
Pietraszewska-Bogiel A, Lefebvre B, Koini MA, et al. Interaction of Medicago truncatula lysin motif receptor-like kinases, NFP and LYK3, produced in Nicotiana benthamiana induces defence-like responses[J]. PLoS One, 2013, 8(6): e65055. DOI:10.1371/journal.pone.0065055 |
| [41] |
Liang Y, Cao Y, Tanaka K, et al. Nonlegumes respond to rhizobial Nod factors by suppressing the innate immune response[J]. Science, 2013, 341(6152): 1384-1387. DOI:10.1126/science.1242736 |
| [42] |
Skorupska A, Janczarek M, Marczak M, et al. Rhizobial exopolysaccharides:genetic control and symbiotic functions[J]. Microb Cell Fact, 2006, 5(1): 7. DOI:10.1186/1475-2859-5-7 |
| [43] |
Kawaharada Y, Nielsen MW, et al. Differential regulation of the Epr3 receptor coordinates membrane-restricted rhizobial colonization of root nodule primordia[J]. Nat Commun, 2017, 8: 14534. DOI:10.1038/ncomms14534 |
| [44] |
Sun Y, Li L, Macho AP, et al. Structural basis for flg22-induced activation of the Arabidopsis FLS2-BAK1 immune complex[J]. Science, 2013, 342(6158): 624-628. DOI:10.1126/science.1243825 |
| [45] |
Shimizu T, Nakano T, Takamizawa D, et al. Two LysM receptor molecules, CEBiP and OsCERK1, cooperatively regulate chitin elicitor signaling in rice[J]. Plant J, 2010, 64(2): 204-214. DOI:10.1111/j.1365-313X.2010.04324.x |
| [46] |
Boller T, Felix G. A renaissance of elicitors:perception of microbe-associated molecular patterns and danger signals by pattern-recognition receptors[J]. Annu Rev Plant Biol, 2009, 60(1): 379-406. DOI:10.1146/annurev.arplant.57.032905.105346 |
| [47] |
Jones JD, Dangl JL. The plant immune system[J]. Nature, 2006, 444(7117): 323-329. DOI:10.1038/nature05286 |
| [48] |
Flor HH. Current Status of the Gene-For-Gene Concept[J]. Annu Rev Phytopathol, 2003, 9(1): 275-296. |
| [49] |
Jia Y, Mcadams SA, Bryan GT, et al. Direct interaction of resistance gene and avirulence gene products confers rice blast resistance[J]. EMBO J, 2014, 19(15): 4004-4014. |
| [50] |
Dangl JL, Mcdowell JM. Two modes of pathogen recognition by plants[J]. Proc Natl Acad Sci USA, 2006, 103(23): 8575-8576. DOI:10.1073/pnas.0603183103 |
| [51] |
Mackey D, Holt BF, Wiig A, et al. RIN4 interacts with Pseudomonas syringae type Ⅲ effector molecules and is required for RPM1-mediated resistance in Arabidopsis[J]. Cell, 2002, 108(6): 743-754. DOI:10.1016/S0092-8674(02)00661-X |
| [52] |
Ade J, Deyoung BJ, Golstein C, et al. Indirect activation of a plant nucleotide binding site-leucine-rich repeat protein by a bacterial protease[J]. Proc Natl Acad Sci USA, 2007, 104(7): 2531-2536. DOI:10.1073/pnas.0608779104 |
| [53] |
Shao F, Golstein C, Ade J, et al. Cleavage of Arabidopsis PBS1 by a bacterial type Ⅲ effector[J]. Science, 2003, 301(5637): 1230-1233. DOI:10.1126/science.1085671 |
| [54] |
Nelson MS, Sadowsky MJ. Secretion systems and signal exchange between nitrogen-fixing rhizobia and legumes[J]. Front Plant Sci, 2015, 6: 491. |
| [55] |
Guan SH, Gris C, Cruveiller S, et al. Experimental evolution of nodule intracellular infection in legume symbionts[J]. ISME J, 2013, 7: 1367-1377. DOI:10.1038/ismej.2013.24 |
| [56] |
Marie C, Broughton WJ, Deakin WJ. Rhizobium type Ⅲ secretion systems:legume charmers or alarmers[J]. Curr Opin Plant Biol, 2001, 4(4): 336-342. DOI:10.1016/S1369-5266(00)00182-5 |
| [57] |
Xin DW, Liao S, Xie ZP, et al. Functional analysis of NopM, a novel E3 ubiquitin ligase (NEL) domain effector of Rhizobium sp. strain NGR234[J]. PLoS Pathog, 2012, 8(5): e1002707. DOI:10.1371/journal.ppat.1002707 |
| [58] |
Bartsev AV, Boukli NM, Deakin WJ, et al. Purification and phosphorylation of the effector protein NopL from Rhizobium sp. NGR234[J]. FEBS Lett, 2003, 554(3): 271-274. DOI:10.1016/S0014-5793(03)01145-1 |
| [59] |
Zhang L, Chen XJ, Lu HB, et al. Functional analysis of the Type 3 effector nodulation outer protein L (NopL) from Rhizobium sp. NGR234[J]. J Biol Chem, 2011, 286(37): 32178-32187. DOI:10.1074/jbc.M111.265942 |
| [60] |
Bartsev AV, Deakin WJ, Boukli NM, et al. NopL, an effector protein of Rhizobium sp. NGR234, thwarts activation of plant defense reactions[J]. Plant Physiol, 2004, 134(2): 871-879. DOI:10.1104/pp.103.031740 |
| [61] |
Skorpil P, Saad MM, Boukli NM, et al. NopP, a phosphorylated effector of Rhizobium sp. strain NGR234, is a major determinant of nodulation of the tropical legumes Flemingia congesta and Tephrosia vogelii[J]. Mol Microbiol, 2005, 57(5): 1304-1317. DOI:10.1111/j.1365-2958.2005.04768.x |
| [62] |
Dai WJ, Zeng Y, Xie ZP, et al. Symbiosis-promoting and deleterious effects of NopT, a novel type 3 effector of Rhizobium sp. strain NGR234[J]. J Bacteriol, 2008, 190(14): 5101-5110. DOI:10.1128/JB.00306-08 |
| [63] |
Fotiadis CT, Dimou M, et al. Functional characterization of NopT1 and NopT2, two type Ⅲ effectors of Bradyrhizobium japonicum[J]. FEMS Microbiol Lett, 2012, 327(1): 66-77. DOI:10.1111/j.1574-6968.2011.02466.x |
| [64] |
Jiménez-Guerrero I, Pérez-Montaño F, Medina C, et al. NopC is a Rhizobium-specific type 3 secretion system effector secreted by Sinorhizobium (Ensifer) fredii HH103[J]. PLoS One, 2015, 10(11): e0142866. DOI:10.1371/journal.pone.0142866 |
| [65] |
Miwa H, Okazaki S. How effectors promote beneficial interactions[J]. Curr Opin Plant Biol, 2017, 38: 148-154. DOI:10.1016/j.pbi.2017.05.011 |
| [66] |
Sprent JI. Evolving ideas of legume evolution and diversity:a taxonomic perspective on the occurrence of nodulation[J]. New Phytol, 2007, 174(1): 11-25. DOI:10.1111/j.1469-8137.2007.02015.x |
| [67] |
Hirsch A. Developmental biology of legume nodulation[J]. New Phytol, 1992, 122(2): 211-237. DOI:10.1111/j.1469-8137.1992.tb04227.x |
| [68] |
Timmers AC, Soupene E, Auriac MC, et al. Saprophytic intracellular rhizobia in alfalfa nodules[J]. Mol Plant Microbe Interact, 2000, 13(11): 1204-1213. DOI:10.1094/MPMI.2000.13.11.1204 |
| [69] |
Maunoury N, Kondorosi A, Kondorosi E, et al. Cell Biology Of Nodule Infection And Development[M]. Nitrogen-fixing Leguminous Symbioses. Springer Netherlands, 2008.
|
| [70] |
Mergaert P, Uchiumi T, Alunni B, et al. Eukaryotic control on bacterial cell cycle and differentiation in the Rhizobium-legume symbiosis[J]. Proc Natl Acad Sci USA, 2006, 103(13): 5230-5235. DOI:10.1073/pnas.0600912103 |
| [71] |
Mergaert P, Nikovics K, Kelemen Z, et al. A novel family in Medicago truncatula consisting of more than 300 nodule-specific genes coding for small, secreted polypeptides with conserved cysteine motifs[J]. Plant Physiol, 2003, 132(1): 161-173. |
| [72] |
Haag AF, Baloban M, Sani M, et al. Protection of Sinorhizobium against host cysteine-rich antimicrobial peptides is critical for symbiosis[J]. PLoS Biol, 2011, 9(10): e1001169. DOI:10.1371/journal.pbio.1001169 |
| [73] |
Berrabah F, Bourcy M, Eschstruth A, et al. A nonRD receptor-like kinase prevents nodule early senescence and defense-like reactions during symbiosis[J]. New Phytol, 2014, 203(4): 1305-1314. DOI:10.1111/nph.12881 |
| [74] |
Sinharoy S, Torres-Jerez I, Bandyopadhyay K, et al. The C2H2 transcription factor regulator of symbiosome differentiation represses transcription of the secretory pathway gene VAMP721a and promotes symbiosome development in Medicago truncatula[J]. Plant Cell, 2013, 25(9): 3584-3601. DOI:10.1105/tpc.113.114017 |
| [75] |
Yu HX, Xiao A, Dong R, et al. Suppression of innate immunity mediated by the CDPK-Rboh complex is required for rhizobial colonization in Medicago truncatula nodules[J]. New Phytol, 2018, 220(2): 425-434. DOI:10.1111/nph.15410 |
| [76] |
Berrabah F, Salem EHA, Garmier M, et al. The multiple faces of the Medicago-Sinorhizobium symbiosis[J]. Methods Mol Biol, 2018, 1822: 241-260. |
| [77] |
Yu H, Bao H, Zhang Z, et al. Immune signaling pathway during terminal bacteroid differentiation in nodules[J]. Trends Plant Sci, 2019, 24(4): 299-302. DOI:10.1016/j.tplants.2019.01.010 |




