2. 西华师范大学环境科学与工程学院,南充 637009
2. College of Environmental Science and Engineering, China West Normal University, Nanchong 637009
食用菌子实体的形成和发育的分子机理对食用菌的产量和品质具有关键的影响。近年来,我国食用菌产业发展迅速,香菇、杏鲍菇等已实现机械化生产,但目前一些重要食用菌仍主要依赖于野生资源,野生资源过度开发及环境的变化导致野生食用菌类资源量急剧下降,甚至一些种类濒临灭绝[1-2]。转录组测序能够反应不同生命阶段的基因的表达、功能,能对基因进行注释及标记[3],已广泛应用于探索大型真菌发育相关基因及关键分子机制。荣成博等[4]通过转录组测序发现编码苯丙氨酸裂解酶的基因(CL4509)和编码乙醇氧化酶的基因与白灵侧耳的子实体发育相关。吴小梅等[5]分析了双孢蘑菇3个发育时期的转录组测序结果,发现双孢蘑菇子实体发育与氨基酸代谢相关,能通过MAPK信号途径传递环境胁迫信号。施肖堃等[6]分析双孢蘑菇As2796子实体4个发育阶段的差异基因,发现疏水蛋白在其子实体发育与后熟过程中有重要的作用。李帆等[7]分析了刺芹侧耳3个发育时期的转录组测序结果,发现菌丝期的碳代谢和氨基酸代谢、原基期的脂肪酸代谢和抗氧化酶、子实体时期的环境因子影响刺芹侧耳发育。探索食用菌子实体的形成和发育的分子机制有助于实现野生食用菌的人工栽培生产。
棒瑚菌(Clavariadelphus pistillaris(Fr.)Donk)属于担子菌门,钉菇目,棒瑚菌科,棒瑚菌属,子实体呈棒状,不分枝,顶部钝圆。主要分布于我国四川、云南等地[8]。棒瑚菌属真菌能与壳斗科栎属植物形成菌根,保证树木的正常生长,维持生态平衡[9],在生态学上有很大的应用潜力。棒瑚菌属真菌含有丰富的微量元素,味道鲜美,可食用,因此它们适合作为营养食品和功能食品[10]。研究发现,从平截棒瑚菌中(Clavariadelphus truncatus.(Quel.)Donk.)提取的棒瑚菌酸(Clavaric acid)能作为法呢基转移酶(FPTase)的抑制剂[11],其菌丝体培养物具有广泛的抗菌性能[12]。棒瑚菌属是具有巨大发展潜力的天然药物资源。棒瑚菌属共发现了22种,在我国仅发现9种,国内外学者对棒瑚菌属的研究主要在形态学、解剖学、显微化学和开发应用等方面[13-14]。目前,棒瑚菌尚未实现人工栽培,野生产量稀少,其独特的形态在大型真菌中独树一帜。因此,分析棒瑚菌子实体不同发育时期的基因表达情况、差异表达基因,探索其子实体不同发育时期关键的功能基因和代谢通路,对棒瑚菌资源的保护与开发应用具有一定的指导意义。
本文通过RNA-Seq测序技术,比较分析采自四川省阿坝藏族羌族自治州小金县棒瑚菌子实体幼菇期和成熟期差异表达的基因,探索棒瑚菌发育过程中与子实体生长发育有关的功能基因及代谢机制,为棒瑚菌子实体发育的进一步研究及食药用资源开发提供一定的理论基础。
1 材料与方法 1.1 材料本实验的棒瑚菌采自四川省阿坝藏族羌族自治州小金县,选取幼菇期(CP-1)与成熟期(CP-2)的棒瑚菌子实体作为研究材料。用无菌水将新鲜完整子实体清洗(重复3次),用无菌剪刀将子实体剪碎后,立即转移至液氮中保存供下一步实验。
1.2 方法 1.2.1 样品总RNA的提取及检测按照OMEGA(OMEGA’s fungal total RNA extraction kit)真菌总RNA提取试剂盒的使用说明提取棒瑚菌子实体中总RNA。称取50 mg经液氮研磨加入裂解液,离心; 转移上清加入0.5倍体积无水乙醇,离心弃滤液; 加Buffer Ⅰ离心,弃去滤液; 加入Buffer Ⅱ离心弃去滤液(重复2次); 加入DEPC Water离心。通过1%的琼脂糖电泳和Agilent 2100检测RNA样品的完整性,用NanoDrop检测RNA的纯度,OD260/OD280应在1.8-2.2之间,OD260 / OD230 > 2[15]。
1.2.2 生物信息学分析样品总RNA检测合格后送至北京诺禾致源生物信息科技有限公司进行cDNA文库构建,库检合格后进行RNA-Seq测序。
将原始测序数据(Raw data)进行过滤筛选,得到分析数据Clean reads。利用Trinity[16]对分析数据Clean reads进行拼接,获得最终转录本序列,选取每个基因中最长的转录本作为非重复独立基因Unigene。将非重复独立基因Unigene在KOG/COG数据库进行功能注释。利用RSEM[17]软件进行基因表达定量分析(FPKM > 0.3)。采用DESeq[18]进行基因差异性表达分析,阈值为padj < 0.05,并对差异基因进行GO[19]富集分析和KEGG[20]富集分析。
2 结果 2.1 转录组的数据质量分析CP-1与CP-2进行RNA-Seq测序后分别获得原始数据(Raw reads),去除带接头的reads和低质量的reads后分别获得8.78 G(CP-1)和8.28 G(CP-2)分析数据(Clean reads)。综合3次测序结果的平均值,其中CP-1的Q20(%)为97.60%,Q30(%)为94.12%,GC含量为57.75%,CP-2的Q20(%)为97.65%,Q30(%)为94.03%,GC含量为57.70%,碱基错误率均为0.01%。拼接分析数据Clean reads后一共获得151 367个转录本(Transcript),101 291个不同长度的非重复独立基因Unigene,占总转录本的66.92%。以上结果说明该测序结果可靠性较高,可用于下一步分析研究(表 1,图 1)。
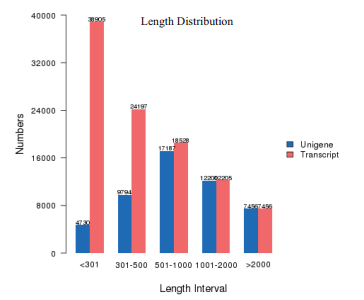
|
| 图 1 拼接转录本与Unigenes序列长度分布图 |
转录组基因KOG功能注释结果(表 2)显示,共有19 204个Unigene在KOG数据库成功注释,共涉及26类。非重复独立基因Unigene数量最多的集中在仅通用功能预测(2 375条),翻译后修饰、蛋白质周转、伴侣蛋白(2 194条)和翻译、核糖体结构、生物发生(2 030条)等,最少的是细胞运动(10条)。该结果显示,CP-1和CP-2中基因所涉及到的KOG功能类别比较全面复杂,包括蛋白质合成相关过程。值得注意的是,有800条Unigene的功能是未知的,且存在未命名蛋白(1条)。由此可见,棒瑚菌还有大量基因有待于进一步研究。
用Goseq方法对CP-1和CP-2的差异基因进行了GO富集分析,统计后结果(图 2)显示,共产生2 169个生物过程(Biological process),517个细胞成分(Cellula component)和1 076个分子功能(Molecular function)。差异基因GO富集结果提示,棒瑚菌在不同生长阶段(CP-1和CP-2)的生物代谢存在一定的差异,同时提示,生物过程(Biological process)中的新陈代谢过程、细胞成分(Cellula component)中的细胞和细胞组分和分子功能(Molecular function)中的氧化还原酶活性是棒瑚菌子实体发育过程中的关键基因门类。
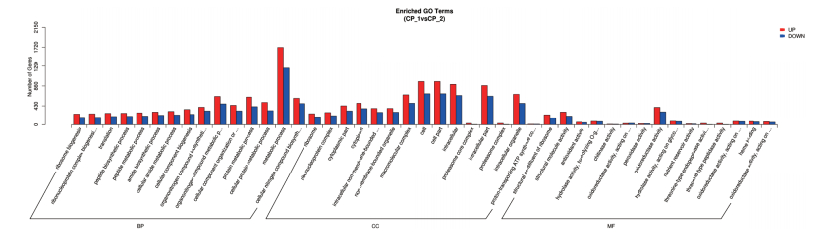
|
| 图 2 差异基因GO富集分析 |
采用RSEM软件对基因表达进行定量分析,FPKM超过60的基因高水平表达,而在0-0.3 FPKM的基因为低水平表达或根本不表达。基因表达水平分析结果显示,棒瑚菌在不同生长阶段(CP-1和CP-2)基因表达水平的分布总体相似,但仍存在一定的差异。在棒瑚菌子实体幼菇期(CP-1)有39 140个基因表达(FPKM ≥ 0.3),约占基因总数的76.38%,其中,2 458个基因高表达(FPKM > 60),约占基因总数的4.96%;而在棒瑚菌子实体成熟期(CP-2),有35 188个基因表达(FPKM ≥ 0.3),约占基因总数的68.5%,其中,2495个基因高表达(FPKM > 60),约占基因总数的4.86%。
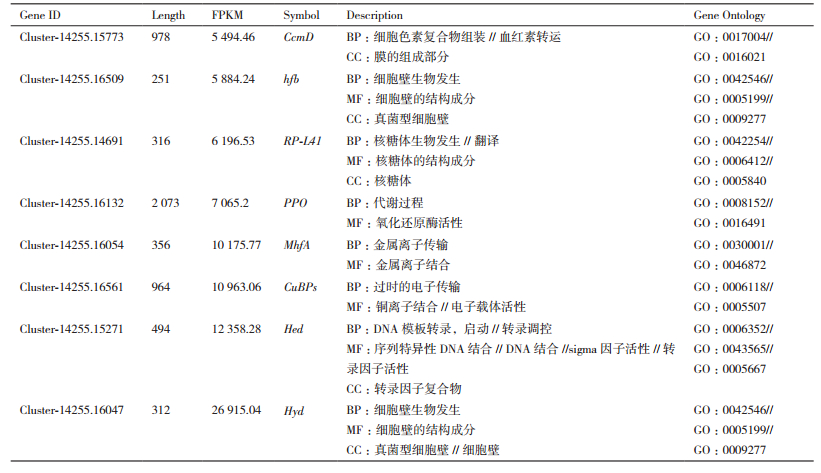
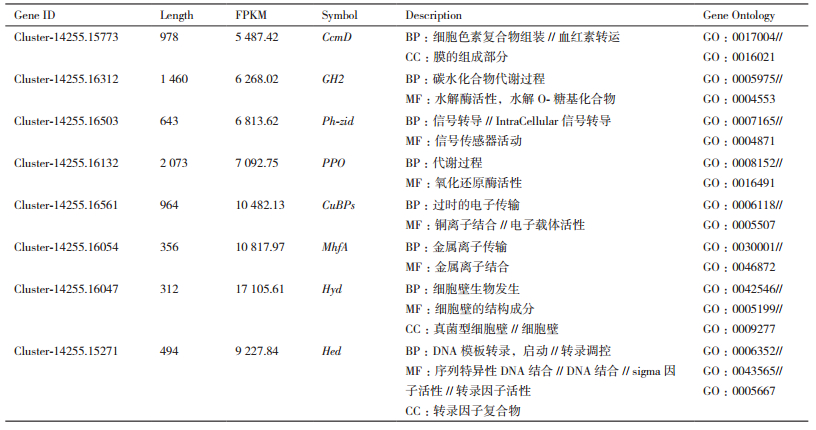
进一步分析发现,棒瑚菌子实体幼菇期(CP-1)和成熟期(CP-2)分别均有8个基因为极高表达(FPKM > 5 000)。棒瑚菌子实体幼菇期(CP-1)的8个极高表达基因为:CcmD,HFB,RP-L41,PPO,MhfA,CuBPs,Hed,Hyd(表 3),在棒瑚菌子实体成熟期(CP-2)的8个基因极高表达基因为:CcmD,GH2,Ph-zid,PPO,CuBPs,MhfA,Hed,Hyd(表 4)。
编码多酚氧化酶基因PPO和编码疏水蛋白基因Hyd在CP-1和CP-2看均极高表达,hfb和RP-L41仅在CP-1中高表达,hfb为编码真菌疏水蛋白的基因,该基因仅在CP-1中极高表达,说明在棒瑚菌的发育过程中至少有两种疏水蛋白参与细胞壁的生物合成; RP-L41为编码核糖体大亚基L41的基因,参与核糖体的生物合成及转录翻译,提示棒瑚菌在成熟期(CP-2)核糖体的代谢发生了变化。而GH2和Ph-zid仅在CP-2中高表达,编码糖苷水解酶家族2的基因GH2仅在CP-2中极高表达,而编码糖苷水解酶家族2的基因GH2和编码苯丙氨酸拉链的基因Ph-zid仅在CP-2中极高表达,以上筛选出的高表达基因对揭示棒瑚菌在不同生长发育期的关键基因提供了一定的科学依据。
2.4 不同发育时期差异表达基因(DEGs)的分析使用DESeq对CP-1和CP-2的差异表达基因(DEGs)进行分析,并设定显著差异表达的阈值Padj < 0.05。棒瑚菌不同发育时期差异表达基因(DEGs)的分析结果显示,棒瑚菌子实体幼菇期(CP-1)和成熟期(CP-2)的差异基因有6 625个,包括3 949个上调基因和2 676个下调基因(图 3),其中有2 290个基因只在幼菇期(CP-1)中表达,1 231个基因只在成熟期(CP-2)中表达。
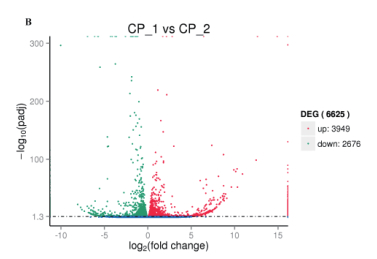
|
| 图 3 CP-1与CP-2基因差异表达分析火山图 |
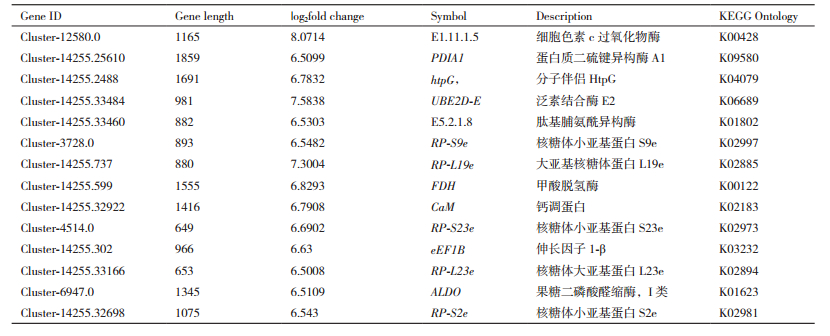
在| log2fold change| > 6.5阈值内的差异基因中,27个基因被上调,包括COX3、CYTB、wrbA、ND1、ARD1、EF3、ND4、SDHA、RP-S3e、RP-L5、RP-S5、EEF1A、ATP1、PDIA1 和 ARF1等; 14个基因被下调,包括E1.11.1.5、PDIA1、htpG、UBE2D-E、CaM、RP-S9e、RP-L19e、ALDO和FDH等(表 5-6)。
进一步分析显示,上调基因中,涉及一些氧化呼吸电子传递链的辅基基因,例如,编码电子传递链中复合体Ⅰ的辅基的基因ND1和ND4,复合体Ⅱ的辅基的基因SDHA,复合体Ⅲ的辅基的基因CYTB,复合体Ⅳ的辅基的基因COX3,ATP合酶辅基的基因ATP1,提示氧化磷酸化途径的基因出现上调。在下调基因中,包括编码钙调蛋白基因CaM和编码甲酸脱氢酶基因FDH。
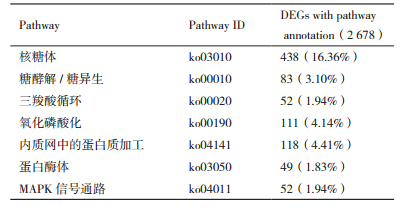
2.6 不同发育时期代谢通路KEGG分析KEGG(Kyoto encyclopedia of genes and genomes)是有关代谢通路的主要公共数据库[28]。棒瑚菌子实体幼菇期(CP-1)和成熟期(CP-2)差异基因KEGG Pathway富集结果(表 7)显示,有2 678个差异表达的基因成功注释在104条通路中。其中,在核糖体(438个差异基因,16.36%)、糖酵解/糖异生途径(83个差异基因,3.10%)、三羧酸循环(52个差异基因,1.94%)、氧化磷酸化(111个差异基因,4.14%)、内质网中的蛋白质加工(118个差异基因,4.41%)、蛋白酶体(49个差异基因,1.83%)和MAPK信号通路(52个差异基因,1.94%)中,差异基因富集显著。
本研究中三羧酸循环(Citrate cycle,TCA cycle)途径(图 4)上调基因,包括pdhc(K00627)、DLST(K00658)、pdhD(K00382)、IDH3(K00030)、CS(K01647)、E4.2.1.2(K01676)、SDHA(K00234)、MDH1(K00025)和ACO(K01681)。MAPK信号通路(MAPK signaling pathway)(图 5)上调基因,包括Cdc42(k04393)、PAK1(k04409)、MKK(K11226)、PKC1(k02677)、MKK1_2(K08294)、MAPK7(K04464)、YPD1(K11232)和GNG(K07973);下调基因包括STE12(K11215)和SSK2(K11230)。

|
| 红色:上调,绿色:下调,黄色:基因家族中既有上调又有下调 图 4 三羧酸循环 |

|
| 红色:上调,绿色:下调,黄色:基因家族中既有上调又有下调 图 5 MAPK信号通路 |
此外,幼菇期(CP-1)与成熟期(CP-2)差异基因还显著富集在核糖体、内质网中的蛋白质加工、蛋白酶体途径,显示棒瑚菌子实体幼菇期(CP-1)与成熟期(CP-2)蛋白质合成途径亦存在差异。
3 讨论大型真菌子实体发育过程涉及物质能量代谢、细胞分化以及信号转导等生命活动。本研究选用四川省小金县棒瑚菌幼菇期(CP-1)和成熟期(CP-2)的子实体进行RNA-Seq测序并进行生物信息学分析,不同发育时期基因表达水平分析发现,编码多酚氧化酶的基因PPO在CP-1和CP-2中均极高表达,可能与棒瑚菌子实体的褐变及抗病性有关[21-22]。编码疏水蛋白的基因Hyd在CP-1和CP-2中均极高表达,有利于孢子的扩散和营养菌丝的分散,还能保护细胞免受恶劣环境的侵害[23-24]。而编码糖苷水解酶家族2的基因GH2仅在CP-2中极高表达,说明GH2作为糖代谢的关键基因参与寡糖和多糖的合成,修饰和降解[25]。编码苯丙氨酸拉链的基因Ph-zid仅在CP-2中极高表达,说明CP-2能在没有细胞外配体的情况下激活和调节酪氨酸激酶的活性[26]。比较分析CP-1和CP-2差异表达的基因,发现上调基因主要富集在氧化呼吸电子传递链,说明棒瑚菌在幼菇期(CP-1)需要大量能量维持正常的生长发育,同时大量的耗氧代谢过程提示,环境含氧量对棒瑚菌的生长发育有重要影响。在下调基因中,编码钙调蛋白基因CaM下调,说明棒湖菌在成熟期(CP-2),细胞内多种生理活动如细胞增殖和细胞周期正在减弱[27]; 编码甲酸脱氨酶基因FDH下调,提示棒瑚菌在发育过程中甲酸含量增加,为维持一碳化合物稳态,成熟期(CP-2)甲酸代谢过程增强[28],初步筛选出了与棒瑚菌生长发育有关的代谢途径,包括三羧酸循环、氧化磷酸化、核糖体途径、蛋白酶体途径以及MAPK信号通路等。
三羧酸循环不仅是细胞能量的来源,而且能产生其他生物大分子合成的前体,更是生物体内3大营养物质代谢的枢纽[29]。本研究中三羧酸循环中的编码丙酮酸脱氢酶的基因PDH、苹果酸脱氢酶的基因MDH、柠檬酸合酶的基因CS、异柠檬酸脱氢酶的基因IDH、琥珀酸脱氢酶的基因SDH均在CP-1中表达上调,说明三羧酸循环在棒瑚菌幼菇期(CP-1)能量代谢的中起关键作用; 而磷酸烯醇丙酮酸羧化激酶基因PCKA在棒瑚菌子实体成熟期(CP-2)中表达表达上调,说明棒瑚菌子实体成熟期(CP-2)的糖异生过程比幼菇期(CP-1)更为活跃,该过程与寡糖和多糖的合成、修饰等生理生化过程有关。MAPK信号通路是细胞内一种重要的信号转导系统,通过MAPK激酶激酶(MAP kinase kinase kinase,MKKK)、MAPK激酶(MAP kinase kinase,MKK)和MAPK依次激活,共同调节细胞的生长、分化以及对环境的应激等多种重要的细胞生理过程[30-31]。我们发现在MAPK信号通路中,细胞分裂控制蛋白42基因(Cdc42)表达上调,提示Cdc42可能参调控与菌丝的极性生长和细胞周期,说明真菌细胞形态转换与Rho家族蛋白基因有关[32-33],以上结果显示,MAPK信号通路在棒瑚菌子实体的生长发育过程中有重要作用。
4 结论在棒瑚菌的生长发育过程中,新陈代谢过程、细胞和细胞组分以及氧化还原酶活性是棒瑚菌子实体发育过程中的关键基因门类。多酚氧化酶,核糖体,糖苷水解酶家族等基因在棒瑚菌子实体的蛋白质和细胞壁合成中起重要作用。氧化磷酸化和三羧酸循环是棒瑚菌子实体幼菇期(CP-1)生长发育的关键代谢途径,且环境含氧量对棒瑚菌的生长发育有重要影响; 同时MAPK信号通路是参与调节棒瑚菌子实体生长发育的关键信号通路,在调控菌丝的极性生长,细胞周期和真菌细胞形态转换中起重要作用。
| [1] |
李玉. 中国食用菌产业发展现状、机遇和挑战——走中国特色菇业发展之路, 实现食用菌产业强国之梦[J]. 菌物研究, 2018, 16(3): 5-11. |
| [2] |
王建瑞, 刘宇, 图力古尔. 山东省大型真菌物种濒危程度与优先保育评价[J]. 生态学报, 2015, 35(3): 837-848. |
| [3] |
贾昌路, 张瑶, 朱玲, 等. 转录组测序技术在生物测序中的应用研究进展[J]. 分子植物育种, 2015, 13(10): 2388-2394. |
| [4] |
荣成博, 赵爽, 宋爽, 等. 白灵侧耳子实体发育相关基因的挖掘[J]. 生物技术通报, 2018, 34(4): 115-120. |
| [5] |
吴小梅, 张昕, 李南羿. 双孢蘑菇子实体不同发育时期的转录组分析[J]. 菌物学报, 2017, 36(2): 193-203. |
| [6] |
施肖堃, 蔡志欣, 郭仲杰, 等. 双孢蘑菇As2796子实体发育转录组测序分析[J]. 福建农业学报, 2018, 33(3): 282-287. |
| [7] |
李帆, 陈利丁, 艾柳英, 等. 刺芹侧耳不同发育时期的转录组差异分析[J]. 菌物学报, 2018, 37(12): 1586-1597. |
| [8] |
卯晓岚. 中国大型真菌[M]. 郑州: 河南科学技术出版社, 2000.
|
| [9] |
Kranabetter JM, Friesen J, Gamiet S, et al. Epigeous fruiting bodies of ectomycorrhizal fungi as indicators of soil fertility and associated nitrogen status of boreal forests[J]. Mycorrhiza, 2009, 19(8): 535-548. DOI:10.1007/s00572-009-0255-0 |
| [10] |
Pereira E, Barros L, Martins A, et al. Towards chemical and nutritional inventory of Portuguese wild edible mushrooms in different habitats[J]. Food Chemistry, 2012, 130(2): 394-403. DOI:10.1016/j.foodchem.2011.07.057 |
| [11] |
Lingham RB, Silverman KC, Jayasuriya H, et al. Clavaric acid and steroidal analogues as Ras- and FPP-directed inhibitors of human farnesyl-protein transferase[J]. Journal of Medicinal Chemistry, 1998, 41(23): 4492-4501. DOI:10.1021/jm980356+ |
| [12] |
Yamac M, Bilgili F. Antimicrobial activities of fruit bodies and/or mycelial cultures of some mushroom isolates[J]. Pharmaceutical Biology, 2006, 44(9): 660-667. DOI:10.1080/13880200601006897 |
| [13] |
赵洁, 杨淑达, 唐丽萍. 棒瑚菌系统分类研究进展[J]. 昆明医科大学学报, 2016, 37(3): 231-233. |
| [14] |
袁明生, 孙佩琼. 中国大型真菌彩色图谱[M]. 成都: 四川科学技术出版社, 2013.
|
| [15] |
Cock PJA, Fields CJ, Goto N, et al. The Sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants[J]. Nucleic Acids Research, 2010, 38(6): 1767-1771. DOI:10.1093/nar/gkp1137 |
| [16] |
Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644-652. DOI:10.1038/nbt.1883 |
| [17] |
Chen G, Shi T, Shi L. Characterizing and annotating the genome using RNA-seq data[J]. Science China Life Sciences, 2017, 60(2): 116-125. DOI:10.1007/s11427-015-0349-4 |
| [18] |
Mccarthy DJ, Chen Y, Smyth GK. Differential expression analysis of multifactor RNA-Seq experiments with respect to biological variation[J]. Nucleic Acids Research, 2012, 40(10): 4288-4297. DOI:10.1093/nar/gks042 |
| [19] |
Young MD, Wakefield MJ, Smyth GK, et al. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biology, 2010, 11(2): R14. DOI:10.1186/gb-2010-11-2-r14 |
| [20] |
Kanehisa M, Sato Y, Kawashima M, et al. KEGG as a reference resource for gene and protein annotation[J]. Nucleic Acids Research, 2016, 44(D1): D457-D462. DOI:10.1093/nar/gkv1070 |
| [21] |
Wang J, Liu B, Xiao Q, et al. Cloning and expression analysis of litchi(Litchi Chinensis Sonn.)polyphenol oxidase gene and relationship with postharvest pericarp browning[J]. PLoS One, 2014(4): e93982. |
| [22] |
LI NY, Cai WM, Jin QL, et al. Molecular cloning and expression of polyphenoloxidase genes from the mushroom, Agaricus bisporus[J]. Agricultural Sciences in China, 2011, 10(2): 185-194. |
| [23] |
Kim HI, Lee CS, Park YJ. Further characterization of hydrophobin genes in genome of Flammulina velutipes[J]. Mycoscience, 2016, 57(5): 320-325. DOI:10.1016/j.myc.2016.04.004 |
| [24] |
黄姗, 王中康, 陈环, 等. 莱氏野村菌疏水蛋白基因Nrhyd的克隆及其表达特征分析[J]. 菌物学报, 2012, 31(3): 350-358. |
| [25] |
Karkehabadi S, Hansson H, Mikkelsen NE, et al. Structural studies of a glycoside hydrolase family 3 β-glucosidase from the model fungus Neurospora crassa[J]. Acta Crystallographica Section F:Structural Biology Communications, 2018, 74(12): 787-796. DOI:10.1107/S2053230X18015662 |
| [26] |
Hayouka Z, Thomas NC, Mortenson DE, et al. Quasiracemate crystal structures of magainin 2 derivatives support the functional significance of the phenylalanine zipper motif[J]. Journal of the American Chemical Society, 2015, 137(37): 11884-11887. DOI:10.1021/jacs.5b07206 |
| [27] |
Boczek NJ, Gomez-Hurtado N, Ye D, et al. Spectrum and prevalence of CALM1-, CALM2-, and CALM3-Encoded calmodulin variants in long QT syndrome and functional characterization of a novel long QT syndrome-associated calmodulin missense variant, E141G[J]. Circulation Cardiovascular Genetics, 2016, 9(2): 136-146. DOI:10.1161/CIRCGENETICS.115.001323 |
| [28] |
李天明, 朱灵桓, 刘天佳, 等. Pseudom onas sp. 101甲酸脱氢酶基因的合成、表达及定点饱和突变[J]. 生物技术通报, 2013(5): 105-110. |
| [29] |
朱钦士. 正转和反转的三羧酸循环[J]. 生物学通报, 2015, 50(1): 16-19. |
| [30] |
Luke NS, Devito MJ, Portier CJ, et al. Employing a mechanistic model for the MAPK pathway to examine the impact of cellular all or none behavior on overall tissue response[J]. Dose Response, 2010, 8(3): 347-367. |
| [31] |
Atay O, Skotheim JM. Spatial and temporal signal processing and decision making by MAPK pathways[J]. The Journal of Cell Biology, 2017, 216(2): 317-330. DOI:10.1083/jcb.201609124 |
| [32] |
Brand AC, Morrison E, Milne S, et al. Cdc42 GTPase dynamics control directional growth responses[J]. Proceedings of the National Academy of Sciences, 2014, 111(2): 811-816. DOI:10.1073/pnas.1307264111 |
| [33] |
Woods B, Lew DJ. Polarity establishment by Cdc42:Key roles for positive feedback and differential mobility[J]. Small GTPases, 2016, 10(2): 130-137. |